PEDV G2 突变株动力学特征比较研究



摘要:[目的]本研究旨在通过对猪流行性腹泻病毒(PEDV)经连续传代后的毒株(PEDV-BJ-150)进行全面研究,探究其生物学特性、遗传变异、致病能力和细胞适应性,以评估其作为潜在减毒疫苗株的潜力。[方法]通过对PEDV-BJ-150 和原始PEDV-BJ-01 毒株进行细胞培养、动物感染试验和基因组测序,评估其在不同细胞系中的增殖能力、病毒滴度、致病性以及基因组的核苷酸序列变异并利用动物模型评估其感染特性和病理变化。[结果]PEDV-BJ-150 经连续传代后在Vero-M 细胞中表现出更高的增殖能力和滴度,尤其在悬浮适应株Vero-M 和贴壁适应株Vero-M 中表现出显著的滴度差异。实验结果显示其在体内感染后的致病性降低,尤其在肺部未检测到病毒存在,鼻翼和小肠部位显示更高的病毒滴度。此外,基因组测序结果表明PEDV-BJ-150 的S 蛋白存在多个位置的氨基酸突变,可能影响其细胞嗜性和致病能力。[结论]本研究发现经连续传代的PEDV-BJ-150 在体内表现出的毒力和致病能力明显降低,表明其有作为减毒疫苗株的潜力。特别是S 蛋白中的连续性氨基酸缺失可能导致病毒致病性降低,这为疫苗研发提供了新的思路。此外,研究发现不同组织对PEDV 的感染特性不同,这对于深入了解病毒传播机制具有重要意义。本研究的创新之处在于发现了PEDV 经连续传代后的致病性变化和S 蛋白突变对病毒特性的影响,为病毒进化和疫苗设计提供了新的见解。这项研究为了解PEDV 的病毒学特性和毒力变化提供了重要线索,对于病毒疫苗研发和应对疫情具有潜在的应用前景。
关键词:猪流行腹泻病毒; 减毒突变株; 动力学特征; 基因组分析
中图分类号:S855.3 文献标识码:A 文章编号:1671-8151(2024)02-0080-10
猪流行性腹泻(PED)是猪急性和高度传染性的肠道疾病,其由猪流行性腹泻病毒(PEDV)引起,其广泛传播严重影响着全球养猪业的经济利益[1-2]。该疾病的主要特征包括乳猪出现呕吐、腹泻、脱水、食欲不振、体重下降以及高死亡率[2-4]。尽管临床上大部分猪流行性腹泻病毒感染多出现在夏季和冬季,但偶尔也可能在春季发生[5]。值得注意的是,在仔猪中发生的猪流行性腹泻病毒感染常伴有极高的死亡率[6-7]。
PEDV 属于Nidovirales 目、冠状病毒科和Al⁃pha 冠状病毒属,为一种单股正链RNA 病毒[8]。PEDV 的基因组长度约为28 kb 核苷酸(nts),在其两端具有5 ′-帽和3 ′-聚腺苷尾;其基因组还包括5 ′和3 ′非翻译区域(UTR),至少有7 个开放阅读框(ORF1a、ORF1b 和ORF2-6)[9-11]。ORF1a 和ORF1b 分别编码复制酶多蛋白1a 和1ab,这些蛋白经病毒蛋白酶自体蛋白水解形成16 种非结构蛋白(Nspl-16),这些酶参与病毒RNA 的转录和复制[12-13]。而ORF2-6 主要编码4 种结构蛋白(刺突蛋白(S)、膜蛋白(M)、包膜蛋白(E)和核衣壳蛋白(N),以及辅助蛋白ORF3;这些蛋白在基因组中按照以下顺序排列:5 ′-ORF(la/lb)-S-ORF3-EM-N-3 ′[13-15]。
1978 年,于比利时首次发现PEDV 毒株CV777[16-17]。2010 年11 月,中国境内发现1 种高致病性PEDV 毒株,在随后几个月内快速传播至全国各地[3, 18]。这一变种随后在美国被发现,并传播至加拿大、墨西哥等地,同时迅速在亚洲多个国家传播,给这些地区造成了巨大的经济损失[19-21]。
疫苗接种被认为是预防猪场PEDV 感染的有效方法[22]。针对经典PEDV 毒株,如CV777、DR13 和KPEDV-9,已开发出多种减毒活化和灭活疫苗,并在许多国家成功上市。然而,在病毒快速传播的过程中,毒株不断变异,导致疫苗株与流行株之间存在抗原和遗传差异[23-25]。因此,迫切需要开发针对新变异株的新型PEDV 疫苗。
本研究通过将分离的PEDV G2 毒株在Vero-M 细胞中进行连续传代,获得了1 株突变毒株,并确定了该毒株在不同细胞系中的生物学特性。此外,使用5 日龄的仔猪评估了这些毒株的致病性。随后利用二代测序技术对经过连续传代后的PEDV-BJ-01 株进行了全基因组序列测定。最终,我们获得了1 株安全的减毒PEDV 毒株,旨在为制备减毒疫苗提供新的理论依据。
1 材料和方法
1. 1 细胞和病毒培养
悬浮适应株Vero-M 在VP-SFM AGT™培养基中培养;悬浮适应株ST 在DMEM/F12 培养基中培养,并在37 ℃、5% CO2、140 r·min-1 的细胞培养箱中培养。贴壁适应株Vero-M 和贴壁细胞株ST,在补充有7% 胎牛血清的低葡萄糖的Dul⁃becco 改良杜尔贝科培养基(DMEM)中培养,37 ℃、5% CO2 的生长培养基中培养。以上细胞均由本实验室保存。
PEDV G2 毒株PEDV-BJ-01 之前由本实验室于病料组织中分离,测序比对后确定为FJzz1 毒株(GenBank:MK288006)。将分离获得的FJzz1毒株接种在单层Vero-M 细胞上,连续传代3 次。当发现明显的细胞病变时收集细胞培养液上清液,并将其命名为PEDV-BJ-01。随后在Vero-M细胞上盲传150 代,收集盲传后病毒液。收集病毒储液,分装储存在−80 ℃冰箱中,将该毒株命名为PEDV-BJ-150。
针对PEDV N 基因设计如下引物,
正向引物PED-N-F(5 ′-TGCGGTTCTCACAGATAGTG)和反向引物PED-N-R(5 ′-AAGTCGCTAGAAAAACACTCAGTAAT)。
将以上引物用于RT-PCR。检测病毒是否存在,并在确认后测定收集到的病毒液的病毒滴度。
1. 2 PEDV 毒株在Vero-M 细胞中的连续传代
将Vero-M 细胞在T25 细胞培养瓶中培养至60%~70% 汇合度。将PEDV 毒株原液稀释在含有10 g·mL-1胰蛋白酶的2 mL 无血清DMEM 培养基中,随后使用磷酸盐缓冲盐水(PBS)冲洗2 次单层培养的Vero-M 细胞。将病毒液感染Vero-M细胞,病毒感染复数(MOI)为0. 01,每隔15 min 震荡混匀,孵育1 h 后,弃去病毒溶液,使用PBS 洗涤细胞培养瓶2 次,加入含有5 g·mL-1 胰蛋白酶的DMEM 培养基5 mL。随后置于37℃、5% CO2 的细胞培养箱中继续培养,当细胞产生可见细胞病变效应(CPE)时,冻融裂解感染的细胞,并在4 ℃下以3000×g 离心1 min 以收获培养液上清。然后将收集的病毒液以同样的方式继续接种Vero-M细胞,直到第150 次传代结束。
1. 3 PEDV 复制动力学测定
使用含有10 μg·mL-1 胰蛋白酶的悬浮细胞培养基将悬浮适应株Vero-M 和悬浮适应株ST 传代至无菌细胞摇瓶中,细胞密度105 Cells·mL-1,体积30 mL。以病毒感染复数(MOI)为0. 01 分别接种2 种病毒。放置于140 r·min-1、37 ℃、5% CO2 温箱中培养,分别于病毒感染后24、48、72、96 h 共4 个时间点收取细胞培养上清液,测定病毒滴度。
将贴壁Vero-M、ST 细胞均匀铺至48 孔细胞培养板中。待细胞长至70%~80% 汇合度时,以多重感染复数(MOI)为0. 01 分别接种2 种病毒。
将病毒和细胞在37 ℃、5% CO2 的温箱中吸附1 h后,弃去病毒液,每孔加入500 μL 无菌PBS 清洗2 次,每孔加入含有10 μg·mL-1 胰蛋白酶的DMEM 培养基250 μL。
将感染后的细胞放置于37 ℃、5% CO2温箱培养,分别于感染后24、48、72 和96 h,收取细胞上清液,分别测定细胞培养上清液中的病毒滴度。期间,观察致细胞病变效应(CPE)。
1. 4 病毒滴度与拷贝数的测定
取正常培养的Vero-M 细胞,将细胞按适当比例稀释后均匀铺至96 孔细胞培养板中,待细胞贴壁生长至汇合度约为70%~80% 左右时接种病毒。将上述收取的病毒液(细胞培养上清液)使用含有10 μg·mL-1 胰蛋白酶的接毒培养液作连续10倍的梯度稀释,从10-1~10-9 连续9 个稀释度,每组重复6 次试验。将稀释好的病毒接种到9 6 孔细胞培养板中,每1 稀释度接种1 纵排,共9 孔,每孔接种1 0 0 μL。并设正常细胞对照。接毒48 h 后观察细胞病变情况并记录结果。按Reed-Muench 法计算结果。
通过RT-qPCR 分析病毒颗粒释放,使用RNA 病毒试剂盒提取细胞培养上清液中的病毒RNA,使用逆转录酶逆转录后进行实时定量聚合酶链反应(RT-qPCR)分析。使用靶向PEDV N基因的RT-qPCR 测定培养液上清中的病毒载量。PEDV 正向引物PEDN-F (5 ′-CGCAAAGACTGAACCCACTAAC-3 ′) ,反向引物PEDN-R(5 ′-TTGCCTCTGTTGTGTTACTTGGAGAT-3 ′)和PEDV 探针(FAM-TGYYACCAYYACCACACGACTCCTGC-BHQ3) , QuantStudio ™ Real-Time PCR 软件用于数据采集和分析。
1. 5 基因组测序与遗传变异分析
使用RNAsimple 总RNA 提取试剂盒(DP419)提取,然后使用HiScript II 1st StrandcDNA Synthesis Kit 反转录试剂盒逆转录成cDNA。以该cDNA 为模板和16 对重叠的引物进行PCR 验证(补充表S1)。PEDV-BJ-150 变异株的基因组序列采用二代测序技术测定。病毒S 蛋白的氨基酸序列使用MegAlign(DNAStar Laser⁃gene)中的Clustal W 方法进行比对。比对完成后通过手动鉴定基因组序列上的不同毒株之间核苷酸/aa 变化的位置。如前所述。利用MEGA 7. 0将基于PEDV-BJ-150 毒株S 基因的和GenBank中提供的另外50 个具有完整S 基因序列的参考毒株,采用邻域连接法(NJ)构建系统发育树。
1. 6 毒株的致病性评价
选取15 头体重相仿的5 d 龄仔猪,随机分配为3 组,分为A 组、B 组和C 组。每组饲养在1 个单独的房间。A 组和B 组的试验动物分别滴鼻感染PEDV-BJ-01 毒株和PEDV-BJ-150 毒株,剂量为104 TCID50。C 组给予等量生理盐水滴鼻。
在试验过程中,每天监测和记录临床症状,包括精神状况和体重的变化。病毒感染后第3 和5天对试验动物实行安乐死,收集鼻甲骨、肺部、小肠样品。将部分收集样品低温研磨后12 000 r·min-1 离心,检测组织上清液的病毒滴度含量。
1. 7 组织病理学和HE 染色分析
在实验中,首先收集小肠肠段并将其在10%福尔马林中室温固定36 h。随后,制备石蜡包埋的组织样本,完成后进行切割、脱蜡、再水化处理。最后,采用标准的苏木精和伊红(Hamp;E)染色方法对样本进行染色,然后进行扫描切片分析[26]。这些步骤用于准备和分析小肠肠段的组织样本。
1. 8 统计分析
所有实验数据均使用Origin2018 统计软件进行分析。使用t 检验评估统计学显著性,当Plt;0. 05 时,认为差异是显著的。
2 结果与分析
2. 1 PEDV-BJ-150 毒株在连续传代后的生物学特性
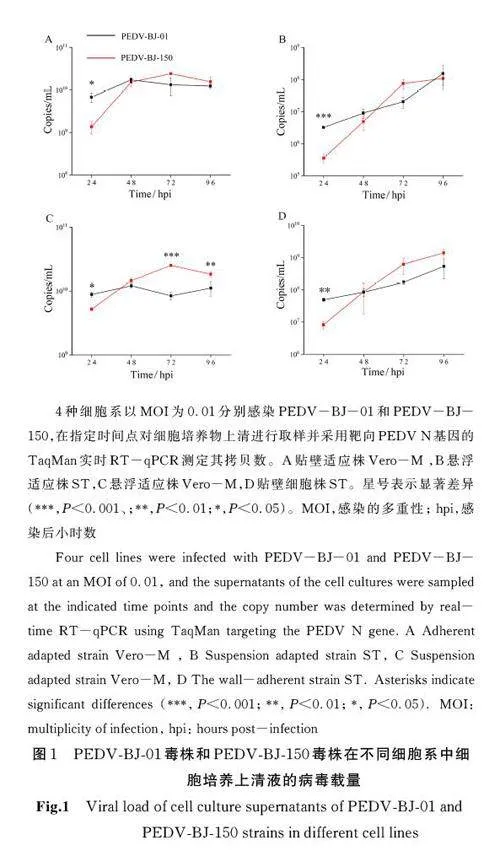
将PEDV-BJ-01 毒株和PEDV-BJ-150 毒株分别以多重感染复数(MOI)0. 01 接种悬浮适应株Vero-M、贴壁适应株Vero-M、悬浮适应株ST、贴壁细胞株ST 共4 种细胞。定时取样以分析体外连续传代过程后其生物学特性的变化。
结果如图1 所示,当不同的细胞系感染2 种毒株时,2 株毒株没有呈现显著的拷贝数差异,仅在感染后的部分时间点呈现统计学差异。随后基于病毒滴度(TCID50)的多步生长动力曲线如图2 所示。当细胞感染两种毒株后,悬浮适应株Vero-M、贴壁适应株Vero-M、悬浮适应株ST 在感染后的各个时间点均发现明显的病毒滴度差异。在ST 贴壁细胞系中2 株毒株的病毒滴度差异较小。2 株毒株在悬浮适应株Vero-M 和贴壁适应株Vero-M 中病毒滴度差异最高。病毒滴度的最大差距约为103。
PEDV-BJ-150 和PEDV-BJ-01 毒株在悬浮适应株ST 中滴度差异较小,病毒滴度的最大差距约102 左右。而在贴壁细胞株ST 中病毒滴度差异相较于其它3 株细胞系,其各个时间点的病毒滴度的差距最小。
总之,这些结果表明PEDV-BJ-150 毒株相比PEDV-BJ-01 毒株在悬浮适应株Vero-M、贴壁适应株Vero-M 中的敏感性和适应性在体外连续传代过程中明显更强。并且PEDV-BJ-150 毒株对于悬浮培养的细胞系敏感程度更高。
2. 2 PEDV-BJ-150 的遗传变异分析
为了进一步分析突变株病毒的遗传变异,在MegAlign 软件中使用Clustal W 对这些变异的全基因组序列进行了比对。结果如图3 表明,相较于PEDV-BJ-01,PEDV-BJ-150 变异株在S 蛋白上有14 个位置的氨基酸发生改变。在其它结构蛋白和非结构蛋白部分也有不同程度的突变。PEDVBJ-150 突变株中S 蛋白的氨基酸变化分散在S1和S2 亚基中。
为了探究PEDV-BJ-150 变异株在连续传代过程后的氨基酸变异,构建了PEDV-BJ-01 和PEDV-BJ-150 中翻译出的S 糖蛋白氨基酸的序列比对。如图4 显示,在体外连续传代后,高传代PEDV-BJ-150 突变株在S1-NTD 区域表现出特
征性的连续aa 缺失(55I56G57E → 55△56△57△)这与Chen 等[27]的研究结果相似。并且在144-145处也发现了氨基酸的缺失(144T145G→144△145△)。在877-878 位氨基酸也发现了相应的改变(877V878L→877R878R)。
在其它位置14、223、265、490、710、773、820、1120、1212、1338、1353 也发现了氨基酸的突变,但这些变化对于病毒的增殖和致病能力的影响仍有待研究。
此外,基于PEDV-BJ-150 突变株的S 基因和另外50 个参考毒株构建了系统发育树,GenBank中提供了完整的基因序列。系统发育分析表明,PEDV-BJ-150 毒株和50 株参考菌株形成2 个基因型:G1 基因型主要包含CV777 和DR13 等经典菌株,而G2 基因型包含PEDV-BJ-150 株和其它变异大流行菌株。
2. 3 感染PEDV 突变毒株仔猪的临床体征
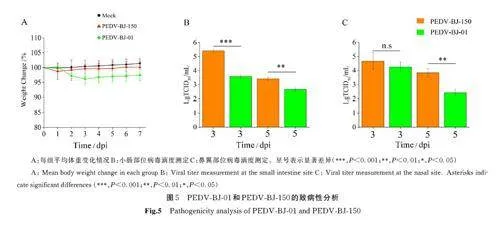
为了评估PEDV-BJ-150 变异毒株的毒力是否发生变化,并分析突变毒株与原始毒株在5 d 龄仔猪体内的致病能力,分别接种了这2 株毒株到试验组A 和试验组B 中,并监测了仔猪每日的体重变化。在接种前,3 组动物的体重差异没有达到统计学上的显著性。但在病毒接种后,A 组的仔猪体重增长速度较慢。相比之下,B 组的大多数动物保持了相对稳定的体重,尽管与对照组相比仍然存在一定程度的下降。整个试验过程中,未观察到任何仔猪死亡的情况。正如图5A 所示,PEDVBJ-150 株对仔猪体重的影响较PEDV-BJ-01 株更小。这些结果表明,PEDV-BJ-150 株在5 d 龄仔猪中具有一定毒性,但在体外连续传代后,其致病能力明显减弱。
2. 4 肠道和肺部不同病毒株的病毒滴度
2 株毒株感染实验动物后,对收集的样本检测病毒滴度,如图5B 和5C 显示,PEDV-BJ-150 与PEDV-BJ-01 在鼻甲骨和小肠组织感染第3 天和第5 天存在一定的滴度差异。而在肺部组织样本2株毒株在不同时间点均没有检测病毒滴度的存在。当感染3 d 后,PEDV-BJ-150 株在小肠部位的滴度约为105. 4TCID50,相应PEDV-BJ-01 株滴度约为103. 5TCID50,二者统计学差异显著。而在感染第5 天,2 株毒株的病毒滴度均有不同程度的下降,二者差异约为10 倍。而在鼻翼部位,感染3d 后,两株毒株的病毒滴度没有统计学差异。有趣的是,当感染第5 天后,2 株毒株的病毒滴度在鼻翼部位产生了统计学差异,2 株毒株产生了约10 倍的滴度差异。这可能是由于2 株毒株在不同感染部位滴度衰减速度不同导致的。当对肺部样本的病毒滴度进行测定时,发现在感染后3 d 和5 d,2 株病毒均没有检测到病毒滴度。
这些结果表明,虽然实验采取滴鼻感染两株毒株,但肺部没有检测到病毒滴度的存在。这些结果表明,PEDV-BJ-150 株在仔猪体内的增殖能力高于PEDV-BJ-01 株;并且滴鼻也是PEDV 的一种有效感染方式。
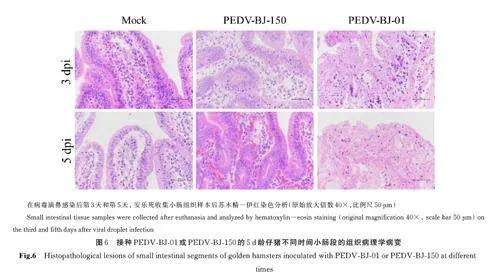
2. 5 感染PEDV-BJ-150 变异株的仔猪组织病理学病变
进行病理学和组织学检查,系统、直观地评价经过连续传代后的PEDV-BJ-01 和PEDV-BJ-150 毒株的致病性。如图6 所示,2 株毒株感染均引起小肠的病理变化。其特征是肠壁变薄,肠腔内有淡黄色液体存在。相比之下,PEDV-BJ-01感染组相比PEDV-BJ-150 毒株显示出更强的病理变化。HE 染色显示,PEDV-BJ-01 感染组在小肠段引起较为严重的组织病理学病变,其特征是肠黏膜结构崩解,溶解消失,仅残留结缔组织和坏死的细胞碎片,黏膜下水肿。在PEDV-BJ-150 感染组中,小肠也检测到了组织病理学病变,但其病变程度略低于PEDV-BJ-01 感染组,其特征为肠黏膜固有层水肿,伴有少量炎细胞浸润,黏膜上皮细胞排列整齐,未见变性,坏死等变化。对照组实验动物的肠黏膜上皮细胞排列整齐,未见变性,坏死等变化,黏膜下层及肌层均未见明显异常变化。
综上所述,经过连续传代后的PEDV-BJ-150毒株的致病性低于PEDV-BJ-01。
3 讨论
PEDV 的爆发给全世界的养猪业造成了巨大的经济损失。目前,疫苗接种是应对病毒传播的有效方法,然而由于PEDV 在传播过程中极易发生氨基酸突变,造成经典减毒疫苗不能提供有效的免疫保护[28-29]。因此,必须加快对该病毒流行毒株的研究进度,以制备新的减毒疫苗株。在世界各地,许多研究人员通过在Vero 细胞中连续传代经典PEDV 毒株,从而人为产生减毒疫苗株[30-32]。在本研究中,原始的PEDV 毒株在经过连续传代后发生大量突变,尤其在其S 蛋白基因中有14 个位置的氨基酸发生了改变,这些位置氨基酸的改变可能导致病毒滴度和致病能力发生了明显改变。与最初分离的PEDV-BJ-01 毒株相比,经过连续传代的PEDV-BJ-150 毒株在悬浮适应株Vero-M、贴壁适应株Vero-M 和悬浮适应株ST 中有着滴度差异,滴度差异在102~103 左右(图2)。并且研究显示,PEDV-BJ-150 毒株在Vero-M 细胞中有着更为迅速的扩增能力,更快的达到病毒滴度的峰值。
之后,为了评估经过连续传代后的PEDV 在体内的致病能力和毒力,分别将PEDV-BJ-01 和PEDV-BJ-150 毒株感染仔猪。结果显示PEDVBJ-150 毒株对于体重的影响能力弱于PEDV-BJ-01 毒株(图5A)。其次,通过测定PEDV-BJ-150和PEDV-BJ-01 毒株在鼻翼、肺部和小肠的滴度。结果显示,PEDV-BJ-150 毒株在鼻翼和小肠部位有着更高的病毒滴度(图5B 和5C),而在肺部,均没有检测到病毒的存在。
另一方面结果显示,接种PEDV-BJ-150 毒株的临床体征和组织病理学病变程度较轻,表明PEDV-BJ-150 毒株在体内的致病能力更弱(图6)。
因此,根据试验结果认为通过在细胞上对PEDV 的连续传代,导致了PEDV-BJ-150 毒株在Vero-M 细胞系上的增殖能力和病毒滴度有着明显提高,并且能够降低PEDV 在体内感染后的严重程度和病毒致病力。
对PEDV-BJ-01 和PEDV-BJ-150 毒株的全部基因组进行了测序,以进一步评估毒力变化。测序结果显示在其S 蛋白的多个位置发生了氨基酸的改变,这些位置的改变可能共同改变了PEDV 的细胞嗜性和毒力[27,33]。在毒株S 蛋白的其它位置发生的氨基酸的改变对于毒株的影响还需验证。总的来说,这些结果表明,PEDV 毒株的细胞嗜性和毒力是基因组中多个核苷酸突变组合的结果,并且可以通过多种分子机制发生。
PEDV 的S 蛋白一直被视为病毒变异的标志[34-35]。S 蛋白在接受免疫压力时很容易变异。S蛋白中的突变(包括缺失和/或插入)可能会改变冠状病毒的致病性和组织嗜性[36-38]。与PEDVBJ-01 毒株相比,PEDV-BJ-150 毒株的S 基因中发生了大量的核苷酸突变和缺失。我们发现PEDV-BJ-150 毒株的S 蛋白序列中存在3 个位置的特征性氨基酸突变(图4)(55I56G57E→55△56△ 57 △ ,144T145G→144 △ 145 △ 和877V878L→877R878R),怀疑S 基因的这些突变可能与病毒致病性有关。
之前的研究称,口服途径为PEDV 的主要感染途径[39-40]。病毒可以通过口腔直接进入肠道,肠道蛋白酶可能帮助病毒在肠道释放[41]。最近的一项研究表明,PEDV 病毒可以被鼻上皮细胞的DC细胞识别和呈递,然后输送到颈部淋巴结的CD3T细胞,通过血液和淋巴循环到达肠道,感染肠上皮细胞[41-42]。本试验通过将PEDV 滴鼻感染5 d 龄仔猪,但在感染后测定试验动物各部位的病毒滴度时,未在肺部检测到病毒滴度的存在,而在肠道等部位检测到了病毒的存在。这印证了之前的结论,鼻部也可以作为PEDV 的感染途径,也印证了PEDV 对于不同组织有着不同的嗜性,但具体的分子机制尚未阐明[43-45]。
在这项研究中,PEDV-BJ-150 毒株在Vero-M 和ST 细胞中传代时表现出不同的滴度变化情况。这一结果表明,PEDV 病毒的细胞适应性在不同的细胞系中存在差异,并且显示出悬浮适应细胞株对于毒株的敏感程度更高。对于经过连续传代的PEDV 在不同细胞系中的细胞嗜性,这种不同敏感程度的机制尚不清楚,还需要进一步实验来验证PEDV 病毒核苷酸序列改变对细胞嗜性的影响。
总的来说,本研究报告了PEDV 病毒经过连续传代后,毒力和致病能力都发生了明显的改变。通过动物实验得出结论,经过连续传代后的PEDV-BJ-150 毒株是安全的,并且有可能作为制备减毒疫苗的候选毒株。
4 结论
综上所述,在本项研究中通过对毒株在Vero-M 细胞中连续传代后获得了一株细胞适应性更好、滴度更高的PEDV 减毒毒株。并且在感染悬浮适应株的细胞系后病毒毒价更高。我们的动物实验结果表明,PEDV-BJ-150 毒株的致病性降低,感染该毒株后,所有试验动物均没有死亡。并且说明滴鼻感染可能也是PEDV 的一种感染方式。此外,全基因组分析的结果显示PEDV-BJ-150 毒株中的氨基酸突变,这可能有助于病毒毒力减弱。因此,减毒菌株可能适用于疫苗制备。
补充表S1 见知网。
参考文献
[1]Lee C. Porcine epidemic diarrhea virus: An emerging and reemerging
epizootic swine virus[J]. Virology Journal, 2015,
12:193.
[2]Li W T, Li H, Liu Y B, et al. New variants of porcine
epidemic diarrhea virus,China,2011[J]. Emerging Infectious
Diseases, 2012, 18(8):1350-1353.
[3]Liu Q, Wang H Y. Porcine enteric coronaviruses: an updated
overview of the pathogenesis, prevalence, and diagnosis[J].
Veterinary Research Communications,2021, 45(2-3):75-86.
[4]Liu X S, Lin C M, Annamalai T, et al. Determination of the
infectious titer and virulence of an original US porcine epidemic
diarrhea virus PC22A strain[J]. Veterinary Research, 2015,
46:109.
[5]Song D, Park B. Porcine epidemic diarrhoea virus: a
comprehensive review of molecular epidemiology, diagnosis,
and vaccines[J]. Virus Genes, 2012, 44(2):167-175.
[6]Weiss S, Witkowski P T, Auste B, et al. Hantavirus in bat,
Sierra Leone[J].Emerging Infectious Diseases, 2012, 18(1):
159-161.
[7]Zhang L P, Liu X S, Zhang Q L, et al. Biological
characterization and pathogenicity of a newly isolated Chinese
highly virulent genotype GIIa porcine epidemic diarrhea virus
strain[J].Archives of Virology, 2019, 164(5):1287-1295
[8]Bosch B J, van der Zee R, de Haan C A M, et al. The
coronavirus spike protein is a class I virus fusion protein:
structural and functional characterization of the fusion core
complex[J].Journal of Virology,2003, 77(16):8801-8811.
[9]Deng F, Ye G, Liu Q Q, et al. Identification and comparison of
receptor binding characteristics of the spike protein of two
porcine epidemic diarrhea virus strains [J]. Viruses,2016,
8(3):55.
[10]Kirchdoerfer R N, Bhandari M, Martini O, et al. Structure
and immune recognition of the porcine epidemic diarrhea virus
spike protein[J]. Structure ,2021, 29(4):385-392.
[11]Li W T, van Kuppeveld F J M, He, Q G, et al. Cellular entry
of the porcine epidemic diarrhea virus[J]. Virus Research
2016, 226:117-127.
[12]Shi W, Fan W L, Bai J, et al. TMPRSS2 and MSPL
facilitate trypsin-independent porcine epidemic diarrhea virus
replication in vero cells[J]. Viruses, 2017, 9(5):114.
[13]Shirato K, Matsuyama S, Ujike M, et al. Role of proteases in
the release of porcine epidemic diarrhea virus from infected cells
[J].Journal of Virology,2011, 85(15):7872-7880.
[14]Turlewicz-Podbielska H, Pomorska-Mol M. Porcine
coronaviruses: overview of the state of the art[J]. Virologica
Sinica, 2021, 36(5):833-851.
[15]Wicht O, Li W T, Willems L, et al. Proteolytic activation of
the porcine epidemic diarrhea coronavirus spike fusion protein
by trypsin in cell culture[J]. Journal of Virology, 2014, 88
(14):7952-7961.
[16]Chen F Z, Zhu Y X, Wu M Z, et al. Comparative genomic
analysis of classical and variant virulent parental/attenuated
strains of porcine epidemic diarrhea virus[J].Viruses, 2015, 7
(10):5525-5538.
[17]Chen P F, Wang K, Hou Y X, et al. Genetic evolution
analysis and pathogenicity assessment of porcine epidemic
diarrhea virus strains circulating in part of China during 2011-
2017 [J] . Infection,Genetics and Evolution:Journal of
Molecular Epidemiology and Evolutionary Genetics in
Infectious Diseases,2019, 69:153-165.
[18]Huang Y W, Dickerman A. W, Pineyro P, et al. Origin,
evolution, and genotyping of emergent porcine epidemic
diarrhea virus strains in the United States[J]. mBio, 2013, 4
(5):e00737-e00713.
[19]Su Y F, Hou Y X, Prarat M, et al. New variants of porcine
epidemic diarrhea virus with large deletions in the spike
protein, identified in the United States, 2016-2017[J] .
Archives of Virology, 2018, 163(9):2485-2489.
[20]Vlasova A N, Marthaler D, Wang Q H, et al. Distinct
characteristics and complex evolution of PEDV strains, North
America, May 2013-February 2014[J]. Emerging Infectious
Diseases, 2014, 20(10):1620-1628.
[21]Wang P, Zhu J Y, Liu X Z, et al. Isolation and recombinant
analysis of variants of porcine epidemic diarrhea virus strains
from Beijing, China[J].Virus Disease, 2019, 30(2): 294-301.
[22]Chattha K S, Roth J A, Saif L J. Strategies for design and
application of enteric viral vaccines[J]. Annual Review of
Animal Biosciences, 2015,3:375-395.
[23]Crawford K, Lager K M, Kulshreshtha V, et al. Status of
vaccines for porcine epidemic diarrhea virus in the United
States and Canada[J].Virus Research, 2016, 226:108-116.
[24]Hou Y X, Wang Q H. Emerging highly virulent porcine
epidemic diarrhea virus: molecular mechanisms of attenuation
and rational design of live attenuated vaccines [J] .
International Journal of Molecular Sciences, 2019, 20(21):
5478.
[25]Jang G, Won H, Lee D U, et al. Assessment of the safety and
efficacy of an attenuated live vaccine based on highly virulent
genotype 2b porcine epidemic diarrhea virus in nursing piglets
[J].Veterinary Microbiology, 2019, 231:120-128.
[26]Jung K, Wang Q H, Scheuer K A, et al. Pathology of US
porcine epidemic diarrhea virus strain PC21A in gnotobiotic
pigs[J]. Emerging Infectious Diseases, 2014, 20(4) :
662-665.
[27]Chen P F, Zhao X W, Zhou S T, et al. A virulent PEDV
strain FJzz1 with genomic mutations and deletions at the high
passage level was attenuated in piglets via serial passage in vitro
[J].Virologica Sinica, 2021, 36(5):1052-1065.
[28]Niu X Y, Wang Q H. Prevention and control of porcine
epidemic diarrhea: the development of recombination-resistant
live attenuated vaccines[J].Viruses, 2022, 14(6):1317.
[29]Zuniga S, Pascual-Iglesias A, Sanchez C. M, et al. Virulence
factors in porcine coronaviruses and vaccine design[J]. Virus
Research, 2016, 226:142-151.
[30]Wu Y, Li W, Zhou Q F, et al. Characterization and
pathogenicity of Vero cell-attenuated porcine epidemic diarrhea
virus CT strain[J].Virology Journal, 2019, 16(1):121.
[31]Lin C. M, Saif L. J, Marthaler D,et al. Evolution,
antigenicity and pathogenicity of global porcine epidemic
diarrhea virus strains[J]. Virus Research, 2016, 226:20-39.
[32]Lin C. M, Hou Y X, Marthaler D. G, et al. Attenuation of an
original US porcine epidemic diarrhea virus strain PC22A via
serial cell culture passage[J] . Veterinary Microbiology,
2017, 201:62-71.
[33]Tao J, Li B Q, Cheng J H , et al. Preparation and
characterization of an attenuated porcine epidemic diarrhea
virus strain by serial passaging[J].Archives of Virology, 2018,
163(11):2997-3004.
[34]Suzuki T, Terada Y, Enjuanes L, et al. S1 subunit of spike
protein from a current highly virulent porcine epidemic diarrhea
virus is an important determinant of virulence in piglets[J].
Viruses, 2018, 10(9):467.
[35]Sato T, Takeyama N, Katsumata A, et al. Mutations in the
spike gene of porcine epidemic diarrhea virus associated with
growth adaptation in vitro and attenuation of virulence in vivo
[J]. Virus Genes, 2011, 43(1):2-78.
[36]Oka T, Saif L. J, Marthaler D, et al. Cell culture isolation and
sequence analysis of genetically diverse US porcine epidemic
diarrhea virus strains including a novel strain with a large
deletion in the spike gene[J]. Veterinary Microbiology, 2014,
173( 3-4):258-269.
[37]Hou Y X, Meulia T, Gao X, et al. Deletion of both the
tyrosine-based endocytosis signal and the endoplasmic
reticulum retrieval signal in the cytoplasmic tail of spike protein
attenuates porcine epidemic diarrhea virus in pigs[J].Journal of
Virology, 2019, 93(2):e01758-e01718.
[38]Hou Y X, Lin C M, Yokoyama M, et al. Deletion of a 197-
amino-acid region in the N-terminal domain of spike protein
attenuates porcine epidemic diarrhea virus in piglets[J].Journal
of Virology, 2017, 91(14):e00227-e00217.
[39]Alonso C, Goede D P, Morrison R. B, et al. Evidence of
infectivity of airborne porcine epidemic diarrhea virus and
detection of airborne viral RNA at long distances from infected
herds[J].Veterinary Research, 2014, 45(1): 73.
[40]Gao Q, Zhao S S, Qin T, et al. Effects of inactivated porcine
epidemic diarrhea virus on porcine monocyte-derived dendritic
cells and intestinal dendritic cells[J]. Research in Veterinary
Science, 2016, 106:149-158.
[41]Li Y C, Wu Q X, Huang L L, et al. An alternative pathway of
enteric PEDV dissemination from nasal cavity to intestinal
mucosa in swine[J]]. Nature Communications, 2018, 9(1):
3811.
[42]Niederwerder M C, Nietfeld J. C, Bai J F, et al. Tissue
localization, shedding, virus carriage, antibody response, and
aerosol transmission of Porcine epidemic diarrhea virus
following inoculation of 4-week-old feeder pigs[J]. Journal of
Veterinary Diagnostic Investigation,2016, 28(6):671-678.
[43]Hou W, Kang W J, Li Y Y, et al. Dynamic dissection of
dynein and kinesin-1 cooperatively mediated intercellular
transport of porcine epidemic diarrhea coronavirus along
microtubule using single virus tracking[J].Virulence, 2021, 12
(1):615-629.
[44]Liu C, Tang J, Ma Y M, et al. Receptor usage and cell entry
of porcine epidemic diarrhea coronavirus [J]. Journal of
Virology, 2015, 89(11):6121-6125.
[45]Luo X L, Guo L J, Zhang J, et al. Tight Junction Protein
Occludin Is a Porcine Epidemic Diarrhea Virus Entry Factor
[J].Journal of Virology, 2017, 91(10):e00202-e00217.
(编辑:吕俊俐)

