西瓜ClKNOX基因家族全基因组鉴定及组织表达分析
陈雅心 户奥锋 张力铭 刘权权 杨路明 朱华玉 刘东明

摘 要:KNOX基因家族是编码同源异型盒蛋白的轉录因子,在植物生长发育和胁迫响应中发挥着重要的调控作用。为进一步挖掘西瓜KNOX转录因子家族成员信息,探究分析其表达模式及基因功能,通过生物信息学方法对西瓜KNOX基因家族成员进行了鉴定,并对其理化性质、基因结构、系统进化及表达模式进行了分析。结果表明,西瓜基因组中共有12个KNOX基因,均定位于细胞核中,符合转录因子属性特征;KNOX1、KNOX2、ELK和Homeobox_KN这4类典型结构域均存在于大多数西瓜KNOX基因中,表明这4类结构域在KNOX基因功能中具有重要作用;系统发育分析将ClKNOX基因家族成员分为Class Ⅰ A、Class Ⅰ B、Class Ⅱ A和Class Ⅱ B 4个亚组,启动子元件分析表明,ClKNOX基因家族含有较多与生长发育和胁迫响应相关的顺式作用调控元件;组织表达分析结果表明,ClKNOX基因具有明显的组织特异表达特性,ClKNOX6和ClKNOX11在根和茎中高表达,而ClKNOX3和ClKNOX10在所有器官中表达量都相对较高,但所有ClKNOX基因在果肉中的相对表达量都较低。研究结果为进一步深入探究ClKNOX基因在生长发育中的功能和组织表达特性奠定了基础。
关键词:西瓜;KNOX基因家族;全基因组鉴定;生物信息学分析;组织表达
中图分类号:S651 文献标志码:A 文章编号:1673-2871(2024)05-018-10
Genome-wide identification and expression analysis of ClKNOX gene family in watermelon
CHEN Yaxin, HU Aofeng, ZHANG Liming, LIU Quanquan, YANG Luming, ZHU Huayu, LIU Dongming
(Horticulture College of Henan Agricultural University, Zhengzhou 450046, Henan, China)
Abstract: KNOTTED-like homeodomain(KNOX)gene family is a class of transcription factors encoding homeobox protein, which plays an important role in plant growth and stress responses. To further investigate the watermelon KNOX transcription factor family members, the bioinformatics methods was utilized to identify and analyze their physical and chemical properties, gene structure, phylogeny, and expression patterns. The study revealed the presence of 12 KNOX genes in the watermelon genome, all of which were identified in the nucleus, the result was consistent with their characteristics as transcription factors. Most KNOX genes in watermelon exhibited the typical domains KNOX1, KNOX2, ELK, and Homeobox_KN, suggesting the significance of these domains in KNOX gene functionality. Phylogenetic analysis categorized ClKNOX gene family members into four subgroups: ClassⅠA, ClassⅠB, ClassⅡA, and ClassⅡB. Analysis of promoter elements indicated that the ClKNOX gene family harbored numerous cis-acting regulatory elements associated with growth, development, and stress response. The results of tissue expression analysis revealed distinct patterns for ClKNOX gene expression. Specifically, ClKNOX6 and ClKNOX11 exhibited high expression levels in roots and stems, whereas ClKNOX3 and ClKNOX10 showed relatively high expression across all organs. However, all ClKNOX genes had low relative expression levels in flesh. These findings provide a basis for future investigations into the functional roles and tissue-specific expression patterns of ClKNOX genes in growth and development.
Key words: Watermelon; KNOX gene family; Genome-wide identification; Bioinformatics analysis; Tissue expression
西瓜[Citrullus lanatus (Thunb.) Matsum. and Nakai]為葫芦科西瓜属一年生蔓生草本植物,富含多种维生素和对人体有益的瓜氨酸、番茄红素等营养成分,无论鲜食还是制成果汁、糖果或蘸酱等都深受世界各地消费者的喜爱。同源异型盒基因家族(homeobox gene family)在动物和植物发育调控中具有重要作用,该基因家族包含高度保守的180个碱基对,编码一个含有60个氨基酸的三螺旋结构域,最早被克隆的homeobox基因来自果蝇[1],随后植物中第一个homeobox基因——Knotted-1在玉米中被分离获得[2]。近年来研究发现,该基因家族广泛存在于陆生植物类群和特定的绿藻门植物中,但在红藻植物中尚未被发现[3-4]。根据是否含有一个三氨基酸的环延伸基序,homeobox类基因被分为TALE和non-TALE两类[5],在动物中已经鉴定出4种类型的TALE基因,分别为MEIS、IRO、TGIF和PBC,而在植物中仅有KNOX(KNOTTED-like homobox)和BELL(BELL-like)两类基因被鉴定[6]。KNOX类基因通常含有4个典型结构域,分别为负责识别下游靶基因启动子序列的C端同源结构域 (Homeobox_KN)、N端的KNOX1和KNOX2以及参与转录抑制核定位信号、促进蛋白质-蛋白质相互作用的ELK结构域。由于MEIS和KNOX家族基因结构相似,KNOX1和KNOX2结构域也称为MEINOX结构域,在他们的同源盒中,在第一和第二螺旋之间还有3个额外的氨基酸 (P-Y-P)[6-7],该类结构域参与抑制靶基因表达和同二聚化。
根据序列相似性、内含子位置、系统发育关系和表达特点,KNOX类基因常分为I类、II类和M类[4,8-9]。I类亚家族包括SHOOT MERISTEMLESS (STM)、KNAT1、KNAT2和KNAT6 等4个成员。作为转录激活因子或抑制因子,I类亚家族基因在分生组织发育、叶形状的控制、激素稳态方面发挥重要作用,常在茎顶分生组织中表达[3]。STM基因在茎顶端分生组织中负责维持并调节花序结构,KNAT1在拟南芥根中也显示出细胞特异性表达模式[10],并与STM类基因具有冗余性[11-13]。KNAT2在茎顶端分生组织和根组织中表达[14-15],而KNAT6在胚胎茎顶端分生组织、茎顶端分生组织边界[14]和根韧皮部组织中表达[16]。II类亚家族基因中,KNAT3、KNAT4和KNAT5在拟南芥根中显示出细胞特异性表达模式[11]。KNAT3在幼果、花序和根中表达量最高,而KNAT4在叶片和幼果中表达量最高[17]。KNAT5在幼芽和根新发育的伸长区表达,在表皮中的表达可用来标记细胞分裂和伸长的边界[11]。KNAT7在拟南芥根的中央部位高度表达[18],在拟南芥和杨树中与次生壁形成有关[19]。M类亚家族包含KNATM基因,在叶片近端-远端模式中起作用,并在成熟器官边界的器官原基近侧域中表达[9]。
我国西瓜种植面积和产量常年居世界第一位[20]。近年来,随着分子生物学技术的不断发展和西瓜参考基因组数据的不断优化,以精准定向聚合优异基因的分子育种已成为西瓜育种技术的主要发展方向。明确重要性状调控基因是实现从传统常规育种向分子标记辅助育种转型的基础。KNOX家族基因具有组织特异性表达的特点,且在植物器官发育调控中具有重要作用。目前KNOX家族基因在葫芦科作物中的报道仅局限于黄瓜和瓠瓜,在黄瓜中有研究表明,KNOX家族基因TKN2和TKN4与APRR2表达模式一致,推测其可能共同调控黄瓜叶绿体代谢机制[21]。在瓠瓜上已经进行了KNOX全基因组水平的鉴定和组织表达分析[22],而针对西瓜还未有系统的研究报道。笔者在全基因组水平上对西瓜KNOX基因家族成员进行了鉴定分析,共筛选获得了12个西瓜KNOX基因,对其理化性质、亚细胞定位、系统进化树、染色体定位和启动子元件进行了分析,并选用遗传性状稳定的西瓜材料WT2来探究其组织表达模式,以期为进一步了解KNOX的进化特征和功能提供参考。
1 材料与方法
1.1 植物材料
试验材料西瓜高代自交系WT2,由河南农业大学园艺学院瓜类作物基因组与分子育种实验室收集保存,性状稳定遗传。试验于2023年5-11月在河南农业大学园艺学院瓜类作物基因组与分子育种实验室进行。
取籽粒饱满的WT2西瓜种子,55 ℃温汤浸种催芽后播种于穴盘中,放置在人工气候箱(28 ℃,16 h光照/8 h黑暗,相对湿度70%)内。于3叶1心时定植在温室大棚中,并合理实施浇水施肥、病虫害防治等栽培管理措施,开花期严格自交授粉。授粉后28 d分别取根、茎、叶、花、果皮和果肉等组织部位,采用随机区组法进行取样,每个样本设置3次重复。所有样品用锡箔纸包裹后放于液氮中速冻,随后存放于-80 ℃超低温冰箱备用。
1.2 方法
1.2.1 ClKNOX基因家族成员的鉴定和理化性质预测 西瓜参考基因组、蛋白序列及注释文件均来自葫芦科基因组数据库(http://cucurbitgenomics.org/organism/21),采用97103-V2版本数据[23]。拟南芥(Arabidopsis thaliana L.)参考基因组、蛋白序列、注释文件及KNOX基因家族成员数据均下载自TAIR网站(https://www.arabidopsis.org/index.jsp)。KNOX基因家族结构域的隐马尔科夫模型文件下载于pfam数据库 (http://pfam.Xfam.org/)[24]。为获得准确的ClKNOX基因鉴定信息,首先在pfam数据库下载KNOX基因家族结构域的隐马尔科夫模型文件,使用hmmer软件对西瓜蛋白数据库进行比对搜索[25];然后以拟南芥KNOX蛋白序列为探针,在西瓜基因组数据库进行搜索。将以上两种方法获得的KNOX蛋白序列在NCBI-CDD网站(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi) 进行保守结构域鉴定,得到含有KNOX结构域的候选蛋白[26]。利用在线工具ProtParamtool (https://web.expasy.org/protparam/)分析西瓜KNOX家族成员的氨基酸数目、蛋白分子质量、等电点等理化特性[27]。利用Cell-PLoc2.0(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)预测各基因的亚细胞定位[28]。
1.2.2 ClKNOX基因的染色体定位和基因结构分析 从葫芦科基因组数据库获得每条染色体的长度和ClKNOX基因的位置信息,结合TBtools软件绘制ClKNOX的染色体定位图。基于葫芦科基因组数据库(http://cucurbitgenomics.org/)中提取的西瓜所有ClKNOX基因的基因组序列和CDS序列信息,利用GSDS 2.0程序(http://gsds. gao-lab.org/)分析ClKNOX的外显子和内含子[29],利用MEME(http://meme-suite.org/tools/meme)分析ClKNOX蛋白的保守基序[30],并通过TBtools软件对基因结构和蛋白保守基序进行可视化分析[31]。
1.2.3 ClKNOX家族的系统进化和启动子顺式作用元件分析 从葫芦科基因组数据库(http://cucurbitgenomics.org/)、拟南芥数据库(https://www.arabidopsis.org/)和水稻基因组数据库(http://rice.plantbiology.msu.edu/)获得西瓜、甜瓜、黄瓜、葫芦、瓠瓜、冬瓜、水稻和拟南芥KNOX家族成员编码的氨基酸序列,利用MUSCLE (http://www.drive5.com/muscle/)软件对所有KNOX蛋白序列进行比对,并利用Jalview (http://www.jalview.org/)软件对比对结果进行编辑[32]。利用IQ-TREE (http://www.iqtree.org/) 的最大似然方法构建KNOX的系统发育树,并将Bootstrap参数设置为1000[33]。从葫芦科基因组数据库中获得ClKNOX基因编码序列上游2000 bp的基因组DNA序列,然后利用PlantCare(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)对ClKNOX基因启动子区域的顺式作用元件进行分析[34],使用TBtools软件对元件的数量与分布进行可视化分析。
1.2.4 ClKNOX基因的组织表达分析 取西瓜各组织样品置于含有液氮的研钵中迅速研磨成粉末,在研磨过程中不断向研钵中加入液氮,防止样品在空气中暴露时间过长造成降解。后续步骤参照北京华越洋RNA提取试剂盒对各组织部位总RNA进行提取,通过凝胶电泳和超微量分光光度计(P200+, Plextech, USA)检测RNA质量,用诺维赞反转录试剂盒通过两步法反转录成cDNA。
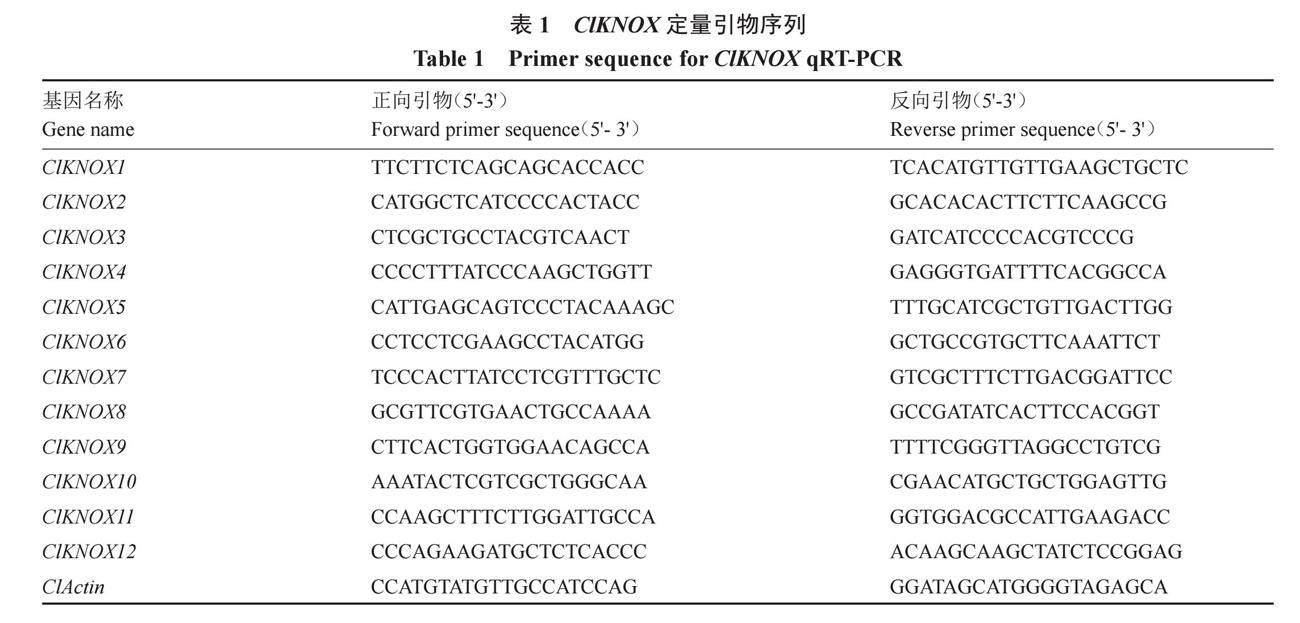
根据葫芦科基因组数据库(http://cucurbitgenomics.org/)和NCBI (https://blast.ncbi.nlm.nih.gov/Blast.cgi)[35]网站设计特异性定量引物(表1)。以ClActin为内参基因,按照荧光定量PCR试剂盒步骤,以西瓜cDNA为模板检测基因的表达量,每个检测样本设置3个生物学重复和3个技术重复,采用StepOneOlusTMReal-Time PCR仪 (Thermo Scientific, Massachusetts, USA)进行荧光定量分析,并按照2-△△Ct的方法计算基因相对表达量[36]。
2 结果与分析
2.1 ClKNOX家族基因的鉴定
KNOX蛋白包含4个保守结构域,从N端到C端依次为KNOX1、KNOX2、ELK和Homeobox_KN。西瓜中这4个结构域的基因数分别为11、9、8和21个,但仅有11个成员同时包含Homeobox_KN结构域和其他类型的结构域。为了获得准确的数据,以9个拟南芥AtKNOX蛋白序列在西瓜蛋白数据库进行blast检索,通过序列同源比对,最终从西瓜基因组数据库中获得了12个ClKNOX成员。综合上述两种分析方法,鉴定到12个西瓜ClKNOX蛋白,根据基因所在染色体位置分别命名为ClKNOX1 ~ ClKNOX12(表2)。理化特性分析结果(表2)表明,西瓜KNOX家族基因编码蛋白分子质量为13.59(ClKNOX9)~51.78 ku(ClKNOX5),编码氨基酸为116(ClKNOX9)~467个(ClKNOX10和ClKNOX5),蛋白等电点为4.44(ClKNOX8)~8.89(ClKNOX9)。亚细胞定位预测结果表明,所有ClKNOX基因都定位在细胞核中(表2),这与KNOX基因的转录因子属性特征相符。
2.2 ClKNOX家族基因的染色体位置和基因结构
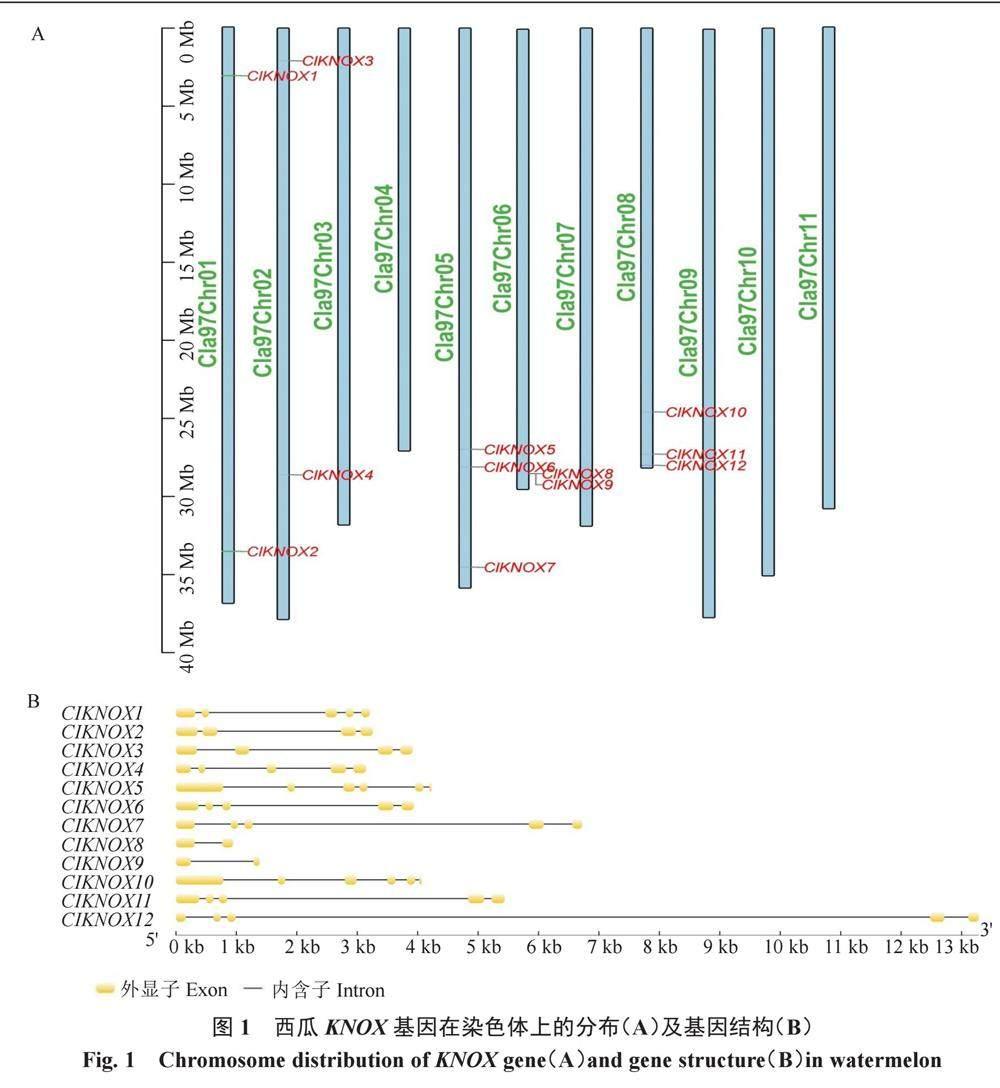
染色体定位结果表明,西瓜ClKNOX基因家族的12个家族成员集中分布于5条不同的染色体上(图1-A)。其中,5号染色体和8号染色体上分布有3个家族成员,成员数量最多,1、2、6号染色体上则分别分布有2个家族成员,其他染色体上没有ClKNOX基因。西瓜ClKNOX基因家族成员中,ClKNOX8和ClKNOX9分布于6号染色体上,且相邻物理距离仅为4.62 kb,推断其为串联重复基因簇。由图1-B可以看出,西瓜ClKNOX家族基因均含有内含子,且内含子数目和长度差异较大。其中,ClKNOX8和ClKNOX9只含有1个内含子,ClKNOX2和ClKNOX3含有3个内含子,ClKNOX1、ClKNOX4、ClKNOX6、ClKNOX7、ClKNOX11和ClKNOX12均含有4个内含子,ClKNOX5和ClKNOX10含有5个内含子。
2.3 ClKNOX家族的系统进化分析
通过对甜瓜、黄瓜、冬瓜、瓠瓜和水稻数据库的检索,并对其保守结构域分析验证,最终分别在各作物上获得的KNOX基因数量为12、11、11、12和13个。将这些基因与获得的12个ClKNOX基因和8个ATKNOX基因编码的氨基酸序列结合,利用MUSCLE、Jalview和IQ-TREE軟件构建KNOX系统发育树。由图2可以看出,所有KNOX基因家族分为Class Ⅰ和Class Ⅱ两个分支,其中KNOX Class Ⅰ又进一步分为ⅠA和ⅠB 2个亚组;KNOX Class Ⅱ分为ⅡA和ⅡB 2个亚组。12个ClKNOX基因中属于ClassⅠA的有5个,其中ClKNOX2和ClKNOX3与AT1G62360.1属于同一分支,亲缘关系最近;ClKNOX6和AT4G08150.1属于同一分支,ClKNOX9和ClKNOX11同属于Class Ⅰ A亚组。Ⅰ B亚组有4个ClKNOX成员,分别是ClKNOX4、ClKNOX7、ClKNOX8和ClKNOX12,与AT1G70510.1和AT1G23380.1同属于Ⅰ B亚组,亲缘关系较近。Class II A亚组的成员最少,仅含有ClKNOX1,该基因与AT1G62990.1亲缘关系最近,Class II B亚组含有2个ClKNOX成员,分别是ClKNOX5和ClKNOX10。
2.4 ClKNOX基因家族保守结构域分析
ClKNOX基因家族的保守结构域分析结果如图3所示,西瓜KNOX基因保守性较强的8类基序分别为Motif 1(RQVLLDWWNSHYKWPYPTEEEKVALAESTGLDQKQINNWFINQRKRHWKP)、Motif 2(IKAKILAHPLYPRLLSAYVDCQKVGAPPEVAPRLEEIRAES)、Motif 3(DPRAEDRELKDELLRKYSGYJSSLKZEFLKKKKKGKLPKDA)、Motif 4 (DPELDQ FMEAYCEMLVKYKEELSRPFREA)、Motif 5(AMEAVMACWEIEQSLQSLTGVSPGEGTGAT MSDDDDDQ)、Motif 6 (SEDMQFVVMDGAHPPYYA)、Motif 7 (TLFLNRIESQLSTLCNGSF)和Motif 8(DANLFDGSLEGHomeobox_KNAMGFGPLIPTESERS)。其中,Motif 1对应Homeobox_KN结构域,Motif 2对应KNOX1结构域,Motif 3对应ELK结构域,Motif 4对应KNOX2结构域。ClKNOX基因家族成员中,除了ClKNOX8和ClKNOX9基因,都含有4个典型的结构域,即KNOX1、KNOX2、ELK和Homeobox_KN;ClKNOX9具有Homeobox_KN和ELK结构域;ClKNOX8只含有KNOX1结构域。4个Motif基序中,Motif 1、Motif 2、Motif 3出现频次为11次,Motif 4的出现频次为10次,由此可见ClKNOX家族基因结构域高度保守。同时,从进化关系上看,亲缘关系较近的同一亚家族的ClKNOX基因在基因结构组成和蛋白结构域分布上也具有较高保守性。
2.5 ClKNOX基因的启动子顺式作用元件分析
顺式作用元件在基因表达调控中具有重要作用,相似功能的基因可能在启动子中具有类似的调控元件。为了更好地探究ClKNOX家族基因的表达调控机制,提取了ClKNOX家族成员的上游2000 bp的基因组序列,并结合PlantCARE数据库对其顺式作用元件进行预测。结果如图4所示,几乎所有ClKNOX基因的启动子区域都存在4种类型的顺式作用元件,除了基本启动子元件外,还包括植物生长发育、非生物胁迫反应、激素反应相关的启动子元件。作为与叶绿体发育紧密相关的基因家族,对其motif统计发现,启动子中多种元件与光反应相关,数量占到了总元件数量的56.45%,如Box 4、GA-motif、MRE、GT1-motif、chs-CMA1a、G-box、GATA-motif、TCT-motif、I-box、TCCC-motif、ATCT-motif、G-box、ATC-motif、3-AF1 binding site、LAMP-element、chs-CMA2a、AE-box、Sp1、TCCC-motif、AAAC-motif和ACE等启动子元件均与光反应有关,表明其基因表达调控受光的影响较大。与逆境胁迫反应相关元件约占20.97%,主要包含TC-rich repeats、ARE、LTR、MBS、WUN-motif等与植物厌氧、低温、干旱、创伤等反应相关的元件;与激素响应相关的元件约占14.92%,主要包含ABRE、TATC-box、P-box、TGA-box、TCA-element等与脱落酸、赤霉素、生长素、水杨酸等植物激素相关的元件;植物生长发育相关元件约占7.26%,其中多为MSA-like、GCN4_motif、CAT-box、O2-site、AACA_motif、AT-rich element等响应细胞周期调控、胚乳发育、分生组织表达等生长发育过程的元件。综上所述,ClKNOX基因可能在调节植物生长发育和非生物胁迫响应过程中发挥重要作用。
2.6 ClKNOX基因家族的组织表达分析
为探索ClKNOX家族基因在西瓜中的表达模式,采用荧光定量qRT-PCR的方法分析了其在西瓜WT2不同组织(根、茎、叶、花、果)中的表达量。由图5可以看出,KNOX家族基因在西瓜不同组织中的表达差异较大。在根中,ClKNOX6和ClKNOX11的表达量较高,其次为ClKNOX3和ClKNOX10,其余基因的相对表达量都较低;在茎中,ClKNOX6的相对表达量最高,ClKNOX11、ClKNOX3、ClKNOX2的相对表达量次之;在叶片中,ClKNOX3和ClKNOX10的相对表达量较高,其余基因都只有微弱表达;在花中,ClKNOX10和ClKNOX3的相对表达量较高,其次为ClKNOX6和ClKNOX2;在果皮中,ClKNOX10的相对表达量最高,其余基因的相对表达量都较低;在果肉中,大多ClKNOX基因的相对表达量都较低,ClKNOX8和ClKNOX12不表达。总体而言,ClKNOX6和ClKNOX11具有相似的表达模式,均在根和茎中具有相对较高的表达量,在其他器官中的表达量均较低。ClKNOX3和ClKNOX10在所有检测组织中表达量均相对较高,而ClKNOX5和ClKNOX7在所有组织中几乎都不表达。
3 讨论与结论
作为一类在植物中广泛存在的转录因子家族,KNOX基因家族在植物的发育和形态建成中起着重要的调控作用,其家族成员在不同的组织和发育阶段中表达具有差异性。随着高通量测序技术的发展,KNOX家族基因已在多个物种中广泛研究,但目前关于西瓜ClKNOX基因家族尚未有研究报道。笔者结合KNOX基因家族的典型结构域特征和西瓜基因组数据库信息,首次对ClKNOX基因家族进行了鉴定,并对其基因结构、蛋白質保守结构域、系统进化和基因表达模式进行分析,为进一步研究ClKNOX基因的功能奠定了基础。
KNOX基因广泛存在于植物基因组中,且在不同物种中的数量差异较大,拟南芥中有8个[37],瓠瓜中有12个[22],水稻中有13个[38],杨树中有15个[39],玉米[40]和苹果[41]中各有22个,笔者共鉴定出12个ClKNOX基因,数量与同为葫芦科植物的瓠瓜相同,与拟南芥、苹果、玉米的数量差异较大,这种现象通常是由基因进化过程中的部分重复和串联重复等造成的[42]。进化分析结果表明,ClKNOX中存在多个序列相似度较高的基因,推断其为同源基因。但染色体位置分析结果显示仅有2个ClKNOX基因为串联重复,推断ClKNOX基因家族数目的扩增主要由部分重复导致。亚细胞定位预测结果表明,12个ClKNOX基因全部定位于细胞核,这与瓠瓜等作物相似[22],并符合其转录因子的属性,表明其在生长发育中可能发挥着调控功能。
拟南芥KNOX基因Class Ⅰ亚家族有4个成员 (KNAT1、 KNAT2、 KNAT6 和STM),西瓜Class Ⅰ亚家族有9个成员基因;拟南芥KNOX基因Class Ⅱ亚家族也有4个成员 (KNAT3、KNAT4、 KNAT5 和KNAT7),西瓜Class Ⅱ亚家族基因成员为3个,说明在进化过程中西瓜KNOX的Class Ⅰ亚家族成员发生数量的扩增,而Class Ⅱ亚家族成员没有发生大量扩增,由此推断Class Ⅰ亚家族基因可能更容易发生基因重复。前人研究表明,基因结构与基因表达方式有密切的对应关系[8]。Class Ⅰ类成员(ClKNOX3、ClKNOX6、ClKNOX11)主要在根和茎中表达水平较高,在其他组织的表达水平均较低,这与其他植物KNOXⅠ类基因的表达模式相符合,推测这些基因在西瓜分生组织的发育和调控中起着重要作用。西瓜12个KNOX基因家族成员中,9个都含有KNOX1、KNOX2、ELK和Homeobox_KN这4个典型的结构域,但ClKNOX9只含有Homeobox_KN和ELK结构域,而ClKNOX8只含有KNOX1结构域。缺失部分保守结构域的ClKNOX9和ClKNOX8可能是假基因,假基因的存在可能是基因复制不完全的结果,通常不具有完整的基因功能,但这些假基因的存在又为将來新基因的进化提供了可能[43-44]。
WT2作为野生型亲本,通过基因定位技术配置遗传群体,已经获得了调控西瓜黄化[45]、绿花[46]、短下胚轴和果皮腹纹[47]等基因,是研究基因组织表达特性的理想材料。前期研究表明,KNOX基因在植物不同组织器官中的表达具有差异性[22,40,48]。笔者在本研究中发现,ClKNOX基因家族成员在西瓜不同组织中的相对表达量同样具有多样性和较大的差异性。ClKNOX3和ClKNOX10在西瓜所有器官中表达量均相对较高,推测这两个基因可能参与西瓜各主要器官的生长发育进程。而其他少数ClKNOX基因(如ClKNOX6和ClKNOX11)在根和茎中的表达水平相对较高,在叶、花和果实中的表达量普遍较低,这一表达水平与小麦[38]、瓠瓜[22]、大豆[49]等相似。ClKNOX6在根和茎中表达量最高,系统发育分析表明,ClKNOX6与瓠瓜Lsi04G014450位于同一进化支,而在瓠瓜中的研究表明,Lsi04G014450在根和茎中的表达量同样最高[22],两基因具有相似的表达模式,推测该基因可能参与了西瓜根系的生长发育和茎顶端分生组织的分化。ClKNOX10是ClKNOX家族成员中唯一在果皮中表达量较高的基因,推测其可能参与西瓜果皮的颜色调控。ClKNOX5和ClKNOX7在所有组织中几乎都不表达,推测其可能存在功能缺失。ClKNOX基因在不同组织中表达模式的差异性暗示了其功能表达的差异性,这为进一步解析ClKNOX基因在西瓜生长发育和形态建成中的作用奠定了重要基础。
综上所述,ClKNOX基因家族的组织表达模式和启动子元件分析结果表明,该基因家族成员在进化过程中可能发生了功能分化,虽然大多数ClKNOX基因都含有KNOX1、KNOX2、ELK和Homeobox_KN这4类典型结构域,但不同ClKNOX基因在不同组织中表达模式的多样性暗示了其功能的差异性,ClKNOX3、ClKNOX6和ClKNOX11可能与根和茎的发育相关,而ClKNOX3和ClKNOX10则可能与叶片和花的发育更加密切,不同ClKNOX基因在不同组织的特异性表达和模式为进一步深入探究ClKNOX基因在生长发育中的功能奠定了基础。
参考文献
[1] GEHRING W J,AFFOLTER M, BURGLIN T.Homeodomain proteins[J].Annual Review of Biochemistry,1994,63:487-526.
[2] VOLLBRECHT E,VEIT B,SINHA N,et al.The developmental gene Knotted-1 is a member of a maize homeobox gene family[J].Nature,1991,350(6315):241-243.
[3] HAKE S,SMITH H M S,HOLTAN H,et al.The role of KNOX genes in plant development[J].Annual Review of Cell and Developmental Biology,2004,20:125-151.
[4] GAO J,YANG X,ZHAO W,et al.Evolution,diversification,and expression of KNOX proteins in plants[J].Frontiers in Plant Science,2015,6:882.
[5] LEE J H,LIN H W,JOO S,et al.Early sexual origins of homeoprotein heterodimerization and evolution of the plant KNOX/BELL family[J].Cell,2008,133(5):829-840.
[6] BURGLIN T R.Analysis of TALE superclass homeobox genes(MEIS,PBC,KNOX,Iroquois,TGIF) reveals a novel domain conserved between plants and animals[J].Nucleic Acids Research,1997,25(21):4173-4180.
[7] CHEN H,ROSIN F M,PRAT S,et al.Interacting transcription factors from the three-amino acid loop extension superclass regulate tuber formation[J].Plant Physiology,2003,132(3):1391-1404.
[8] KERSTETTER R,VOLLBRECHT E,LOWE B,et al.Sequence analysis and expression patterns divide the maize knotted1-like homeobox genes into two classes[J].The Plant Cell,1994,6(12):1877-1887.
[9] MAGNANI E,HAKE S.KNOX lost the OX:The Arabidopsis KNATM gene defines a novel class of KNOX transcriptional regulators missing the homeodomain[J].Plant Cell,2008,20(4):875-887.
[10] LONG J A,MOAN E I,MEDFORD J I,et al.A member of the KNOTTED class of homeodomain proteins encoded by the STM gene of Arabidopsis[J].Nature,1996,379(6560):66-69.
[11] TRUERNIT E,SIEMERING K R,HODGE S,et al.A map of KNAT gene expression in the Arabidopsis root[J].Plant Molecular Biology,2006,60(1):1-20.
[12] DOUGLAS S J,CHUCK G,DENGLER R E,et al.KNAT1 and ERECTA regulate inflorescence architecture in Arabidopsis[J].Plant Cell,2002,14(3):547-558.
[13] VENGLAT S P,DUMONCEAUX T,ROZWADOWSKI K,et al.The homeobox gene BREVIPEDICELLUS is a key regulator of inflorescence architecture in Arabidopsis[J].Proceedings of the National Academy of Sciences,2002,99(7):4730-4735.
[14] BELLES-BOIX E,HAMANT O,WITIAK S M,et al.KNAT6:An Arabidopsis homeobox gene involved in meristem activity and organ separation[J].Plant Cell,2006,18(8):1900-1907.
[15] HAMANT O,NOGUE F,BELLES-BOIX E,et al.The KNAT2 homeodomain protein interacts with ethylene and cytokinin signaling[J].Plant Physiology,2002,130(2):657-665.
[16] DEAN G,CASSON S,LINDSEY K.KNAT6 gene of Arabidopsis is expressed in roots and is required for correct lateral root formation[J].Plant Molecular Biology,2004,54(1):71-84.
[17] SERIKAWA K A,MARTINEZ-LABORDA A,ZAMBRYSKI P.Three knotted1-like homeobox genes in Arabidopsis[J].Plant Molecular Biology,1996,32:673-683.
[18] BIRNBAUM K,SHASHA D E,WANG J Y,et al.A gene expression map of the Arabidopsis root[J].Science,2003,302(5652):1956-1960.
[19] LI E Y,BHARGAVA A,QIANG W Y,et al.The Class II KNOX gene KNAT7 negatively regulates secondary wall formation in Arabidopsis and is functionally conserved in Populus[J].New Phytologist,2012,194(1):102-115.
[20] 張磊,武竞春,张海英,等.西瓜重要性状功能基因研究进展[J].园艺学报,2023,50(12):2748-2764.
[21] LIU H,JIAO J Q,LIANG X J,et al.Map-based cloning,identification and characterization of the w gene controlling white immature fruit color in cucumber(Cucumis sativus L.)[J].Theoretical and Applied Genetics.2016,129(7):1247-1256.
[22] 徐晓燕,黄小忠,刘万仙,等.瓠瓜KNOX基因家族全基因组鉴定及组织表达分析[J].西北农林科技大学学报(自然科学版),2024,52(3):113-122.
[23] GUO S G,ZHANG J G,SUN H H,et al.The draft genome of watermelon(Citrullus lanatus)and resequencing of 20 diverse accessions[J].Nature Genetics,2013,45(1):51-58.
[24] MISTRY J,CHUGURANSKY S,WILLIAMS L,et al.Pfam:The protein families database in 2021[J].Nucleic Acids Research,2021,49(D1):412-419.
[25] FINN R D,CLEMENTS J,EDDY S R.HMMER web server:Interactive sequence similarity searching[J].Nucleic Acids Research,2011,39(2):29-37.
[26] MARCHLER-BAUER A,LU S,ANDERSON J B,et al.CDD:A conserved domain database for the functional annotation of proteins[J].Nucleic Acids Research,2010,39(1):225-229.
[27] GASTEIGER E,GATTIKER A,HOOGLAND C,et al.ExPASy:The proteomics server for in-depth protein knowledge and analysis[J].Nucleic Acids Research,2003,31(13):3784-3788.
[28] CHOU K C,SHEN H B.Cell-PLoc:A package of web-servers for predicting subcellular localization of proteins in various organisms[J].Nature Protocols,2008,3(2):153-162.
[29] HU B,JIN J,GUO A Y,et al.GSDS 2.0:An upgraded gene feature visualization server[J].Bioinformatics,2015,31(8):1296-1297.
[30] BAILEY T L,JOHNSON J,GRANT C E,et al.The MEME suite[J].Nucleic Acids Research,2015,43(W1):39-49.
[31] CHEN C J,WU Y,LI J W,et al.TBtools-II:A “one for all,all for one” bioinformatics platform for biological big-data mining[J].Molecular Plant,2023,16(11):1733-1742.
[32] EDGAR R C.MUSCLE:Multiple sequence alignment with high accuracy and high throughput[J].Nucleic Acids Research,2004,32(5):1792-1797.
[33] MINH B Q,SCHMIDT H A,CHERNOMOR O,et al.IQ-TREE 2:New models and efficient methods for phylogenetic inference in the genomic era[J].Molecular Biology and Evolution,2020,37(8):2461.
[34] LESCOT M,DEHAIS P,THIJS G,et al.PlantCARE,a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences[J].Nucleic Acids Research,2002,30(1):325-327.
[35] WOFFELMEN C.DNAMAN for Windows,Version 5.2.10:Lynon Biosoft[D].Leiden,The Netherlands:Institute of Molecular Plant Sciences,Leiden University,2004.
[36] LIVAK K J,SCHMITTGEN T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2?ΔΔCT method[J].Methods,2001,25(4):402-408.
[37] 宋晓峰,鲁成凯,董晓娜,等.花生KNOX基因家族的全基因组鉴定及组织表达分析[J].山东农业科学,2023,55(8):1-10.
[38] 宋宁宁,梁慧慧,安义伟,等.小麦KNOX基因家族鉴定与生物信息学分析[J].分子植物育种,2022,20(7):2105-2117.
[39] XIONG H,SHI A,WU D,et al.Genome-Wide identification,classification and evolutionary expansion of KNOX gene family in rice (Oryza sativa) and populus (Populus trichocarpa)[J].American Journal of Plant Sciences,2018,9(6):1071-1092.
[40] 赵长江,宋巨奇,都梦翔,等.玉米KNOX基因家族鉴定及组织和逆境表达分析[J].西北植物学报,2021,41(7):1109-1119.
[41] JIA P,XING L B,ZHANG C G,et al.MdKNOX15,a class I knotted-like transcription factor of apple,controls flowering and plant height by regulating GA levels through promoting the MdGA2ox7 transcription[J].Environmental and Experimental Botany,2021,185:104411.
[42] FLAGEL L E,WENDEL J F.Gene duplication and evolutionary novelty in plants[J].New Phytologist,2009,183(3):557-564.
[43] LIU J J,EKRAMODDOULLAH A K M.Isolation,genetic variation and expression of TIR-NBS-LRR resistance gene analogs from western white pine (Pinus monticola Dougl.ex.D.Don.)[J].Molecular Genetics and Genomics,2004,270(5):432-441.
[44] NAIR R A,THOMAS G.Isolation,characterization and expression studies of resistance gene candidates (RGCs) from Zingiber spp.[J].Theoretical and Applied Genetics,2007,116(1):123-134.
[45] 段世享.西瓜植株黃化基因Clyp的图位克隆与功能验证[D].郑州:河南农业大学,2023.
[46] 孙东玲.西瓜绿花基因ClGF和“星月西瓜”基因MS的定位研究[D].郑州:河南农业大学,2022.
[47] 王登科.西瓜果皮覆纹基因(ClGS)和短下胚轴基因(Clsh)的精细定位[D].郑州:河南农业大学,2022.
[48] 叶明辉,赵朋,牛洋,等.马铃薯同源异形框基因家族的鉴定和表达分析[J].农业生物技术学报,2021,29(2):224-239.
[49] 李方正,杨素欣,吴春霞,等.大豆KNOX基因家族的结构和表达分析[J].植物学报,2012,47(3):236-247.

