甾醇脱氢酶的基因挖掘、分子改造及其催化合成熊去氧胆酸
游智能 张仙 李春秀 许建和
摘 要 本文介绍近年来利用以甾醇脱氢酶为核心元件的生物合成法制备熊去氧胆酸的研究进展,并提出了该技术进一步发展所面临的挑战和未来的研究方向,旨在为熊去氧胆酸的生物合成研究提供参考。
关键词 熊去氧胆酸 甾醇脱氢酶 生物合成
中图分类号:Q81; Q819 文献标志码:A 文章编号:1006-1533(2024)07-0016-08
引用本文 游智能, 张仙, 李春秀, 等. 甾醇脱氢酶的基因挖掘、分子改造及其催化合成熊去氧胆酸[J]. 上海医药, 2024, 45(7): 16-23.
基金项目:国家自然科学基金项目(21871085、31971381)
Gene mining and molecular modification of hydroxysteroid dehydrogenase and its catalytic synthesis of ursodeoxycholic acid
YOU Zhineng1, ZHANG Xian1, LI Chunxiu2, XU Jianhe2
(1. Shanghai Bioforany Biotechnology Co., Ltd., Shanghai 200030, China; 2. School of Biotechnology, East China University of Science and Technology, Shanghai 200237, China)
ABSTRACT The research progress in the biosynthesis of ursodeoxycholic acid with hydroxysteroid dehydrogenase as the core element in recent years is reviewed, and the challenges and future research directions for further development of this technology are proposeed, aiming to provide reference for the ursodeoxycholic acid biosynthesis.
KEY WORDS ursodeoxycholic acid; hydroxysteroid dehydrogenase; biosynthesis
熊去氧膽酸(ursodeoxycholic acid, UDCA)是传统名贵中药熊胆粉的主要活性成分,其化学名为3α,7β-二羟基-5β-胆甾烷-24-酸。UDCA由于药用价值高、安全性好,广泛用于利胆溶石和治疗各种肝胆疾病[1],临床需求量日渐增大,致使对其药理机制和合成制备相关的研究也趋增多。1997年,UDCA还成为第一个被美国FDA批准用于治疗原发性胆汁性肝硬化的药物[2]。最近有研究发现,UDCA类药物可通过其本身具有的抑制转录因子法尼醇受体的作用来关闭血管紧张素转化酶受体通道,而该通道是新型冠状病毒及其各种变体感染人体细胞的主要通道。换言之,UDCA类药物在预防新型冠状病毒感染方面也有很大的临床应用潜力[3]。
当前,UDCA主要有3个来源,分别是“活熊取胆”、化学合成和生物合成。其中,“活熊取胆”是从饲养的黑熊或棕熊体内抽取熊胆汁并提取天然UDCA的方法,这种方法既违背自然伦理且不可持续,也不利于国家对于濒危动物(黑熊是国家二级保护动物)的保护。利用化学方法人工合成UDCA的原料主要有2类:一类是来源于动物的胆汁酸类化合物,包括鹅去氧胆酸(chenodeoxycholic acid, CDCA)、胆酸(cholic acid, CA)、石胆酸(lithocholic acid, LCA)、猪胆酸(hyocholic acid, HCA)和猪去氧胆酸(hyodesoxycholic acid, HDCA);另一类是来源于植物的非胆汁酸类化合物,主要包括黄体酮和雄甾烯二酮。然而,化学合成方法通常需要进行多个保护与脱保护反应步骤,且化学反应条件相对苛刻,并会用到有毒和危险试剂(如CrO3和吡啶)。随着生物技术的迅猛发展,利用生物方法合成UDCA的研究越来越多。
生物催化和生物转化具有选择性高、反应条件温和、绿色环保等优点,是最有望拓展化学工业绿色可持续发展的技术,而酶则是生物催化技术的核心元件[4-5]。如图1所示,当前工业上以CDCA为起始原料全酶法合成 UDCA和以CA为起始原料化学-酶法合成UDCA过程中均要用到甾醇脱氢酶(hydroxysteroid dehydrogenases, HSDHs)。近年来,国内外学者在对UDCA酶法合成过程中所需关键酶HSDHs的基因挖掘、分子改造及其合成应用等方面进行了大量的研究,并取得了一些可喜的研究进展。
1 HSDHs的分类与反应机制
HSDHs属于NAD(P)H依赖型氧化还原酶,可催化胆汁酸甾体结构骨架上羰基的还原或羟基的氧化,二者互为可逆过程[6]。HSDHs具有很好的立体选择性和区域选择性,故通常按照它们作用的选择性来进行分类,如7α-HSDH能够专一性地将胆汁酸甾体结构骨架上的7α-羟基氧化为羰基或将C7位羰基还原为7α-羟基,7β-HSDH则可专一性地将胆汁酸甾体结构骨架上的C7位羰基还原为7β-羟基或将7β-羟基氧化为羰基[7]。UDCA酶法合成过程中所需的关键酶主要有3α-HSDH(EC 1.1.1.50)、12α-HSDH(EC 1.1.1.176)、7α-HSDH(EC 1.1.1.159)和7β-HSDH(EC 1.1.1.201)。
3α-HSDH最早發现于哺乳动物细胞中,后在肠道细菌中也检测到了3α-HSDH活性[6]。目前,来源于大鼠肝脏、Comamonas testosteroni和Eggerthella sp. CAG: 298的3α-HSDH的基因序列已被成功克隆并异源表达[8-9]。大鼠肝脏来源的3α-HSDH属于醛酮还原酶超家族(aldoketo reductase superfamily, AKR)[10]。His-Tyr-Lys是AKR的催化三联体,它们位于AKR的N端,Tyr和Lys为催化残基,Asp负责降低催化残基的酸度系数,His接收来自于Tyr的电子[10]。AKR在Lys和Tyr的作用下将底物醇的[H]传递给NAD(P)+,即可完成底物醇氧化转化为酮的过程[10]。
除了大鼠肝脏来源的3α-HSDH属于AKR,来源于Comamonas testosteroni的3α-HSDH以及12α-HSDH、7α-HSDH和7β-HSDH均属于短链脱氢酶超家族(shortchain dehydrogenase/reductase superfamily, SDR)[11]。SDR的催化三联体是Ser-Tyr-Lys,其催化的反应过程遵循顺序Bi-Bi机制,即辅酶NAD(P)H先与酶蛋白结合,随后底物再进入酶的活性口袋中;Ser通过与底物形成氢键识别并稳定底物,NAD(P)H的[H]转移至底物的羰基碳上形成中间态,同时Tyr作为催化酸将[H]转移至底物的羰基氧上形成产物醇,失去的[H]将从溶剂中获取,Lys则通过静电相互作用降低Tyr的酸度系数;反应结束后,产物醇从酶的活性口袋中被释放出来,随即NAD(P)+与酶分离[12]。
2 HSDHs的基因挖掘
尽管许多HSDHs已被发现并初步应用于UDCA的酶法合成研究,但具有基因序列报告的HSDHs依然较少。不少研究致力于发现新的HSDHs基因,以期获得性能更优的天然HSDHs。传统的从土壤中筛选具有相应催化能力的目标野生型菌株,再从中获取酶基因的方法,不仅步骤多、周期长,而且效率低下,不能满足当前的快速发掘生物催化剂的要求。随着生物信息学的迅速发展以及基因与蛋白质数据库的快速扩容,利用已报告的有活性的功能序列作为探针,在宏大的基因组数据中搜索并钓取潜在的目标酶基因,随后将其克隆表达获得酶制剂,再通过功能检测即可望筛选得到具有相关催化活性的目标酶。这种基于公开数据库的基因组挖掘技术具有操作简便、周期短等优点,在工业用新酶开发中得到了广泛应用。
2015年,Zheng等[13]利用来源于Collinsella aerofaciens和Clostridium absonum的2种7β-HSDH的蛋白序列作为探针,在美国国家生物技术信息中心(National Center for Biotechnology Information, NCBI)数据库中进行Blastp比对分析,最终从Ruminococcus torques挖掘得到1种新的Rt7β-HSDH,后者的比活性为8.6 U/mg。这是第一次利用数据库挖掘方法发现的新型HSDH。2017年,Song等[14]利用宏基因组技术,在黑熊的排泄物中鉴定出5种新型7α-HSDH(S1-a-1、S1-a-2、H1-a-1、H1-a-2和Y1-a-1)和1种新型7β-HSDH(Y1-b-1),它们均来源于黑熊的肠道微生物,丰富了7α/7β-HSDH酶库。
2019年,Tonin等[15]利用数据库挖掘方法,分别从Stenotrophomonas maltophilia和Lactobacillus spicheri中克隆出Sm7α-HSDH和Ls7β-HSDH,它们的比活性分别为430和3.1 U/mg。值得一提的是,Ls7β-HSDH是第一种具有基因序列报告的天然NADH依赖型7β-HSDH。在这之前,所有具有基因序列报告的7β-HSDH均为NADPH依赖型。此外,虽然来源于Xanthomonas maltophilia的7β-HSDH和7α-HSDH均从野生菌株中被分离、纯化出来并被表征为NADH依赖型,但它们的基因序列并未公开[16]。
2019年,Chen等[17]利用Ec7α-HSDH和Cae7β-HSDH的蛋白序列作为探针,在NCBI数据库中进行同源序列搜索,最终挖掘得到来源于Brevundimonas sp.的Bs7α-HSDH和来源于Clostridium sp. Marseille的Cm7β-HSDH,它们的比活性分别为471和26.5 U/mg。
2020年,Bertuletti等[18]利用宏基因组技术,从极端微生物中克隆出8种新型HSDHs,包括1种12α-HSDH、3种7α-HSDH和4种7β-HSDH,并研究了这些酶的底物混乱性。研究结果不仅扩大了HSDHs酶库,而且拓展了HSDHs的应用范围(不再局限于胆汁酸类化合物的生物合成)。
相较于7α-HSDH和7β-HSDH,对12α-HSDH的基因挖掘研究则较少。2019年,Tonin等[19]从Eggerthella lenta挖掘到一种新的El12α-HSDH,后者是第一种有蛋白序列报告的NADH依赖型12α-HSDH,其比活性达到76 U/mg。同年,Shi等[20]提出了一种结构导向的基因挖掘(structure-guided genome mining, SGGM)策略:首先通过分析比较NADPH依赖型Cl12α-HSDH(图2A)和NADH依赖型El12α-HSDH(图2B)的辅酶结合口袋空间结构的差异,并对保守的辅酶结合基序进行比对,解析比较12α-HSDH与NADP+和NAD+的作用机制,再基于此从NCBI数据库中定向挖掘到一种来源于Rhodococcus ruber的NADH依赖型Rr12α-HSDH(图2C),其比活性达289 U/mg,为目前报告的活力最高的12α-HSDH。
SGGM策略較传统的基因挖掘方法更加理性,准确性也更高。基于SGGM策略,2023年,Huang等[21]从Candidatus Ligilactobacillus excrementigallinarum挖掘到一种新的NADH依赖型Cle7β-HSDH,其比活性达9.6 U/mg,是当时已报告的活力最高的NADH依赖型7β-HSDH。
3 HSDHs的分子改造
天然酶分子往往不能耐受工业反应器中苛刻的操作环境,高温、高pH、高浓度的底物或产物和有机溶剂等均可能会抑制酶的催化性能[22]。因此,需要对天然酶进行分子改造,以获得符合工业应用要求的优良突变体。
目前,对HSDHs的分子改造研究主要集中在对7α-HSDH和7β-HSDH的改造方面。2017年,Bakonyi等[23]通过定点突变(A37D),将来源于Clostridium difficile的Cd7α-HSDH的辅酶偏好性由NADP+改造为NAD+,但酶活性下降了近10倍。这是首次对HSDHs的辅酶特异性实施改造。2019年,Huang等[24]对来源于Clostridium absonum的Cab7α-HSDH进行定向进化,所获双突变体(Q255L/C260S)的比活性由母本的5.7 U/mg提高至36.8 U/mg,且突变体对底物的耐受性也有很大的提高。2021年,Liu等[25]发现,对于来源于Brucella melitensis的Bm7α-HSDH,一个点突变(I258M)就可同时提高酶的催化效率、对辅酶的亲和力和热稳定性。Liu等[26]随后还研究了末端修饰对Bm7α-HSDH的催化效率和热稳定性的影响,为HSDHs的分子改造提供了新的思路。2023年,Pan等[27]对St-2-2 7α-HSDH的255位异亮氨酸的改造研究亦发现,仅这一个点的突变就可以改变酶的底物偏好、催化活性和热稳定性。
2017年,Zheng等[28]首次提出了多目标协同进化(multiobjective directed evolution)策略,对来源于Ruminococcus torques的Rt7β-HSDH的活性、热稳定性和最适pH同时进行进化,所获双突变体(T189V/ V207M)的比活性较母本提高5.5倍,热稳定性提高3倍,且最适pH从6.5转变为7.5。随后,You等[29]又通过对Rt7β-HSDH辅酶保守结构域的序列比对和辅酶结合位点的结构分析,建立了快速、定向创制NADH依赖型HSDHs的辅酶特异性反转之小巧突变库设计(cofactor specificity reversal: small-and-smart library design)方法,实现了Rt7β-HSDH辅酶偏好性由NADPH到NADH约953 000倍的理性反转,将酶对NADH的比活性提高了223倍,由此获得了第一种NADH依赖型非天然7β-HSDH工程酶。
2019年,Tonin等[15]利用定点饱和突变技术,通过对来源于Clostridium absonum的Cab7β-HSDH的T17、E18、G39、R40和R41 5个位点进行突变分析,最终得到一种四重突变体(T17A/G39D/R40L/R41N),该突变体的辅酶偏好性由母本的NADPH依赖型被改造为 NADH依赖型,但其比活性下降了3.2倍。
迄今为止,尚未见有对3α-HSDH和12α-HSDH的分子改造研究报告。
4 HSDHs在UDCA合成中的应用
通过对HSDHs的基因挖掘和分子改造,研究人员得到了一系列具有一定工业应用潜力的酶催化剂,并将它们应用于UDCA及其衍生物的酶法合成。2019年,Tonin等[15]和Zhang等[30]各自将7α-HSDH催化的氧化反应和7β-HSDH催化的还原反应耦联起来,构建成一个氧化还原自给自足的双酶“借氢”级联反应,实现了辅酶的内部自循环而不需要额外构建辅酶循环系统,可将CDCA直接转化为UDCA(图3)。Xu等[31]和Ji等[32]则分别利用这一氧化还原自给自足的双酶“借氢”级联反应体系,将来源于鸡胆粉的牛磺鹅去氧胆酸直接转化为牛磺熊去氧胆酸。但是,由于受到化学平衡的限制,最终的转化率只能达到70%~92%,产物分离纯化的难度较大。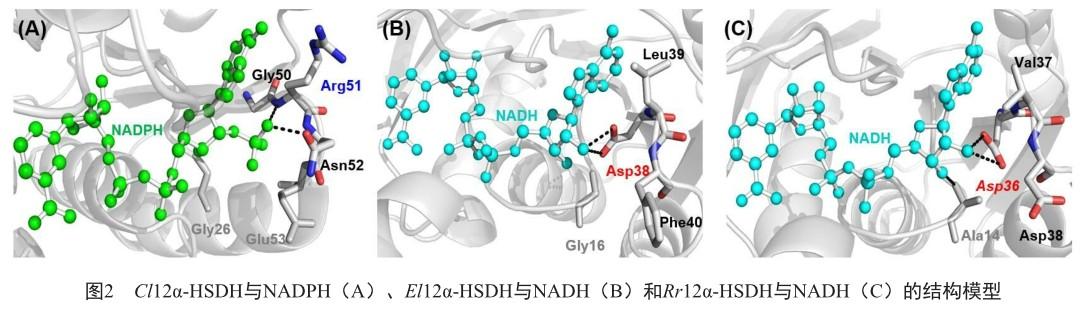
为了将CDCA完全转化为UDCA,通常需要构建两步辅酶相互独立的酶法级联反应体系,每一步都耦联合适的辅酶再生系统,以促进辅酶的循环再生和底物的完全转化。2015年,Zheng等[13]通过构建两步辅酶相互独立的酶法级联反应体系,成功地将底物CDCA转化为UDCA:如图4A所示,选用乳酸脱氢酶(lactate dehydrogenase, LDH)/丙酮酸作为氧化反应的辅酶再生系统,CDCA在7α-HSDH的作用下完全转化为中间产物7-羰基-LCA;同时,以葡萄糖脱氢酶(glucose dehydrogenase, GDH)/葡萄糖作为还原反应的辅酶循环系统,中间产物7-羰基-LCA在7β-HSDH的作用下转化为UDCA。2018年,Zheng等[33]又进一步地将氧化反应过程中的2种酶(7α-HSDH和LDH)和还原反应过程中的2种酶(7β-HSDH和GDH)分别共固定化,进而设计出一种填充床式串联反应器,实现了从CDCA到UDCA的连续酶法转化,UDCA的时空产率(space-time yield, STY)达到88.5 g/(L·d)(图4B)。

2019年,Chen等[17]报告了一种通过“一锅”级联反应体系将CDCA转化为UDCA的方法,即利用黄素还原酶(flavin reductase, FR)/黄素单核苷酸(flavin mononucleotide, FMN)和醇脱氢酶(alcohol dehydrogenase, ADH) /异丙醇系统分别再生NAD+和NADPH,二者互不干扰。如图5所示,以“一锅两步”反应模式,可以将30 mmol/L CDCA完全转化为UDCA,分离得率为92%;以“一锅一步”反应模式,只能将 12.5 mmol/L CDCA完全转化为UDCA,分离得率为78%。造成这种差异的原因可能是高浓度的产物对反应体系中的酶产生了抑制作用。
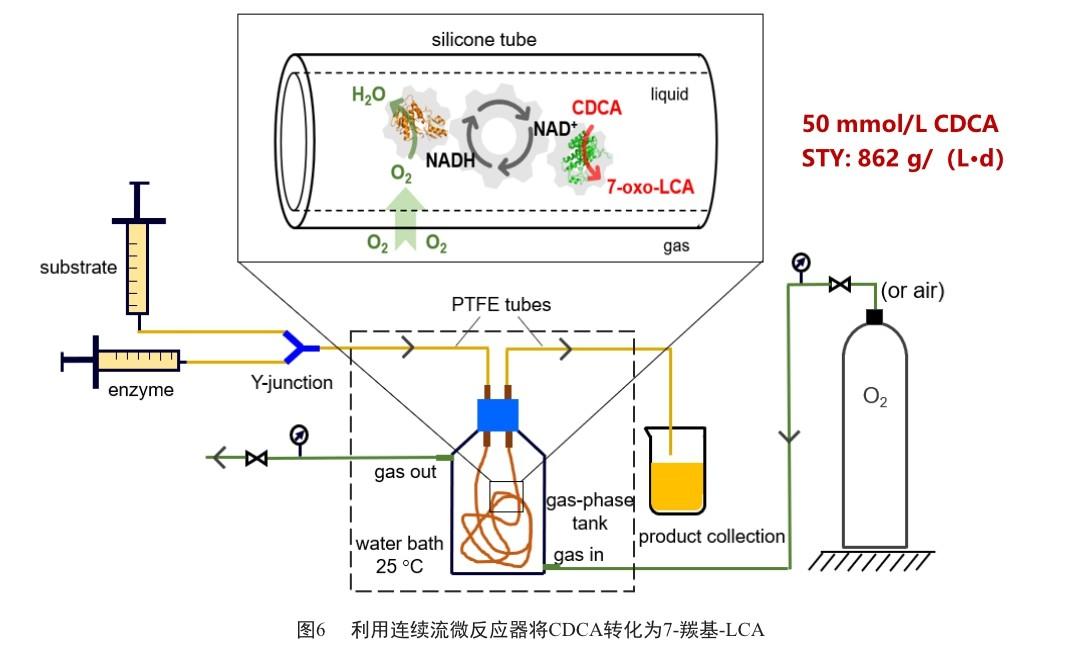
2022年,Li等[34]将7α-HSDH催化的反应中所使用的辅酶再生系统LDH/丙酮酸替换为更绿色清洁的NAD(P)H氧化酶/氧气,同时通过构建微反应器系统提高反应体系的供氧水平(图6),由此强化NAD(P)H氧化酶的再生效率和7α-HSDH的反应效率。当底物载量为50 mmol/L时,该反应体系可以在30 min内将92%的CDCA转化为7-羰基-LCA,STY达到862 g/(L·d),较搅拌式批反应器提高约96倍,且辅酶的总转换数也提高了10倍,酶消耗数降低7倍。同年,Li等[35]又构建了一个填充床式连续流反应器系统(图7),利用共固定化的 7β-HSDH和异丙醇脱氢酶(isopropanol dehydrogenase, IPADH)将7-羰基-LCA转化为UDCA,最终STY达到1 040 g/(L·d)。此外,通过辅酶的回收再利用,成功地将UDCA的生产成本降低了约21%。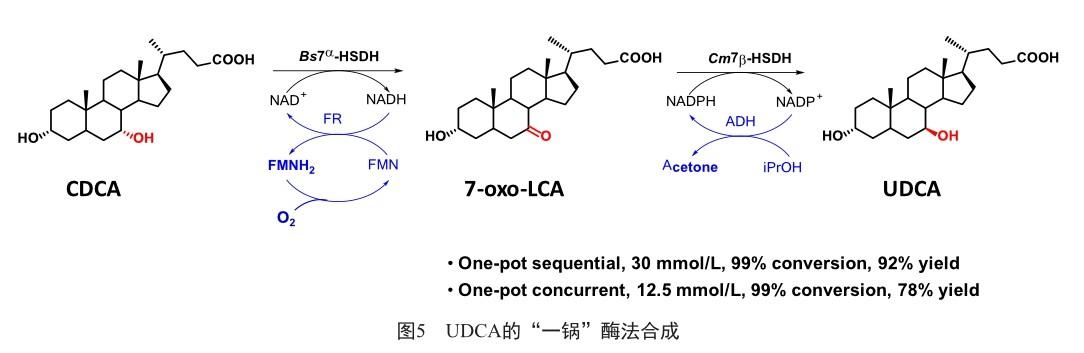
迄今报告的以CDCA为底物经过两步酶法合成UDCA的研究,虽然最高STY可以達到942 g/(L·d),但底物CDCA的浓度最高只有100 mmol/L,导致辅酶的总转换数较低,实际应用时会出现辅酶NADP+所占成本过高等问题[28]。造成这种现象的原因是由于HSDHs在反应条件下极其不稳定,特别是胆汁酸类化合物会导致 HSDHs严重失活,使之无法实现高浓度的底物转化[29]。
5 小结
UDCA既是传统中药熊胆粉的有效活性成分,也是临床上用于治疗胆固醇性结石和胆汁淤积性肝胆病的首选药物。随着全球对UDCA需求量的逐渐增大,从熊胆汁中获取UDCA显然已不能满足市场的需求,更何况“活熊取胆”方法并不可取(违背伦理、不可持续,且不利于黑熊、棕熊等濒危动物保护)。利用化学方法合成UDCA需要经过多个羟基保护/脱保护步骤,步骤繁琐、条件苛刻、总收率不高,还会用到危险和有毒试剂,产生对环境有害的废物。利用以酶为核心元件的生物方法替代或改进传统的UDCA化学合成工艺,可以缩短合成路线,避免或减少有害且昂贵化学试剂的使用,降低能耗并减少污染物的排放。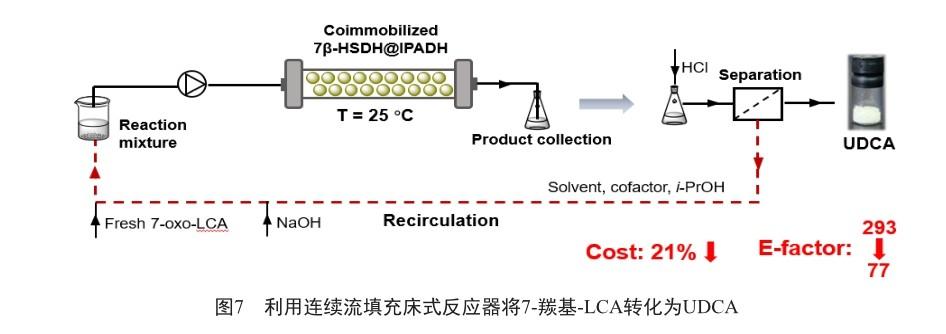
目前,将HSDHs应用于UDCA生物合成所用的原料主要为CDCA。以CDCA为底物,通过两步酶法(7α-HSDH和7β-HSDH)就可以将其转化为UDCA。然而,现已报告的HSDHs大多无法耐受高浓度的底物/产物,工业生产条件下容易失活,故反应过程中往往需要添加大量的酶催化剂和辅酶以加快反应速率,让酶在较短时间内将底物转化完全,但这样也就产生了生产成本过高的问题。由此可见,HSDHs在工业生产条件(高底物/产物浓度)下的不稳定性是当前酶法制备UDCA的关键限制性因素。
针对上述问题,未来应从以下3个方面进行更为深入的研究:①解析HSDHs的晶体结构,借助分子动力学模拟和光谱分析技术探索底物/产物使酶失活的机制,分析酶在高底物/产物浓度下维持稳定构象的关键性残基和结构域,揭示酶耐受性的结构基础和分子作用机制;②对酶分子中的关键残基或结构域进行(半)理性设计,并对其应用性能进行研究,创制新型高耐受性HSDHs,为UDCA的生物合成提供高效的工业酶元件;③利用创制的优质酶突变体,为UDCA的生物合成构建一个绿色、高效、经济的酶催化生产工艺体系,从而为最终实现其工业化生产奠定坚实的基础。
参考文献
[1] Festi D, Montagnani M, Azzaroli F, et al. Clinical efficacy and effectiveness of ursodeoxycholic acid in cholestatic liver diseases [J]. Curr Clin Pharmacol, 2007, 2(2): 155-177.
[2] Feng Y, Siu K, Wang N, et al. Bear bile: dilemma of traditional medicinal use and animal protection [J]. J Ethnobiol Ethnomed, 2009, 5: 2.
[3] Brevini T, Maes M, Webb GJ, et al. FXR inhibition may protect from SARS-CoV-2 infection by reducing ACE2 [J]. Nature, 2023, 615(7950): 134-142.
[4] 许建和, 郁惠蕾. 生物催化剂工程:原理及应用[M]. 北京:化学工业出版社, 2016: 15-17.
[5] 陳琦, 李春秀, 郑高伟, 等. 工业蛋白质构效关系的计算生物学解析[J]. 生物工程学报, 2019, 35(10): 1829-1842.
[6] Eggert T, Bakonyi D, Hummel W. Enzymatic routes for the synthesis of ursodeoxycholic acid [J]. J Biotechnol, 2014, 191: 11-21.
[7] Tonin F, Arends IWCE. Latest development in the synthesis of ursodeoxycholic acid (UDCA): a critical review [J]. Beilstein J Org Chem, 2018, 14: 470-483.
[8] Hoog SS, Pawlowski JE, Alzari PM, et al. Three-dimensional structure of rat liver 3α-hydroxysteroid/dihydrodiol dehydrogenase: a member of the aldo-keto reductase superfamily [J]. Proc Natl Acad Sci U S A, 1994, 91(7): 2517-2521.
[9] Grimm C, Maser E, M?bus E, et al. The crystal structure of 3α-hydroxysteroid dehydrogenase/carbonyl reductase from Comamonas testosteroni shows a novel oligomerization pattern within the short chain dehydrogenase/reductase family[J]. J Biol Chem, 2000, 275(52): 41333-41339.
[10] Di Costanzo L, Penning TM, Christianson DW. Aldo-keto reductases in which the conserved catalytic histidine is substituted [J]. Chem Biol Interact, 2009, 178(1-3): 127-133.
[11] Ferrandi EE, Bertuletti S, Monti D, et al. Hydroxysteroid dehydrogenases: an ongoing story [J]. European J Org Chem, 2020, 2020(29): 4463-4473.
[12] Buysschaert G, Verstraete K, Savvides SN, et al. Structural and biochemical characterization of an atypical shortchain dehydrogenase/reductase reveals an unusual cofactor preference [J]. FEBS J, 2013, 280(5): 1358-1370.
[13] Zheng MM, Wang RF, Li CX, et al. Two-step enzymatic synthesis of ursodeoxycholic acid with a new 7β-hydroxysteroid dehydrogenase from Ruminococcus torques [J]. Process Biochem, 2015, 50(4): 598-604.
[14] Song C, Wang B, Tan J, et al. Discovery of tauroursodeoxycholic acid biotransformation enzymes from the gut microbiome of black bears using metagenomics [J]. Sci Rep, 2017, 7: 45495.
[15] Tonin F, Otten LG, Arends IWCE. NAD+-dependent enzymatic route for the epimerization of hydroxysteroids [J]. ChemSusChem, 2019, 12(13): 3192-3203.
[16] Pedrini P, Andreotti E, Guerrini A, et al. Xanthomonas maltophilia CBS 897.97 as a source of new 7β- and 7α-hydroxysteroid dehydrogenases and cholylglycine hydrolase: improved biotransformations of bile acids [J]. Steroids, 2006, 71(3): 189-198.
[17] Chen X, Cui Y, Feng J, et al. Flavin oxidoreductase-mediated regeneration of nicotinamide adenine dinucleotide with dioxygen and catalytic amount of flavin mononucleotide for one-pot multi-enzymatic preparation of ursodeoxycholic acid[J]. Adv Synth Catal, 2019, 361(11): 2497-2504.
[18] Bertuletti S, Ferrandi EE, Marzorati S, et al. Insights into the substrate promiscuity of novel hydroxysteroid dehydrogenases[J]. Adv Synth Catal, 2020, 362(12): 2474-2485.
[19] Tonin F, Alvarenga N, Ye JZ, et al. Clean enzymatic oxidation of 12α-hydroxysteroids to 12-oxo-derivatives catalyzed by hydroxysteroid dehydrogenase [J]. Adv Synth Catal, 2019, 361(11): 2448-2455.
[20] Shi SC, You ZN, Zhou K, et al. Efficient synthesis of 12-oxochenodeoxycholic acid using a 12α-hydroxysteroid dehydrogenase from Rhodococcus ruber [J]. Adv Synth Catal, 2019, 361(20): 4661-4668.
[21] Huang B, Yang K, Amanze C, et al. Sequence and structure-guided discovery of a novel NADH-dependent 7β-hydroxysteroid dehydrogenase for efficient biosynthesis of ursodeoxycholic acid [J]. Bioorg Chem, 2023, 131: 106340.
[22] 許建和, 孙志浩, 宋航. 生物催化工程[M]. 上海: 华东理工大学出版社, 2008: 25-27.
[23] Bakonyi D, Hummel W. Cloning, expression, and biochemical characterization of a novel NADP+-dependent 7α-hydroxysteroid dehydrogenase from Clostridium difficile and its application for the oxidation of bile acids [J]. Enzyme Microb Technol, 2017, 99: 16-24.
[24] Huang B, Zhao Q, Zhou JH, et al. Enhanced activity and substrate tolerance of 7α-hydroxysteroid dehydrogenase by directed evolution for 7-ketolithocholic acid production [J]. Appl Microbiol Biotechnol, 2019, 103(6): 2665-2674.
[25] Liu Z, Zhang R, Zhang W, et al. Ile258Met mutation of Brucella melitensis 7α-hydroxysteroid dehydrogenase significantly enhances catalytic efficiency, cofactor affinity, and thermostability [J]. Appl Microbiol Biotechnol, 2021, 105(9): 3573-3586.
[26] Liu Z, Zhang R, Zhang W, et al. Effects of terminal modification on the catalytic efficiency and thermostability of Brucella melitensis 7α-hydroxysteroid dehydrogenase [J]. Syst Microbiol Biomanuf, 2023, 3(3): 469-478.
[27] Pan Y, Tang S, Zhu L, et al. Design of St-2-2 7α-HSDH mutants for altering substrate preference and thermostability[J]. Mol Catal, 2023, 548: 113423.
[28] Zheng MM, Chen KC, Wang RF, et al. Engineering 7β-hydroxysteroid dehydrogenase for enhanced ursodeoxycholic acid production by multiobjective directed evolution [J]. J Agric Food Chem, 2017, 65(6): 1178-1185.
[29] You ZN, Chen Q, Shi SC, et al. Switching cofactor dependence of 7β-hydroxysteroid dehydrogenase for costeffective production of ursodeoxycholic acid [J]. ACS Catal, 2019, 9(1): 466-473.
[30] Zhang X, Fan D, Hua X, et al. Large-scale production of ursodeoxycholic acid from chenodeoxycholic acid by engineering 7α- and 7β-hydroxysteroid dehydrogenase [J]. Bioprocess Biosyst Eng, 2019, 42(9): 1537-1545.
[31] Xu Y, Yang L, Zhao S, et al. Large-scale production of tauroursodeoxycholic acid products through fermentation optimization of engineered Escherichia coli cell factory [J]. Microb Cell Fact, 2019, 18(1): 34.
[32] Ji Q, Tan J, Zhu L, et al. Preparing tauroursodeoxycholic acid (TUDCA) using a double-enzyme-coupled system [J]. Biochem Eng J, 2016, 105: 1-9.
[33] Zheng MM, Chen FF, Li H, et al. Continuous production of ursodeoxycholic acid by using two cascade reactors with coimmobilized enzymes [J]. Chembiochem, 2018, 19(4): 347-353.
[34] Li HP, You ZN, Liu YY, et al. Continuous-flow microreactorenhanced clean NAD+ regeneration for biosynthesis of 7-oxolithocholic acid [J]. ACS Sustainable Chem Eng, 2022, 10(1): 456-463.
[35] Li HP, Yang BY, Su Y, et al. Sustainable and robust closedloop enzymatic platform for continuous/semi-continuous synthesis of ursodeoxycholic acid [J]. ACS Sustainable Chem Eng, 2022, 10(50): 16916-16923.

