基于CiteSpace 的SCoT分子标记技术研究热点和前沿分析
庞敏霞 姚国祥 戚勇 谢雄杰

摘要 采用文献计量学可视化分析的方法分析SCoT分子标记技术研究进程与现状,揭示研究热点及演化过程。检索中国知网(CNKI)和Web of Science (WOS)核心合集数据库中2000—2022年相关文献,通过Citespace 6.2.R2 软件分析文献作者、机构、关键词、引文情况。结果表明,CNKI检索出297篇文献,WOS检索出319篇文献,SCoT分子标记技术研究发文量呈逐年增加趋势。中文文献大都呈散点状,没有形成复杂的网络合作关系,发文量以广西大学何新华教授居多,其次是陕西理工大学的张羽教授。英文文献发文量以Islamic Azad University的Etminan Alireza教授居多,但没有形成复杂的网络合作关系;而来自四川农业大学的张宇(Zhang Yu)教授与其他团队交流紧密,形成较大的研究团队和合作网络。高频关键词有种质资源、亲缘关系、体系优化、genetic diversity(遗传多样性)、scot (start codon targeted)(靶向起始密码子)、polymorphism(多态性)等,近年频率突现关键词为指纹图谱、猕猴桃、葛根、variability(可变性)、gene flow(基因流)、genotype(基因型)、traits(特性)等。英文分析高被引文章大都为遗传多样性分析文献,引文关键词为callus(愈伤组织)、spectral fingerprinting(光谱指纹)。综上所述,中文和英文文献对SCoT分子標记技术研究的关键词相似,前沿热点稍有不同,作者机构间应继续加强合作,促进同专业的学术交流,共同推动SCoT分子标记技术的应用发展。
关键词 SCoT分子标记技术;CiteSpace;研究热点;前沿分析;可视化分析
中图分类号 S-058 文献标识码 A 文章编号 0517-6611(2024)09-0212-08
doi:10.3969/j.issn.0517-6611.2024.09.046
开放科学(资源服务)标识码(OSID):
Research Hotspots and Frontier Trends Analysis of SCoT Molecular Marker Technology Based on CiteSpace
PANG Min-xia1,2,YAO Guo-xiang1,QI Yong1 et al
(1.Zhejiang Yunzhitang Biotechnology Co.,Ltd.-Zhejiang Jianjiuhe Pharmaceutical Group Co.,Ltd.,Yuyao,Zhejiang 315402;2.College of Environmental & Resource Sciences of Zhejiang University,Hangzhou,Zhejiang 310012)
Abstract We analyzed the progress and status of research on SCoT Molecular Marker Technology by the method of literature metrology visualization,and the research hotspots and evolution process was revealed.Relevant literatures from 2000 to 2022 were retrieved from CNKI and Web of Science.The authors,research institutions,keywords and citations of literatures in this field were analyzed by using CiteSpace 6.2.R2. The results showed that 297 literatures were retrieved from CNKI,and 319 literatures retrieved from WOS.The number of publications on SCoT molecular marker technology was increasing year by year.The Chinese literature was mostly in scattered form without forming a complex network of cooperative relationships.The number of publications was mostly led by Prof. He Xinhua from Guangxi University,followed by Prof. Zhang Yu from Shaanxi University of Technology.The number of publications in English literature was mostly led by Prof.Etminan Alireza from Islamic Azad University,but without forming a complex network of cooperative relationships.On the other hand,Prof. Zhang Yu from Sichuan Agricultural University had close communication with other teams,forming a larger research team and cooperation network.High-frequency keywords included germplasm resources,genetic relationship,system optimization,genetic diversity,SCoT,polymorphism,etc.In recent years,keywords with sudden change in frequency included fingerprint map,kiwifruit,kudzu root,variability,gene flow,genotype,and traits,etc.The highly cited articles in English were mostly genetic diversity analysis literature,with citation keywords such as callus and spectral fingerprinting.In summary,the keywords of Chinese and English literature on SCoT molecular marker technology research were similar,with slight differences in the frontier hotspots.Collaborations among institutions and authors should be strengthened to promote academic communication in the same field and jointly promote the application and development of SCoT molecular marker technology.
Key words SCoT Molecular Marker Technology;CiteSpace;Research hotspots;Frontier trends;Visualization analysis
基金项目
国家重点研发计划项目(2017YFC1702203);宁波市自然科学基金项目(202003N4301)。
作者简介 庞敏霞(1989—),女,浙江天台人,副研究员,博士,从事中药药理与新产品开发研究、农业资源与环境研究。*通信作者,高级经济师,硕士,从事铁皮石斛产业化研究。
收稿日期 2023-06-02
随着分子生物学技术的发展,DNA分子標记的种类逐渐丰富,除传统的RAPD、AFLP、SSR、ISSR分子标记外,近年来新发展的SCoT(Start Codon Targeted Polymorphism,目标起始密码子多态性标记)分子标记是一种依据植物基因中ATG翻译起始位点侧翼序列的保守性,设计单引物并对基因组进行扩增的新型目的基因靶向标记[1-2],被认为是一种很有前途的靶向植物基因起始密码子的标记。目前SCoT分子标记方面的中英文研究较多,但相关文献计量学可视化分析尚鲜见报道。
CiteSpace(Citation Space,引文空间)软件是一款着眼于分析科学文献中蕴含的潜在知识,并在科学计量学、数据和信息可视化背景下逐渐发展起来的多元、分时、动态的引文可视化分析软件[3]。该软件包含合作图谱、共现图谱、共引图谱和突现词探测等多种功能图谱,可用于识别科学文献趋势,进行研究热点分析和预测未来研究趋势,并且从能绘制精美、色彩鲜明、富有美感图片等优势在文献剂量学中被广泛应用[4-5]。鉴于此,笔者采用文献计量学可视化分析的方法对近年来发表的SCoT分子标记相关中英文文献进行分析,以更直观的方式方便大家了解SCoT分子标记的研究进程与现状,揭示研究热点及演化过程,为学者更好地应用SCoT分子标记提供依据和思路。
1 资料与方法
1.1 数据来源
检索中国知网(CNKI)和 Web of Science核心合集TM(以下简称WOS)数据库,检索时间设定为 2000年1月1日—2022年12月31日。CNKI 上采用专业检索方式,以检索式“SU%=((SCoT) OR (SCoT分子标记) )AND((种质资源) OR (亲缘关系) OR (遗传多样性))”为检索主题,共检索到文献 659篇,对检索结果进行筛选,去除与主题无关的、会议等类型后得到297篇;WOS 上采用高级检索方式多次检索对比,以检索式“(((((TS=(SCoT)) OR TS=(SCoT Molecular Marker)) OR TS=(SCoT Molecular Marker Technology)OR TS=(start codon targeted)) )AND (((TS=(germplasm resource)) OR TS=(genetic relationship)) OR TS=(the genetic diversity)))NOT (TS=(scots pine))”为检索主题,共检索到文献325篇,精选论文、综述、English后319篇,最终纳入文献616篇。
1.2 数据处理
将CNKI 上的检索结果导出为 Refworks 格式,并用 CiteSpace 的“Data-CNKI”功能转换为可分析格式;将WOS 上的检索结果以纯文本格式导出包括作者、标题、摘要、关键词、类别和来源出版物等全记录及引用的参考文献在内的数据信息。应用Citespace 6.2.R2 软件,绘制对应的知识图谱。
1.3 统计分析
应用 Microsoft Excel 2010 统计相关文献数据信息,包括发表时间、作者、研究机构、关键词、文献类型等。用 CiteSpace 6.2.R2软件进行关键词共现分析,机构、作者合作网络可视化分析;通过突现检测识别前沿领域,通过构建文献网络定位关键文献,通过时间线视图描述关键词的发展进程和趋势。参数设置:时间分区为2000年1月至2022年12月,时间切片为1年,节点类型选择“作者”和“机构”,选择标准设置为g-index(k=25);节点类型选择“关键词”,选择标准设置为TopN=50(中文)、30(英文),修剪方式选择Pathfinder中“Pruning sliced networks”和“Pruning the merged network”;控制面板中选择Burstness,将γ设置为0.4(中文)、0.7(英文),其他默认值不做修改。在CiteSpace生成的科学知识图谱中,节点字体越大说明该作者的发文量越多;具有较高频数和中心性的关键词代表着该领域的研究热点;Q值(模块值)>0.3表示结构显著;S值(轮廓值)>0.5表示聚类合理,S值>0.7表示聚类结果可信度高;Strength表示突现强度,强度越高越能反映该领域的研究前沿和发展趋势[6]。
2 结果与分析
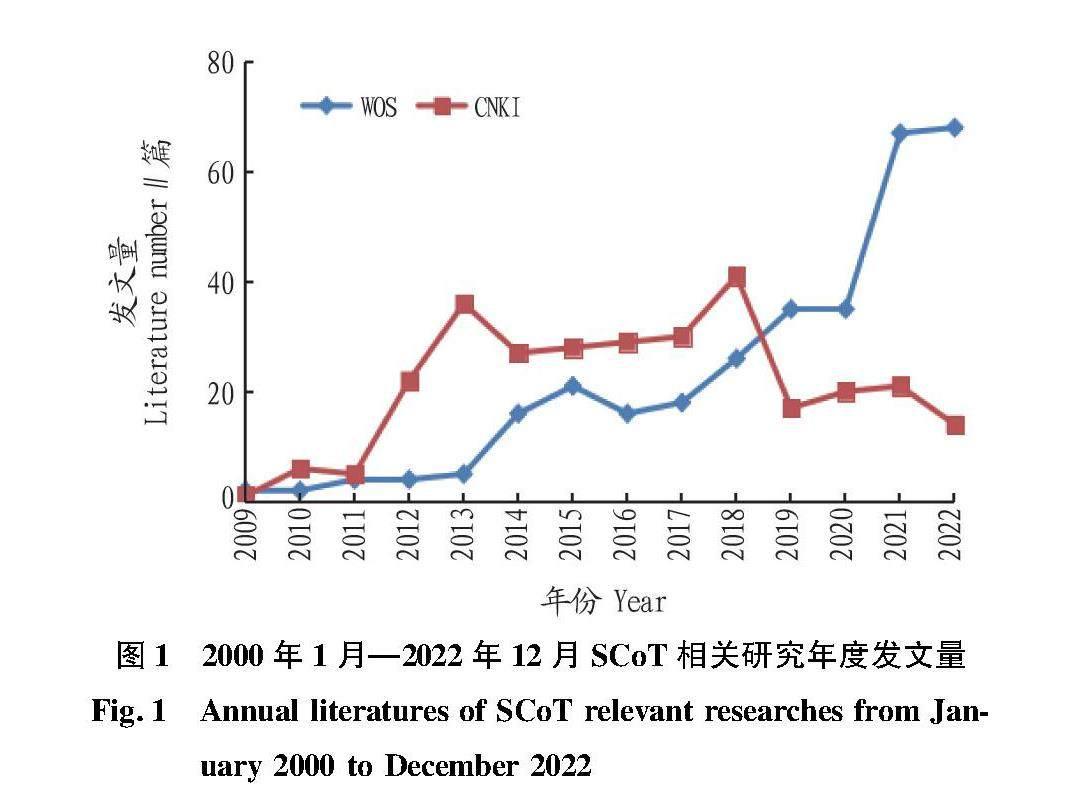
2.1 年度发文趋势
对纳入的616篇SCoT相关文献按照发表年份进行统计,中文文献从2009年开始才有应用SCoT技术进行种质资源、亲缘关系或遗传多样性相关研究文献的报道,在2013和2018年达到高峰,分别发表36、41篇相关文献;在2019年以后呈下降趋势(图1)。英文文献也是在2009年才开始有文献报道,发文量总体呈逐年上升趋势,在2022年达到高峰,发表68篇相关文献,这说明利用SCoT分子标记技术的应用逐渐深入,研究水平也逐渐提高。
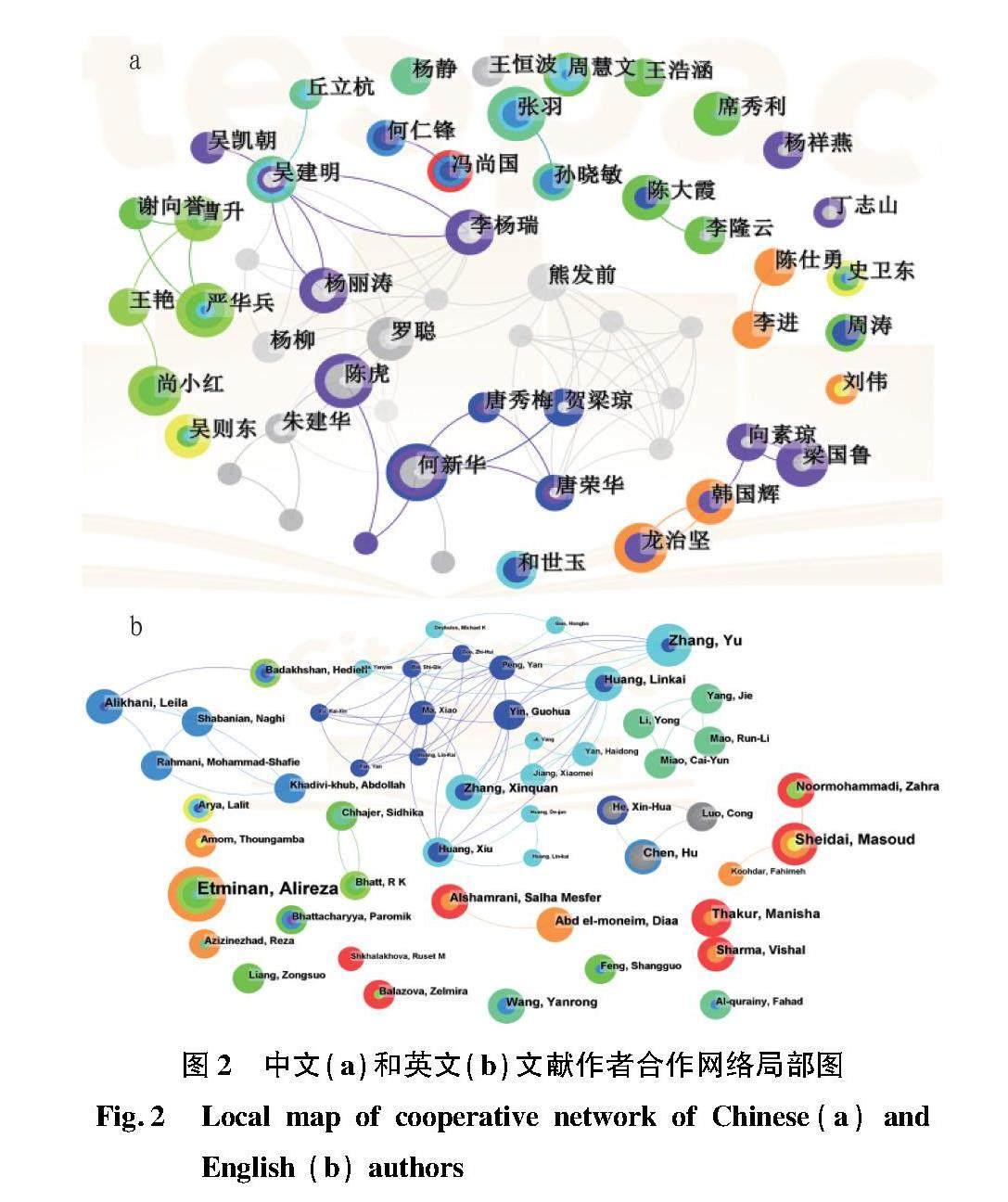
2.2 作者合作网络
对涉及867位中文文献作者和1 276位英文文献作者进行统计。作者合作网络局部图(图2)是根据发文量和合作关系绘制的,节点大小代表了发文量的多少,不同节点的大小代表作者的发文频次存在差异,图中的节点越大,代表作者的发文频次越高;颜色为时间年轮,颜色越鲜艳说明近年发文越活跃;连线代表作者间合作关系,连线越粗表示作者合作次数越多。中文主要研究学者有何新华(7)、张羽(6)、陈虎(6)、严华兵(6),括号内数值代表频数;何新华团队自2009年就开始应用SCoT分析技术开展研究,在中文文献中是最早的,引用该技术后对龙眼 SCoT-PCR 反应体系进行优化[7];何教授在中英文期刊均发表高水平论文,利用SCoT和ISSR标记研究芒果品种的遗传多样性[8]等。英文主要学者有Etminan Alireza(10)、Zhang Yu(6)、Sheidai Masoud(6)。其中,来自四川农业大学的张宇(Zhang Yu)教授与其他团队交流紧密,形成较大的研究团队和合作网络。作者频数结果见表1。
2.3 机构合作网络
中文研究机构主要有广西大学(20)、广西作物遗传改良生物技术重点开放实验室(15)、广西大学
农学院(8)、广州中医药大学(8)。从机构属性看,大部分研
究机构属于综合类系统;从时间年轮看,大部分机构发文量
近10年增长明显,其中以广西大学系统最为显著,其次是广西作物遗传改良生物技术重点开放实验室系统。根据节点离散程度可知,研究机构以高校为主,当地研究所/院与高校合作密切,不同系统独立研究明显(图3a)。英文研究机构主要有Islamic Azad University(38)、Egyptian Knowledge Bank (EKB)(38)、Indian Council of Agricultural Research (ICAR)(20)、Agricultural Research Center - Egypt(14)、Cairo University(13)、Shahid Beheshti University(11)(图3b)。Islamic Azad University(伊斯兰阿扎德大学)是世界上规模较大的大学之一,根据荷兰莱顿(Leiden)大学发布的CWTS大学排名,伊斯兰阿扎德大学在伊朗排名第1,Etminan Alireza教授就是来自这所大学。机构频数表结果见表2。
2.4 关键词共现网络
文献的关键词反映了一篇文章的核心内容,对文章的关键词进行分析,往往被用来探寻和确定一个研究领域的热点问题。将同义词、近义词、缩写词、大小写字母等合并。中文文獻关键词共现图谱网络共有382个节点,517条边,密度为0.007 1(图4a)。其中,频数位于前5的关键词是“分子标记”“种质资源”“亲缘关系”“体系优化”和“正交设计”,主要涉及关于SCoT分子标记方法的应用及其研究对象;英文文献关键词共现图谱网络共有460个节点,1 771条边,密度为0.016 8(图4b)。其中,频数位于前5的关键词是“genetic diversity”(遗传多样性)、“scot (start codon targeted)”(靶向起始密码子)、“polymorphism”(多态性)、“markers”(标记)和“diversity”(差异性),主要涉及关于SCoT分子标记方法的应用及与其他研究方法的比较。关键词频数结果见表3。
2.5 关键词聚类分析
对关键词进行聚类形成热点研究领域,聚类数字越小,所包含的关键词越多,每个聚类都是多个紧密相关的词组成;聚类内部节点之间的连线表示共现关系,连线越多,表明该领域的关键词之间共现程度越高;聚类跨聚类的连线较多,表明这些研究方向间的共被引程度高。中文文献关键词网络节点共有382个,连线527条,Q值为0.880 5,S值为0.961 6,共形成#0亲缘关系、#1指纹图谱、#2聚类分析、#3分子标记、#4种质资源、#5大豆等14个大聚类群组(图5a),#1与#3、#5与#8差异表达、#7表型性状与#11优化聚类图谱内的连线大多数是在聚类内部的,但仍存在一些跨聚类的连线,这些研究方向间的共被引程度较高。其中#4种质资源、#6遗传图谱、#10遗传结构、#12龙眼4个聚类几乎没有跨聚类的共现,因此可以从种质资源、遗传图谱、遗传结构方面作为研究的切入点。英文文献关键词网络节点共有450个,连线1 741条,Q值为0.815 7,S值为0.930 5,共形成#0 molecular marker(分子标记)、#1 elymus sibiricus(老芒麦)、#2 pcoa(principal coordinate analysis,主坐标分析)、#3 cbdp(CAAT box-derived polymorphism,CAAT盒多态性标记)、#4 molecular polymorphism(分子多态性)、#5 solanum nigrum(龙葵)等14个大聚类群组(图5b),#0与#1#2#4、#3与#8、#6与#10聚类图谱内的连线大多数是在聚类内部的,但仍存在一些跨聚类的连线,这些研究方向间的共被引程度较高。其中#5、#7遗传保真度、#9非生物胁迫、#11甜菊糖苷、#16超氧化物歧化酶这5个聚类几乎没有跨聚类的共现,因此可以从遗传保真度、非生物胁迫、超氧化物歧化酶方面作为研究的切入点。文献关键词聚类信息结果见表4、5。
2.6 关键词突现分析(前沿内容)
突现词分析可以筛选某个时间段频数突然增加的关键词,表明相关领域的新趋势或者转折点,用以指导未来研究方向。由图6可知,中文文献中突现强度排名前3的突现词是“甘蔗”“聚类分析”和“铁皮石斛”,近几年研究前沿突现词是“指纹图谱”“猕猴桃”和“葛根”。英文文献中突现强度排名前3的突现词是“variability”(可变性)、“cluster analysis”(聚类分析)和“sequences”(排列顺序),近几年研究前沿突现词是“variability”(可变性)、“gene flow”(基因流)、“genotype”(基因型)、“traits”(特性)。
2.7 文献共被引
文献共被引指2篇(或多篇论文)同时被后来1篇或多篇论文所引证,则称这2篇文献构成共被引关系,引用越多,强度越大,越能说明文献在该领域的指导地位。共被引用频次前12位的文献主要来源MOL BIOL REP、BIOTECHNOL BIOTEC EQ等影响力中等的期刊,引用最高的作者为Etminan A,其次为Guo DL,中国作者在国际杂志上发表高影响力的文章占比也逐年增加,这些文献最显著的特征是SCoT分子标记在遗传多样性分析中的应用研究,并且出版时间新(最晚的是2016年),说明SCoT分子标记应用不断更新,技术不断成熟。对共被引文献进行关键词的时间线分析,得到12个聚类。聚类#0 polygonati rhizoma(何首乌)、#3 mangifera indica L.(芒果)、#5 durum wheat(硬粒小麦)、#7 orchardgrass(鸭茅)涉及SCoT分子标记应用的研究对象,其中#2 callus(愈伤组织)、#14 spectral fingerprinting(光谱指纹)为近几年兴起的引用热点词聚类,预示着SCoT分子标记在愈伤组织及光谱指纹等领域的研究是前沿热点。
3 讨论
3.1 发文特征
研究采用可视化分析方法,以2000—2022年CNKI和WOS数据库中有关SCoT分子标记技术的616篇核心文献为研究对象,通过年度发文趋势、作者合作及机构合作网络、关键词共现网络、关键词聚类与突现分析及文献共被引分析,绘制出SCoT分子标记技术应用的研究主题与网络图。
结果发现,2000—2022年间,从2009年开始才有了关于SCoT分子标记技术研究文献的报道,这与其诞生的时期吻合,并且该技术一经开发便有学者开展应用研究,涉及种质资源、亲缘关系或遗传多样性相关研究。中文文献在2013和2018年达到高峰,分别发表36、41篇相关文献;在2019年以后呈下降趋势;英文文献发文量总体呈逐年上升趋势,在2022年达到高峰,发表68篇相关文献。这说明利用SCoT分子标记技术的应用逐渐深入,研究水平也逐渐提高。作者合作网络图与机构合作网络图很好地反映了中英文文献的特点,中文文献大都呈散点状,没有形成复杂的网络合作关系,发文量以广西大学何新华教授居多,其次是陕西理工大学的
张羽教授。张羽教授主要从事植物分子标记的构建及应用研究,在水稻、油菜、小麦、茶叶及魔芋等植物的分子遗传研究方面有丰富的科研经验,在《HEREDITAS》《中国水稻
科学》《遗传》《西北农林科技大学学报(自然科学版)》《湖南农业大学学报(自然科学版)》《植物遗传资源学报》《分子植物育种》等学术期刊发表论文50余篇,并且与其他团队交流紧密,形成较大的研究团队和合作网络。英文文献发文量以Islamic Azad University的Etminan Alireza教授居多,但没有形成复杂的网络合作关系;而来自四川农业大学的张宇(Zhang Yu)教授与其他团队交流紧密,形成较大的研究团队和合作网络。
3.2 演化进程
中、英文文献的关键词分析显示SCoT分子标记的不同演化进程,中文文献主要侧重于种质资源、亲缘关系、体系优化等方面的研究,并主要针对药用植物、水果、豆类进行探索。英文文献主要侧重于遗传多样性、多态性、差异性等方面的研究,并主要针对谷物、作物进行探索,少有针对药用植物探索,大多为中国学者。关键词聚类分析显示可以从种质资源、遗传图谱、遗传结构、遗传保真度、非生物胁迫、超氧化物歧化酶等方面作为研究的切入点。关键词突现分析显示,SCoT分子标记的发展历程,并进一步预测研究的热点方向:聚类分析、指纹图谱、可变性、基因流、基因型、特性等。文献共被引显示,SCoT分子标记在愈伤组织及光谱指纹等领域的研究是前沿热点。
现代研究表明,RAPD、ISSR、SSR和SCoT等基于DNA的分子标记已被用于组织培养植物的遗传保真度分析[9-11]。在过去的10年中,SCoT标记被单独或与其他传统分子标记,如RAPD和ISSR一起用于检测许多植物物种的组培植物的体细胞无性系变异和评估遗传同质性。大多数涉及腋芽增殖的微繁殖方法在组织培养的植物中显示出100%的单态性[12],而通过愈伤组织再生的植物在体外繁殖的植物中表现出多态性[13]。可见,通过愈伤组织或原生质体等未分化细胞进行植株再生對无性系繁殖是不可靠的,与体细胞无性系变异相比更敏感[9,14]。而在少数情况下,SCoT标记也能够检测腋芽增殖直接再生植株的低水平多态性。这可能是由于培养细胞或组织中预先存在的遗传变异或影响体外再生的不同因素引起的变异[15]。
3.3 小结 该研究基于文献计量学可视化分析方法,对近几年来SCoT分子标记研究的现状、热点和发展趋势等进行简要分析,研究提示SCoT分子标记在愈伤组织及光谱指纹等领域的研究是前沿热点;自2009年被发现以来,SCoT分子标记因具有简单、高效、经济、不需要事先获得基因组序列信息、能定位基因或特定性状等优点,在遗传和基因组研究以及不同植物类群的作物改良计划中具有优势;并且其在基因组信息有限、研究较少的非模式植物中也具有很高的应用价值。SCoT分子标记已广泛应用于组织培养植物的遗传多样性分析、遗传和系统发育关系、种/品种鉴定、连锁图谱构建、关联图谱分析、差异基因表达、遗传保真度等遗传和基因组学研究中。
参考文献
[1]
COLLARD B C Y,MACKILL D J.Start codon targeted(SCoT) polymorphism:A simple,novel DNA marker technique for generating gene-targeted markers in plants[J].Plant Mol Biol Rep,2009,27(1):86-93.
[2] 马利奋,尹跃,赵建华,等.17 个枸杞品种的 SCoT 遗传多样性[J].浙江农业学报,2018,30(10):1665-1670.
[3] 李杰,陈超美.CiteSpace:科技文本挖掘及可視化[M].3版.北京:首都经济贸易大学出版社,2022:2-3.
[4] 冯涛,宋佳敏,孙立丽,等.基于CiteSpace知识网络图谱的黄芩研究进展[J].中国现代应用药学,2022,39(19):2549-2556.
[5] CHEN C M.CiteSpace II:Detecting and visualizing emerging trends and transient patterns in scientific literature[J].J Am Soc Inf Sci Technol,2006,57(3):359-377.
[6] 陈悦,陈超美,胡志风,等.引文空间分析原理与应用:CiteSpace 实用指南[M].北京:科学出版社,2014:24.
[7] 陈虎,何新华,罗聪,等.龙眼SCoT-PCR反应体系的优化[J].基因组学与应用生物学,2009,28(5):970-974.
[8] LUO C,HE X H,CHEN H,et al.Genetic diversity of mango cultivars estimated using SCoT and ISSR markers[J].Biochem Syst Ecol,2011,39(4/5/6):676-684.
[9] RAI M K,PHULWARIA M,HARISH,et al.Genetic homogeneity of guava plants derived from somatic embryogenesis using SSR and ISSR markers[J].Plant Cell Tissue Organ Cult,2012,111(2):259-264.
[10] AGARWAL T,GUPTA A K,PATEL A K,et al.Micropropagation and validation of genetic homogeneity of Alhagi maurorum using SCoT,ISSR and RAPD markers[J].Plant Cell Tissue Organ Cult,2015,120(3):313-323.
[11] KUDIKALA H,JOGAM P,SIRIKONDA A,et al.In vitro micropropagation and genetic fidelity studies using SCoT and ISSR primers in Annona reticulata L.:An important medicinal plant[J].Vegetos,2020,33(3):446-457.
[12] SETH S,RATH S C,ROUT G R,et al.Somatic embryogenesis in Abutilon indicum (L.) Sweet and assessment of genetic homogeneity using SCoT markers[J].Plant Biosyst IntJ Deal Aspects Plant Biol,2017,151(4):704-714.
[13] RAHMANI M S,PIJUT P M,SHABANIAN N,et al.Genetic fidelity assessment of in vitro-regenerated plants of Albizia julibrissin using SCoT and IRAP fingerprinting[J].Vitro Cell Dev Biol Plant,2015,51(4):407-419.
[14] RANI V,RAINA S N.Genetic fidelity of organized meristem-derived micropropagated plants:A critical reappraisal[J].Vitro Cell Dev Biol Plant,2000,36(5):319-330.
[15] BAIRU M W,AREMU A O,VAN STADEN J.Somaclonal variation in plants:Causes and detection methods[J].Plant Growth Regul,2011,63(2):63:147-173.

