青蒿查尔酮合成酶(AaCHS)基因家族鉴定及光调控分析







摘要:[目的]为了解青蒿(Artemisia annua L.)查尔酮合成酶(chalcone synthase,CHS)基因家族的分子进化特性及其在不同组织部位的表达情况。[方法]从Pfam 数据库下载CHS 蛋白HMM 模型,并使用HUMMER 3. 0、Blastp 和CD-search 鉴定出青蒿基因组中的CHS 基因家族成员。采用TBtools、EXPASy、CELLO v. 2. 5、MEGA 7. 0、MEME、SOPMA、SWISSMODEL 和PlantCARE 等软件对其氨基酸与碱基序列进行生物信息学分析,通过8 个不同组织部位的转录组数据对 AaCHS 基因家族成员进行表达分析。[结果]青蒿基因组中共有16 个AaCHS 基因家族成员,蛋白结构分析显示AaCHS 蛋白质结构并不稳定,将其分为4 个组,基因结构分析显示所有AaCHS 基因蛋白均具有外显子,数量为2~4,内含子数量为0~2,其中8 个AaCHS 基因不具有内含子。启动子顺式作用元件分析表明该基因家族成员启动子上含有大量光响应、植物激素响应元件;CD-search 验证结果发现每个AaCHS 蛋白均有Chalsti-synt_N 结构域。[结论]AaCHS 家族成员表达分析结果表明,16 个AaCHS 基因在不同组织和光处理下表达不同。GO 富集结果得到GO 二级分类的生物过程和分子功能,生物过程共富集了80 条序列;细胞组分共富集了19 条序列,其中二级分类过程细胞质中富集了11 条序列,数量最多,与亚细胞定位预测结果一致;推测AaCHS 基因在细胞质中调控黄酮类化合物的累积。通过青蒿光调控差异表达分析,推测AaCHS7、AaCHS10 在青蒿受到红光光处理中起正向调控作用。
关键词:青蒿; CHS 基因家族; 光调控; 基因表达; 差异基因表达
中图分类号:R932;S567.219 文献标识码:A 文章编号:1671-8151(2024)01-0001-13
青蒿(Artemisia annua L.)是菊科的成员,具有相当大的药用、观赏和经济价值[1]。青蒿素以青蒿为提取来源,是目前治疗疟疾的最有效的药物。疟疾是由原生动物寄生虫疟原虫引起的毁灭性疾病[2]。根据世界卫生组织2015 年的数据,约有33亿人面临感染疟疾的风险,12 亿人处于高风险[3]。有研究表明青蒿素和黄酮类化合物在治疗包括疟疾在内的各种疾病方面具有协同作用[4-5]。黄酮类化合物作为植物抗毒素或抗氧化剂,具有清除活性氧(ROS)的能力[6],并保护植物免受非生物和生物胁迫的损害,包括UV 照射、冷胁迫、病原体感染和昆虫取食[7-9]。许多青蒿素和类黄酮生物合成途径的酶的增加,通常与产物丰度增加相关联[10]。但在青蒿中对于它们的生物合成途径及调控的研究报导还比较少,所以探究青蒿中黄酮化合物合成途径关键基因及其表达具有重要意义。
查尔酮合成酶(CHS)是植物聚酮合酶超家族的成员,其还包括芪合酶(STS)、吖啶酮合酶、吡喃酮合酶、联苄基合酶和对香豆酰三乙酸合酶[11]。黄酮类化合物在植物防御中起着重要的作用,而CHS 作为黄酮类化合物生物合成的关键酶基因,在调节该途径中起着重要的作用。此外,查尔酮合成酶为在开花植物中具有重要作用的黄酮类化合物提供起始材料,如提供花色素、抗生素、UV 保护剂和驱虫剂[12]。由于CHS 在类黄酮生产中的重要性,在植物中已经得到了很好的研究,如矮牵牛(Petunia hybrida)CHS 基因家族的每个成员由2个外显子组成,由1 个大小和序列可变的内含子分隔,内含子位于保守位置[13];玉米中的2 个CHS 基因,具有高度同源的外显子序列,在编码区上游5 ′和下游3 ′序列以及内含子中有很大差异[14];菊花(Chrysanthemum nankingense)中鉴定出16 个CnCHS 基因家族成员,大多数含有2 个外显子和1个内含子,具有Chal-sti-synt_N 和Chal-stisynt_C 结构域,其中CnCHS10 在花器官中的表达水平高于营养组织[15]。芒果(Mangifera indicaL.)中MiCHS4、MiCHS16 和MiCHS17 响应UVB和可见光处理[16]。
光作为一个重要的环境调控因子,不仅是植物光合作用的主要能量来源,同时也在调节植物生长发育过程中发挥关键作用[17]。有研究表明不同光质处理可使AaCHS 表达上调,刺葡萄(Vitisdavidii)中发现VdCHS2 和VdCHS3 在绿光、蓝光、暖黄光处理下表达量先升高后降低[18];非洲菊(Gerbera hybrida)中发现蓝光和红光促进CHS 基因表达[19]。
目前,虽然对于CHS 的研究已经取得了一些进展,但是AaCHS 基因家族分析未见报道,其在青蒿中的功能尚不清楚。本研究基于青蒿已公布的参考基因组,鉴定了AaCHS 基因家族成员。本研究验证了这些成员的序列特征、蛋白质保守基序、顺式作用元件和编码序列。同时,还分析了CHS 蛋白结构域及其系统进化关系,并绘制了在青蒿不同组织器官和光调控条件下的表达量热图。最后,筛选出在不同光调控条件下差异表达的CHS 基因。研究结果对挖掘具有特殊功能的青蒿中CHS 基因家族成员具有重要意义。
1 材料与方法
1. 1 AaCHS 基因家族成员鉴定与序列特征分析
在NCBI 数据库中下载青蒿数据库(PRJNA416223)的全基因组文件、GFF 注释文件、CDS 序列和蛋白序列。从Pfam 数据库获取CHS 结构域种子文件(PF00195),然后在Linux 系统中使用HMMER 3. 2. 1 软件创建Profile HMM,与青蒿的蛋白序列进行比对,去除冗余序列。接下来筛选出质量较高的基因家族候选基因,要求E-valuelt;1×10−20,并使用clustalW2 对这些高质多序列比对。随后利用hmmbuild 构建隐马可夫模型,并使用该新建模型进一步筛选完整的CHS 基因家族序列,筛选得到AaCHS 蛋白序列。在NCBI 下载拟南芥CHS 的蛋白质序列文件,利用Blastp 软件包进行同源比对,筛选目标物种中identity大于75% 的序列,可筛选得到AaCHS 蛋白序列。最后将Blastp 同源比对的数据和HMMER 筛选过后的数据取交集,得到AaCHS 蛋白。将候选蛋白序列用NCBI 中的CD-Search 功能进行预测,对候选转录因子序列进行检验,最终鉴定出AaCHS 基因家族成员。利用在线工具ExPAS 软件对AaCHS 基因家族成员的蛋白质长度、等电点、pH 值等基本信息进行了分析[20];利用CELLOv. 2. 5 软件进行亚细胞定位预测[21];利用SOPMA对AaCHS 基因蛋白质α 螺旋、β 转角、延长线、随机线圈进行分析[22] ;利用SWISSMODEL 对AaCHS 基因蛋白质三级结构预测[23]。
1. 2 AaCHS 基因家族成员顺式作用元件分析
用TBtools 软件从青蒿的完整基因组序列中提取了16 个AaCHS 基因起始密码子上游2000 bp的启动子序列。使用PlantCARE 在线数据库对这些启动子进行了顺式作用元件的分析[24],并最终在Tbtools 软件中进行了可视化展示。
1. 3 AaCHS 家族成员保守结构域分析
使用MEGA7 中ClustalW 工具对AaCHS 蛋白进行多序列比对,然后利用Weblogo 软件获取其标识。最后,通过在线网站MEME 和TBtools软件对AaCHS 蛋白保守基序motif 进行分析[25]。
1. 4 AaCHS 家族成员系统发育树的构建
在MEGA7 中,以多序列比对的结果先计算遗传距离,之后在MEGA7 软件的phylogeny(系统进化)中使用邻接法(NJ)法,设置其为Poisson model模型,此外设置bootstrap(n=1000)进行系统发育分析[26]。
1. 5 AaCHS 基因家族成员表达分析
从NCBI 数据库中下载青蒿不同部位的转录组数据(SRP129502)以及青蒿光调控转录组数据(Pro-ject: PRJNA435470)。使用Trimmomatic 软件去除测序数据中质量较低的序列,并利用FastQC 软件将测序数据转换为Fastq 格式。使用hisat2重新构建索引文件,并将其与转录组数据在Fastq 格式下进行比对。最后,通过定量分析计算得到TPM 值,进行log10(TPM+1)后生成基因表达量矩阵[27]。最后在TBtools 中对该矩阵进行归一化处理并进行可视化分析。
1. 6 AaCHS 基因家族成员GO 注释
根据eggNOG-mapper 在线网站,对青蒿蛋白序列进行GO 注释,所得注释文件以差异基因作为筛选范围,得到AaCHS 基因功能注释文件,再通过Rstudio 进行可视化。
1. 7 AaCHS 基因家族成员差异基因分析
差异表达基因在Linux 系统下使用R 语言,使用BiocManager 中edgeR 软件包进行差异表达分析,所得差异表达基因的P-Value 去除NA 值,并对筛选出的AaCHS 基因家族成员进行标记,使用enhancedvolcano R 包进行可视化。
2 结果与分析
2. 1 AaCHS 基因家族成员鉴定及理化性质分析
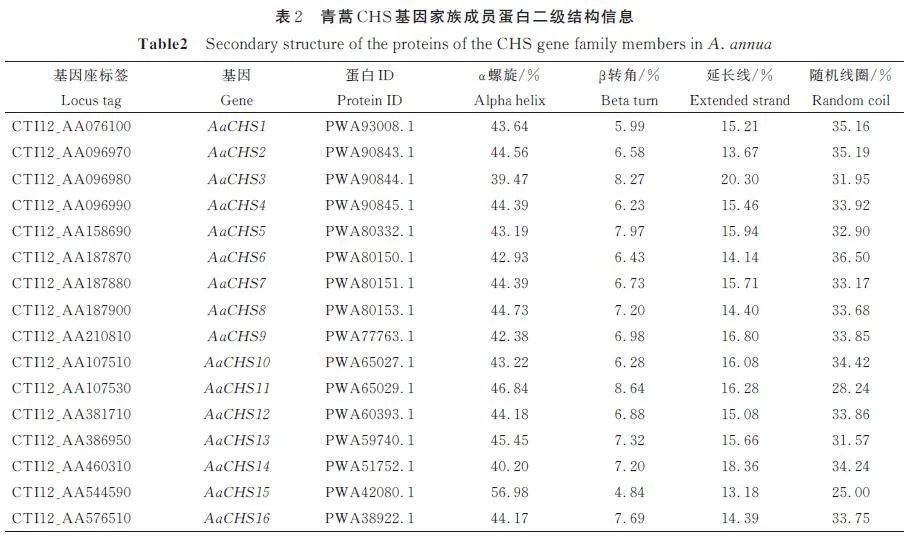
AaCHS 基因家族成员鉴定以Pfam 数据库下载的CHS 结构域序列(PF00195)靶点进行筛选,与Blastp 2 次筛选求交集,最后使用NCBI 中CDsearch功能筛选得到16个CHS 蛋白(表1),根据命名规则将其基因依次命名为AaCHS1~AaCHS16[28]。对该基因家族成员蛋白长度、分子量、等电点、亲水性平均值、脂肪指数、亚细胞定位分析,结果表明AaCHS15 蛋白长度最长为516 个氨基酸,AaCHS3 蛋白长度最短为266 个氨基酸;分子量为29 392. 96~56 149. 6 Da;亲水性平均值从−0. 149(AaCHS16)到0. 432(AaCHS15);脂肪指数最高的为99. 97 (AaCHS2) ,最小的为89. 60(AaCHS12)。亚细胞定位预测显示所有AaCHS蛋白都定位在细胞质中(表1)。根据AaCHS基因家族成员蛋白二级结构信息表(表2),可见α 螺旋为39. 47%~56. 98%;β 转角4. 84%~8. 64%;延长线13. 18%~20. 30%;随机线圈25. 00%~36. 50%。由二级结构可知,β 转角均小于50%,可知AaCHS基因家族成员结构疏松。根据AaCHS 基因家族成员三级结构预测图(图1),再次证明了AaCHS蛋白结构。
2. 2 AaCHS 基因家族成员系统进化分析
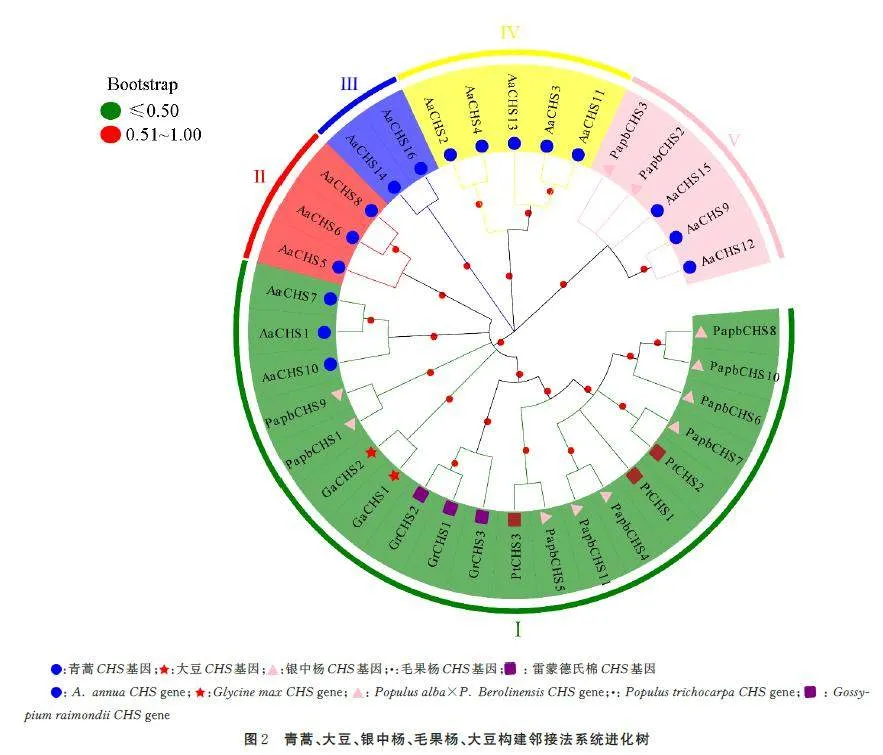
为了研究不同植物之间CHS 基因家族成员的系统进化关系,我们使用邻接法(NJ)对包括青蒿(Artemisia annua)16 条、雷蒙德氏棉(Gossypiumraimondii)3 条、毛果杨(Populus trichocarpa)3 条、银中杨(Populus alba×P. Berolinensis)11 条和大豆(Glycine max)2 条在内的5 个物种共35 条CHS蛋白序列进行了系统进化分析(图2)。通过这项分析,我们将CHS 蛋白划分为5 个亚族:亚族Ⅰ(图2 中绿色部分)中包含CHS 蛋白数量最多,其中包含雷蒙德氏棉、大豆和毛果杨全部CHS 蛋白,还包含青蒿CHS 蛋白(AaCHS1、AaCHS7、AaCHS10);亚族Ⅱ(图2 中红色部分)中只包含青蒿CHS 蛋白(AaCHS5、AaCHS6、AaCHS8);亚族Ⅲ(图2 中蓝色部分)中只包含剩余的青蒿CHS 蛋白(AaCHS14、AaCHS16),亚族Ⅴ(图2 中粉色部分)中银中杨蛋白和青蒿CHS 蛋白(AaCHS9、AaCHS12、AaCHS15)聚为一支,雷蒙德氏棉、大豆和毛果杨都属于双子叶植物。因此这些物种的CHS 基因家族成员之间存在较为密切的进化关系,并且归类在同一类别。
2. 3 AaCHS 基因结构、顺式作用元件及鉴定分析
利用TBtools 软件对AaCHS 基因家族成员的编码序列基因结构进行了分析,结果显示16 个AaCHS 基因中有8 个没有非编码区,而所有16 个基因均包含CDS 外显子(图3d),经CD-search 鉴定后发现16 个AaCHS 成员均有Chal-sti-synt_N结构(图3c)。通过使用PlantCARE 在线网站对青蒿CHS 基因家族成员的启动子区域进行分析,研究了转录起始位点上游约2000 bp 的序列。根据分析结果,发现了77 个顺式作用元件(图3d),其中也包括常见元件TATA-box、CAAT-box 以及Abox,这些元件中部分元件与生长发育、逆境胁迫响应、植物激素以及光响应相关(图4),其中与生长发育相关的元件有6 种,包含有参与玉米醇溶蛋白代谢调节(O2-site)、调节各种细胞过程(CCAAT-box)等;与逆境胁迫有关的元件有4种,包括低温响应顺式作用元件(LTR)、抗病和胁迫响应元件(TC-rich repeats)、厌氧诱导作用元件(ARE);与植物激素有关的元件有7 种,其中包括参与ABA 调节的基因表达(ABRE)、水杨酸顺式作用元件(TCA-element)、茉莉酸响应元件(CGTCA-motif)等;光响应元件种类最多有16种,其中包括调控靶基因的表达(MRE)、植物响应外界环境刺激的通用调控元件(G-box)。利用MEME 在线网站在AaCHS 基因家族成员中找到12 个保守基序(Motif 1~Motif 12),通过对图5 中的保守基序分析,结果表明:根据进化关系,在亚族Ⅱ 中,3 个CHS 基因(AaCHS5、AaCHS6 和AaCHS8)有相同的10 个保守结构域;亚族Ⅰ中的3 个CHS 基因(AaCHS1、AaCHS7 和AaCHS10)有相同的11 个保守结构域;而在亚族Ⅳ 中,AaCHS2、AaCHS4、AaCHS11 和AaCHS13 均具有相同的11 个保守基序,亚族Ⅴ中AaCHS15 仅有2 个保守结构域,AaCHS9 和AaCHS12 中除了Motif7 外的7 个保守基序均相同;亚族Ⅲ 中AaCHS14 和AaCHS12 除了Motif 11 的10 个保守结构域均相同(图3b)。
2. 4 AaCHS 家族成员表达分析
为了研究AaCHS 基因家族成员的表达模式,我们对不同部位组织(嫩叶、老叶、根、表皮、花蕾、种子、花、毛状体)青蒿转录组测序结果进行分析(图6a)。研究结果显示,AaCHS10 和AaCHS16属于亚族Ⅲ,其在嫩叶、老叶、表皮、花蕾、种子、花和毛状体均显著上调;在青蒿嫩叶中AaCHS14 显著上调。此外对AaCHS 基因家族成员进行光调控分析(图6b),结果表明,AaCHS16 在白光和远红光条件下均显著上调,在蓝光条件下表达上调;AaCHS10 在蓝光、红光和白光条件下均有表达,在黑暗下几乎不表达,这表明蓝光、红光和白光会促进AaCHS10 的表达;AaCHS6 在白光条件下表达均上调,这些结果能为进一步研究 AaCHS 基因家族成员的功能提供借鉴。
2. 5 AaCHS 基因家族成员GO 注释
用eggNOG-mapper 在线网站对青蒿蛋白序列进行GO 注释,以AaCHS 基因蛋白为筛选范围,AaCHS 基因进行功能注释。选取P-valuelt;0. 05且富集Count 最多的前12 个GO 二级分类过程,AaCHS 基因在GO 的二级分类的生物过程(biologicalprocess,BP)、细胞组分(cellular component,CC)2 个大类并将其注释(图7)。从图5 可见,生物过程共富集了80 条序列;细胞组分共富集了19 条序列,其中包括的细胞质(cytoplasm)富集了11 条序列,数量最多;而在生物过程中富集了细胞成分组装(cellular component assembly)、细胞发育过程(cellular developmental process)、解剖结构的形态发生(anatomical structure morphogenesis)、细胞成分的生物生成(cellular component biogenesis)、内膜系统(endomembrane system)、多细胞生物体的发育(multicellular organism development)、细胞成分组织(cellular component organization)、多细胞生物体的过程(multicellular organismal process)、细胞成分的组织或生物发生(cellular componentorganization or biogenesis)、解剖结构的发展(anatomical structure development)、发育过程(developmental)均富集了最多的8 条序列。
2. 6 AaCHS 基因家族成员差异基因分析
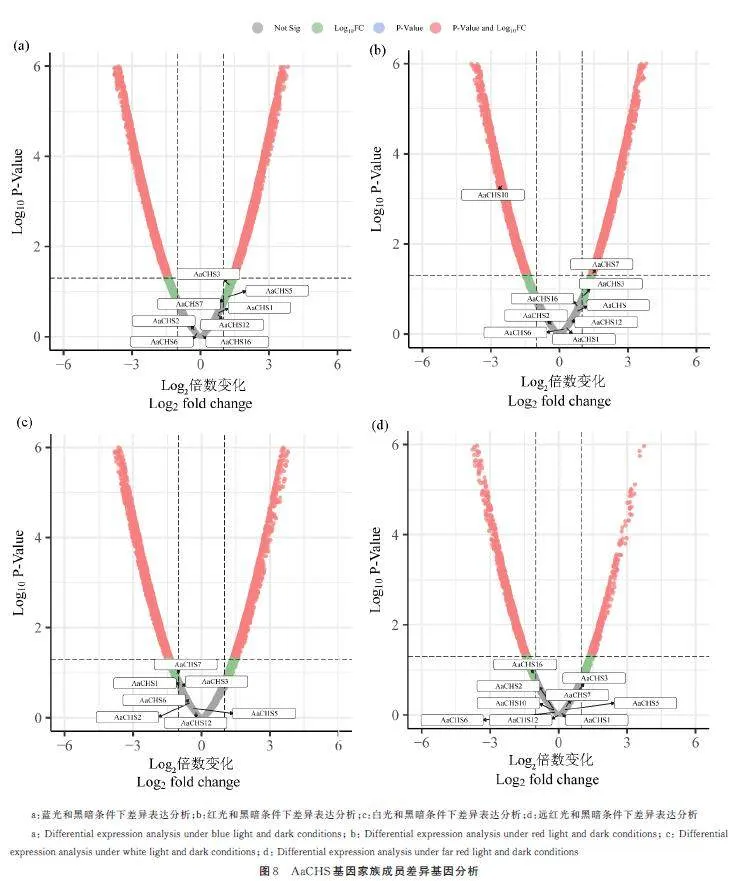
基于青蒿在不同光照下的调控,将青蒿在蓝光、红光、白光、远红光调控的转录组测序数据作为实验组,黑暗条件作为对照组,依次进行差异基因表达分析并将其分为4 组,第1 组为蓝光和黑暗条件下差异表达分析;第2 组为红光和黑暗条件下差异表达分析;第3 组为白光和黑暗条件下差异表达分析;第4 组为远红光和黑暗条件下差异表达分析。将分组过后的2 组青蒿器官进行差异表达分析;在Linux 系统下使用R 语言,利用BiocManager中的edgeR 软件包对差异表达分析,将所得差异表达基因的P-Value 去除NA 值,将既是差异表达基因又是AaCHS 基因家族成员筛选出来,使用enhancedvolcanoR 包将其可视化,以Log10FCgt;2. 0且P-Valuegt;0. 05 为阈值筛选差异表达基因。分析结果发现,如图8b,AaCHS7、AaCHS10 是红光和黑暗条件下差异表达分析下的正向差异表达基因,其Log10FC 值均大于2. 0 且P-Value 值大于0. 05;而在另外3 组(图8a、图8c、图8d)中均未发现差异表达基因。
3 讨 论
黄酮类化合物是一类植物次生代谢产物。黄酮类生物合成途径包括类苯丙素途径和类黄酮途径。查尔酮合成酶(CHS)是苯丙素类化合物生物合成途径中的关键酶,在植物中普遍存在。它在类苯丙素途径的初始步骤中起作用,并使3 个丙二酰-CoA 分子与1 个4-香豆酰-CoA 分子缩合以产生柚皮素查尔酮,其是各种类黄酮的前体[28-31]。因此,研究查尔酮合成酶基因对于进一步分析影响青蒿中类黄酮化合物合成的环节十分必要。本研究对青蒿中CHS 基因家族进行了识别,并进一步分析了AaCHS 基因家族成员的理化性质结构、基因序列、进化关系及不同组织部位AaCHS 基因表达模式。在青蒿中共发现16 个AaCHS 基因家族成员,本研究还对AaCHS 蛋白一级结构进行了分析,包括蛋白质大小、分子量、等电点、平均亲水性系数及亚细胞定位分析,结果发现16 个AaCHS 蛋白均定位于细胞质;大多数为酸性不亲水蛋白。对AaCHS 蛋白二级结构进行了分析,包括α 螺旋、β 转角、延长线、随即线圈分析,结果发现16 个AaCHS 蛋白的β 转角均低于50%,说明16 个AaCHS 蛋白结构疏松;对AaCHS 蛋白三级结构可视化分析,证明了这一观点。通过邻接法(NJ)对5 个植物物种(包括青蒿、雷蒙德氏棉等)的35条CHS 蛋白序列进行系统进化分析后发现,所有AaCHS 被划分为5 个亚族,并与银中杨CHS 蛋白亲缘关系更近,推测他们具有相似的蛋白结构[32];对AaCHS 基因序列分析发现:16 条AaCHS 基因有7 条没有内含子,剩余基因的内含子数量为0~2个,16 条基因均包含CDS 外显子,含有2~5 个外显子,何梦媛等[33]与本研究结果类似。另外,在所有青蒿AaCHS 基因上游启动子区域内最常见的元件是光反应元件,其次是植物激素响应元件,最后是生长发育和逆境相关元件。本研究在AaCHS 基因中找到12 个保守基序(Motif 1~Motif12),通过对其分析发现:根据进化关系,在亚族Ⅱ 中,3 个CHS 基因(AaCHS5、AaCHS6、AaCHS8)有相同的10 个保守结构域;亚族Ⅰ中的3 个CHS 基因(AaCHS1、AaCHS7、AaCHS10)有相同的11 个保守结构域;而在亚族Ⅳ 中,AaCHS2、AaCHS4、AaCHS11 和AaCHS13 均具有相同的11 个保守基序;亚族Ⅲ 中AaCHS14 和AaCHS12 除了Motif 11 的10 个保守结构域均相同;亚族Ⅴ 中AaCHS15 仅有2 个保守结构域,AaCHS9 和AaCHS12 中除了Motif 7,其它7 个保守基序均相同。在亚族Ⅰ 、Ⅱ 、Ⅲ 和Ⅳ 中,除了AaCHS3 外的AaCHS 家族成员均具有相同的10个保守结构域,根据进化树划分的亚族成员,其保守结构域存在高度保守性,推测它们有共同的起源,此外不同亚族成员间也发生了进化,如图3b 中的Motif 11。
为了深入探究AaCHS 基因在不同组织部位中可能发挥的功能,通过挖掘NCBI 数据库中青蒿8 个不同组织部位(嫩叶、老叶、根、表皮、花蕾、种子、花、毛状体)转录组数据,我们筛选出了16 个AaCHS 基因家族成员,并发现它们在各个组织中均没有表达下调情况,与Shen 等[34]研究结果类似,证明了我们筛选到的AaCHS 基因家族成员是可靠的。生物信息学分析显示,AaCHS 蛋白序列高度保守,它们的Motif 分布也支持了系统进化树的结果。根据系统进化树,AaCHS10 和AaCHS16在嫩叶、老叶、表皮、花蕾、种子、花和毛状体均显著上调;在青蒿嫩叶中AaCHS14 基因显著上调;AaCHS2、AaCHS3、AaCHS4、AaCHS11 和AaCHS13 聚在一起,其表达水平类似,推测它们可能有共同的起源并且序列保守[35],Hassani 等[36]研究发现查尔酮合成酶在青蒿的芽中显著表达,与本研究所得结论一致。为进一步了解不同光质对AaCHS 表达的调控,对蓝光、黑暗、红光、远红光、白光5 个条件下AaCHS 家族基因表达模式进行了分析。远红光处理下,AaCHS1~AaCHS15的表达量都很低,推测在青蒿生长发育和逆境适应过程中,它们可能并未发挥作用,但这一推测需要进一步验证。此外,我们还发现,与黑暗条件相比,光诱导可以显著提升植物体内CHS 基因表达量。比如,与黑暗处理相比,蓝光诱导能够显著增加AaCHS10 基因的表达量,Kitazaki 等[37]通过分析莴苣叶的代谢途径发现,在蓝光下CHS 表达量显著上调,这与本研究所得结果一致。白光诱导则能显著提高AaCHS10 和AaCHS16 基因的表达量。而在红光处理下,AaCHS16 的表达受到抑制,赖恭梯等[18]研究发现刺葡萄经红光处理后CHS 表达水平降低,与本研究所得结果一致。CHS 与植物体内的类黄酮等次生代谢物的生成有关,其活性与类黄酮含量紧密相连[38]。因此,可以推测,作为环境因素之一的光质,可能是通过影响植物体内CHS 基因表达量,进而影响青蒿中黄酮类物质的生成。这些结果也为深入研究AaCHS基因的功能,及光调控下黄酮化合物生物合成的调控机制提供了参考。
根据eggNOG-mapper 在线网站,对AaCHS基因进行GO 注释,以AaCHS 基因蛋白作为筛选范围,对AaCHS 基因进行功能注释,将其分为生物过程(biological process,BP)、细胞组分(cellularcomponent,CC)2 个大类,其中生物过程共富集了90 条序列;细胞组分共富集了19 条序列,其中包括的细胞质(cytoplasm)富集了11 条序列,数量最多,与亚细胞定位预测结果一致;推测AaCHS 基因在细胞质中调控黄酮类化合物的累积。为了研究AaCHS 基因家族成员在不同光照下的差异表达,将青蒿在蓝光、红光、白光、远红光调控的转录组测序数据作为实验组,而黑暗条件则作为对照组。根据差异基因表达火山图结果显示,AaCHS7、AaCHS10 是红光和黑暗条件下差异表达分析下的正向差异表达基因。推测它们在青蒿受到红光处理中起正向调控作用。此外,蔡伟等[39]对鱼腥草中的2 种材料(鱼腥草6 号和7 号)进行了差异表达分析,并筛选出4 个基因。本研究采用相同的筛选方法,即通过比较试验组与对照组之间的差异表达情况来确定目标基因。然而,在蓝光、白光、远红光以及黑暗条件下,未筛选出AaCHS 差异表达基因。
4 结论
本研究在青蒿基因组中发现了16 个CHS 基因家族成员,并且这些家族成员有12 个保守的基序。通过序列分析,发现AaCHS 家族相对较为保守,其蛋白质结构也比较松散;其含有大量激素、逆境、生长和光响应元件;有8 条没有非编码区,16条均包含CDS 外显子;CD-search 验证结果发现每个AaCHS 蛋白均有Chal-sti-synt_N 结构域;系统进化分析将青蒿(16 条)、雷蒙德氏棉(3 条)、毛果杨(3 条)、银中杨(11 条)、大豆(2 条)在内的5 个物种CHS 蛋白划分为5 个亚族。对AaCHS 基因家族成员表达分析,AaCHS10 和AaCHS16 在嫩叶、老叶、表皮、花蕾、种子、花和毛状体均显著上调;在青蒿嫩叶中AaCHS14 基因显著上调。对AaCHS 进行光调控分析,发现与黑暗处理相比,蓝光诱导能显著提高AaCHS10 基因的表达量,白光诱导则能显著提高AaCHS10 和AaCHS16 基因的表达量。AaCHS 基因进行GO 功能注释,富集结果分为2 个大类BP 和CC,其中包括的细胞质(cytoplasm)富集了11 条序列,数量最多,与亚细胞定位预测结果一致,推测AaCHS 基因在细胞质中调控黄酮类化合物的累积。通过差异表达分析,AaCHS7、AaCHS10 是红光和黑暗条件下差异表达分析下的正向差异表达基因,推测其在青蒿受到红光处理中起正向调控作用。
参考文献
[1]Vidic D, Čopra-Janićijević A, Miloš M, et al. Effects ofdifferent methods of isolation on volatile composition ofArtemisia annua L [J]. International Journal of AnalyticalChemistry, 2018,2018:9604183.
[2]Hao X L, Zhong Y J, Nützmann H W, et al. Light-inducedartemisinin biosynthesis is regulated by the bZIP transcriptionfactor AaHY5 in Artemisia annua [J]. Plant and CellPhysiology, 2019, 60(8): 1747-1760.
[3]WHO Expert Committee on the Selection, Use of EssentialMedicines, World Health Organization. The Selection and Useof Essential Medicines [M]. World Health Organization, 2014:1-88.
[4]Bilia A R, Lazari D, Messori L, et al. Simple and rapid physicochemicalmethods to examine action of antimalarial drugs withhemin: its application to Artemisia annua constituents [J]. LifeSciences, 2002, 70(7): 769-778.
[5]Liu C S C, Yang S L, Roberts M F, et al. Antimalarial activityof Artemisia annua flavonoids from whole plants and cellcultures[ J]. Plant Cell Reports, 1992, 11: 637-640.
[6]Cavaiuolo M, Cocetta G, Ferrante A. The antioxidants changesin ornamental flowers during development and senescence[J].Antioxidants, 2013, 2(3): 132-155.
[7]Iwashina T. Flavonoid function and activity to plants and otherorganisms [J]. Biological Sciences in Space, 2003, 17(1):24-44.
[8]Zhang P P, Du H Y, Wang J, et al. Multiplex CRISPR/Cas9-mediated metabolic engineering increases soya bean isoflavonecontent and resistance to soya bean mosaic virus [J]. PlantBiotechnology Journal, 2020, 18(6): 1384-1395.
[9]Pourcel L, Routaboul J M, Cheynier V, et al. Flavonoidoxidation in plants: from biochemical properties to physiologicalfunctions[ J]. Trends in Plant Science, 2007, 12(1): 29-36.
[10]Colinas M, Goossens A. Combinatorial transcriptional controlof plant specialized metabolism [J]. Trends in Plant Science,2018, 23(4): 324-336.
[11]Flores-Sanchez I J. Polyketide synthases in Cannabis sativa L[M]. Amsterdam:PrintPartners Ipskamp B.V., 2008:1-169.
[12]Hahlbrock K, Scheel D. Physiology and molecular biology ofphenylpropanoid metabolism [J]. Annual Review of PlantPhysiology and Plant Molecular Biology, 1989, 40: 347-369.
[13]Koes R E, Spelt C E, van den Elzen P J M, et al. Cloning andmolecular characterization of the chalcone synthase multigenefamily of Petunia hybrida[ J]. Gene, 1989, 81(2): 245-257.
[14]Franken P, Niesbach ‐ Klösgen U, Weydemann U, et al. Theduplicated chalcone synthase genes C2 and Whp (whitepollen) of Zea mays are independently regulated; evidence fortranslational control of Whp expression by the anthocyaninintensifying gene in [J]. The EMBO Journal, 1991, 10(9):2605-2612.
[15]Zhu L L, Ding Y Q, Wang S X, et al. Genome-wideidentification, characterization, and expression analysis of CHSgene family members in Chrysanthemum nankingense [J].Genes, 2022, 13(11): 2145.
[16]Hu H F, Shi B, Zhu W C, et al. Genome-wide identification,characterization and expression analysis of mango (Mangiferaindica L.) chalcone synthase (CHS) genes in response to light[J]. Horticulturae, 2022, 8(10): 968.
[17]贾昆鹏.植物激素独脚金内酯和茉莉酸信号与光信号互作的分子机制研究[D]. 上海:上海交通大学,2014.
Jia K P. The molecular mechanism of cross-talking betweenlight- and phytohormones strigolactone- and Jasmonatesignaling[ D]. Shanghai:Shanghai Jiao Tong University, 2014.
[18]赖恭梯,阙秋霞,潘若,等.刺葡萄查尔酮合成酶基因CHS 对不同光质的响应及转录因子调控分析[J]. 生物技术通报,2022,38(11):129-139.
Lai G T, Que Q X, Pan R, et al. Response of Chalconesynthase gene(CHS)to different light quality and transcriptionfactor regulation in Vitis davidii [J]. Biotechnology Bulletin,2022, 38(11): 129-139.
[19]Meng X C, Xing T, Wang X J. The role of light in theregulation of anthocyanin accumulation in Gerbera hybrida[ J].Plant Growth Regulation, 2004, 44(3): 243-250.
[20]Mariethoz J, Alocci D, Gastaldello A, et al. Glycomics@ExPASy: bridging the gap [J]. Molecular amp; CellularProteomics, 2018, 17(11): 2164-2176.
[21]Yu C S, Lin C J, Hwang J K. Predicting subcellularlocalization of proteins for Gram ‐ negative bacteria by supportvector machines based on n ‐ peptide compositions [J]. ProteinScience, 2004, 13(5): 1402-1406.
[22]Geourjon C, Deléage G. SOPMA: significant improvementsin protein secondary structure prediction by consensusprediction from multiple alignments [J]. Bioinformatics, 1995,11(6): 681-684.
[23]Waterhouse A, Bertoni M, Bienert S, et al. SWISSMODEL:homology modelling of protein structures andcomplexes [J]. Nucleic Acids Research, 2018, 46(W1):W296-W303.
[24]Lescot M, Déhais P, Thijs G, et al. PlantCARE, a databaseof plant cis-acting regulatory elements and a portal to tools forin silico analysis of promoter sequences [J]. Nucleic acidsresearch, 2002, 30(1): 325-327.
[25]Chen C J, Chen H, Zhang Y, et al. TBtools: an integrativetoolkit developed for interactive analyses of big biological data[J]. Molecular Plant, 2020, 13(8): 1194-1202.
[26]Tamura K, Stecher G, Kumar S. MEGA11: molecularevolutionary genetics analysis version 11 [J]. MolecularBiology and Evolution, 2021, 38(7): 3022-3027.
[27]Bray N L, Pimentel H, Melsted P, et al. Near-optimalprobabilistic RNA-seq quantification [J]. NatureBiotechnology, 2016, 34(5): 525-527.
[28]Jin J F, Wang Z Q, He Q Y, et al. Genome-wideidentification and expression analysis of the NAC transcriptionfactor family in tomato (Solanum lycopersicum) duringaluminum stress[ J]. BMC Genomics, 2020, 21: 288.
[29]Jez J M, Bowman M E, Noel J P. Structure-guidedprogramming of polyketide chain-length determination inchalcone synthase [J]. Biochemistry, 2001, 40(49): 14829-14838.
[30]Kreuzaler F, Ragg H, Heller W, et al. Flavanone synthasefrom Petroselinum hortense: Molecular weight, subunitcomposition, size of messenger RNA, and absence ofpantetheinyl residue [J]. European Journal of Biochemistry,1979, 99(1): 89-96.
[31]Jez J M, Ferrer J L, Bowman M E, et al. Structure andmechanism of chalcone synthase-like polyketide synthases [J].Journal of Industrial Microbiology and Biotechnology, 2001,27(6): 393-398.
[32]何锐杰,方庭,余伟军,等.西番莲查尔酮合成酶(CHS)基因家族全基因组鉴定及表达模式[J]. 应用与环境生物学报,2022,28(4):1066-1075.
He R J, Fang T, Yu W J, et al. Genome-wide identificationand expression analysis of the CHS gene family in passion fruit[J]. Chinese Journal of Applied and Environmental Biology,2022, 28(4): 1066-1075.
[33]何梦媛,姚华,李国治,等.甘草CHS 基因家族鉴定、表达特性分析及其与甘草查尔酮A 积累的关系研究[J]. 植物生理学报,2022,58(1):141-154.
He M Y, Yao H, Li G Z, et al. Identification of CHS genefamily and analysis of its expression characteristics in relation tothe accumulation of licochalcone A in Chinese licorice(Glycyrrhiza uralensis) [J]. Plant Physiology Journal,2022,58(1):141-154.
[34]Shen Q, Zhang L D, Liao Z H, et al. The genome ofArtemisia annua provides insight into the evolution ofAsteraceae family and artemisinin biosynthesis[J]. MolecularPlant, 2018, 11(6): 776-788.
[35]戢强强,陈伟达,陈敬东,等.青蒿苯丙氨酸解氨酶(AaPAL)基因家族鉴定及光调控分析[J]. 分子植物育种,2023,21(11):3544-3551.
Ji Q Q, Chen W D, Chen J D, et al. Family identification andphotoregulation analysis of phenylalanine ammonia lyase(AaPAL) gene of Artemisia annua [J]. Molecular PlantBreeding, 2023, 21(11): 3544-3551.
[36]Hassani D, Fu X, Shen Q, et al. Parallel transcriptionalregulation of artemisinin and flavonoid biosynthesis[J]. Trendsin Plant Science, 2020, 25(5): 466-476.
[37]Kitazaki K, Fukushima A, Nakabayashi R, et al. Metabolicreprogramming in leaf lettuce grown under different lightquality and intensity conditions using narrow-band LEDs[J].Scientific Reports, 2018, 8(1): 7914.
[38]孟祥春. 非洲菊花生长、花色素苷积累及CHS、DFR 基因表达的光调控研究[D]. 广州:华南师范大学,2004.
Meng X C. Flower growth,anthocyanin accumulation andCHS、DFR gene expression regulated by light in Gerberahybrida[D]. Guangzhou:South China Normal University,2004.
[39]蔡伟,肖敬忠,余小丽,等. 鱼腥草bZIP 基因的鉴定与分析[J]. 中草药,2023,54(13):4295-4305.
Cai W, Xiao J Z, Yu X L, et al. Identification and analysis ofbZIP gene in Herba Houttuynia [J]. Chinese Traditional andHerbal Drugs, 2019,54(13):4295-4305.
(编辑:吕俊俐)

