医学领域知识融合研究进展
彭 琳,宋 珺,熊玲珠,杜建强,叶 青,刘安栋
江西中医药大学计算机学院,南昌 330004
长期以来,医学领域的知识分散在各种信息载体中,未充分利用其价值。为发挥信息技术对医学传承创新发展的支撑作用,研究人员将知识图谱(knowledge graph,KG)引入医学领域,以提升电子病历、医案、古籍等知识的研究与利用效果。然而,不同知识图谱的来源多样,呈现多样性和异质性,可能导致数据重复和冗余,同时医学知识间存在互补。因此,如何有效融合和充分利用多源的医学知识,并减少研究中的重复工作,是一项意义深远的任务。
知识融合作为知识图谱研究中的核心问题之一,能够将不同知识图谱融合为一个统一、一致且简洁的形式,以实现应用间的互操作性[1]。在医学领域,知识融合旨在利用融合技术将分散在各个知识图谱或不同数据源中的医学知识进行对齐与合并,形成一个更全面的医学领域知识图谱,在提高知识质量、扩大规模、提高医学知识利用率和共享性等方面具有促进作用。
根据匹配对象的不同,知识融合可分为本体匹配(ontology matching)、实体对齐(entity alignment)和实体链接(entity linking)三类。本体匹配是指将不同本体之间的概念进行对齐和匹配;实体对齐旨在找到不同知识图谱中对应的实体;实体链接则用于将文本中的实体与外部知识图谱中的实体进行关联。由于技术发展迅速,本文主要调研了近5年医学领域知识融合任务的相关文献,并发现以下现象:(1)描述医学本体匹配的文献数量逐渐减少,且均不足另外两类文献的三分之一。(2)近年来,关于本体匹配的文献研究进展不多,本体更多用于辅助实体对齐或实体链接,以增强知识融合的准确性。(3)医学实体链接文献相对最多,但随着技术的发展以及各类知识图谱的构建,医学实体对齐的研究在近几年不断增长。因此,本文将聚焦于实体对齐和实体链接这两个核心任务。
目前,医学领域中与知识融合相关的综述较少,French 等[2]梳理了1980 年至2022 年生物医学实体链接的发展状况;Shi等[3]从技术角度分析了生物医学实体链接的发展历程,并探讨了应用于不同场景的数据集特征以及不同方法在各类数据集上的效果。上述两篇文献仅综述了实体链接任务,未涉及实体对齐任务。同时,本文紧扣“问题-方法”的思路,从任务的问题切入,通过分析现有研究方法给出相应的解决方案,具体结构如图1 所示。首先,系统梳理医学领域知识融合的定义、评价指标及数据集;归类医学领域知识融合中存在的问题。然后,按照问题、技术两个维度,综述了近年来医学领域知识融合中实体对齐、实体链接任务的相关方法,重点对最新研究进展进行对比和深入分析;在此基础上,针对每类问题,总结现有研究工作的解决思路与策略。最后,根据前文的分析,给出了医学领域知识融合的未来研究方向。
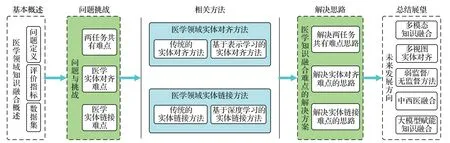
图1 组织结构图Fig.1 Organizational structure diagram
1 医学领域知识融合概述
1.1 问题定义
知识图谱是一个有向图,一般表示为G={E,R,T},其中,E、R、T分别代表知识图谱中的实体、关系、三元组的集合[4]。实体对齐、实体链接的定义如下:
定义1实体对齐(entity alignment,EA)也称实例匹配、实体消解,旨在将不同数据源或知识图谱中指代相同事物的实体进行匹配。具体而言,给定两个知识图谱G1={E1,R1,T1},G2={E2,R2,T2},目标是找到它们之间等价的实体对,其中“≡”表示等价关系,即实体ei与实体ej指向同一个事物。一般情况下,会给定一组已对齐实体对作为训练集,称为种子对齐(seed alignment)。医学实体对齐任务示例如图2(a)所示。
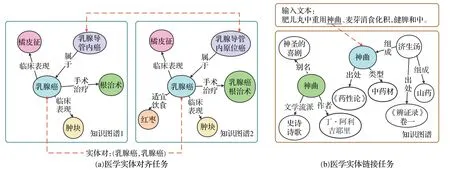
图2 医学实体对齐与实体链接任务示意图Fig.2 Schematic of medical entity alignment and entity linking tasks
定义2实体链接(entity linking,EL)有时也被称为实体消歧,侧重于将文本中的实体提及(mention)链接到知识图谱或知识库中对应的实体。其中,“实体提及”简称为“提及”,指用自然语言文本表示实体的语言片段;“实体”通常指代知识图谱或知识库中的实体对象。具体地,给定一个特定的医学领域知识图谱G={E,R,T},其中包含N个实体E={e1,e2,…,eN},同时,给定一个包含一组被识别的实体提及M={m1,m2,…,mM}的医学文本D,目标是找到实体提及mj∈M对应的知识图谱中的实体ei∈E。图2(b)展示了医学实体链接任务的示意图。
1.2 评价指标
医学领域知识融合任务采用的评价指标可分为两类:第一类指标为精确率P(precision)、召回率R(recall)、F1 值(F1-measure);另一类指标为Hits@k、MR、MRR。其中,这两类均可作为实体对齐任务的评价指标,实体链接任务则常用第一类作为评价指标。
(1)P、R、F1
以下公式中,TP表示模型正确预测的正样本数目、FP为模型错误预测的负样本数目、FN则代表被模型错误预测的正样本数目。
其中,这三个指标的数值越大,模型效果越好。并且,F1 值是用来评估不同模型的综合指标,综合考虑了P和R 的调和平均值。
对于医学实体对齐任务,P 表示正确预测的实体对数量与所有预测实体对数量之比,即正确对齐实体的比例;R 表示正确预测的实体对数量与所有真实存在的实体对数量之比,即正确对齐实体的覆盖率。
对于医学实体链接任务,P 衡量了链接到知识图谱的实体中有多少是正确的,即模型给出的链接中有多少是准确的;R 衡量了模型是否能够找到文本中的大部分实体提及并将它们正确地链接到知识图谱中,即模型有多少能够找到的实体提及被正确链接。
(2)Hits@k、MR、MRR
Hits@k:表示前k个命中率,即对齐结果中在前k名的正确对齐实体所占的比例,其中k是一个预先设定的整数。
MR(mean rank):平均排名,即正确对齐实体排名的平均值。
MRR(mean reciprocal rank):平均倒数排名,即计算正确对齐实体排名的倒数的平均值。该指标反映模型对于不同实体间相似度的区分能力。
其中,Hits@k、MRR 越大模型的效果越好,而MR 越小模型的效果越好。
1.3 数据集
基于知识图谱构建的实体对齐数据集中,DBP15k[5]是由DBpedia 不同语言版本链接而成的大型通用实体对齐数据集,包含了DBP15KZH-EN、DBP15KJA-EN和DBP15KFR-EN三个子版本;D-W-100K[6]的数据来自DBpedia和Wikidata,用于支持多领域知识图谱的实体对齐任务;MED-BBK-9K[7]是腾讯天衍实验室构建的基于两个医疗知识图谱的实体对齐数据集,包含9 162 个一对一实体对。
医学领域实体链接数据集中,NCBI 疾病语料库是由Doğan 等[8]构建的科学领域医学实体链接语料库,包含793篇生物医学文献摘要,常见数据集划分为593/100/100;COMETA[9]是一个医学社交媒体领域的实体链接数据集,由Reddit论坛上的医学实体提及和SNOMED CT[10]术语组成。医学知识融合数据集见表1。
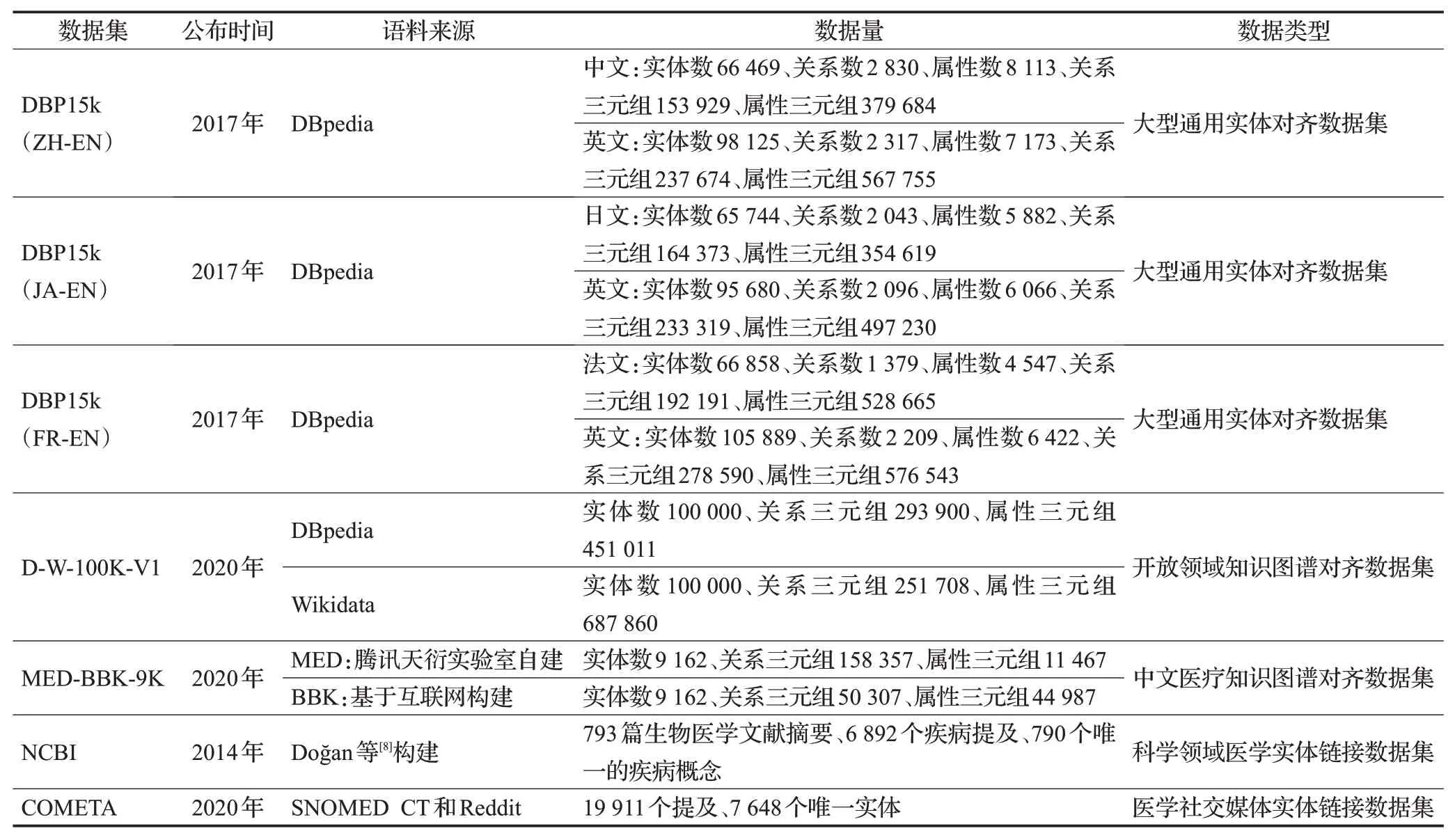
表1 医学领域知识融合数据集Table 1 Knowledge fusion datasets for medical field
2 问题与挑战
实体对齐和实体链接均为医学领域知识融合的重要任务,但它们的关注点有所不同。(1)任务目标上,实体对齐致力于解决不同医学数据源或知识图谱中相同实体的对应问题,例如,将不同数据源中描述的相同疾病“高血压”和“高血压病”进行匹配,以便在整合后的知识图谱中建立一致的关联;而实体链接旨在将医学文本中识别到的实体提及,如疾病、症状、治疗等,链接到外部知识源中的规范实体,从而丰富实体的语义信息,其中外部知识源通常为预定义的知识图谱。(2)在评测方面,实体对齐侧重于匹配质量和数据源整合,解决语义差异和质量问题;而实体链接更关注正确的实体消歧和上下文理解,以确保链接准确性。(3)应用场景中,实体对齐用于整合多源知识,以实现跨数据源查询和分析,如整合医学知识图谱以提供统一查询接口;实体链接则将非结构化文本与结构化的知识图谱关联,是医疗智能问答、基于知识图谱的信息检索、内容推荐等应用的基础。
相比于通用领域的知识融合,医学领域由于其专业性与复杂性特点给知识融合研究带来了诸多挑战。下文将具体分析医学领域知识融合任务所面临的共有难点,以及实体对齐和实体链接各自的难点。
2.1 医学知识融合中的共同难点
2.1.1 多样性与歧义性
多样性与歧义性问题分别表现出“多词一义”与“一词多义”的现象。(1)多样性问题:不同的研究机构和研究人员对于命名的习惯存在差异,同一个医学实体往往具有多个不同的名称,例如,“乳腺导管内癌”和“乳腺导管内原位癌”,“帕金森病”和“帕金森氏症”,“龋齿”和“烂牙”。这种实体命名多样性增加了知识融合任务的复杂性。(2)歧义性问题:同一个实体名称可以表示不同的含义,例如,“神曲”在中药领域是中药材,在文学领域属于一部文学作品;“山楂”既可表示为药物,也能表示为饮食。歧义性问题需要结合上下文信息进行消歧。示例如图3所示。

图3 多样性和歧义性问题示例Fig.3 Examples of diversity and ambiguity issues
2.1.2 标注数据的缺乏
在实体对齐中,通常需要大量人工标注的预对齐实体对,以便连接两个知识图谱。同样,在实体链接过程中也需标注数据进行训练。然而,由于医学领域的专业性,需依赖专业的医学人员进行标注,因此,获取高质量的标注数据困难且昂贵,这导致知识融合任务中标注数据的匮乏。
2.1.3 计算效率的问题
医学领域的知识图谱规模庞大,涉及大量的实体和关系。如何在合理的时间内高效完成实体对齐或实体链接计算,是一个具有挑战性的问题。因此,需要设计高效的算法和优化策略,以提高知识融合的计算效率。
2.2 医学领域实体对齐的难点
2.2.1 知识图谱异质性
知识图谱之间的结构异质性包含实体邻域异质性及关系异质性。(1)实体邻域异质性:目前许多研究建立在待对齐的实体对具有相似的邻域结构这一假设之上,但由于结构异质性的存在,两个知识图谱的对齐实体不一定具有相似或相同的邻域结构,如图4 所示,中心实体“乳腺导管内癌”与“乳腺导管内原位癌”仅有实体“乳腺癌”这一共同邻居,其余邻居均不同。(2)关系异质性:在现实生活中,不同来源的知识图谱通常具有关系独立性,即在一个知识图谱中存在的某个关系未必存在于另一个知识图谱中。例如,图4中知识图谱1存在的“适宜饮食”关系在知识图谱2中不存在。
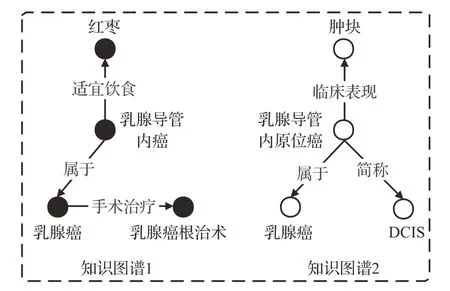
图4 知识图谱异质性示例Fig.4 Examples of knowledge graph heterogeneity
2.2.2 利用潜在的信息
在传统的实体对齐任务中,通常直接对医学实体进行对齐,而忽略了实体相关的潜在信息,如结构信息、属性信息和实体描述信息等。这种方法导致实体对齐任务的准确率较低,并容易产生大量的噪声和错误数据,例如,“心脏病”和“心脏衰竭”在名称上相似但不等价,若不考虑其他信息可能导致错误的匹配。因此,需要考虑如何更好地利用有效信息以提高医学实体对齐准确性。
2.3 医学领域实体链接的难点
2.3.1 未见实体问题
在医学领域,存在大量专有名词、罕见实体和新兴概念,如新的医学术语、疾病、治疗方法等,这些实体可能未在训练数据或知识图谱中出现。因此,未见实体问题可细分为两类:(1)训练集中罕见实体,其出现的频率较低,难以在有限数据中进行充分学习;(2)知识图谱中缺乏对应提及,导致文本中部分实体提及无法在知识图谱中找到对应项,通常称为“NIL(unlinkable mentions)实体”,如“Curry-Jones 综合征”[11]在2017 年前未被添加到SNOMED CT中。
2.3.2 短文本问题
医学领域的一些文本往往篇幅较短,例如,临床记录、病历摘要中简洁的描述,如“患者接受了青霉素治疗”;提及级别的文本很短,可能不足8个字符,如“腿扭伤后遗症”“左胧骨骨折上端”。这类短文本的语义信息有限,导致其缺乏足够的上下文信息来进行准确的实体识别和链接。因此,在医学实体链接中,短文本上下文信息不丰富的问题是常见的挑战。
3 医学领域实体对齐方法
随着研究的不断深入,医学实体对齐方法经历了传统方法和基于表示学习方法两大发展阶段。其中,传统方法包括基于词典和规则、基于相似性计算等方法,而基于表示学习的方法涵盖了翻译模型和深度模型等技术。各方法的区别、优缺点及适用范围,如表2所示。
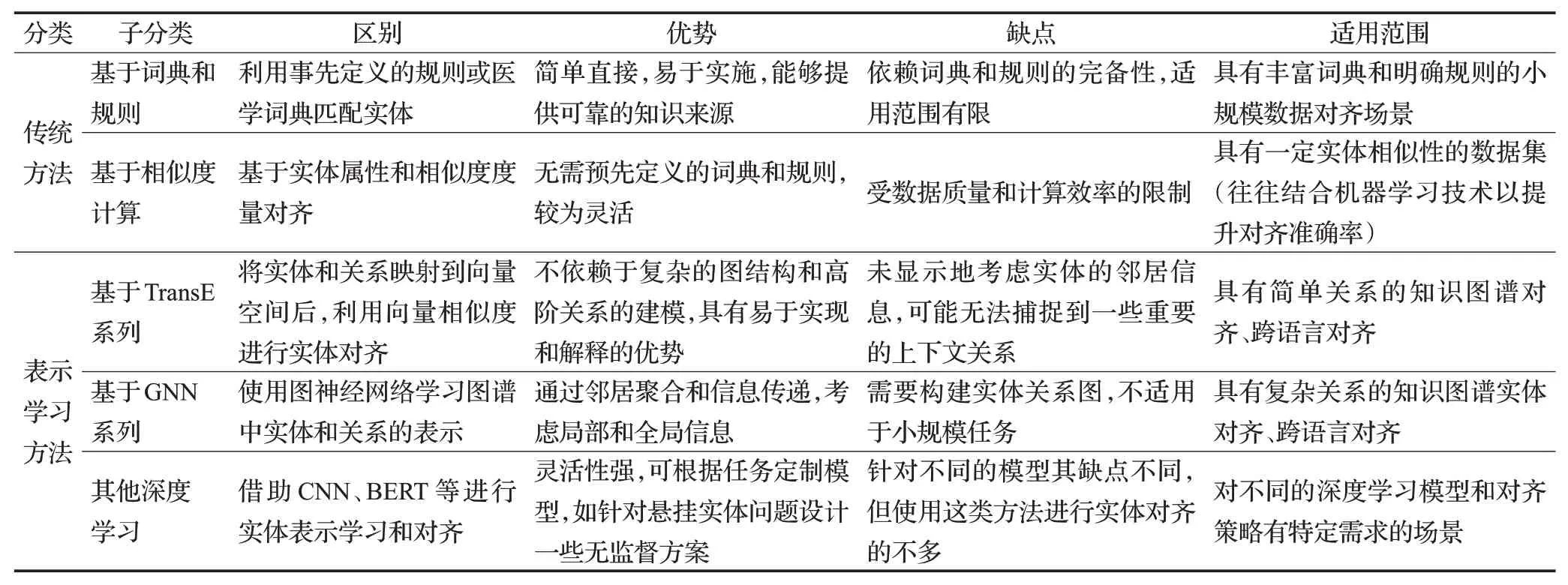
表2 医学实体对齐方法分类Table 2 Classification of medical entity alignment methods
3.1 传统的实体对齐方法
3.1.1 基于词典和规则的方法
早期的医学实体对齐方法大多采用词典和规则的方式。这类方法通过事先构建医学词典或定义规则来匹配实体,其优点是简单直接,易于实施。由于不同数据源的差异,医学实体对齐中存在大量实体名称不一致和术语描述不规范的情况。为此,王明强[12]以不孕症相关古籍为数据来源,基于知识规范化规则和行业标准,并根据不同知识元素特点,通过异名字符串匹配与人工校验相结合的方式实现实体对齐,解决了中医古籍中异名、简称和错误的情况。翟东升等[13]则针对不同术语的特点,制定不同规则以实现实体对齐,包括利用词典映射中医药别名,借助Uniprot 数据库构造包含靶点和基因的映射词典,通过字符串匹配标准化表述不一致的药性、味和归经信息等。
上述方法的共同特点是单纯依赖自定义规则或术语词典进行匹配,容易导致语义、语法等信息的缺失。为此,胡正银等[14]基于SPO三元组模型,通过UMLS[15]超级词表和多维映射技术,实现了多源异构领域知识的实体对齐。刘道文等[16]则尝试将ICD[17]术语体系作为桥梁,利用同义词与上下位关系对齐互联网医疗数据和电子健康档案真实数据,弥补了真实数据中疾病与科室关系的不足。然而,基于词典和规则的方法对词典、规则的依赖性高,且无法处理未知实体或术语,扩展性较差。
3.1.2 基于相似度计算的方法
为了克服基于词典和规则方法的局限性,研究人号提出了基于相似性计算的实体对齐方法。这类方法考虑实体的属性、关系和语义信息,通过计算相似度度量来确定实体对齐关系,较为灵活。Gong 等[18]提出将预处理与实体匹配方法相结合,以融合基于Web挖掘的多源糖尿病数据。而针对医学实体名称的独特性,An[19]设计了一个三阶段实体解析算法TSER,同时处理多源异构乳腺癌数据实体对齐过程中的标准库规模大、措辞相似的不同实体、同一实体的名称可能有大量字面差异等问题。
基于相似度计算的方法可以灵活应对不同的实体对齐需求,但受到数据质量和计算效率的限制,且在处理大规模数据时存在效率问题。随着传统机器学习的广泛应用,学者们开始将机器学习技术引入到相似度计算中,以进一步提升对齐准确性。宋文欣[20]通过构建同义医疗实体库应对多词一义问题,并使用三种无监督实体对齐方法来判断待对齐实体与候选实体间的相关程度,以缓解数据标注的不足,但由于数据广泛且差异大导致对齐效果不佳。同时,以上方法均未考虑现实应用中正负例样本占比不一致所引发的数据不平衡问题。针对这一问题,蔡娇[21]采用基于不平衡数据的机器学习方法,从单分类、数据、算法三个角度探究不同分类模型对遗传病领域实体对齐的影响,实验表明,对不平衡数据集进行处理能够提升对齐效果。传统机器学习方法可以通过训练模型来学习实体对齐的规律,具有一定的泛化能力,但需要手动调整模型参数和特征工程,这一过程相对繁琐且耗时。
3.2 基于表示学习的实体对齐方法
传统的实体对齐方法通常需要手工提取实体属性和关系特征,并要求对齐的实体在知识图谱中具有相同的属性和关系,因此容易受到知识图谱稀疏性和异质性的限制。而基于表示学习的方法可以自动学习实体的连续表示,通过学习实体的共同邻居或关系路径等信息来建模实体间的相似度。目前,基于表示学习的实体对齐方法以翻译表示(translating embedding,TransE)[22]系列模型、图神经网络(graph neural network,GNN)[23]系列模型为主。
3.2.1 基于TransE系列模型的方法
TransE由Bordes等[22]在2013年提出,被公认为知识图谱表示学习领域的里程碑。该模型基于距离度量思想,关注如何通过最小化实体在不同知识图谱中的关系表示间的距离来进行实体对齐。孙倩南[24]将TransE 运用到医院多个呼吸科室疾病数据库实体对齐任务中,但由于建模的两个知识图谱规模和信息差异较大,导致向量学习不够准确,进而影响了基于联合知识嵌入的实体对齐方法效果。在大多数方法中,未充分利用本体资源。为提高对齐准确性,Xiang 等[25]提出采用本体语义来增强知识图谱实体对齐的OntoEA 方法,该方法通过迭代共同训练策略整合基于TransE的实体嵌入、本体嵌入、类别冲突矩阵、成员关系嵌入和初始对齐嵌入模块,减少了类别冲突和误报问题,并在多个基准测试中获得良好效果。然而,引入本体会产生新的挑战,如本体中的类别冲突很难处理。虽然TransE计算效率高,易于实现,但仍然存在一些局限性,如图5 展示了TransE 的一系列改进方法。
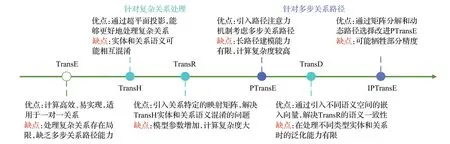
图5 基于TransE系列方法发展历程Fig.5 Development process of TransE series methods
TransH[26]、TransR[27]和TransD[28]方法旨在解决TransE处理复杂关系的限制。TransH 利用关系特定的超平面投影实体的向量表示,使得一个实体在不同关系中有不同的表示。而TransR改进了TransE和TransH中实体和关系在同一语义空间中映射可能导致实体和关系语义相互混淆的问题,其通过引入新矩阵表示关系的转换,将实体和关系分别映射到不同的语义空间中。由于医学知识图谱的复杂性,往往存在“一对多”关系。为此,Fang等[29]以TransR为基础模型之一,通过提取电子病历和网络中与垂体瘤相关的数据,依次对齐尾、头实体,并结合分类模型来学习和预测使用的字符、语义和结构三种特征,实验证明该分步对齐方法具有良好效果。TransD在TransR的基础上进一步改进,对头、尾实体使用不同的转换矩阵,以更好地处理复杂关系并降低计算复杂度。
PTransE[30]和IPTransE[31]方法弥补了TransE 缺乏考虑多步关系路径能力的问题。PTransE结合了TransE和TransH 的思想,引入基于路径的注意力机制,通过分配不同的注意力权重来捕捉多步关系的语义信息。在医学实体对齐任务中,PTransE 方法考虑到了关系信息中的多步关系路径,例如<小儿肺炎,表现为,发热><发热,对应药品,布洛芬><小儿肺炎,对应药品,布洛芬>。借助PTransE 的支持,程瑞[32]提出了一种联合关系信息和属性信息的迭代EA 方法,以解决现有方法忽略实体属性信息或将属性与关系信息混淆处理的问题。然而,PTransE 存在长路径建模能力不足、路径注意力的计算复杂度较高等问题。为了解决这些问题,IPTransE被提出,它采用矩阵分解和动态路径选择来改善PTransE的性能。
3.2.2 基于GNN系列模型的方法
基于TransE 系列实体对齐方法的优点在于其简单而直观的表示学习框架,通过学习实体间的关系向量进行对齐,取得了一定的效果。这类方法不依赖于复杂的图结构和高阶关系的建模,具有计算效率高、易于实现和解释的优势。但其未显示地考虑实体的邻居信息,因此可能无法捕捉到一些重要的上下文关系。随着图神经网络(graph neural network,GNN)的兴起,研究人员发现GNN 能够更好地利用图结构信息,通过多层次的邻居聚合和信息传递,更全面地考虑实体的上下文信息和全局信息,从而实现更准确、鲁棒的实体对齐。因此,研究重点逐渐转向基于GNN 系列的实体对齐方法,以进一步提升对齐任务的性能和灵活性。
图卷积网络(graph convolutional network,GCN)[33]是最先被广泛应用于实体对齐的图神经网络模型之一。Zhang等[34]通过结合语义和结构嵌入来衡量实体间的相关性,在采用预训练语言模型获得实体语义嵌入的同时,结合GCN捕获实体下位词和同义词的结构嵌入,以完成电子病历与术语库之间的实体对齐。然而,GCN在处理图数据时每个节点对邻居节点赋予了相同权重,使得GCN 无法充分考虑不同邻居节点的重要性差异。针对GCN 中固定权重聚合方式的不足,Velickovic 等[35]在2018 年提出图注意力网络(graph attention network,GAT)。GAT通过引入注意力机制使每个节点能够自适应地分配不同权重给邻居节点,从而更灵活地关注与当前节点相关性更高的邻居节点,增强了图神经网络的表达能力和学习能力。廖开际等[36]重视实体邻居的利用,借助多种注意力机制和图卷积神经网络清除实体冗余并赋予实体权重,通过实体邻域信息增强实体嵌入以完成多源乳腺癌数据的实体对齐,解决了实体邻域异质性问题。
上述方法未直接讨论或解决GNN由于多层堆叠引起的噪声问题,也较少关注实体与关系间的良性互动。对此,邬萌[37]提出RD-HRGCNs模型,首先,通过构造原始实体图的对偶关系图和使用图注意力机制,将关系信息融入实体表示中,弥补了GCN 忽略实体间关系信息的不足;其次,利用带有高速路神经网络门控(highway gates)的双层RGCN进一步捕获医疗实体的邻域结构信息并过滤噪声。次年,李丽双等[38]针对中文电子病历知识图谱间结构异质性的特点,设计了一个名为DvGNet的双视角并行图神经网络模型,该模型分别采用实体交互和关系交互视角缓解实体邻域异质性和关系异质性,并利用门控机制聚合嵌入表示以解决噪声传播问题,进而提高了模型的性能和效果。基于GNN系列的实体对齐方法可以更好地捕捉图结构中的关系和特征,因此整体效果略优于基于TransE系列的方法。在GNN模型中引入更丰富的知识来帮助实体对齐,仍然是一个值得探索的问题。
3.2.3 其他深度学习方法
除了基于TransE、GNN系列的方法,还有其他一些深度学习方法被应用于实体对齐任务。随着2018 年BERT(bidirectional encoder representation from transformers)[39]预训练语言模型的发布,研究者们也尝试将BERT 模型应用于实体对齐,并取得了良好效果。通过将两个实体的文本描述输入BERT,可以获得它们的语义表示,并对这些表示进行比较以判断是否对应同一实体。刘旭利等[40]将医疗实体视为短句,从而把病人事件图谱中医疗实体与医疗知识库实体的对齐问题转化为两个句子之间的语义关系多分类问题,通过使用基于字符特征和语义相似性的BERT 分类算法实现了知识融合任务。上述方法将一对术语作为序列输入到BERT中,并不生成单个术语的嵌入,可能会丢失术语级语义信息。针对这一问题,Ma 等[41]设计了SiBERT 模型,利用词嵌入序列生成术语级嵌入以增强相似性计算中实体的特征,并引入迁移学习机制对模型进行预训练有效缓解对数据的依赖,旨在将电子病历中治疗、检查、疾病和症状四类实体与ICD 标准术语进行对齐。该模型采用孪生网络使其训练速度比CNN和BERT更快,但由于模型训练数据量较小且数据标注难度大,在应用上存在局限。
在当前深度学习盛行的时代,也不乏有研究者将传统方法与深度学习相融合,并提出新的框架,旨在提升医学实体对齐的效果。例如,Tang等[42]提出了一种融合Gromov-Wasserstein 距离的无监督实体对齐框架,通过联合优化实体语义和图结构充分利用知识图谱的结构信息,并结合三阶段渐进优化算法以应对相关的计算挑战,在多个数据集上取得了良好的结果,但该框架在处理悬挂实体时的能力有限。悬挂实体是近年来实体对齐新问题,指在不同知识图谱之间找不到对应的实体,这些实体的存在可能会影响知识图谱的完整性和准确性,即并非所有实体都有对应的等价实体。为了解决这一问题,Luo 等[43]基于UMLS 构建了一个具有实体对齐(EA)任务和悬挂实体检测(DED)任务的医学跨语言知识图谱数据集MedED。此外,提出了一种联合EA 和DED的无监督方法UED,该方法挖掘文字语义信息,为EA生成虚拟实体对和全局引导对齐信息,然后利用EA结果来协助DED,在解决悬挂实体对齐问题的同时消除了对种子对的需求。然而,所提出的方法依赖于文字语义信息的可用性,而该信息并不总是可靠的。对此,Xu 等[44]为充分利用知识图谱中图结构信息,提出一种弱监督框架WOGCL,其在模型、训练和推理三个方面进行了改进。具体来说,通过引入门控图注意力网络来捕捉局部和全局图结构相似性,设计了对比学习和最优传输学习的两个学习目标;通过最优传输计划获得可区分的实体表示,提出了基于PageRank 的方法来计算高阶结构相似性。WOGCL仅使用纯图结构信息,解决了跨语言知识图谱中的悬挂实体对齐问题并增强了实体对齐框架的可推广性。如表3 总结了基于表示学习的医学领域实体对齐方法。
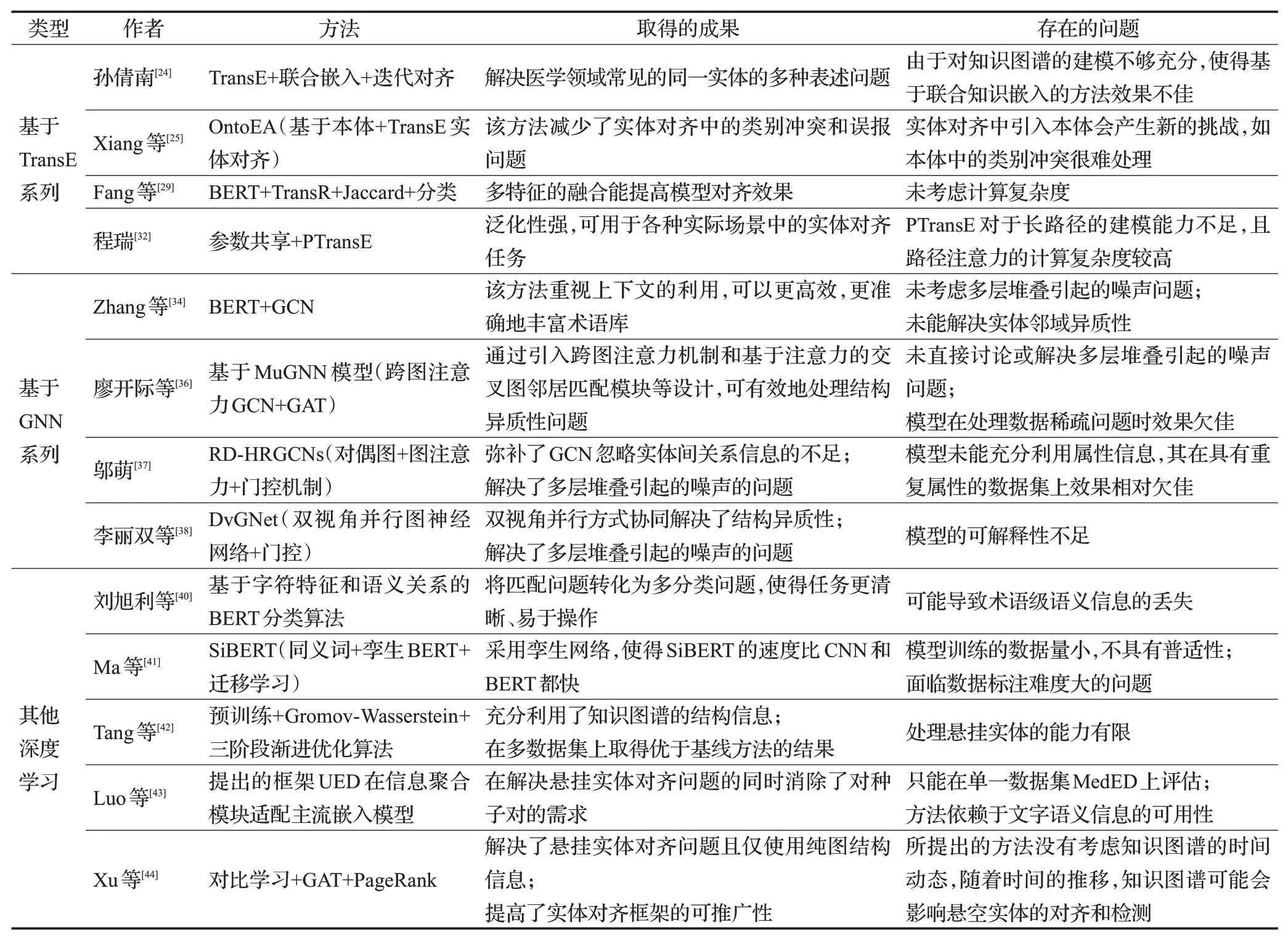
表3 基于表示学习的医学实体对齐方法Table 3 Medical entity alignment method based on representation learning
4 医学领域实体链接方式
实体链接各方法的优缺点及适用范围,如表4所示。
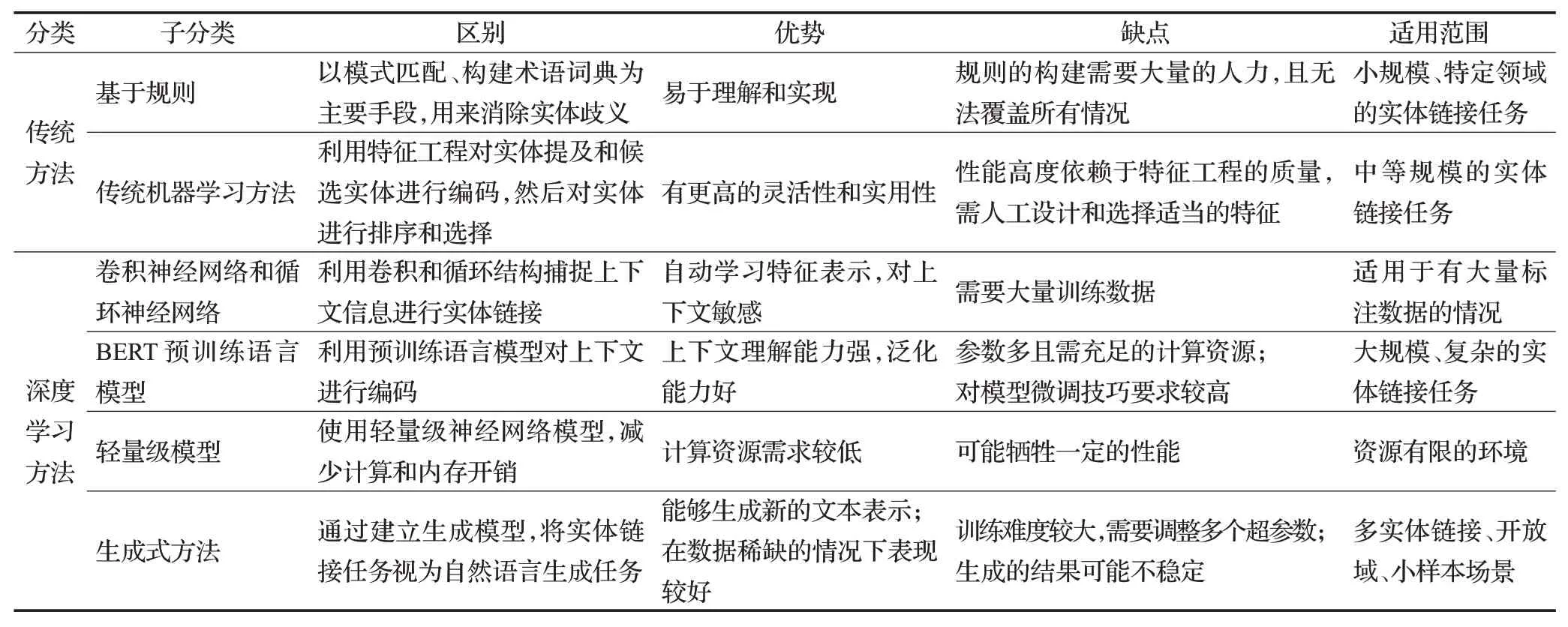
表4 医学实体链接方法分类Table 4 Classification of medical entity linking methods
4.1 传统的实体链接方法
4.1.1 基于规则的方法
早期的医学实体链接方法基于规则,以模式匹配、构建术语词典为主要手段,主要用来消除医学实体的歧义。模式匹配方法通常根据设定的拼写规则、指示词、构词规则和前后缀字符串等来定义模板,然后应用这些模板进行精确或部分匹配。例如,Li等[45]提出的基于规则的模型通过创建一组句法规则或语义约束来解决医学实体歧义性问题。然而,这种方法很难处理实体的别名、缩写词等多样形式。基于词典的方法将实体链接问题转化为词典匹配问题,该方法使用构建完善的词典来识别和匹配实体,其中包含大量词汇缩写、变体、同义词和昵称等条目。Xiu 等[46]和Liu 等[47]利用构建的词典扩展不匹配的实体提及,并采用基于排名的余弦相似度方法消歧,分别构建了消化系统肿瘤知识图谱、类风湿关节炎知识图谱。但由于医学实体名称的多样性,这类方法需要维护一个庞大的词典,并且难以处理新词。
不同的技术可以用来辅助这两种方法,以便更好地解决医学实体链接中的问题。例如,为了解决中文医学实体链接中可能出现的错别字或音译差异、措辞相似但不同的实体等困难,Sun 等[48]设计了多级相似性匹配方法。尽管基于规则的方法易于理解和实现,但存在以下问题:规则的构建需要大量的人力,且无法覆盖所有情况;规则的设计往往依赖于实体的形态特征,因此在区分形态上相似但语义不同的语境时变得困难;一个领域中设计的实体规则无法直接迁移至另一个医学领域,例如,专为电子病历设计的规则并不适用于古籍。
4.1.2 基于传统机器学习的方法
基于传统机器学习的方法主要利用特征工程编码实体提及和候选实体,然后通过相似度计算对实体进行排序和选择,进而实现实体链接任务。相较于基于规则的方法,该方法灵活性和实用性更高。将机器学习应用于医学实体链接的最早尝试之一是DNorm[49]。该方法引入了成对学习排序的思想,通过学习文本中疾病实体提及与知识库中概念名称的相似性来进行评分,不仅能够处理同义词、多义词和非一对一的关系,而且可以专门用于解决缩写和词序变化问题。然而,监督方法依赖于大量标注数据,且难以处理未见实体的情况。
相比于监督方法,无监督方法不需要标注数据,能够自动发现实体间潜在的关联关系。Wu等[50]同样针对临床缩写问题,开发了一个临床缩写识别和消歧的框架CARD。与DNorm思想不同的是,CARD利用机器学习方法识别缩写,并基于聚类生成缩写的可能含义,然后利用基于概要和向量空间模型进行词义消歧。Angell等[51]也设计了一个基于聚类的模型,考虑了文档内和跨文档间的实体提及关系,通过聚类多个提及并共同预测链接,以更好地应对医学文本中存在的模糊或不明确指代的挑战,但其性能受到聚类算法的选择和参数设置的影响。
随着研究的深入,研究人员尝试在医学实体链接中应用更多的机器学习算法。其中,PageRank 算法最初为评估搜索引擎结果相关性而设计,在使用基于图的表示时也常用于实体链接。例如,Duque 等[52]利用基于共现信息的图自动构建知识图谱,并采用PageRank 进行词义消歧,该方法不仅考虑了歧义词的上下文信息,还能在不依赖大量外部资源的情况下提高消歧的准确性。而为了弥补知识图谱中缺失的领域知识,Ruas等[53]提出一种将关系抽取用于实体链接的框架,通过自动提取的关系来构建消歧图,然后利用PageRank 和本体信息选择每个实体的最佳候选,从而提高实体链接性能。传统机器学习方法在性能和准确性方面相较于基于规则的方法取得了显著进展,但其性能高度依赖于特征工程的质量,需要人工设计和选择适当的特征。此外,该方法的语义信息抽取和表示能力有限,难以处理复杂语义关联和上下文依赖。
4.2 基于深度学习的实体链接方法
近年来,随着深度学习的发展,神经网络因其出色的泛化能力而在医学实体链接中备受关注,并取得了良好的链接效果。这些模型可以自动学习特征和表示,具备强大的表达能力,并能有效地捕捉文本中的复杂语义和上下文关系。
4.2.1 卷积神经网络和循环神经网络
早期基于深度学习的实体链接技术主要采用卷积神经网络(convolutional neural network,CNN)[54]。Luo等[55]通过引入匹配张量和多视图CNN 模型,结合多任务共享结构,以解决中文医学短文本规范化和非标准表达问题。但由于卷积核的大小限制,CNN 只能学习到文本中的局部信息。为了克服该局限,具有参数共享和记忆性的循环神经网络(recurrent neural network,RNN)[56]被提出,并逐渐取代CNN的地位。
随后,RNN 的变种之一,长短期记忆网络(long short-term memory,LSTM)[56]因解决了RNN 存在的梯度消失、爆炸问题而成为许多自然语言处理应用的主要模型,并广泛应用于医学实体链接任务中。在后续研究中,Bi-LSTM[57]弥补了LSTM 处理上下文信息时的缺陷,可以更好地捕捉双向上下文信息。Yan 等[58]提出了一种无监督方法,使用多实例学习来提高链接的准确性。通过构建一个中文医疗实体链接数据集,利用Bi-LSTM作为编码器获取实体和上下文的表示,并使用排序网络对候选实体进行评分,在解决标注数据难以获取的同时缓解了知识库稀疏性。考虑到实体链接基于命名实体识别,部分学者尝试对医学命名实体识别和实体链接任务进行联合建模。然而,在上述方法中,Yan等[58]将这两项任务视为独立步骤导致错误级联和相互支持不足,而Luo 等[55]简单地联合建模两项任务却无法编码复杂特征。为了解决这些问题,Zhao 等[59]提出了一种基于Word2Vec、CNN 和Bi-LSTM 方法的深度神经多任务学习框架。该框架通过显式反馈策略共同建模这两项任务,并通过多任务学习提供的通用表示增强任务之间的相互作用,从而提高了联合执行效率。
4.2.2 BERT预训练语言模型
最近,基于BERT的预训练语言模型在许多医学实体链接任务中得到了有效应用。Liu等[60]提出了一种自对齐的预训练模型SAPBERT。该模型利用从统一医学语言系统(UMLS)中通过聚类提取的同义词集对BERT进行微调,以应对医学领域特定的细粒度语义关系的挑战,并有效提升了医学实体链接的性能。同年,Liu等[61]在SAPBERT 基础上引入领域特定知识,尝试完成跨语言医学实体链接任务。该研究通过建立一个包含10种语言的跨语言评估基准,并提出跨语言转移方法,展示了如何将领域特定知识从英语传递到资源匮乏的语言,在目标语言中实现了性能的提升。
为了更好地利用BERT技术,研究人员结合特定任务需求进行改进,并引入数据增强、对抗训练进一步提高链接性能。Dong等[62]专注于发现不在知识库(out-of-KB)中的实体提及,提出一种基于BERT 的实体链接改进方法BLINKout。该方法通过将这些提及与特殊的NIL 实体进行匹配,能够有效识别知识库之外的提及;采用经过微调的NIL实体表示、同义词增强和特定领域的语言模型,以增强同义词对实体链接的作用。而针对实体提及表达的一致性问题,Li等[63]引入Stacking-BERT模型,利用基于BERT 的原始排名模型和Stacking-BERT 排名模型来捕获语义信息,并通过堆叠机制选择最佳映射对,以实现将临床术语自动映射到中文ICD编码。并采用对抗训练和数据增强的技巧,有效地提高了模型在小样本上的效果。
4.2.3 轻量级模型
然而,BERT模型参数过多且需要大量的计算资源,这限制了其在资源有限的场景中的应用。研究人员发现,当输入的单词顺序被打乱或者注意力范围受限制时,现有基于BERT 模型的医学实体链接性能变化很小。这意味着在解决这类特定任务时,使用像BERT这样的大型模型可能存在不必要的计算成本。为此,文献[64-66]提出了用于医学实体链接的轻量级神经网络模型,这些模型分别采用具有注意机制的对齐层、残差卷积神经网络、基于内部和外部实体的注意力3种不同的方式,以降低模型的复杂性和资源消耗,实现了与基于BERT 模型的先前工作相媲美的性能,并且在资源有限的场景下具有更高的效率。其中,Abdurxit 等[66]通过整合自注意力和交叉注意力模块,能够更好地捕捉医学提及和候选实体之间以及实体之间的信息。
4.2.4 生成式方法
不同于上述方法的思想,生成式方法试图通过建立一个生成模型,将实体链接(EL)任务视为自然语言生成(NLG)任务。在该任务中,模型接收一个包含提及的文本作为输入,然后生成相应链接的实体名称作为输出。Yuan 等[67]尝试将生成式方法应用于医学领域实体链接中,通过知识图谱引导的预训练和同义词感知微调来提升生成式EL 效果,解决了缺乏大规模人工标注和多个同义词的问题。De等[68]提出的自回归实体链接模型也采用了生成式方法,通过生成观察到的提及-实体对来学习。该模型采用浅层LSTM 解码器实现并行计算,并引入判别性修正项来改进解码器的排序,解决了以往自回归生成方法中计算成本高、无法并行解码和需要大量数据训练的问题。然而,该方法需要候选集或知识图谱的支持,并且缺乏对生成样本的重新排序能力。
为了解决上述问题,Mrini 等[69]在De 等[68]的基础上进一步改进,提出一种编码器-解码器的自回归实体链接模型。通过将模型与提及检测和实体匹配预测这两个辅助任务一起进行训练,并学习在推理时重新排序生成的样本,取得了多个数据集上的最佳性能,同时解决对预定义候选集依赖问题。此外,消融实验证明了每个辅助任务对主任务性能提升的重要性,以及重新排序对性能提升起到的关键作用。尽管在医学领域应用生成式方法进行实体链接任务引起了相当大的关注,但生成式EL方法在训练过程需要大量的计算资源才能达到高性能。因此,需要进一步研究以克服这一挑战。表5总结了基于深度学习的医学实体链接方法。
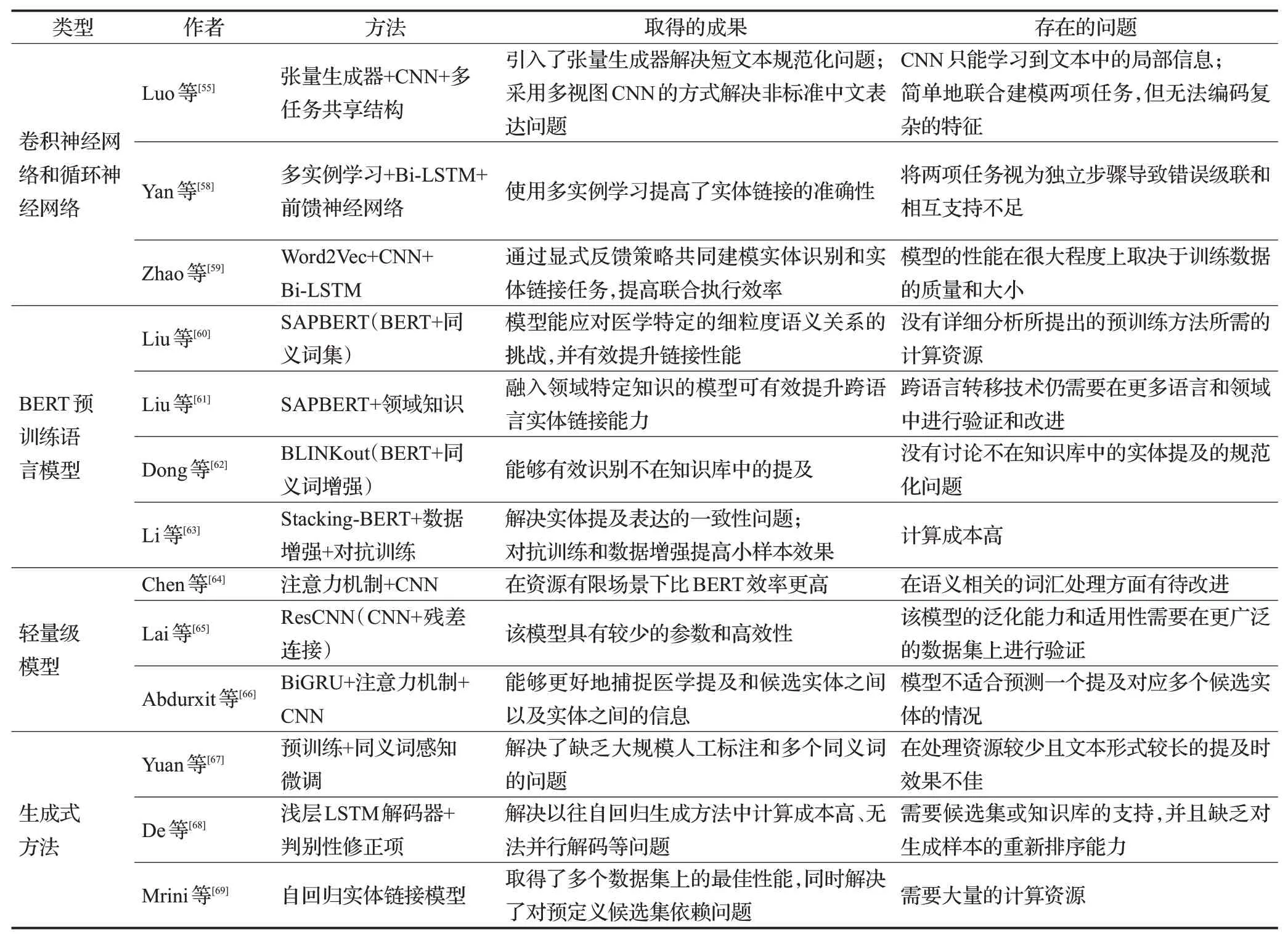
表5 基于深度学习的医学实体链接方法Table 5 Medical entity linking methods based on deep learning
5 医学知识融合难点的解决方案
5.1 解决两任务共有难点思路
5.1.1 多样性与歧义性
实体命名多样性与歧义性是医学领域知识融合中的常见问题。实体对齐任务中,实体命名多样性表现为两个不同知识图谱中的实体在进行一对一对齐时可能由于命名差异而出现错误;而在实体链接任务中,则是多个实体提及链接到知识图谱中的一个标准实体。以下是解决该问题的主要方法:
(1)借助同义词词典。包括维护症状词汇字典[70]、构建同义医疗实体库[20]、构造医学词根库[38]等。例如,Xie 等[70]通过将疾病实体映射到ICD-10 以维护症状词汇字典,并使用基于编辑距离的相似性函数对实体名称进行匹配,解决实体命名多样性问题。
(2)语义相似度匹配。利用自然语言处理技术计算实体名称的语义相似度,将语义上相似的实体进行对齐,从而弥补命名差异带来的问题。例如,An[19]通过对乳腺癌疾病术语的核心词拆分候选,并结合编辑距离、BERT+Cosine和BERT+ESIM等多种语义相似性计算方法,最终融合得到相似性并对其排序。
(3)迁移学习与预训练模型。刘龙航[71]设计融入多种传统特征的BERT 匹配模型对临床术语和疾病实体进行处理,提高实体对齐准确性。Ma 等[41]引入迁移学习机制并利用同义词词典对模型进行预训练,提高模型对多样性实体命名的适应能力。
(4)规范化和标准化。无论是实体对齐还是实体链接,都可以通过规范化和标准化方法统一不同数据源的实体命名,以缓解多样性带来的难题。例如,Yuan 等[72]提出基于知识图谱和对比学习的CODER 方法,利用医学知识图谱中的术语和关系三元组进行对比学习,实现电子病历术语规范化,从而解决多样性问题。
相对于多样性问题,医学领域的歧义性问题常见于实体链接任务。王莹[73]基于融入医疗实体的上下文语义信息特征对待消歧实体和候选实体进行余弦相似度计算,以完成实体消歧过程,从而提高实体准确性。例如,消歧完成后,对“山楂”一词,查询实体关系“药物治疗”时被细化为“山楂·药物”,查询实体关系“适宜饮食”时,该词则细化为“山楂·饮食”。
5.1.2 标注数据的缺乏
针对标注数据缺乏问题,主要解决方法如下:
(1)自监督学习。其核心思想是通过设计辅助任务从数据中生成伪标签,然后利用这些伪标签进行模型训练。如Zhang等[74]利用基于领域知识的自监督方法,并使用对比学习训练上下文编码器,弥补了标注数据缺乏的问题,但其自监督质量需进一步增强。
(2)主动学习。这是一种智能的数据采样方法,它通过挑选对当前模型不确定的样本,请求专家进行标注,从而有针对性地增加标注数据,提高模型性能。如Oberhauser等[75]通过使用主动采样策略和基于BERT的双编码器技术,能够支持医学专家进行数据标注,并链接到大型知识图谱中的实体,有效减轻标注负担。
(3)对抗训练。在医学领域知识融合任务中,对抗训练可以通过增强模型的稳健性来减少标注数据的需求。例如,Wiatrak等[76]通过设计和应用基于快速梯度符号方法(fast gradient sign method,FGSM)的对抗正则化方法,并结合基于代理的度量学习损失,模拟硬负样本的采样以增强训练对抗性,从而有效地解决了候选检索阶段的标注数据缺乏问题。
(4)无监督方法。无监督实体对齐能够在没有任何先验知识或者人工标注的情况下,自动地将两个知识图谱中相同实体进行匹配。Qi 等[77]首次尝试将传统概率推理和语义嵌入技术相结合,提出了PRASE方法,以消除种子对的需求。首先,使用概率推理选择高置信实体映射作为种子对,然后基于这些种子对训练语义嵌入模块,并不断迭代更新。相比之下,无监督实体链接方法的实现相对多样,包括基于共现图的无监督技术[52]、利用现有知识库并基于自注意力机制的方法[78]、多实例学习结合ICD-10 聚类方法[58]、利用潜在细粒度信息的模型[79]、基于聚类的实体链接模型[51]等。
5.1.3 计算效率的问题
针对计算效率问题,主要从注意力机制、模型结构、嵌入表示、编解码器四个方面进行改进。
(1)优化注意力机制:Abdurxit等[66]结合内部和外部实体注意力机制,整合医学实体提及和候选实体之间以及实体之间的信息,实现更好的性能表现和更高的推理速度。
(2)模型结构优化:改进模型的结构,设计更高效、轻量级的神经网络模型,如使用残差卷积神经网络ResCNN[65]或基于CNN 的轻量级神经模型[64],以减少参数数量和计算资源的需求,从而提高计算效率。
(3)改进嵌入表示:可以采用不同的方法来优化嵌入表示,例如,Chen等[64]引入更多特征来改进嵌入表示;Ma等[41]基于孪生式BERT网络,通过直接计算每个术语级别的嵌入表示相似性,提高了准确性并降低了计算成本。
(4)优化编解码器:Bhowmik 等[80]和De 等[68]将医学实体链接任务看成一个端到端的过程,分别从编码器和解码器角度进行了改进,以提高处理速度。Bhowmik等[80]提出基于BERT 的双编码器模型,通过提及编码器和候选编码器的协同作用,可以一次同时处理文档中的多个实体提及;而De 等[68]则通过引入判别性修正项和浅层解码器,实现高效的并行解码计算。如表6总结了医学领域知识融合共有难点的解决方案。
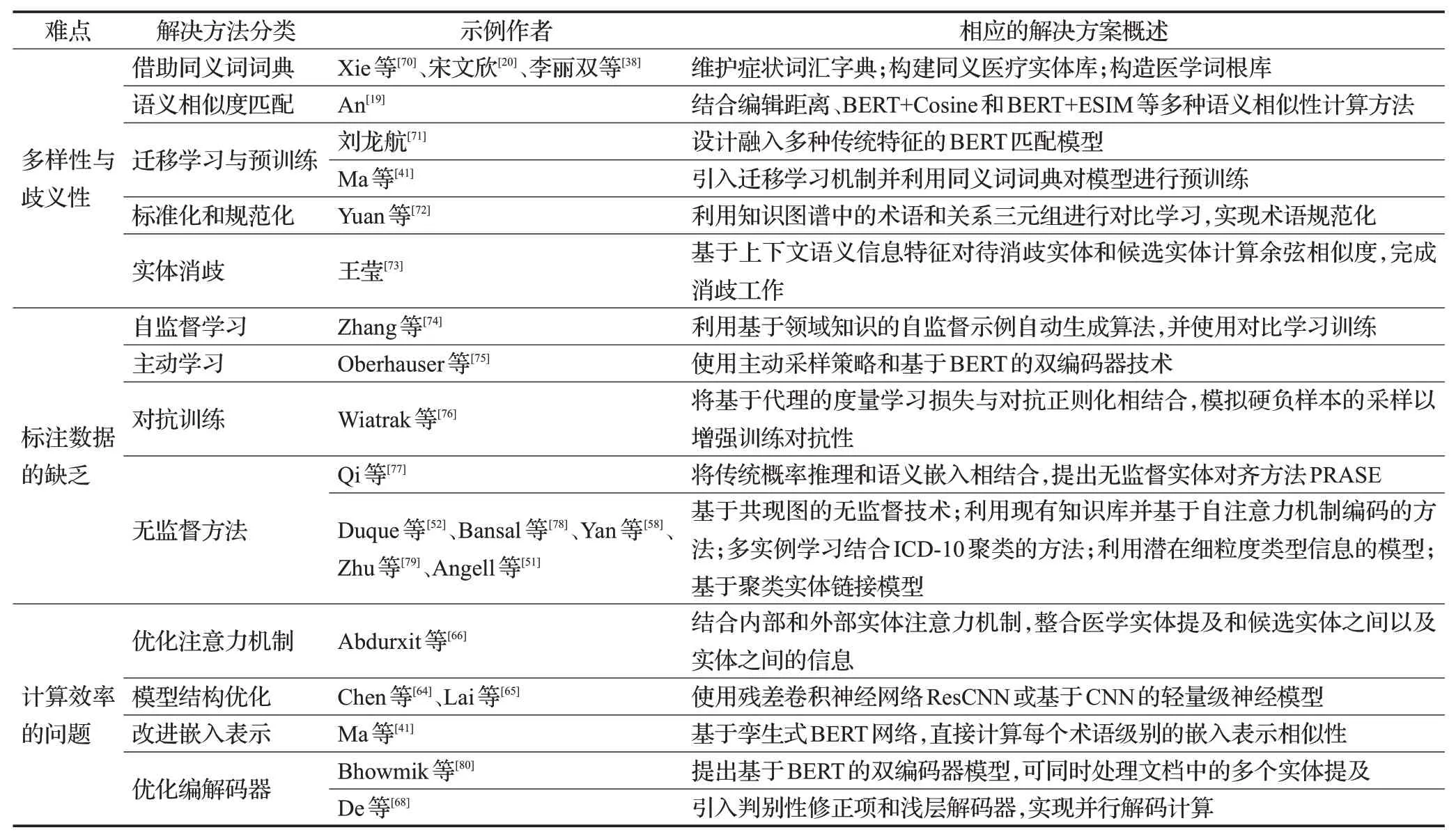
表6 医学领域知识融合共有难点的解决方案Table 6 Challenging solutions for knowledge fusion in medical domain
5.2 解决实体对齐难点的思路
5.2.1 知识图谱异质性
针对知识图谱之间的结构异质性,主要有引入图注意力机制、引入远距离邻居以及多视图学习三种方法。通用领域已有不少研究,在图神经网络中引入图注意力机制可以对不同邻域赋予不一样的权重,以缓解实体邻域异质性。例如,KGNN[81]、MuGNN[82]和NMN[83]等模型均引入了图注意力机制,但在具体方法上略有不同。其中,KGNN 通过联合训练基于TransE 的模型和基于GAT 的模型来处理跨语言知识图谱的异质性;MuGNN通过多个通道对知识图谱进行编码,实现了自注意力和跨图注意力,从不同的角度增强了实体嵌入的结构信息;NMN通过采样和匹配实体的邻域,来选择信息丰富的邻域并捕捉邻域之间的差异,从而更准确地估计实体之间的相似度。Sun等[84]提出的AliNet模型同时考虑了直接邻居和远距离邻居,该模型通过引入远距离邻居来扩展实体邻域结构的重叠部分,并使用注意机制和门控策略来聚合多跳邻域信息,以减轻不同知识图谱中实体邻域异质性。
近年来,医疗领域对该难点的研究逐渐受到关注,廖开际等[36]将MuGNN 模型应用于医学领域,通过引入跨图注意力机制和基于注意力的跨图邻域匹配模块等设计,来缓解实体邻域异质性,从而有效地处理异质性问题。然而,以上研究主要聚焦于实体邻域异质性,却忽略了关系异质性对结构异质性的影响。对此,李丽双等[38]采用多视图学习的方式,尝试从实体和关系两个不同的图谱视角学习嵌入表示,并通过门控机制将它们结合起来,以全面缓解电子病历知识图谱的结构异质性。
5.2.2 利用潜在的信息
现有研究通过考虑属性信息、结构信息和语义信息来提高医学实体对齐的准确性。一些研究[37,44]通过充分利用其中一种信息来达到不错的对齐效果,而另一些研究[29,32,77]则考虑多种信息以融合多维特征。例如,程瑞[32]提出了两种联合实体对齐方法,用于解决现有方法忽略实体属性信息或将其与关系信息混淆处理,以及使用相同模型对知识图谱中不同信息建模限制精度提升这两大问题。其中一种是迭代方法,它充分结合了关系信息和属性信息;另一种方法则基于GCN和TransE,先对结构、关系和属性信息分别建模,再有效地联合利用这些信息。为了解决现有方法未充分利用图结构信息的问题,Xu 等[44]采用门控图注意力网络来捕捉局部和全局图结构相似性,同时,利用对比学习和最优传输学习的目标,以获得可区分的实体表示。通过这种方式,能够仅利用结构信息就完成医学实体对齐任务。而Tang 等[42]在其最新研究中通过联合优化实体语义和知识图谱结构的方式,充分利用了知识图谱的结构信息,而不是仅仅将其隐式编码到嵌入中,实验表明,该方法在多个基准数据集中取得不错的效果。需要注意的是,信息的利用并非越多种类越好,有时过多信息反而会产生噪声,从而干扰实体对齐。因此,应根据不同数据集特点及特定领域需求,充分利用有效信息。
5.3 解决实体链接难点的思路
5.3.1 未见实体问题
下面分别对未见实体中的两类问题提供解决思路。
针对训练集中罕见实体问题,Varma 等[85]使用跨领域数据集集成方法,将通用文本知识库中的结构化知识转移到医学领域,增强医学实体的结构资源,从而提高对罕见实体的泛化能力;Ujiie等[86]结合了跨度表示和字典匹配特征,其利用神经网络将从跨度表示中获得的上下文分数和字典匹配分数加权和,来预测每个跨度的疾病概念;Angell等[51]采用基于聚类的推理方法实现医学实体链接。通过推理方法构建图,并利用聚类将相似节点聚合形成提及组,若其中一个提及正确链接到实体,整个组即正确分类,间接解决未见实体问题。
而对于知识图谱中缺乏对应提及问题,即不可链接实体的预测,可以采用后修剪和阈值法、利用深度学习模型等方法。例如,Yuan等[87]探索了医学实体链接中的部分知识图谱推理问题,并提出了后修剪和阈值法这两种修复方法以解决无法链接提及(NIL)问题,从而提高部分知识图谱推理的性能;Dong 等[62]提出了BLINKout方法,利用BERT 模型对实体链接进行改进,将未见实体的提及与特殊的NIL 实体匹配,以识别在UMLS、SNOMED CT等语料库中不存在的提及。
5.3.2 短文本问题
在解决医学领域尤其是中文的短文本问题上,研究人员专注于以下两个方面:(1)如何扩展短文本内容;(2)如何巧妙运用语义匹配模式之间的微妙差异。Luo等[55]提出了多视图卷积神经网络的多任务框架,通过引入匹配张量将短文本比较扩展到字符、词和句子层面建模提及与实体交互,解决中文临床诊断和手术名称的短文本问题。Li等[63]使用基于BERT的融合模型将中文实体链接视为句对分类任务,以捕捉临床实体提及的语义信息,并通过生成硬负样本增强模型的特征学习能力,该方法巧妙运用了语义匹配的细微差别。Vretinaris等[88]将文本片段中的实体提及表示为查询图以捕捉它们的相互关系,其中不同的节点类型表示药物、不良反应、症状和发现,然后运用图神经网络将医学实体链接问题建模成图匹配问题,实现了扩展短文本内容并充分利用语义匹配的微妙差异。尽管其是在英文医学文本上的应用,但思路对中文具有借鉴作用。这些方法为解决复杂领域的实体链接短文本问题带来了新的视角和可能性。表7 总结了医学领域实体对齐与实体链接各自难点的解决方案。
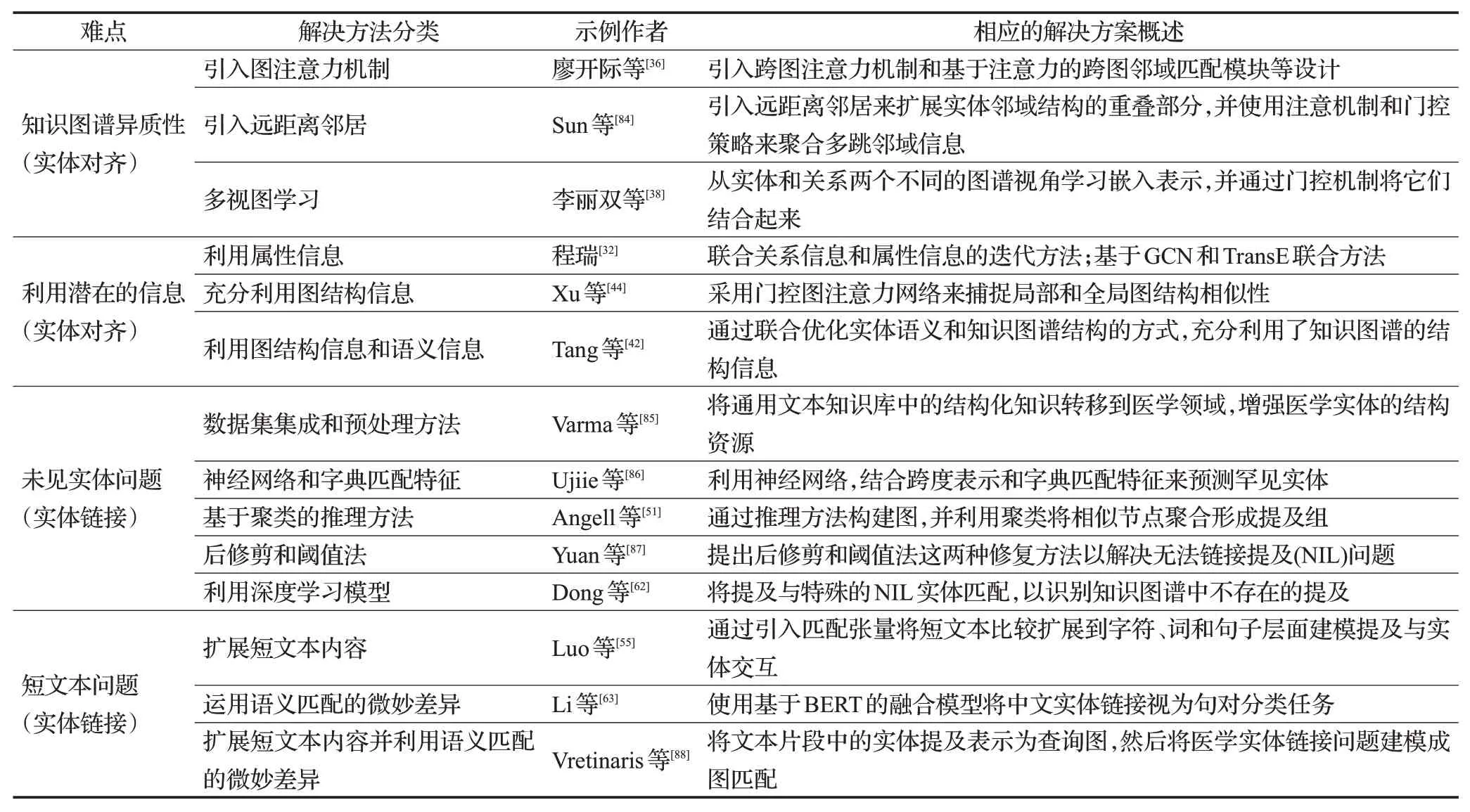
表7 医学领域实体对齐与实体链接各自难点的解决方案Table 7 Challenging solutions for entity alignment and entity linking in medical domain
6 总结与展望
通过对医学领域实体对齐和实体链接研究现状的分析,发现国内医学实体对齐的研究由于领域知识图谱规模的限制和公开语料库的稀缺,其起步相对较晚;相比之下,医学实体链接的研究更为多样与丰富。深度学习的兴起促进了知识融合任务的发展,使得医学领域知识融合中存在的实体命名多样性、利用潜在的信息等问题得到了较好的解决。然而,标注语料的缺乏、模型的计算效率、知识图谱异质性等问题仍需不断研究,以探索更为高效的解决方案。根据本文的研究,未来医学领域的知识融合将涵盖以下几个方面:
(1)多模态知识融合。医学领域涉及临床报告、医学影像和音视频等多种类型的信息,不同模态的信息之间存在互补性。例如,中医临床辨证依赖于四诊合参,通过望闻问切获取舌面象、脉象、体质信息和症状信息等多模态数据,这些数据的融合能更全面地评估患者的疾病状况。目前,多模态知识融合主要包括特征级的早期融合、基于决策的晚期融合、混合融合三种方法。然而,不同模态间的信息贡献存在差异,多数研究却采用固定的权重融合各模态信息。未来可以考虑:如何确定不同模态数据之间的权重以保证数据一致性;如何设计模型提取特征以提高各模态信息的利用率。
(2)多视图实体对齐。由于医学知识图谱中的实体存在各种特性,传统的单一视图对齐方法难以满足精准对齐的需求。多视图对齐可以将知识图谱的不同特性划分为不同的视图,如实体名称视图、属性视图、关系视图等,从而从特定视图中学习到实体嵌入。通过联合优化多个视图的信息,可以提高实体对齐的性能。因此,多视图实体对齐值得进一步研究。
(3)弱监督或无监督方法。为了适应医学领域样本少的特点,已有一些研究人员针对标注数据的缺乏难点进行了探索,但由于特定领域的复杂性,小样本或零样本问题将长期存在,这依旧是研究热点。因此,未来可以期待更多研究专注于弱监督或无监督方法,以降低数据标注成本,并实现医学知识融合过程的自动化和高效化。
(4)中西医融合。中医与西医可以相互补充,将两者的知识进行融合,构建跨领域的医学知识图谱,有利于融会贯通中西医学的优势,为下游应用提供全面的支持。此外,中西医融合也促进中医的现代化和标准化,推动中医知识的共享和传播。然而,由于中西医在思维方式、诊断方法、治疗途径等方面存在差异,这将给知识融合研究带来新的挑战。因此,如何有效对齐中医实体与西医实体成为亟待解决的难题。
(5)大模型赋能知识融合。随着“ChatGPT”“GPT-4”等大模型掀起的新浪潮,使得利用大模型增强医学领域知识融合成为可能,例如,使用大模型作为编码器和解码器来实现知识融合与补全。此外,医学领域也涌现出一些专用大模型,如“华佗GPT”“灵医Bot”以及专注于中医领域的“岐黄问道·大模型”,它们的出现可进一步提升医学领域模型训练的准确性。未来,利用大模型进行医学术语定义补全、实体标准化对齐、同义词的提取与融合等研究将成为医疗领域的重要突破。

