基于U-Net改进的内窥镜息肉图像分割算法
邓晓青 李征 王雁林
息肉图像的分割在临床医疗和计算机辅助诊断技术等领域具有广泛的研究和应用价值,但是就目前的研究和应用需求来看,准确的息肉分割仍然是一项挑战. 针对内窥镜息肉图像中出现的息肉与黏膜边界不清晰、息肉的大小和形状差异较大等影响分割质量的问题,该文提出了一种基于U-Net改进的息肉图像分割算法(SBF-Net). 首先,在U-Net架构上引入了边界特征加强模块(BFEM),考虑到息肉边界和内部区域的关键线索,该模块利用编码器高层特征生成额外的边界补充信息,在解码器阶段进行融合,提升模型处理边界特征的能力. 其次,该模型的解码器(GFBD)采用了从上至下逐步融合特征的方式,将编码器阶段的输出特征经过局部加强(LE)模块之后再逐步融合边界特征,这种多尺度特征融合方式有效缓解了编码器和解码器之间的语义差距问题. 最后,在后处理阶段采用测试时数据增强(TTA)来进一步对分割结果进行细化. 该模型在CVC-300、CVC-ClinicDB、Kvasir-SEG、CVC-ColonDB和ETIS-LaribPolypDB等5个公开数据集上进行了对比实验和消融实验,实验结果证明了该文所改进方法的有效性,并在内窥镜息肉图像上表现出更好的分割性能和更强的稳定性,为息肉图像的处理和分析提供了新的参考.
内窥镜息肉图像; 息肉分割; U-Net; 边界加强
TP391.41 A 2024.013004
An improved endoscopic polyp image segmentation algorithm based on U-Net
DENG Xiao-Qing 1, LI Zheng 1,2 , WANG Yan-Lin 1
(1.College of Computer Science, Sichuan University, Chengdu 610065, China; 2. Tianfu Engineering-Oriented Numerical Simulation & Software Innovation Center, Sichuan University, Chengdu 610207, China )
The segmentation of polyp images has extensive research and application value in the fields of clinical treatment and computer-aided diagnostic technology, but accurate polyp segmentation is still a challenge in terms of current research and application needs. In order to solve the problems that affect the segmentation quality of endoscopic polyp images, such as the unclear boundary between polyps and mucous membranes, and the large difference in the size and shape of polyps, this paper proposed an improved U-Net polyp segmentation algorithm. Firstly, the boundary feature enhancement module was introduced on the U-Net architecture. Considering the key clues of polyp boundary and internal area, this module used the high-level features of the encoder to generate additional boundary supplementary information, which is fused at the decoder stage to improve the ability of the model to process boundary features. Secondly, the decoder of the model adopts the method of gradually fusing features from the top to the bottom. After the output features of the encoder stage are passed through local emphasis module, the boundary features are gradually fused. This multi-scale feature fusion method effectively reduces the semantic gap between the encoder and the decoder. Finally, test-time augmentation was used in the post-processing stage to further refine the segmentation results. The model has been compared and ablated on five public datasets: CVC-300, CVC-ClinicDB, Kvasir-SEG, CVC-ColonDB and ETIS-LibPolypDB. The experimental results prove the effectiveness of the modified method, and it shows better segmentation performance and stronger stability in the endoscopic polyp image, which provides a new reference for the processing and analysis of the polyp image.
Endoscopic polyp image; Polyp segmentation; U-Net; Boundary strengthening
1 引 言
结直肠癌是世界上第三大最常见的癌症类型 [1] ,大多数结直肠癌通常是由于结直肠息肉未及时发现和治疗而转变成为恶性肿瘤所引起的 [2] . 研究表明,在息肉转为恶性肿瘤之前,可以通过结肠镜检查和治疗使结直肠癌的发病率和死亡率得到有效降低. 结直肠息肉检查中,内窥镜检查是最重要也是最常用的一种方式,内窥镜检查是利用结肠镜深入到患者的结直肠内部,通过内窥镜拍摄大量的图像或者实时视频的方式获取病灶的信息,这种方式不仅可以确定结直肠息肉的位置,还能明确息肉的大小、体积和质地等信息,为诊断治疗提供参考,使医生能够在其发展成结直肠癌之前将其切除 [3] ,迄今为止,结肠镜检查仍然是最有效的结直肠癌预防技术. 但是,此过程高度依赖于临床医生的技能和经验,检查效果非常不稳定,这也是息肉检测错误率和漏检率高的原因之一,漏检率甚至高达25% [4] . 用于息肉分割的计算机辅助诊断系统可以通过提高准确性和减少人工干预来减少医生的工作量并改善临床工作流程 [5] ,因此,能够在早期检测到所有可能的息肉并进行自动和准确的分割,这对于预防结直肠癌具有重要意义 [6] .
早期的息肉图像分割方法大多依赖于阈值、形状和边缘检测等传统方法,虽然实现简单,但是精度不高 [7] . 图像分割是医学图像处理的重要研究方向之一,随着深度学习技术的迅速发展,各种深度神经网络模型被广泛应用于医学图像分割领域 [8] ,并取得了显著的成果. Ronneberger等人 [9] 提出的U-Net是一个用于医学图像分割的全卷积网络,因其能够采用多级特征来重建图像的预测结果而引起了医学图像分割领域的广泛关注,该网络使用跳跃连接将编码器的每一层特征都拼接至对应的解码器端,以实现对浅层特征中的空间信息的最大利用. Zhou等人 [10] 提出的UNet++是U-Net的改进版本,与U-Net不同的是,UNet++在编码器和解码器之间使用了密集的跳跃连接,通过这种连接间接地融合了多个层次的特征,这对于医学图像中各种形状和大小不同的病灶分割效果良好. Jha等人 [11] 提出的ResUnet++是在基于U-Net结构的基础上增加了SE(Squeeze and Excitation)模块、ASPP(Atrous Spatial Pyramidal Pooling)模块、残差模块和注意力块来增强模型的特征提取能力,实现更快更准确的息肉分割. Wei等人 [12] 提出了浅层注意力网络SANet,使用颜色交换的方式来消除颜色对分割结果的影响,以及一种概率校正策略来减少负样本的干扰并提高正样本的预测能力. 这些方法专注于分割息肉的整体区域,忽略了边界和内部区域的关系,而这对于准确的分割非常重要. 因此,Fan等人 [13] 提出了PraNet,该网络采用了两阶段分割方法,使用反向注意模块来恢复息肉的边界和内部结构从而实现更加准确的息肉分割. 受此启发,Kim等人 [14] 提出的UACANet整合了前景、背景以及不确定区域的特征来细化边界的线索. 然而这些方法提出的息肉边界和内部结构信息都未在特定的监督下生成,因而隐藏层的特征透明度和辨别力不高. BDG-Net [15] 模型添加了边界分布图来指导解码器分割,但是未考虑来自编码器的浅层特征与解码器的深层特征存在的信息差距问题,并且未充分利用编码器的浅层信息.
基于强化息肉边界的关键线索以及减少特征层之间信息差距的思想,本文提出了一种基于U-Net改进的内窥镜息肉图像分割网络,称为逐步融合边界特征网络(Stepwise Boundary Fusion Net,SBF-Net).其整体上基于U-Net框架,在编码器阶段利用高层特征生成息肉边界补充信息,提升模型对边界处特征的学习能力,辅助解码器恢复出更准确的分割结果. 在解码器阶段,将编码器的输出特征经过局部加强(Local Emphasis, LE) [16] 模块后与当前阶段的上一层输出特征和息肉边界特征进行逐步融合,最后将TTA [17] (Test-Time Augmentation)应用于模型的后处理阶段以实现对分割结果的进一步细化.
2 SBF-Net模型结构
2.1 SBF-Net模型
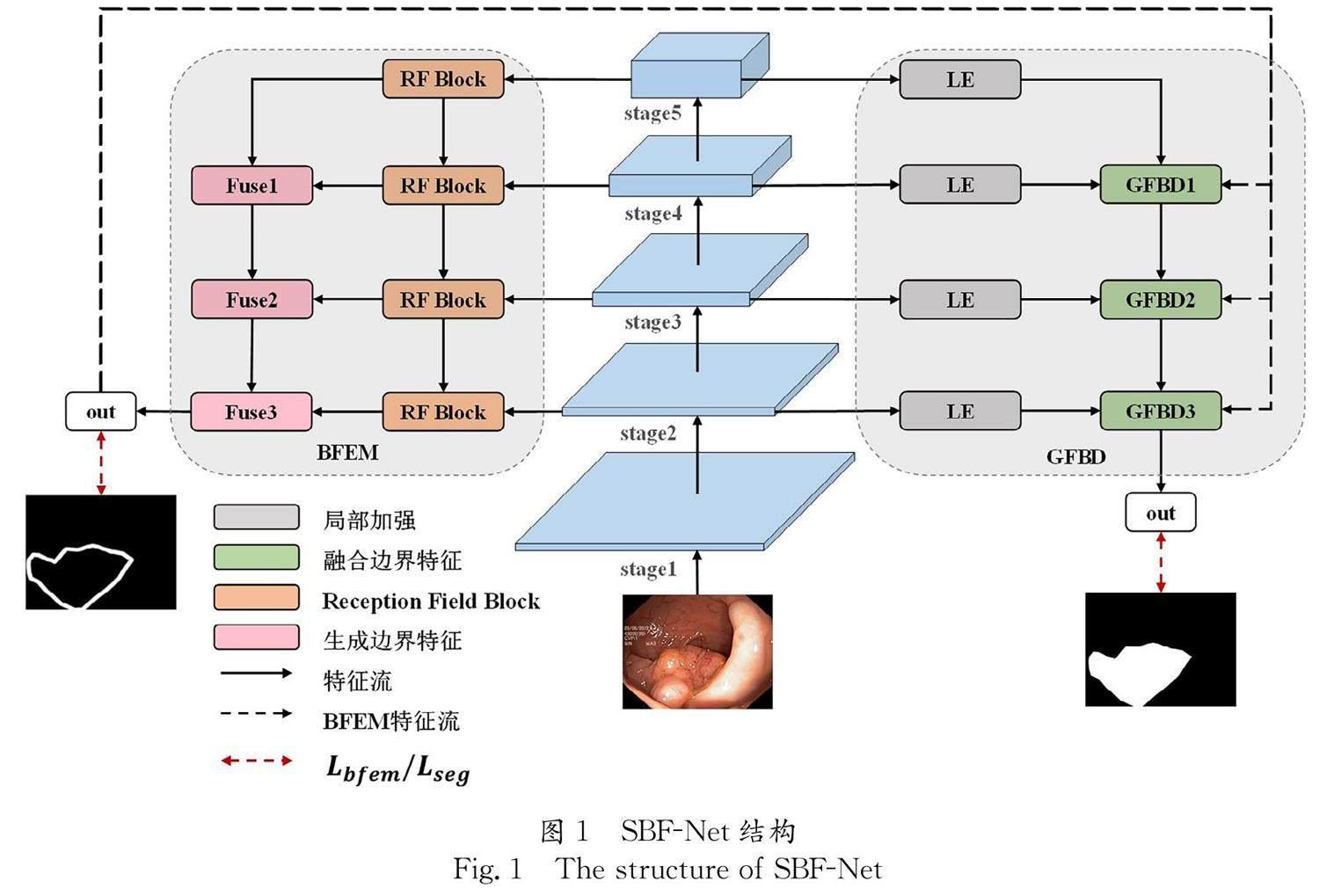
SBF-Net的整体架构如图 1所示,该网络主体上是U型网络结构,中间部分是编码器,右侧是逐步融合边界特征的解码器(Gradually Fused Boundary Feature Decoder, GFBD),跳跃连接部分加入了局部加强(LE)模块,编码器左侧部分是边界特征加强模块(Boundary Feature Enhancement Module, BFEM). 由于内窥镜息肉图像中息肉的颜色与周围黏膜非常相似,因此边界的区分非常不明显 [18] .所以为了让模型更好地学习边界特征,本文参考了BDG-Net [15] 中以高斯分布近似真实的息肉边界分布.但是为了充分利用浅层特征的有效信息,本文将编码器最后四个阶段的输出用于生成额外的边界补充信息.同时,考虑到U型网络中可能产生的语义差距问题,本文的解码器使用局部加强的跳跃连接来融合编码器特征、息肉边界特征和当前阶段的上一层输出特征,使高维特征和低维特征之间融合的信息差距更小,同时还能更好地减少了息肉边界处的信息损失,恢复出高分辨率的预测结果.
2.2 边界特征加强模块
医学图像与普通图像的差异较大,例如在内窥镜息肉图像中,部分病灶边缘模糊不清,边缘与周围黏膜组织的过渡区域附近存在细小的纹理及噪声,在息肉病灶局部区域,息肉与周围邻近像素点间存在较高的颜色相似性和亮度,但整体区域的一致性却各有不同. 目前许多优秀的息肉分割算法在息肉内部区域的分割效果良好,但是息肉边界上往往无法得到很好的处理 [13] .为了更好地让模型学习边界处的特征,本文提出了一种基于残差结构和通道注意力机制的边界特征加强模块,即图1中BFEM部分,为充分利用编码器浅层的细节信息,该模块由编码器的最后4个阶段生成.
BDG-Net中添加了边界分布生成模块来生成息肉边界分布图指导解码器实现更准确的分割,但是该模块在保持模型轻量的情况下只融合了编码器的高三层特征,缺少局部细节信息,恢复出的息肉边界精度有限.本文将编码器四个阶段的输出进行融合输出息肉边界,在提升精度的同时分割的息肉结果拥有更丰富的边缘细节.考虑到浅层特征可能包含更多噪声,BDG-Net中简单的卷积融合并不能很好地抑制息肉黏膜周围的干扰信息,因此本文引入了通道注意力机制来融合多尺度特征,以尽可能抑制边界处的无用噪声,增强有用特征.本文重新设计了边界生成的方式,BFEM的设计旨在加强该模块的学习能力和特征复用能力以生成包含更多细节信息的息肉边缘,总体上是基于ResNet [19] 中提出的残差机制以及通道注意力机制 [20] 的思想. 首先,使用RF Block [21] 对来自编码器的特征进行处理,RF Block是在Inception [22] 的基础上引入了不同膨胀系数的卷积层,可以在减少特征图的通道数的同时,增大模型的感受野,辅助BFEM提取出包含更多全局信息的特征. 其次,为了使得深层网络中包含更多的特征信息,并缓解其可能存在的退化问题,我们添加了如图2所示的残差连接,使模型在原有特征的基础上学习出新的特征,从而提升特征复用能力. 最后,由于内窥镜息肉图像中在息肉病灶以外的区域也可能表现出诸如毛细血管、褶皱等复杂的结构特征,这些信息会对息肉边界的分割造成干扰.因此我们在BFEM融合过程中引入了通道注意力机制的挤压激励(Squeeze and Excite, SE)模块,以达到强化有效特征,抑制边界处无效噪声的目的. BFEM以此机制作为一个学习单元,可以使模型恢复出更加准确的息肉边缘,BFEM的具体融合过程如图 2所示.
首先,我们将编码器最后两个阶段的输出特征进行融合,低层特征表示为 e l ,高层特征表示为 e h , e l 经过RF Block后进行 C 3 BR模块处理得到 e ′ l , C 3 BR 包括3×3卷积层(Convolution)、批次归一化 [23] (Batch Normalization)和Relu [24] 激活函数层, e ′ l 再传入SE模块对不同通道的特征进行加权,在恒等映射和残差相加之前对残差分支进行了“特征重标定”. 然后与经过RF Block后的 e l 进行残差连接得到 e ″ l . 类似地, e h 进行同样的处理得到 e ″ h ,接着将 e ″ h 进行双线性插值上采样后与 e ″ l 相加得到融合了高层和低层特征的 e lh ,接着 e lh 经过 C 3 BR处理,再传入SE模块后与输入特征 e lh 进行残差连接,最后再作上采样处理得到最终的输出 e out . 输出特征 e out 与上一阶段编码器特征继续同样的融合过程,直到融合至编码器的第二个阶段为止. 最终的输出 e out 可以描述为式(1)所示, e lh 表达式如式(2)所示, e ″ l 和 e ″ h 表达式分别如式(3)和式(4)所示, e ′ h 和 e ′ l 的生成表达式分别如式(5)和式(6)所示. 其中, RFB · 表示RF Block, SE(·) 表示挤压激励操作, Up(·) 表示双线性插值上采样.
e out =Up SE C 3BR e lh +e lh (1)
e lh =Up e″ h +e″ l (2)
e″ h=SE e′ h +RFB e h (3)
e″ l=SE e′ l +RFB e l (4)
e′ h=C 3BR RFB e h (5)
e′ l=C 3BR RFB e l (6)
由于内窥镜息肉图像存在的反光、粗边缘等原因,息肉与黏膜界限很难确定.为了更加精确地定位息肉边界附近的像素是否属于息肉,使模型训练出更加真实的边界分布,本文使用高斯分布来近似息肉边界像素的分布,以此作为息肉与黏膜的理想界限. 高斯分布是一种随机分布,用于描述测量值在真值附近的分布,距离真值越远,观测实例就越稀少.所以本文参考了BDG-Net的真实边界分布图的生成方式,假设息肉的边界是遵循均值为0,标准差为 σ 的高斯分布 [15] ,息肉边界分布的真实值可以近似为一元高斯分布,表达式如下式所示.
Gs(b ij )= 1 2π σ e - ε (b ij ) 2 2σ 2 (7)
式中, ε b ij 表示 i,j 像素点到边界的最短欧式距离. 首先将息肉分割标签 mask 先进行预处理转换为前景为1,背景为0的二值化图像,然后分别计算出前景点和背景点到息肉边界像素点的距离,得到距离转换图,并整合前景和背景到边界的距离,我们将式(7)作用于距离转换图像即可估计出边界附近的像素点属于边界的概率映射,最终的息肉边界分布可以表示为如下式所示.
BFEM mask,σ =
λ·G s trans mask +trans 1-mask ,σ (8)
式中, mask 表示息肉分割的标签图像; λ 表示增强因子,用于增强特征响应,本文实验中将 λ 设为 σ 2 ; trans 表示距离转换操作.
2.3 逐步融合边界特征解码器
由于U-Net在医学图像分割领域的优秀性能,现已广泛用于各种病灶分割.虽然U-Net因其能够融合多尺度特征,充分利用图像的空间语义信息,在小型医学数据集上也能表现良好.但是类似于U-Net这样的编码器-解码器结构的一个主要问题就是来自编码器的浅层特征和来自解码器的深层特征存在较大的语义差距.这种来自编码器和解码器之间的语义鸿沟,限制了分割性能,甚至有一些跳跃连接会对分割产生负面效应.
基于减少特征层语义差距的思想,本文提出的解码器采用了U型网络中从上至下的方式逐步恢复高分辨率的息肉分割结果,在逐步融合边界特征的过程中尽可能使特征融合平滑进行,如图 1中GFBD部分. 首先,我们通过在编码器输出的特征上引入了局部加强的非线性变换来加强跳跃连接,以缩小编码器和解码器之间的语义差距,利用卷积的固定感受野来混合每个像素点相邻的图像块,从而增加相邻图像块到图像中心的相关权重,以强调每个图像块关键的局部信息. 其次,整个解码器端采用U-Net中从上到下的方式逐步融合特征金字塔中不同层次的特征,避免直接聚合具有显著深度差异的特征影响分割性能. 最后,将BFEM生成的息肉边界特征在解码器端辅助GFBD生成更加准确的分割结果. 这种特征融合方式旨在保持轻量和高效,并且可以使融合的高维和低维特征之间的信息差距更小.
以图 1中GFBD2为例,解码器融合息肉边界特征过程如图 3所示. GFBD2融合过程的输入包括上一阶段解码器的输出 d i-1 、BFEM输出的息肉边界 b 和经过LE的编码器输出特征 e′ i ,输出是 d i . GFBD1和GFBD3的结构与此类似,由于GFBD1为解码器端首个融合单元,该部分输入包括 e′ i-1 、 e′ i 和 b ,在GFBD3中,在输出 d i 后还经过了一个由卷积层和两倍双线性插值上采样组成的分割头来预测最终的分割结果.
LE模块是两个3×3卷积和Relu激活函数,再加上一个双线性插值上采样组成,编码器的输出 e i 经过LE后得到 e′ i . 接着 e′ i 再与 d i-1 和 b 进行连接,最后再经过 C 1 BR 块得到输出特征 d i , C 1 BR包括1×1卷积、批次归一化和Relu激活函数. 解码器融合各个特征的操作输出 d i . d i 作为GFBD3的输入继续下一阶段融合. d i 表达式可以描述为式(9)所示, LE · 表示局部加强,表达式如式(10)所示, Down · 表示下采样,本文中采用的是双线性插值法, Conv · 表示3×3卷积, Concat(·) 表示连接操作.
d i=C 1BR Concat LE e i ,d i-1 , Down b (9)
LE(e i)=Up( Relu(Conv (Relu( Conv (e i))))) (10)
2.4 测试时数据增强
测试时数据增强(TTA)广泛应用于各类视觉任务上提升精度,但同时会带来测试时间的增加,在非实时的视觉任务中应用更多. TTA是一种通过对测试数据集进行裁剪、翻转、旋转等增强手段 [17] ,对每个测试图像进行增强,并对增强后的图像进行预测,将预测结果的均值作为最终的输出预测. 在本文中,我们在测试时应用到了水平翻转和旋转的增强方法.
2.5 损失函数
本文的损失函数由边界特征加强模块的损失 L bfem 与分割损失 L seg 的和来作为最终的损失 L . L bfem 采用BDG-Net的辅助损失 [15] 用于计算边界特征加强模块的中间结果与真实值之间的损失,如式(11)所示, f b ij 用于忽略损失较低的部分,表达式如式(12)所示.
L bfem = ∑ (i,j) b ij -b ^ ij 2·f b ij (11)
f b ij =δ b ij -b ^ ij 2<κ (12)
其中, b ij 表示息肉边界的预测值; b ^ ij 表示理想的真实值. k 表示忽略损失区间的阈值, k 的初始值设置为0002,随着训练次数的增加,损失越来越低, k 的值也会逐渐降低. 当预测点与真实值之间的欧式距离的平方小于 k 时, δ · 的值为0,即忽略掉这部分损失,当大于 k 时, δ · 的值为1.最终的损失函数 L 如式(13)所示. L seg 用于计算最终分割结果和真实值之间的损失,由加权二元交叉熵(Binary Cross Entropy,BCE)和加权交并比(Intersection over Union,IoU)损失组成,如式(14)所示.
L=L bfem +L seg (13)
L seg = L wbce + L wiou (14)
3 实验结果和分析
3.1 实验环境
本文提出的模型基于PyTorch 1.12.1实现. 在实验中,使用了Intel(R) Core(TM) i7-4790K CPU @ 4.00 GHz 的处理器,并在两张NVIDIA GeForce GTX 1080 Ti(11 GB)设备上进行训练,使用Adam(Adaptive Moment Estimation)优化器 [25] 来调整模型参数,初始学习率为10 -4 ,随着训练次数的增加逐渐降低学习率.
3.2 数据集
为了更加全面地评估模型的性能,本文在CVC-300 [26] 、CVC-ClinicDB [27] 、Kvasir-SEG [28] 、CVC-ColonDB [29] 和ETIS-LaribPolypDB [30] 等5个公开数据集进行了对比实验. CVC-300数据集是EndoScene [26] 数据集的一部分,我们选取了60张图像作为测试集,EndoScene包含了来自36例患者的44个结肠镜检查序列的912张图像,分辨率为574×500. CVC-ClinicDB包含了来自31个结肠镜检查序列的612张图像,分辨率为384×288. Kvasir-SEG是胃肠息肉图像及其相应标签的数据集,由医生手动标注,然后由经验丰富的胃肠病学家验证,图像的分辨率各不相同. CVC-ColonDB数据集中包含了来自15个结肠镜检查序列的380张图像,图像大小为574×500. ETIS-LaribPolypDB数据集包含了从34个结肠镜视频中采集的一共196张图像,图像大小为1225×966,是几个数据集中分辨率最高的,并且由于ETIS-LaribPolypDB是早期的息肉图像,所以该数据集中息肉与黏膜极其相似,并且形状和大小多样化,这使得该数据集更具有挑战性.
我们参考了PraNet [13] 的数据集分割方案,为了使实验结果更加客观,这与现在许多息肉分割模型所采用的数据集保持一致,在CVC-ClinicDB选取了550张图像,在Kvasir-SEG数据集中选取了900张图像,一共1450张图像作为训练集,CVC-ClinicDB剩余的62张图像和Kvasir-SEG剩余的100张图像作为测试集. 同时,为了验证模型的泛化能力,我们还在CVC-300、CVC-ColonDB和ETIS-LaribPolypDB这三个模型未见过的数据集上也进行了测试.
3.3 评估指标
实验中使用了医学图像分割领域中使用频率最高的度量指标Dice系数(Dice Similarity Coefficient, DSC)以及语义分割中的系统性能评价指标平均交并比MIoU(Mean Intersection over Union). Dice系数常用于医学图像分割领域中计算两个样本的相似度,阈值为[0,1],分割的结果最好时为1,最差时为0,计算公式如式(15)所示. MIoU是用于计算真实值和预测值两个集合的交集与并集之比,如式(16)所示. 其中, A 表示模型的预测结果集; B 表示原始息肉图像的真实标签结果集; TP 表示被模型预测为正类的正样本数; FN 表示被模型预测为负类的正样本数; FP 表示被模型预测为正类的负样本数.
Dice(A,B)= 2× A∩B A + B = 2×TP FP+FN+2×TP (15)
MIoU(A,B)= A∩B A ∪B = TP TP+FP+FN (16)
3.4 与现有方法的对比实验
为了验证本文提出的SBF-Net的性能,本文将SBF-Net在息肉图像上的分割结果与U-Net [9] ,UNet++ [10] ,PraNet [13] ,SANet [12] ,UACANet-S [14] 和BDG-Net [15] 模型分别在五个公开数据集上进行对比. 算法对比结果如表 1所示.
我们提出的经过SBF-Net模型经过TTA后在CVC-ClinicDB和Kvasir- SEG数据集上均取得了最优的Dice和MIoU. 在CVC-ClinicDB数据集中,SBF-Net在Dice和MIoU上分别达到了9232%和88.53%,经过TTA处理后(SBF-Net+TTA)分别达到了92.60%和88.81%,均优于对比方法. 在Kvasir-SEG数据集上,SBF-Net在MIoU上达到了86.79%,优于所有对比方法,Dice系数达到了91.04%,BDG-Net的Dice仅高于SBF-Net约0.004 5,达到几乎可以忽略的优势,并且SBF-Net经过TTA处理后Dice和MIoU分别达到了91.60%和87.92%,高于BDG-Net和其他对比方法.
为了评估SBF-Net的稳定性和泛化能力,我们将SBF-Net和SBF-Net+TTA在CVC-300、CVC-ClonDB和ETIS-LaribPolypDB三个模型未见过的数据集上进行了测试,算法对比结果如表2所示.
由表2可知,我们提出的模型在CVC-300、CVC-ColonDB和ETIS-LaribPolypDB上也表现出优秀的性能,Dice和MIoU均高于其他方法. 其中,ETIS-LaribPolypDB数据集为早期息肉图像,大部分息肉还未出现明显的凸起,因此表现为小型的扁平状,我们的模型分割结果仍然取得了显著提高. 该测试结果可以反映出我们提出的方法具有更加优秀的泛化能力和准确的预测能力.
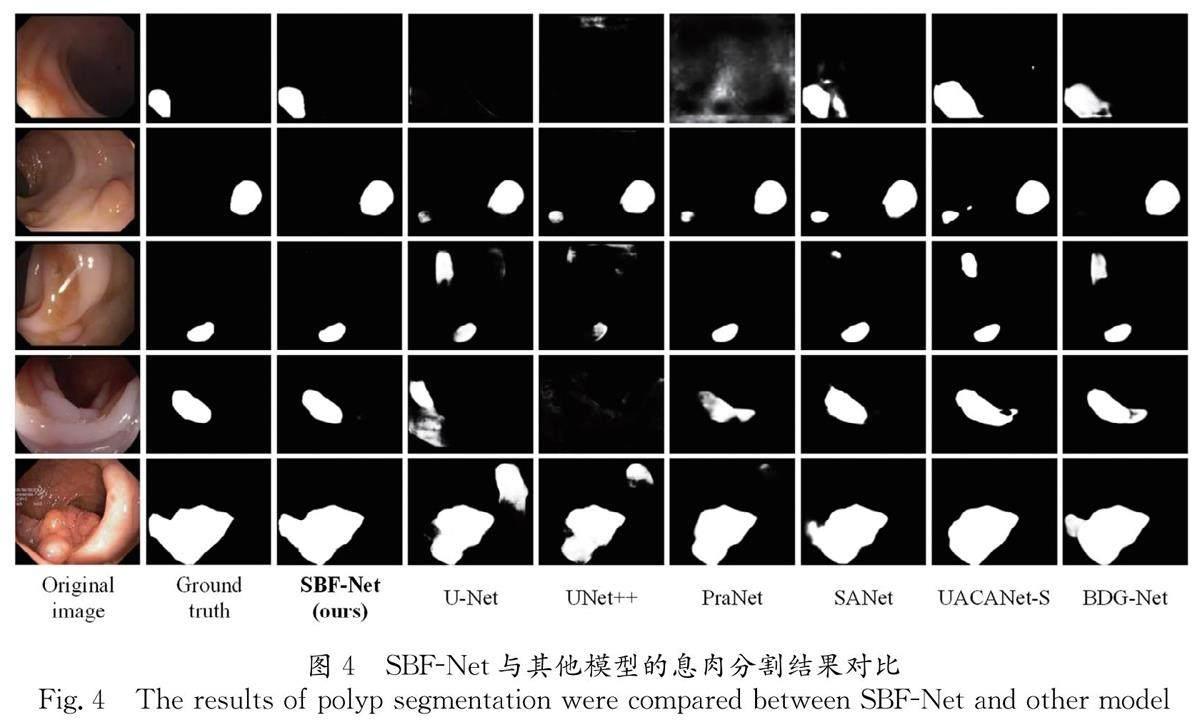
图4展示了SBF-Net与其他分割模型在部分息肉图像上的分割结果对比. 图4每一列分别是原息肉图像、真实标签图、SBF-Net、U-Net、UNet++、PraNet、SANet、UACANet-S和BDG-Net网络的分割结果图.
从图4可以看出,SBF-Net的分割结果相比于其他方法更接近于真实标签. 如图4中第1行所示,息肉由于过度曝光导致U-Net、UNet++和PraNet模型根本无法识别,剩余的几个方法的分割结果也与真实标签出现较大差异,SBF-Net通过加强局部特征并融合多尺度特征的解码器能够有效捕获更多深层和浅层信息,对过度曝光区域的处理更好. 如图4第2行和第3行所示,由于周围黏膜与息肉颜色极其相似,许多对比方法都将黏膜误识别为息肉,我们的模型可以在关注关键局部信息的同时,还抑制了其他无用信息,并且在息肉外部无伪影出现. 如图4第4和第5行所示,SBF-Net都比其他算法表现出更好的边界处理效果,在其他对比方法中均出现了过度分割或分割不足的情况,我们的模型通过边界加强模块让模型对边界处特征有更好的学习能力,有效地对息肉边缘进行了更准确的分割.
3.5 消融实验
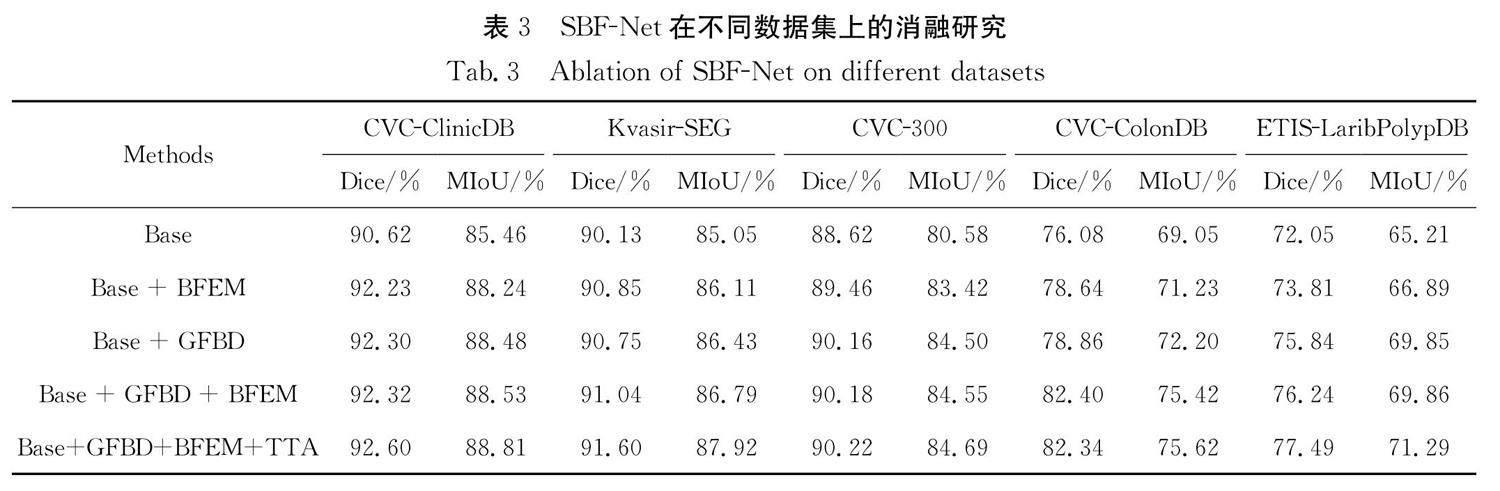
为了验证边界特征加强模块、逐步融合边界特征解码器和TTA的有效性,我们设计了从Base模型逐渐过渡到SBF-Net模型的消融实验来评价各个模块对实验结果的影响,并分别在五个数据集上进行了对比,消融实验结果如表 3所示. 本文主要做了5组实验,第1组是使用EfficientNet-B5编码器 [31] 加上一个语义分割头SegmentationHead作为Base模型. 第2组是在Base的基础上加上U-Net的解码器,并加入边界特征加强模块(BFEM). 第3组是在Base的基础上加入了逐步融合边界特征解码器(GFBD). 第4组则是为了研究边界特征加强模块与逐步融合边界特征解码器的组合是否有效,即本文提出的模型SBF-Net. 第5组为加上TTA作为后处理的SBF-Net. 通过对比可以发现,本文提出的边界特征加强模块和逐步融合边界特征解码器对模型有着积极的影响,从第一组到第4组实验相对于前一组实验均有不同程度的提升. 第4组(SBF-Net)相比于第1组(Base) 在CVC-ColonDB和ETIS-LaribPolypDB数据集上的Dice和MIoU有4.19%~6.37%的明显上升,在其他数据集上也有显著上升. 而经过TTA处理后的SBF-Net模型也在大部分数据集上都有精度上的提升. 实验结果表明,本文方法生成的息肉分割图像在分割质量和泛化能力上都具有优秀的表现.
4 结 论
针对内窥镜息肉图像分割中存在的边界分割过度或不足,以及在不同图像上息肉形状差异较大导致分割效果差等问题,本文提出了一种基于U-Net改进的SBF-Net模型. 该模型通过引入边界特征加强模块来提升模型对边界特征的学习能力,在编码器的跳跃连接中间加入一个特征加强模块用来加强关键的局部特征和浅层特征,在解码器阶段,通过逐步融合编码器特征、上一阶段的输出特征和息肉边界特征,指导生成更加准确的分割,并减少高维和低维特征之间的信息差距,改善了U-Net中直接并行聚合深度差异较大的不同阶段特征而产生的信息差距问题,最后再使用TTA来进一步提高模型在某些数据集上的整体预测性能. 在5个公开数据集上的实验结果表明,SBF-Net的Dice和MIoU在5个数据集上都在不同程度上优于其他方法,较为客观地说明本文方法在分割性能和泛化能力上更有优势. 但是我们在测试时发现经过TTA处理的SBF-Net在推理效率上有显著降低,因此我们未来将进一步对本文方法进行优化,以及考虑使用数据预处理等手段来改善因内窥镜图像高光对分割造成的不良影响等.
参考文献:
[1] Bray F, Ferlay J, Soerjomataram I, et al . Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries [J]. CA-Cancer J Clin, 2018, 68: 394.
[2] Gschwantler M, Kriwanek S, Langner E, et al . High-grade dysplasia and invasive carcinoma in colorectal adenomas: a multivariate analysis of the impact of adenoma and patient characteristics [J]. EUr J Gastroen Hepat, 2002, 14: 183.
[3] Haggar F A, Boushey R P. Colorectal cancer epidemiology: incidence, mortality, survival, and risk factors [J]. Clin Colon Rect Surg, 2009, 22: 191.
[4] Leufkens A M, Van Oijen M G H, Vleggaar F P, et al . Factors influencing the miss rate of polyps in a back-to-back colonoscopy study[J]. Endoscopy, 2012, 44: 470.
[5] Rahim T, Usman M A, Shin S Y. A survey on contemporary computer-aided tumor, polyp, and ulcer detection methods in wireless capsule endoscopy imaging [J]. Comput Med Imag Grap, 2020, 85: 101767.
[6] Jia X, Xing X, Yuan Y, et al . Wireless capsule endoscopy: a new tool for cancer screening in the colon with deep-learning-based polyp recognition [J]. P IEEE, 2019, 108: 178.
[7] Wang Y G, Xi Y Y, Pan X Y. Method for intestinal polyp segmentation by improving DeepLabv3+ network [J]. FCST, 2020, 14: 1243.[王亚刚, 郗怡媛, 潘晓英. 改进 DeepLabv3+ 网络的肠道息肉分割方法[J]. 计算机科学与探索, 2020, 14: 1243.]
[8] Li D, Wang Y, Ma Z Q, et al . Ultrasound image segmentation based on Dense ASPP model [J]. J Sichuan Univ(Nat Sci Ed), 2020, 57: 741.[李頔, 王艳, 马宗庆, 等. 基于DenseASPP模型的超声图像分割[J]. 四川大学学报(自然科学版),2020, 57: 741.]
[9] Ronneberger O, Fischer P, Brox T. U-net: Convolutional networks for biomedical image segmentation[C]//International Conference on Medical Image Computing and Computer-assisted Intervention. Cham: Springer, 2015.
[10] Zhou Z, Rahman S M M, Tajbakhsh N, et al . Unet++: A nested u-net architecture for medical image segmentation [M]//Deep learning in medical image analysis and multimodal learning for clinical decision support. Berlin: Springer, 2018: 3.
[11] Jha D, Smedsrud P H, Riegler M A, et al . Resunet++: an advanced architecture for medical image segmentation [C]//2019 IEEE International Symposium on Multimedia (ISM). [S. l.]: IEEE, 2019.
[12] Wei J, Hu Y, Zhang R, et al . Shallow attention network for polyp segmentation[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer, 2021.
[13] Fan D P, Ji G P, Zhou T, et al . Pranet: parallel reverse attention network for polyp segmentation[C]//International Conference on Medical Image Computing and Computer-assisted Intervention. Cham: Springer, 2020.
[14] Kim T, Lee H, Kim D.Uacanet: uncertainty augmented context attention for polyp segmentation[C]//Proceedings of the 29th ACM International Conference on Multimedia. New York: ACM Press, 2021.
[15] Qiu Z, Wang Z, Zhang M, et al . BDG-Net: boundary distribution guided network for accurate polyp segmentation[C]//Medical Imaging 2022: Image Processing. [S. l.]: SPIE, 2022.
[16] Wang J, Huang Q, Tang F, et al . Stepwise feature fusion: local guides global[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer Nature Switzerland, 2022.
[17] Jha D, Smedsrud P H, Johansen D, et al . A comprehensive study on colorectal polyp segmentation with ResUNet++, conditional random field and test-time augmentation [J]. IEEE J Biomed Health, 2021, 25: 2029.
[18] DongB, Wang W, Fan D P, et al . Polyp-pvt: polyp segmentation with pyramid vision transformers [EB/OL]. [2022-11-20]. https://arxiv.org/abs/2108.06932.
[19] He K, Zhang X, Ren S, et al . Deep residual learning for image recognition[C]//Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. [S. l.]: IEEE, 2016.
[20] Hu J, Shen L, Sun G. Squeeze-and-excitation networks [C]//Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition.Piscataway: IEEE, 2018.
[21] Liu S, Huang D. Receptive field block net for accurate and fast object detection[C]//Proceedings of the European Conference on Computer Vision (ECCV). Cham: Springer, 2018.
[22] Szegedy C, Liu W, Jia Y, et al . Going deeper with convolutions [C]//Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. [S. l.]: IEEE, 2015.
[23] Ioffe S, Szegedy C. Batch normalization: accelerating deep network training by reducing internal covariate shift [C]//International Conference on Machine Learning. New York: PMLR, 2015.
[24] Glorot X, Bordes A, Bengio Y. Deep sparse rectifier neural networks [C]//Proceedings of the Fourteenth International Conference on Artificial Intelligence and Statistics. Fort Lauderdale, USA: JMLR, 2011.
[25] Kingma D P, Ba J. Adam: a method forstochastic optimization [EB/OL]. [2022-11-24]. https://arxiv.org/abs/1412.6980v6.
[26] Vázquez D, Bernal J, Sánchez F J, et al . A benchmark for endoluminal scene segmentation of colonoscopy images[J]. J Healthc Eng, 2017, 2017: 4037190.
[27] Bernal J, Sánchez F J, Fernández-Esparrach G, et al . WM-DOVA maps for accurate polyp highlighting in colonoscopy: validation vs. saliency maps from physicians[J]. Comput Med Imag Grap, 2015, 43: 99.
[28] Jha D, Smedsrud P H, Riegler M A, et al . Kvasir-seg: a segmented polyp dataset[C]//International Conference on Multimedia Modeling. Cham: Springer, 2020.
[29] Tajbakhsh N,Gurudu S R, Liang J. Automated polyp detection in colonoscopy videos using shape and context information [J]. IEEE T Med Imaging, 2015, 35: 630.
[30] Silva J,Histace A, Romain O, et al . Toward embedded detection of polyps in wce images for early diagnosis of colorectal cancer [J]. Int J Comput Ass Rad, 2014, 9: 283.
[31] Tan M, Le Q.Efficientnet: rethinking model scaling for convolutional neural networks [C]//International Conference on Machine Learning. New York: PMLR, 2019.
收稿日期: 2023-01-05
基金项目: 国家重点研发计划项目(2020YFA0714003); 国家重大项目(GJXM92579); 四川省科技厅重点研发项目(2021YFQ0059)
作者简介: 邓晓青(1999-), 女, 四川资阳人, 硕士研究生, 研究方向为计算机视觉. E-mail: dengxq0123@163.com
通讯作者: 李征. E-mail: lizheng@scu.edu.cn

