水稻OsGRF6 基因的克隆及调控初生根发育功能初步分析
余青青 ,单玉爽 ,尚汇鑫 ,曾 川 ,张泽勇*
(1 厅市共建甘薯及特色豆科作物种质创新与利用四川省重点实验室,四川南充 637000;2 细胞活动与逆境适应教育部重点实验室,兰州 730000;3 重庆三峡农业科学院,重庆万州 404001)
水稻是全球重要的主粮作物之一,根系通过对植物营养和水分吸收以及激素合成直接影响植物的生长和产量,因此,改良作物根系是提高作物产量的重要手段[1]。水稻根系主要由主根,侧根和不定根组成,与拟南芥相比有很大的差异。此外,水稻根发育的调控机理尚不清楚,有待进一步研究。
生长素、细胞分裂素和乙烯等植物激素在根发育过程起着重要的调节作用[2-5]。生长素参与调控水稻根的发育,生长素合成缺失的突变体表现出水稻根发育缺陷的表型。如COW1隶属于水稻YUCCA基因家族,调节依赖于色氨酸的IAA 生物合成,功能缺失oscow1-1突变体表现出不定根变少的表型[6]。此外,IAA 酰胺合成酶通过催化氨基酸与IAA 结合调节IAA 平衡,过表达OsGH3.2和Os-GH3.8的转基因植株表现出IAA 缺失的表型,转基因植株表现不定根和根毛变少的表型[7-9]。功能获得性突变体osgh3.13表现出侧根和不定根减少的表型[10]。而过表达生长素运输相关基因Os-AUX1的转基因植株则表现促进侧根和根毛发育的表型[11]。OsPIN1和OsPIN2编码生长素极性运输蛋白,通过调节生长素的分布参与调节水稻根伸长和侧根发育[12-13],进一步研究表明,OsPIN1和OsPIN2的表达分别受OsSPL14和OsSPL3转录因子调控[14-16]。生长素响应因子OsARF12和Os-ARF25的缺失会导致水稻主根变短[17]。进一步研究表明,生长素参与调控不定根发育是通过LOBdomain 转录因子CROWN ROOTLESS1(CRL1)来实现的,与野生型相比,crl1突变体表现不定根数目明显减少的表型[18]。
细胞分裂素与生长素相互拮抗共同调控水稻根的发育[19]。OsIPT1编码异戊二烯转移酶,调控细胞分裂素的合成,过表达OsIPT1基因抑制水稻根的发育[20],而敲除编码细胞分裂素激酶基因LONELY GUY(LOG)的突变体植株表现出根变短,且发育延迟的表型[21]。相反,过表达编码细胞分裂素氧化酶的基因OsCKX4的转基因植株则表现出促进根生长且不定根数目增多的表型[22]。除了细胞分裂素合成途径参与调控水稻根发育之外,细胞分裂素信号途径相关的基因也参与调控水稻根的发育,如OsRR4、OsRR5和OsRR2,过表达Os-RR4和OsRR5可以促进水稻根的伸长和侧根的发育[23],而OsRR2则参与调控水稻不定根的发育,过表达OsRR2的转基因植株表现不定根数目增加的表型[24]。生长素和细胞分裂素拮抗调节水稻根发育的分子机理较为复杂。生长素途径和细胞分裂素途径调控根发育的途径存在许多结点基因,如Os-CRL5的表达能被生长素诱导,而OsCRL5调节水稻不定根的发育又是通过调节OsRR1基因的表达来实现[25],细胞分裂素途径的基因OsCK X4能通过调节下游的生长素响应基因ARF25和细胞分裂素响应基因OsRR2和OsRR3来调控水稻不定根的发育[17]。WUSCHEL相关的基因也在生长素和细胞分裂素调节水稻根发育过程中起着重要的枢纽作用:一方面OsWOX11能通过调节OsCKX4的表达参与水稻不定根的发育[26];另一方面OsWOX11还能通过抑制细胞分裂素OsRR2的表达,调控水稻不定根中细胞的增殖[27]。
植物生长调节因子(growth regulating factor,GRF)是植物体内非常重要的转录因子,参与调节植物生长发育和对非生物胁迫的响应。At GRF1参与调控细胞大小和细胞数目进而影响叶片和种子大小[28]。OsGRF6调节水稻花,叶夹角,种子和株高的发育[29-30],OsGRF1调节水稻抽穗时间,OsGRF4参与调节水稻种子大小的发育,而OsGRF7则通过调节赤霉素,吲哚三乙酸代谢和独脚金内酯进而影响水稻的株型和分蘖[31-32]。GRF 类转录因子调节植物对非生物胁迫的应答方面,At GRF7能直接与DREB2A启动子的S区结合,抑制DREB2A的表达,降低植物对盐耐受[33]。过表达水稻GRF 上游的调节基因OsmiR396c降低了水稻对盐碱的耐受性[34],而降低OsGRF4的表达则能提高水稻对冷的耐受性[35]。此外,osgrf6功能缺失突变体也表现出对冷耐受的表型[36]。
植物小RNA396家族参与调控植物根的发育,作为小RNA396家族的靶基因,GRF家族调控水稻根发育方面的研究鲜见报道,因此,研究以OsGRF6为对象,通过对OsGRF6的亚细胞定位、转录活性、OsGRF6转基因材料和突变体材料的表型和候选靶基因分析,为深入研究OsGRF6参与调控水稻根发育提供新的线索。
1 材料和方法
1.1 试验材料
水稻材料:试验所用材料为粳稻品种中花11,并以中花11为受体材料,构建了OsGRF6的过表达株系和基因敲除突变体。菌种:大肠杆菌感受态(DH5α)、农杆菌感受态(EH105,GV3101)、酵母菌(Y2 H,Golden)。
载体:基因克隆载体(p EASY-Blunt simple Cloning Vector,TransGen)、过表达载体(pCAMBIA1300,Cambia)、亚细胞定位载体(p BI221-GFP,Cambia)、转录激活活性载体[GAL4-BD,GAL4(4X)-D1-3(4X)∷GUS,中科院植物研究所种康院士馈赠]、基因敲除载体(百格生物)。
试验仪器:PCR 仪、荧光定量PCR(Bio-Rad CFX96 Real-Time PCR Detection System,Bio-Rad)。
1.2 总RNA提取及cDNA合成
将水稻‘ZH11’置于30℃萌发3 d后,播种于温室中,3 周后取材,准备抽取RNA。参照TRNzol-A+reagent说明书提取RNA。参照天根公司的反转录试剂盒(EasyScript one-step gDNA Removal and cDNA Synthesis Super Mix)说明书进行cDNA 合成,cDNA 可以立即使用或存放于-20℃备用。
1.3 OsGRF6 基因的扩增及转基因材料获得
在MSU-RGAP 数据库中对OsGRF6(LOC_Os03g51970)进 行BLAST 搜 索,获 得OsGRF6对应的全长cDNA 序列,根据序列信息对该基因的ORF序列设计引物:OsGRF6 F:5′-ATGCAGGGTGCAATG-3′和OsGRF6 R:5′-GGGGTACCCACCAGGCGGATG-3′,以ZH11的cDNA 为模板克隆该基因。克隆体系(25μL):dd H2O 17μL,10×PCR buffer 3μL,d NTP 2μL,引物F/R 1μL,cDNA 1μL,PCR 酶1μL 进行,反应程序:94℃5 min,94℃10 s,55℃1 min,68℃90 s,35个循环,68℃5 min,4℃保存。PCR 反应结束后,将扩增产物于1%的琼脂糖凝胶上电泳,回收,获得目的基因片段克隆到p EASY-Blunt simple cloning vector载体上,转化大肠杆菌,挑取单菌落,送测序,测序正确后,保存质粒供后续载体构建使用。根据p CAMBIA1300过表达载体上的信息,设计含有BamHⅠ和KpnⅠ引物OsGRF6 F:5′-GGATCCATGCAGGGTGCAATG-3′和OsGRF6 R:5′-GGTACCGGGGTACCCACCAGGCGGATG-3′,以含OsGRF6基因的载体p EASY-Blunt simple为模板,按上述方法进行PCR 扩增、菌检和测序。将正确的OsGRF6基因序列通过重组连接到超表达载体p CAMBIA1300上,提取质粒,转化农杆菌EHA105,通过农杆菌EHA105转化‘ZH11’(由百格生物转化),获得转基因植株。OsGRF6基因敲除突变体购于百格生物公司。
1.4 OsGRF6组织表达模式分析
将水稻‘ZH11’种子用15%次氯酸钠消毒15 min,用蒸馏水清洗5~6 次后,30℃萌发2~3 d后,种于液体培养盆中,2周后,取水稻苗不同部位材料按1.2节所述方法,提取RNA,反转录cDNA冷藏备用。其他部位水稻植株组织取于大田种植的水稻,提取RNA,获得cDNA 冷藏备用。以水稻中ubiquitin基因为内参基因,根据OsGRF6编码区的序列设计定量PCR 引物OsGRF6-qPCR F:5′-CGGACGGACGGCAAG-3′和OsGRF6-qPCR R:5′-GGTTCATGTGTCGCTCACAG-3′,Ubi-qPCR F:5′-AACCAGCTGAGGCCCAAGA-3′和Ubi-qPCR R:5′-AACCAGTCCATGAACCCGG-3′。反应总体系为15μL:Top Green qPCR Super Mix(2×)8 μL、Prime F/R(2μmol/L)1μL、cDNA 1μL、dd H2O 5μL。反应条件为:94℃30 s,94℃5 s,56℃15 s,72℃10 s,循环40次。利用计算Os-GRF6基因的相对表达量,最后用Origin 7.0作图。
1.5 OsGRF6的转录激活活性分析
根据转录激活活性载体信息设计含有SmaⅠ和SalⅠ的上下游引物:OsGRF6-BD F:5′-TCCCCCGGGGATGCAGGGTGCAATGGCCA-3′ 和OsGRF6-BD R:5′-GCGTCGACCACCAGGCGGATGC-3′,以OsGRF6的质粒为模板,参照1.2节所述,进行PCR 扩增,回收目的片段。将目的片段构建到GAL4-BD 上,提取质粒与报告基因质粒[GAL4(4X)-D1-3(4X)∷GUS],共转拟南芥原生质体,弱光培养24 h后,测定GUS活性。GUS活性测定:取5μL 裂解产物加到1.5 m L Eppendorf管中,接着加入45μL MUG 底物混匀,37℃恒温箱中孵育60 min。结束后,加入950μL,0.2 mol/L Na2CO3溶液停止反应,用PromegaGlo Max发光检测仪测定MUG 荧光[37]。
1.6 OsGRF6的亚细胞定位
以获得含有OsGRF6的质粒为模板,根据pBI221-GFP的载体预测信息设计上下游含有XbaⅠ和KpnⅠ的引物OsGRF6-GFP F:5′-TCTAGAATGCAGGGTGCAATGGCCA-3′;GRF6R:5′-GGTACCCACCAGGCGGATGCTCGGAT-3′。参照1.2节,进行PCR 扩增。回收目的基因片段,将目的基因片断克隆到植物表达载体p BI221GFP中,然后将反应液转化大肠杆菌DH5α感受态细胞,转化完成的大肠杆菌液涂布在含有氨苄霉素的平板上生长,接着挑取单菌落,进行PCR 检测和测序验证,最后提取经测序验证正确的OsGRF6-GFP 质粒。将OsGRF6-GFP融合载体转化水稻原生质体中,弱光下培养24 h,用莱卡激光共聚焦显微镜观察荧光信号。
1.7 酵母单杂交验证OsGRF6与CGGCA motif结合
将CAGCA motif构建到p Ab Ai载体上,以AD 空+p Ab Ai-motif 和AD-OsGRF6+p Ab Aimotif分别转入酵母细胞,于加180 ng/m L Ab A 和不加Ab A 的双缺(-Ura,-Leu)培养基上,生长2~3 d后,观察菌斑生长情况,并拍照。
2 结果与分析
2.1 OsGRF6 基因克隆、氨基酸序列对比及进化树分析
以“ZH11”为模板,对OsGRF6的CDS 进行PCR 扩增,获得大小约1 300 bp 的条带,构建到p EASY-Blunt simple cloning vector载体上进行测序分析,测序结果显示,OsGRF6的CDS全长1 371 bp,编码456个氨基酸(图1)。

A.PCR 扩增OsGRF6;M.DL2000 DNA marker;1.OsGRF6 基因;B.OsGRF6蛋白序列。图1 OsGRF6 基因的克隆及蛋白序列A.Cloning O sGRF6 gene by PCR.M,DL2000 DNA marker.1,OsGRF6 gene.B.Protein sequence of OsGRF6.Fig.1 Cloning of OsGRF6 gene and its protein sequence
通过CDD 网站,对OsGRF6 蛋白进行保守功能结构域预测。结果表明,OsGRF6蛋白的第11~45位氨基酸,存在1个典型的GRF转录因子QLQ保守结构域,在第77~118位氨基酸存在1个WRC结构域,符合GRF家族的特征,表明OsGRF6蛋白属于植物的GRF家族。
将OsGRF6氨基酸序列与拟南芥的GRF家族蛋白序列进行比对。OsGRF6 蛋白与拟南芥GRF蛋白类似,均存在1个高度保守的QLQ 结构域和WRC结构域(图2)。
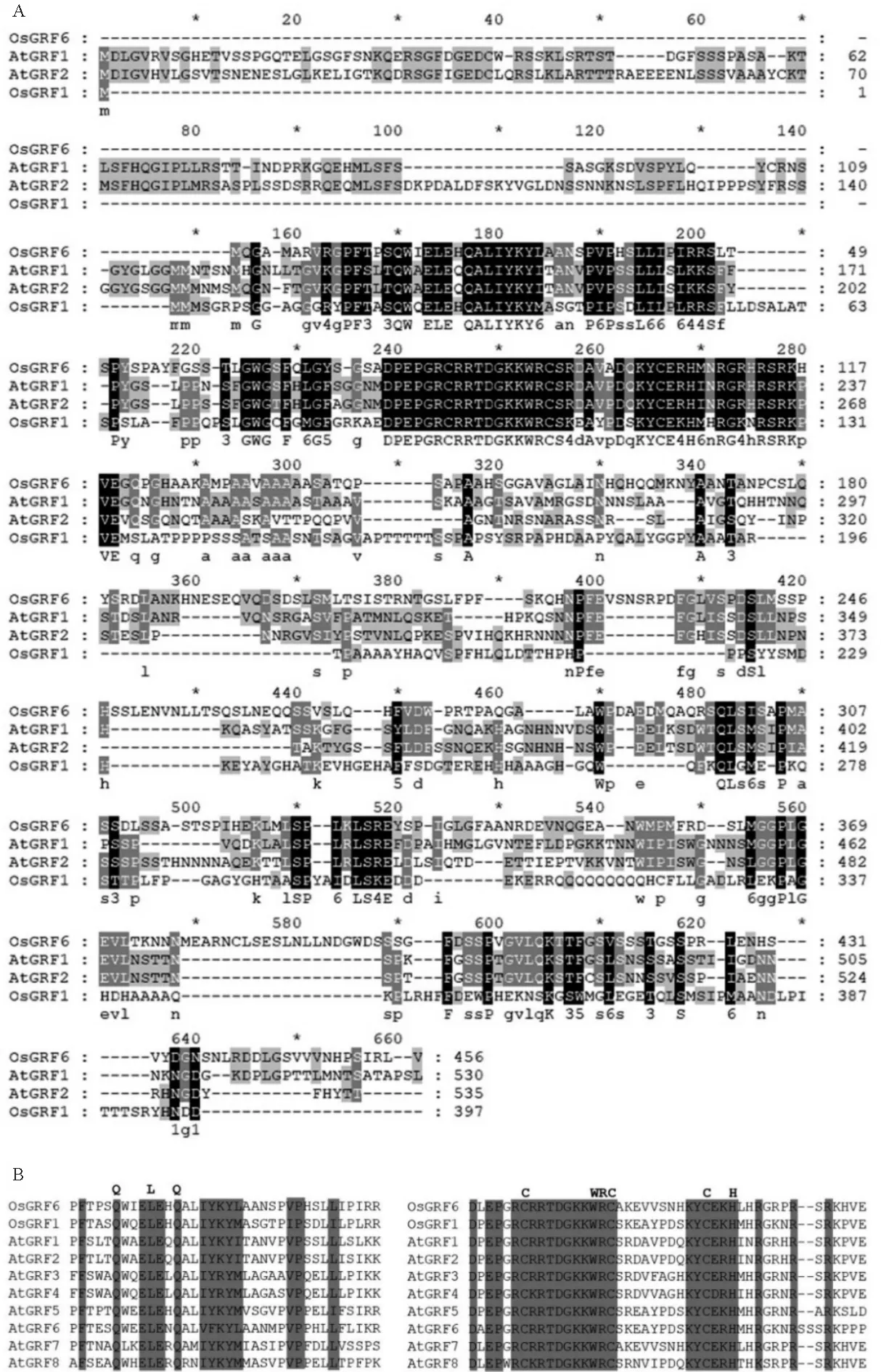
图2 OsGRF6与OsGRF1,AtGRF1和AtGRF2同源蛋白序列比对(A)及OsGRF6保守结构域分析(B)Fig.2 Homologous protein sequence alignment of OsGRF6 with OsGRF1,AtGRF1,and AtGRF2(A)and analysis of the conservered domain of OsGRF6(B)
利用MEGA 7.0软件将OsGRF6与其他GRF蛋白构建系统进化树。结果表明,OsGRF6与单子叶植物水稻中OsGRF7和OsGRF8蛋白同源性较高,亲缘关系最近,其次与双子叶植物拟南芥比较发现OsGRF6 与AtGRF1、AtGRF2 亲缘关系较近(图3)。

图3 OsGRF6进化树分析Fig.3 Phylogenetic tree of OsGRF6
2.2 OsGRF6 启动子作用元件分析
从水稻信息网(RAP-DB)获得了ZH11中Os-GRF6起始密码子上游2 000 bp的启动子区域序列,利用在线软件PlantCARE对其含有的顺式作用元件进行分析,结果显示:启动子区域除了常见的TATA-box和CAAT-box 外,还包含光响应元件10 个(GATA-motif、Sp1、G-box、ATCT-motif、TCT-motif、GT1-motif),植物激素顺时作用元件6个(TGACG-motif、TCA-element、CGTCA-motif、Aux RR-core),非生物胁迫应答元件3 个(ARE、LTR),植物分生组织表达相关元件1 个(CATbox),昼夜节律调节元件1个(circadian),见表1。
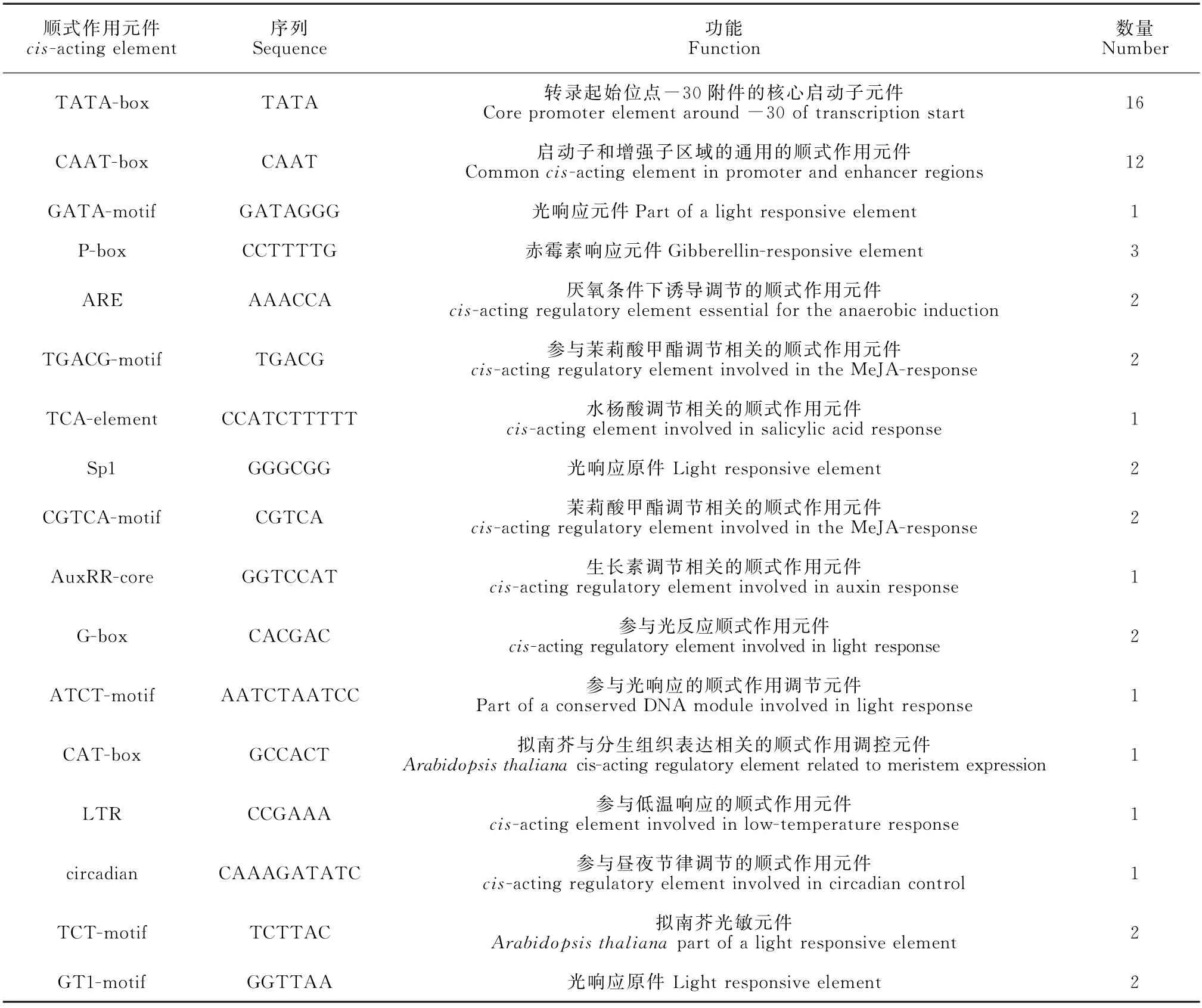
表1 OsGRF6 启动子顺式元件分析Table 1 Analysis of cis-acting elements of OsGRF6 promotor
2.3 OsGRF6 的组织表达模式、亚细胞定位和转录活性分析
通过水稻eFP网站(http://www.bar.utoronto.ca/efprice/cgi-bin/efp Web.cgi? dataSource=ricestigma_mas)分析OsGRF6在水稻不同组织的表达量发现,OsGRF6在水稻叶、种子、花、根和顶端分生组织中都有比较高的表达,其中以花和顶端分生组织中表达量最高,根和种子中表达量次之,叶中表达量相对较少(图4,A)。进一步用qPCR 对网站分析结果进行了验证,结果显示与网站分析结果一致(图4,B)。为了确定OsGRF6 在水稻细胞中的表达位置,通过构建p BI221-OsGRF6-GFP质粒,转化原生质体细胞,结果显示,OsGRF6 绿色荧光与核定位基因H2B 的红色荧光重叠,表明OsGRF6定位细胞核(图4,C),并且通过转化拟南芥原生质体,检测GUS活性分析显示BD-OsGRF6的实验组GUS活性明显高于对照组BD,说明OsGRF6具有转录激活活性(图4,D、E)。

A.eFP网站上OsGRF6 组织表达模式分析;B.qPCR 分析OsGRF6 在水稻叶、种子、穗、茎、根中的相对表达量;C.OsGRF6的亚细胞定位分析;D.OsGRF6 转录活性分析载体构建模式图;E.OsGRF6 转录激活活性分析,BD-HOS15 为正对照,采用Student's t test进行显著性分析(*P<0.05)。图4 OsGRF6 组织表达模式分析、亚细胞定位及转录激活活性分析A.Analysis of the expression pattern of OsGRF6 in eFP.B.Expression levels of OsGRF6 in leaves,seeds,panicles,shoots,and roots.C.Subcellular localization of OsGRF6 in rice protoplast cells.D.Vector construction for analyzing the transcriptional activation activity of OsGRF6.E.Analysis of OsGRF6 transcriptional activation activity.BD-HOS15 as the positive control.Statistical analysis was performed with Student's t-test(*P<0.05).Fig.4 Analysis of the expression pattern,subcellular localization,and transcriptional activation activity of OsGRF6
2.4 OsGRF6 的转基因材料及突变体鉴定和根发育表型分析
为了确定OsGRF6在水稻发育过程中的功能,构建了OsGRF6的过表达转基因材料和基因编辑敲除突变体材料,并对其根发育表型进行初步分析。对转基因材料进行分子鉴定,分析显示突变体材料在OsGRF6第1个外显子有11bp的缺失,并且该缺失使得OsGRF6的氨基酸产生了移码突变(图5,A)。通过分析水稻萌发后3 d和6d的水稻根长发现,在osgrf6突变体中水稻初生根根长度与野生型ZH11相比,明显短于野生型(图5,B),qPCR 检测发现在转基因OE 材料中,OsGRF6的表达量与野生型ZH11相比显著上调(图5,C),而过表达转基因植株的主根长度与野生型和突变体相比则明显变长(图5,D)。
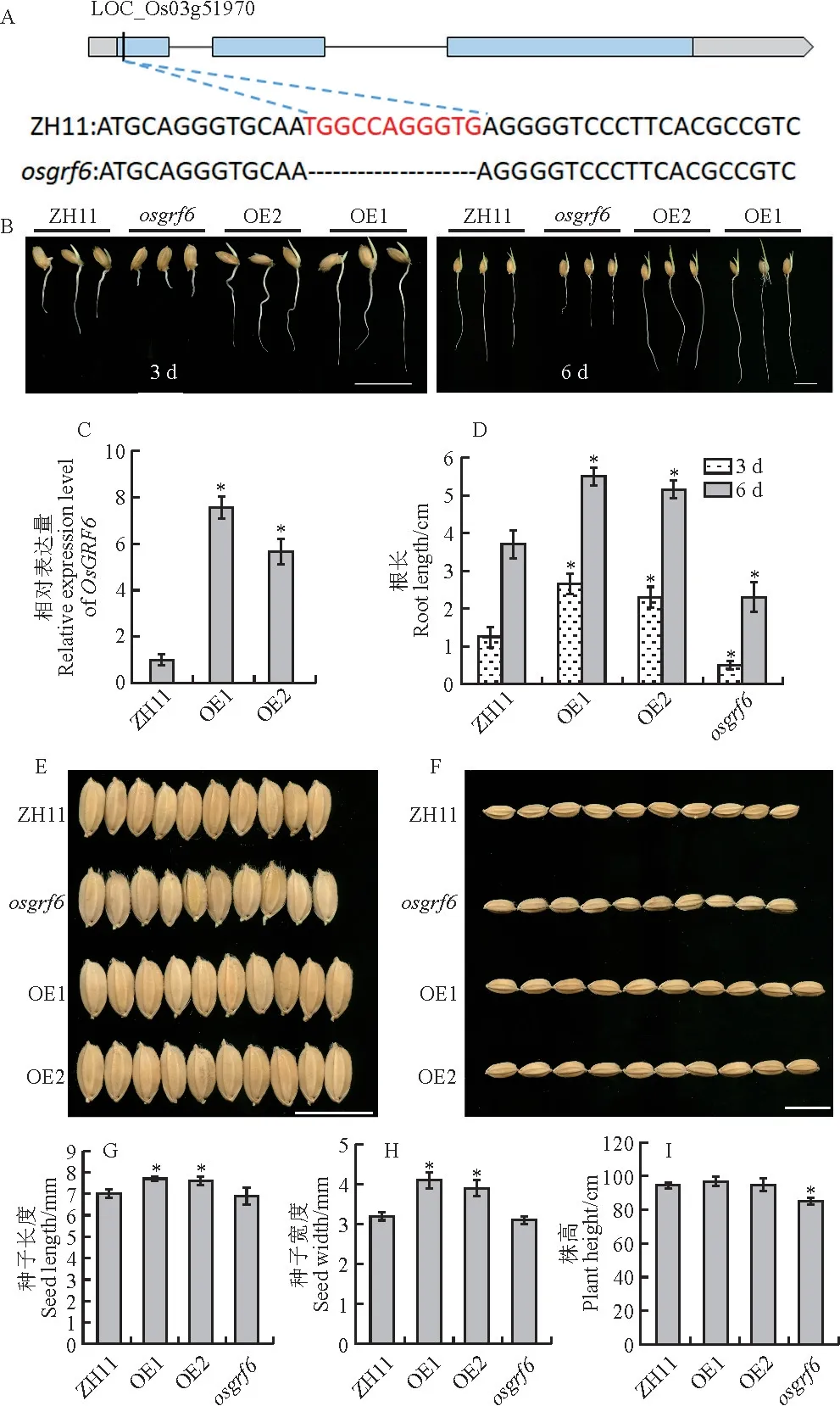
ZH11.中花11;osgrf6.OsGRF6 突变体;OE1.过表达OsGRF6 转基因材料1;OE2.过表达OsGRF6 转基因材料2;A.OsGRF6 基因结构分析和osgrf6 突变体鉴定,浅灰色表示UTR,浅蓝色表示外显子;B.水稻种子萌发3 d和6 d时初生根表型分析,标尺为1 cm;C.OsGRF6 基因表达量分析;D.根长;E.粒宽,标尺为1 cm;F.粒长,标尺为1 cm;G.水稻种子粒长统计;H.水稻种子粒宽统计;I.水稻穗期株高统计;采用Student's t test进行显著性分析(*P<0.05)图5 OsGRF6 转基因材料和突变体鉴定及初生根、粒型、株高表型分析ZH11,Zhonghua 11.osgrf6,OsGRF6 mutant.OE1,OsGRF6 over-expression line 1.OE2,OsGRF6 over-expression line 2.A.Analysis of OsGRF6 gene and identification of osgrf6 mutant.Light gray represents UTR,light blue represents exons.B.Analysis of primary root length after seed germination for 3 days and 6 days,Bar=1 cm.C.The expression level of OsGRF6.D.Root length.E.Seed width,Bar=1 cm.F.Seed length,Bar=1 cm.G.Analysis of seed length.H.Analysis of seed width.I.Analysis of plant height.Statistical analysis was performed with Student's t-test(*P<0.05).Fig.5 Identification of OsGRF6 transgenic lines and mutants and phenotypic analysis of primary roots,seeds,and plant heights
通过表型分析初步说明OsGRF6能参与调控水稻初生根的伸长。此外,通过对水稻粒型的观察和统计,在ZH11背景下OsGRF6-OE 转基因株系表现出粒宽和粒长都增加的表型。而突变体种子则变化不明显(图5,E-H)。通过统计水稻穗期植株株高发现,突变体植株表现出比野生型株高变矮的表型(图5,I)。
2.5 OsGRF6 可与CGGCA 基序结合调控下游靶基因的表达
为了进一步明确OsGRF6调控水稻主根长度的分子机制,用酵母单杂交对已报道OsGRF6结合的基序CGGCA 进行验证,发现OsGRF6能与CGGCA 在酵母细胞中结合(图6,A),进一步对ChIPseq和miRNA396 RNA-seq数据[38]进行分析发现,在ChIP-Seq获得175个靶基因中,有98个靶基因在RNA-seq数据中表达上调的,66 个表达下调的(图6,B)。OsGRF6在已报道的文献中主要参与激活下游靶基因的表达,因此,对上调的98个靶基因进行进一步分析发现,有10个候选靶基因可能参与调控水稻根的发育(表2)。

表2 OsGRF6参与调控水稻根发育的候选靶基因Table 2 OsGRF6 target genes involved in root development

A.酵母单杂交实验验证OsGRF6能与CGGCA motif结合;B.ChIP-seq和RNA-seq韦恩图。图6 酵母单杂交实验验证OsGRF6与CGGCA motif结合以及利用ChIP-seq和RNA-seq预测OsGRF6的直接靶基因A.OsGRF6 interact swith CGGCA motif in yeast cells.B.Wayne diagram analysis of ChIP-seq and RNA-seq assays.Fig.6 Interaction of OsGRF6 with CGGCA motif by yeast one-hybrid and target gene prediction by ChIP-seq and RNA-seq
除了OsARF7和OsARF4外,其中大部分基因功能都未见报道。通过分析这些基因(拟南芥的同源基因)发现,joka8与拟南芥LRL1同源,参与调控植物根毛的发育[39]。LOC_Os01g55780 与拟南芥GDPD6同源,参与调控磷缺失下,拟南芥根的伸长[40]。LOC_Os07g37454与拟南芥OCT2同源参与调控根的发育[41]。此外,LOC_Os03g58230,LOC_Os07g36610和LOC_Os03g44440的同源基因也参与调控拟南芥根的发育[42-44]。这些候选靶基因可为进一步研究OsGRF6参与调控水稻根的发育提供线索。
3 讨论
GRF转录因子是植物体内一类重要的转录因子。该类转录因子主要有2 个保守的结构域,即QLQ 结构域和WRC 结构域,其中WRC 结构域其主要功能负责与靶基因的启动子区结合,从而调控下游靶基因的转录水平[45]。GRF 家族基因被报道参与调控植物的生长发育和对非生物胁迫的响应。
有研究表明,GRF 家族参与调控植物叶的发育,如At GRF1、At GRF3、At GRF5通过调控叶片细胞的数目调控叶片发育[46-48],过表达AtGRF1、At-GRF5的转基因植株表现出叶片变大的表型。GRF家族转录因子也参与调控植物花的发育,GIF-GRF互作模块在植物花发育调控中起着重要作用。拟南芥中atgif1/2/3突变体表现出雌蕊和胚囊缺失的表型[49]。水稻中osgrf6/osgrf10的纯合突变体表现出小花发育异常和开颖的表型[29]。GRF 主要在生长旺盛的幼嫩组织中表达,且其表达受小RNA396a、RNA396b、RNA396d的负调控[32,50]。此外,OsGRF6和OsGRF4还参与调控水稻种子的发育,过表达OsGRF6的转基因株系种子与野生型相比,表现出种子变大的表型,而突变体则表现出种子变小的表型[35,38]。进一步研究表明,OsGRF6通过调控转录因子Os TAWAWA1和OsMADS34的表达,正调控生长素合成和信号通路,进而调控种子的发育[38]。本文中过表达OsGRF6的转基因植株也表现出种子变大的表型,而突变体植株的种子与野生型差别不明显,可能是由于水稻GRF家族存在功能冗余的现象。此外,与前人的研究比较,水稻转基因背景品种差异也可能是导致突变体种子表型不明显的另一重要原因。拟南芥中小RNA396a参与调控拟南芥根的发育,过表达小RNA396a的转基因植株表现出根变短的表型,进一步研究发现,小RNA396通过抑制转录因子b H LH74的表达水平,进而调控拟南芥根的发育[51]。作为小RNA396家族的靶基因GRF,是否也参与调控植物根的发育,组织表达模式分析显示,GRF转录因子在许多种类植物的根中都有表达,说明GRF家族转录因子可能参与调控植物根的发育[50]。拟南芥中还没有直接证据证明是否miR396通过调控GRF转录因子参与调控植物根的发育。本文通过对水稻OsGRF6组织表达模式分析,也验证了OsGRF6在根中表达量较高,且OsGRF6过表达转基因植株和功能缺失突变体也分别表现出与野生型相比初生根较长和较短的表型。说明OsGRF6能参与调控水稻初生根的伸长。在OsGRF6调控种子大小方面的研究表明,OsGRF6通过生长素途径调控种子的大小,在其ChIP-seq数据中发现多个与植物根发育相关的候选靶基因,其中也有受生长素诱导表达的Os ARF4和OsARF7。可能OsGRF6调控水稻初生根的发育,也是通过调控生长素途径。此外,通过OsGRF6启动子分析表明,OsGRF6启动子区有多个生长素调控的结合元件,可能存在ARF 家族基因对GRF基因的反馈调控,基于以上的研究结论,是否存在这一推论,未来还需从分子和遗传方面做更深入探索。
4 结论
本研究初步解析了OsGRF6参与调控水稻根发育的生物学功能,确定了OsGRF6调控水稻根发育可能通过调节生长素信号通路来实现,挖掘出一些与水稻根发育相关且可能是OsGRF6的靶基因,可为进一步研究OsGRF6的生物学功能提供理论依据。
致谢:感谢中科院植物研究所种康院士和徐云远研究员惠赠OsGRF6的试验材料。

