冠层活性相关性状全基因组关联分析及其对产量性状遗传效应的解析
李法计,程敦公,余晓丛,闻伟锷,刘金栋,翟胜男,刘爱峰,郭军,曹新有,刘成,宋健民,刘建军,李豪圣
冠层活性相关性状全基因组关联分析及其对产量性状遗传效应的解析

1山东省农业科学院作物研究所/小麦玉米国家工程研究中心/农业农村部黄淮北部小麦生物学与遗传育种重点实验室/山东省小麦技术创新中心/济南市小麦遗传改良重点实验室,济南 250100;2菏泽学院农业与生物工程学院(牡丹学院),山东菏泽 274015;3遵义医科大学细胞生物学教研室,贵州遵义 563099;4中国农业科学院作物科学研究所,北京 100081
【目的】冠层活性是反映作物生长发育的重要指标,发掘小麦冠层活性相关基因并分析其与产量性状的关系,为解析产量遗传结构和辅助育种提供理论支撑。【方法】以166份国内外小麦品种为材料,将其种植于河南安阳和安徽濉溪,结合已构建的包含326 570个来源于小麦90K和660K SNP芯片标记的高密度整合物理图谱,对苗期和花后10 d归一化植被指数(NDVI-S和NDVI-10)及花后10 d旗叶叶绿素含量(Chl-10)进行关联分析,并与前期利用相同材料得到的产量相关性状结果进行比较。【结果】NDVI-S、NDVI-10和Chl-10在基因型、环境及基因型×环境互作间变异方差均达极显著(<0.01)差异,广义遗传力(2)分别为0.81、0.81和0.91。分别检测到13、12和15个与NDVI-S、NDVI-10和Chl-10显著相关的位点,其中,有12、11和12个为新位点,5个位点与2个以上性状有关。166份小麦品种含有NDVI-S、NDVI-10和Chl-10优异等位基因数变幅分别为4-11、3-11和4-12,且随着优异等位基因数目增加其对应表型值呈递增趋势。NDVI-S与千粒重、粒长和粒宽显著正相关(<0.01);Chl-10与产量和旗叶宽显著正相关(<0.01),与单位面积穗数、株高和穗下节长显著负相关(<0.01)。检测到7个同时与产量和冠层活性相关的多效性位点。【结论】NDVI-S可用于品种产量性状的选择,检测到的稳定位点和多效性位点可在开发标记后用于育种材料的检测。
小麦;冠层活性;产量;归一化植被指数;叶绿素含量;关联分析
0 引言
【研究意义】高产是小麦育种永恒的目标。作物冠层活性主要受绿色植被覆盖度、叶绿素(chlorophyll,Chl)含量、蒸腾速率和光合速率等相关性状影响,是表征作物群体活力的重要性状,也是作物高产形成的重要基础。归一化植被指数(normalized difference vegetation index,NDVI)是反映作物地上部绿色植被覆盖度的重要参数之一,被广泛应用于不同时期及水肥处理下小麦群体冠层活性研究[1-8]。叶绿素是植物进行光合作用的重要色素,灌浆期叶片Chl含量可直接影响小麦光合速率和灌浆速率[9]。发掘小麦NDVI和Chl相关基因并分析其与产量性状的关系,是解析产量形成机理、进行高产分子遗传改良的重要途径。【前人研究进展】前人将灌浆期NDVI相关QTL(quantitative trait locus)定位在2D、3A、4A、4B、4D、5A、5B、6A和6B染色体上[10-11],将苗期NDVI相关QTL定位在1B、5A和7B染色体上[12],将灌浆期旗叶Chl含量相关QTL定位在1B、2A、2B、2D、3A、3B、4A、4B、4D、5A、6A、6B和6D染色体上[11-22]。此外,前期利用3个RIL群体结合小麦90K SNP芯片对产量和冠层活性相关性状进行了QTL定位,分别检测到6、16和14个与苗期NDVI(NDVI-S)、花后10 d NDVI(NDVI-10)和花后10 d旗叶Chl(Chl-10)含量相关的QTL,其中,有6、10和10个为新QTL;将冠层活性相关性状QTL与产量性状QTL位点进行比较,检测到12个同时包含冠层活性和产量相关性状QTL位点的区段[23-24]。【本研究切入点】目前,利用自然群体进行关联分析发掘冠层活性相关位点的研究报道较少,冠层活性与产量相关性状表型和基因型关系的研究也较为缺乏。【拟解决的关键问题】本研究以166份国内外小麦品种为材料,结合已构建的包含小麦90K和660K SNP芯片的高密度整合物理图谱,对冠层活性相关性状NDVI-S、NDVI-10和Chl-10进行全基因组关联分析,发掘相关遗传位点。同时,结合前期研究结果,从表型和基因型分析冠层活性与产量相关性状的关系,为小麦高产分子遗传改良提供理论支撑和基因资源。
1 材料与方法
1.1 试验材料
选用166份国内外小麦品种作为试验材料。其中,144份为国内黄淮麦区主栽品种,22份为国外引进的种质。品种具体来源见Li等[25]。
1.2 田间试验和性状调查
将试验材料种植于中国农业科学院安阳棉花所白壁试验站和安徽省农业科学院濉溪试验站。在白壁试验站2年度播种期分别为10月6日和10月4日,在濉溪试验站2年度播种期分别为10月10日和10月8日,田间试验采用随机区组设计,3行区,50粒/行,均匀点播,行距20 cm,行长2 m,3次重复。田间管理与当地大田生产一致。
分别于播种后30 d和花后10 d,在每个小区内选取相邻的2行离冠层50 cm高度,利用手持式GreenSeeker(GreenSeeker, Ntech Industries, Inc, Ukiah, CA)进行扫描,作为该品种的NDVI值;于花后10 d,在每个小区内随机选取10片旗叶利用手持式SPAD-502叶绿素测定仪(Minolta, Japan)进行测定,作为该品种的Chl含量值。产量相关性状调查方法见LI等[25]。
1.3 统计分析

1.4 基因型检测、群体结构与亲缘关系及连锁不平衡衰减分析
利用Illumina公司(http://www.illumina.com/)的90K及中国农业科学院作物科学研究所贾继增课题组开发的660K SNP芯片对166份品种的DNA进行检测和基因型分析。SNP基因芯片检测在北京博奥晶典生物技术有限公司(http://cn.capitalbio.com/)进行。参照中国春参考基因组IWGSC v1.0(http://www. wheatgenome.org/)进行本地比对(BLASTn,e<10— 20)[29],构建整合物理图谱[25]。利用Structure v2.3.4软件(http://pritchardlab.stanford.edu/structure. html)[30]进行群体结构分析,然后利用Tassel v5.0进行主成分分析(principal components analysis,PCA)和亲缘关系分析[25]。
以位点间的相关系数平方(2)作为衡量多态性位点两两之间连锁不平衡(linkage disequilibrium,LD)的参数。采用Tassel v5.0计算标记间的LD值。由于A、B和D基因组间标记密度差异较大,分别对A、B、D和全基因组进行LD衰减分析[25]。
1.5 全基因组关联分析
利用Tassel v5.0软件的混合线性模型(MLM),在考虑群体结构(Q)和亲缘关系(K)的情况下,分别对NDVI-S、NDVI-10和Chl-10 3个性状的E1—E5 5个数据集进行GWAS分析,E1:2012—2013安阳;E2:2012—2013濉溪;E3:2013—2014安阳;E4:2013—2014濉溪;E5:E1—E4的BLUE值。根据GWAS分析结果,当<0.001时,认为该标记与性状显著关联。GWAS分析的QQ图(quantile-quantile plot)可表示在当前模型下的分析效果,实际与期望的接近程度反映了该模型对群体结构和亲缘关系对GWAS分析影响的控制效果。曼哈顿图(manhattan plot)是一种表示点与数值间对应关系的散点图,横坐标为连锁群位置即染色体,纵坐标为SNP标记的负常用对数:Y=-log10()。根据图中SNP位点的分布可以观察标记与性状的关联程度。在2个及以上环境下(包含BLUE值)检测到的位点作为显著性位点[25]。
1.6 优异等位基因效应分析
以增加表型值的等位基因作为优异等位基因,统计每个品种多环境下重复被检测到的显著性位点的优异等位基因数目。将含有不同数目优异等位基因的品种进行分组,利用R语言绘制优异等位基因数目对3个性状表型BLUE值的效应。
2 结果
2.1 冠层活性相关性状的统计和广义遗传力
方差分析(表1)表明,NDVI-S、NDVI-10和Chl-10在基因型、环境及基因型×环境互作间变异方差均达极显著(<0.01)差异;除NDVI-10外,NDVI-S和Chl-10在重复间变异方差达极显著(<0.01)水平。NDVI-S、NDVI-10和Chl-10的分别为0.81、0.81和0.91,说明遗传因素是导致3个性状变异的主要原因。
2.2 冠层活性相关性状的频率分布、变异范围、标准偏差和变异系数
频率分布直方图(图1)显示,NDVI-S、NDVI-10和Chl-10 3个性状表型值呈连续分布,表型值分布频率均在变异范围内呈现先增加后降低的趋势。3个性状的表型值在不同品种间变异较大,环境对性状表型值也有一定影响,性状表型数据的平均值接近中间值。其中,NDVI-S的变异系数大于NDVI-10和Chl-10(表2)。
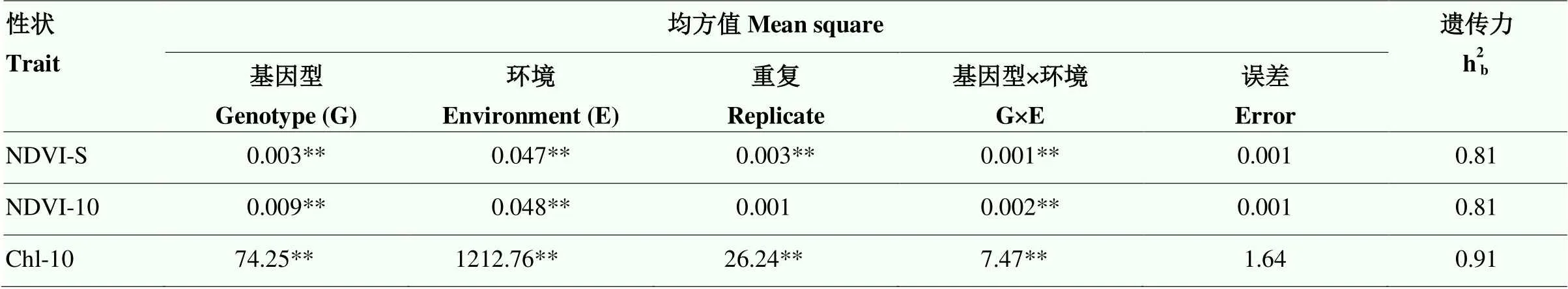
表1 冠层活性相关性状的方差分析和广义遗传力
NDVI-S:苗期归一化植被指数;NDVI-10:花后10 d归一化植被指数;Chl-10:花后10 d旗叶叶绿素含量;**表示在0.01水平差异显著。下同
NDVI-S: normalized difference vegetation index at seedling stage; NDVI-10: Normalized difference vegetation index at 10 days after flowering; Chl-10: Chlorophyll content in flag leaf at 10 days after flowering; ** Significant at<0.01. The same as below
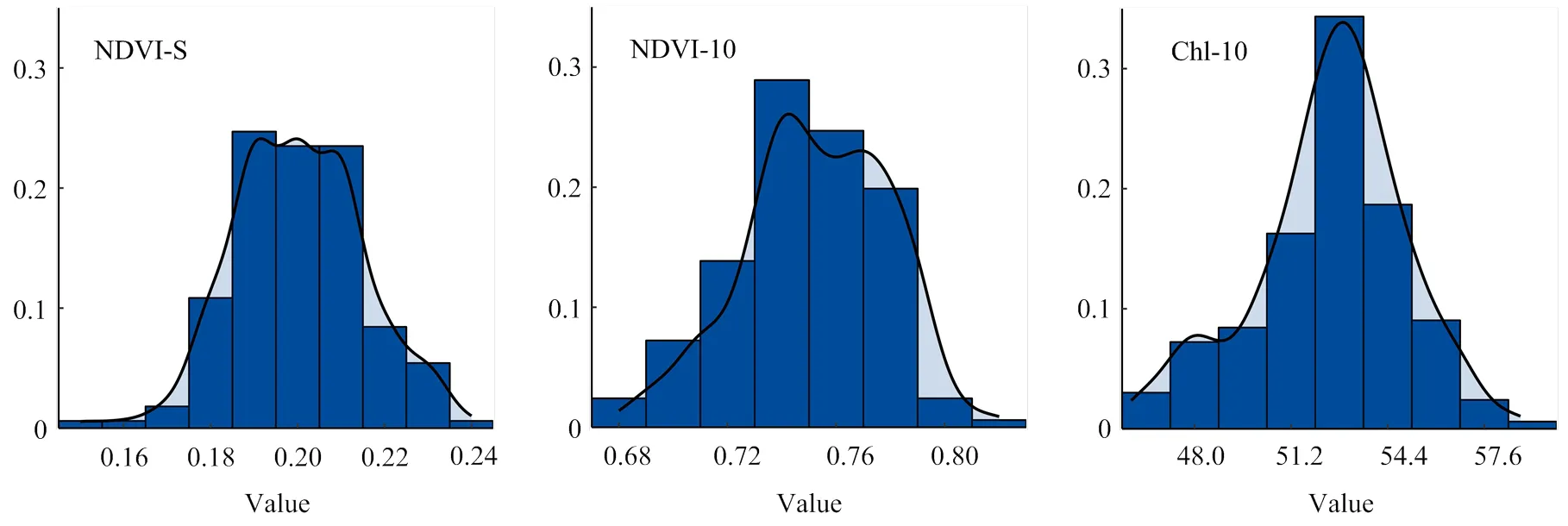
图1 NDVI-S、NDVI-10和Chl-10 BLUE值的频率分布直方图

表2 166份小麦品种冠层活性相关性状的均值、标准差和变异范围
E1:2012—2013安阳;E2:2012—2013濉溪;E3:2013—2014安阳;E4:2013—2014濉溪;E5:最佳线性无偏估计值。下同
E1: 2012-2013 Anyang; E2: 2012-2013 Suixi; E3: 2013-2014 Anyang; E4: 2013-2014 Suixi; E5: Best linear unbiased estimation. The same as below
2.3 冠层活性相关性状显著性位点
经过分析,共检测到40个与NDVI-S、NDVI-10和Chl-10相关的显著性位点(图2和表3)。
检测到13个与NDVI-S相关的显著性位点,分别位于1A、2A、2B、3B、4A、4B、5A、5B、5D、6B、7A和7B(2)染色体上。其中,位于3B()和4B()染色体上的位点均在3个环境及BLUE值下被检测到,分别解释表型变异的6.9%—9.8%和7.0%—11.1%。位于2B()和7B()染色体上的位点分别可解释表型变异的6.9%—12.5%和6.8%—13.5%。
检测到12个与NDVI-10相关的显著性位点,分别位于2A、3A、4A、5A(3)、5B(2)、5D、7A(2)和7B染色体上。其中,位于3A()和4B()染色体上的位点均在3个环境及BLUE值下被检测到,分别解释表型变异的7.3%— 13.7%和7.0%—11.5%。位于7B()染色体上的位点可解释表型变异的6.7%—18.5%。
检测到15个与Chl-10相关的显著性位点,分别位于1A(2)、1B(4)、2B、2D、4A(3)、5A、5B、6A和7A染色体上。其中,位于1B()、2B()、2D()、4A()、5A()和7A()染色体上的位点均在4个环境及BLUE值下被检测到,分别解释表型变异的6.9%—11.7%、6.8%—10.1%、6.9%—11.0%、6.8%—9.7%、6.5%—12.1%和6.7%— 10.3%。
在检测到的冠层活性相关性状显著性位点中,有5个位点与2个或2个以上性状有关,分布在2A、4A(2)、5A和7A染色体上。其中,5A()染色体上的位点与NDVI-S、NDVI-10和Chl-10均显著相关;2A()和7A()染色体上的位点均与NDVI-S和NDVI-10显著相关。
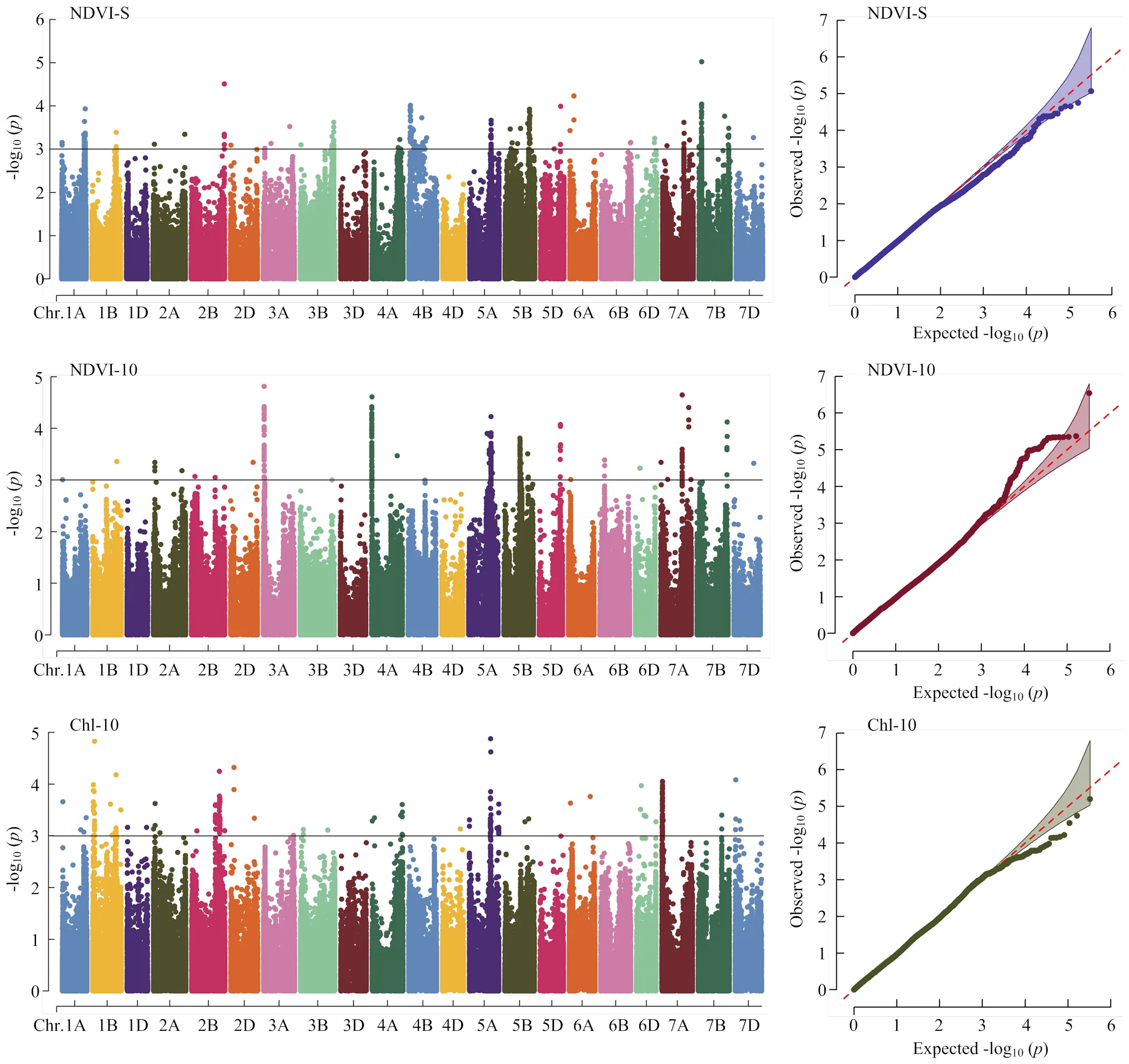
图2 NDVI-S、NDVI-10和Chl-10 BLUE值的曼哈顿图(左)和QQ图(右)

表3 冠层活性相关性状显著性位点及与前人研究比较
a代表性标记;bSNP标记在中国春基因组上的物理位置;c优异等位基因(SNP)下标注横线;d已报道的相关位点附近的QTL、标记或基因
aRepresentative markers;bThe physical positions of SNP markers based on Chinese Spring genome sequences from the International Wheat Genome Sequencing Consortium;cFavorable allele (SNP) is underlined;dThe previously reported QTL, markers or genes near the loci identified in the present study
2.4 优异等位基因遗传效应
把增加表型值的等位基因记作优异等位基因,NDVI-S、NDVI-10和Chl-10在166份小麦品种中优异等位基因变异范围分别为4—11、3—11和4—12。对含有不同等位基因数的品种的表型值分析显示,随着优异等位基因数目的增加,3个性状的表型值均呈递增趋势(图3)。

图3 优异等位基因数目增加对NDVI-S、NDVI-10和Chl-10表型值的作用
2.5 冠层活性与产量相关性状的遗传关系
将冠层活性相关性状与前期研究的产量性状进行相关分析显示,NDVI-S与千粒重(thousand-kernel weight,TKW)、粒长(kernel length,KL)、粒宽(kernel width,KW)、穗干重(spike dry weight,SDW)、株高(plant height,PH)、穗下节长(uppermost internode length,UIL)和旗叶长(flag leaf length,FLL)呈显著正相关(<0.01),与抽穗期(heading date,HD)呈显著负相关(<0.01)(表4)[25]。NDVI-10与穗长(spike length,SL)、PH和UIL呈显著负相关(<0.01)。Chl-10与产量(grain yield, GY)和FLW(flag leaf width,FLW)显著正相关(<0.01),与单位面积穗数(spike number per unit area,SN)、PH和UIL显著负相关(<0.01)。
通过比较冠层活性相关位点与前期利用相同材料检测到的产量性状相关位点的关系,在1A、2A、5A(2)、5B、6B和7A染色体上检测到7个同时与产量和冠层活性相关的多效性位点(表5)[25]。发现GY与NDVI-S、NDVI-10和Chl-10分别有3、3和1个位点一致,SL与NDVI-S、NDVI-10和Chl-10分别有2、3和3个位点一致,PH与NDVI-S、NDVI-10和Chl-10分别有2、3和1个位点一致。
3 讨论
3.1 显著性位点的比较分析
近年来,与小麦发育进程相关的生理性状基因位点被报道越来越多,但不同材料、检测方法得到的位点差异较大。本研究在6B染色体检测到的NDVI-S显著性位点()与前期利用豆麦/石4185构建的重组自交系群体(recombinant inbred line,RIL)检测到的QTL()位置一致[24]。5A染色体上NDVI-10显著相关的位点与前期利用临麦2号/中892 RIL群体检测到的QTL()位置一致[24]。Chl-10显著性位点(2D)与Gao等[11]定位到的位置一致,(5A)与Liu等[21]检测到的位点(5A)位置一致,(6A)与Gao等[11]、Hassan等[20]和杨等[22]分别定位到的、和位置一致。除以上位点外,本研究检测到的其他冠层活性相关位点可能为新位点。另外,已报道的5A染色体上与NDVI和Chl相关的位点约有30个,该染色体可能对调控小麦生理性状起重要作用[11-14,16,21,24]。
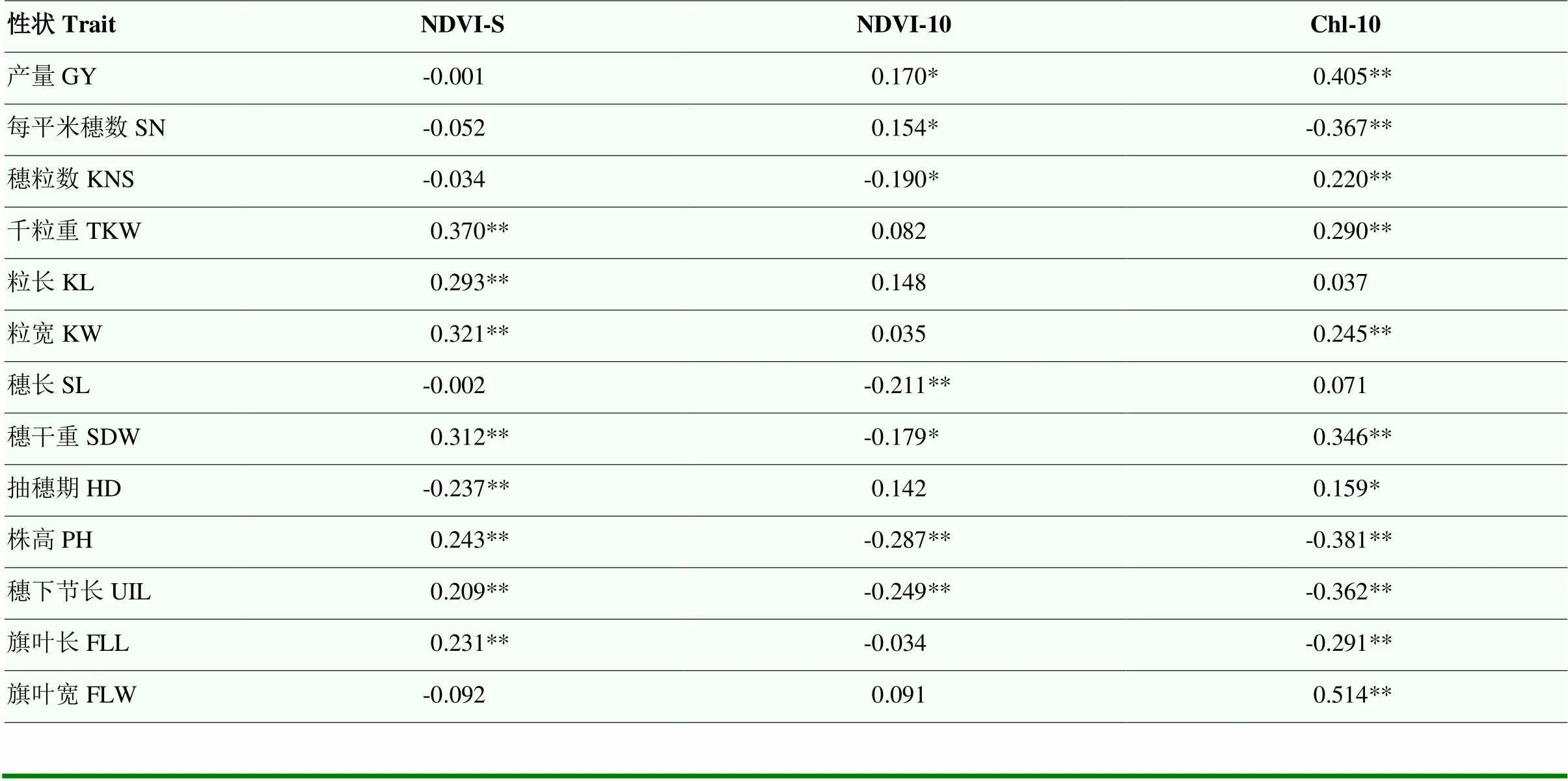
表4 冠层活性相关性状与产量相关性状间的相关分析
GY:产量;SN:每平米穗数;KNS:穗粒数;TKW:千粒重;KL:粒长;KW:粒宽;SL:穗长;SDW:穗干重;HD:抽穗期;PH:株高;UIL:穗下节长;FLL:旗叶长;FLW:旗叶宽;*表示在<0.05水平差异显著
GY: grain yield; SN: spike number per unit area; KNS: kernel number per spike; TKW: thousand-kernel weight; KL: kernel length; KW: kernel width; SL: spike length; SDW: spike dry weight; HD: heading date; PH: plant height; UIL: uppermost internode length; FLL: flag leaf length; FLW: flag leaf width; *indicate significantly different at<0.05
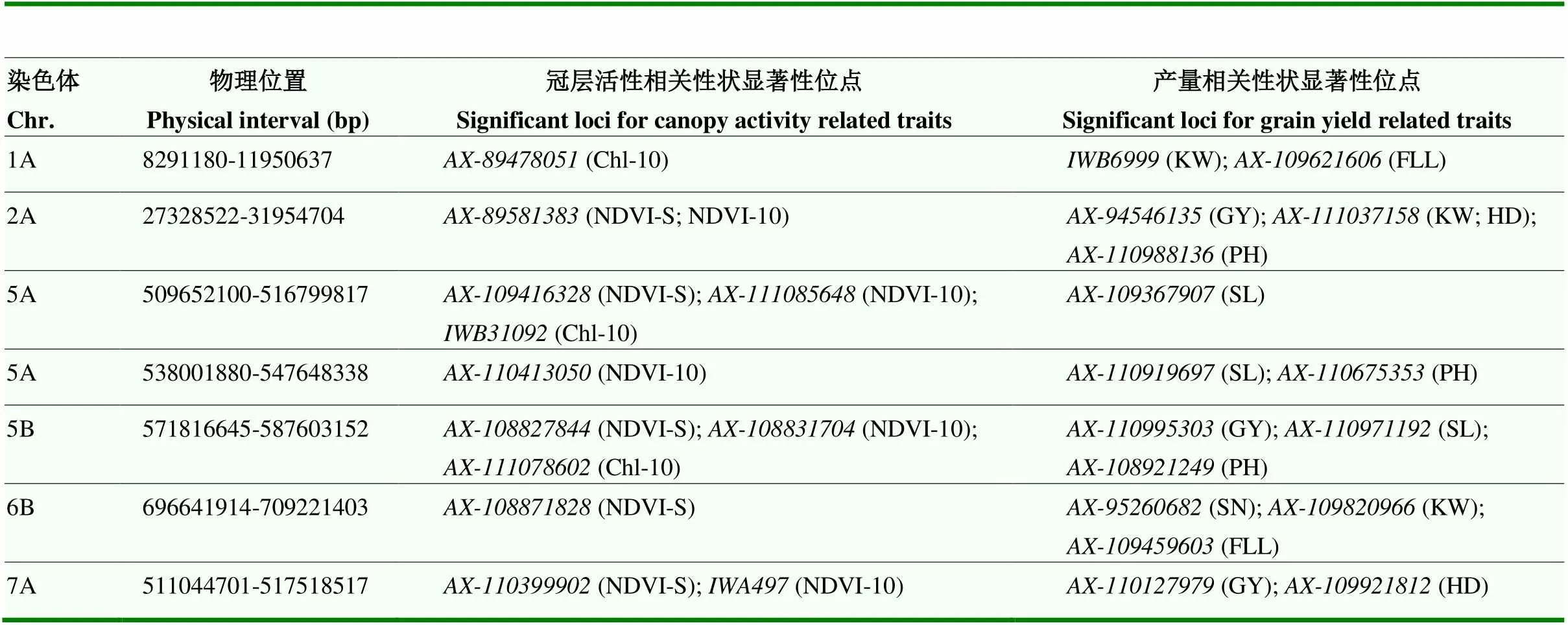
表5 冠层活性和产量相关性状多效性位点
3.2 冠层活性相关性状与产量性状的关系分析
NDVI和Chl含量是表征作物绿色植被覆盖度和光合活力的重要指标,对作物产量形成具有重要影响。国内外学者在作物生长过程中NDVI和Chl含量等冠层活性相关性状与产量性状的遗传关系研究开展了大量工作,其中,灌浆期NDVI已经被越来越多地应用于小麦产量的预测研究[1-7]。Bellundagi等[31]研究发现,出苗后25 d地面绿色植被覆盖率与小麦产量呈极显著(<0.01)正相关,相关系数达0.662,这与本研究中NDVI-S与GY相关性不高的结果存在差异。Shi等利用1个双单倍体(doubled haploid,DH)群体进行研究,发现在雨养条件下返青、拔节和孕穗期NDVI与产量均呈显著(<0.05)或极显著(<0.01)正相关;在充分灌溉条件下,返青、拔节和孕穗期NDVI与TKW在多数环境下呈极显著(<0.01)负相关,与GY在多数环境下呈极显著(<0.01)正相关,开花期和灌浆期旗叶Chl含量与TKW和GY呈负相关,并在半数环境下达显著(<0.05)负相关。前期利用豆麦/石4185、藁城8901/周麦16、临麦2号/中892 3个RIL群体研究发现,NDVI-S与TKW在3个群体中均显著正相关(<0.01),这与本研究结果一致;Chl-10与FLW在3个群体中均显著正相关(<0.01),与株高(PH)显著负相关(<0.01),与本研究结果也一致[23-24]。以上研究表明,试验材料、环境和性状调查时期对冠层活性相关性状与产量性状关系的影响较大,NDVI-S在遗传群体与自然群体中与TKW相关性均较高,可用于育种材料粒重的初步选择。
前人在冠层活性相关性状与产量性状一致性位点发掘方面也取得了一定成效。Hassan等[20]在2B染色体上定位到一个热处理下与旗叶Chl含量和GY同时相关的QTL。Liu等[21]利用1个RIL群体发掘到多个旗叶Chl含量和产量性状多效性位点,其中,1D染色体上—标记区间内的QTL同时与灌浆中期Chl含量和GY有关,3B染色体上—标记区间内的QTL同时与灌浆后期Chl含量和TKW有关,3D染色体上—标记区间内的QTL同时与开花期Chl含量和TKW有关。Shi等[12]利用DH群体,在4D染色体上发掘到1个与拔节期NDVI、灌浆期旗叶Chl含量、TKW和PH相关的QTL区段,该区段与前期利用藁城8901/周麦16和临麦2号/中892 2个RIL群体发掘到的QTL簇位置相近,可能为作用所致[24];此外,在5A染色体上还检测到1个与返青、拔节和孕穗期NDVI、开花期Chl含量和TKW均相关的QTL区段。Xu等[18]利用1个RIL群体在2B、3A、5B和7B染色体上检测到4个与旗叶Chl含量及产量性状同时相关的QTL簇,其中3A和7B染色体上的QTL簇与前期利用3个RIL群体检测到的QTL簇位置不一致[24],5B染色体上的QTL簇与本研究检测到的多效性位点位置也不一致。综上所述,NDVI和Chl含量相关位点与产量性状相关位点存在部分重合现象,说明NDVI和Chl含量对产量形成起重要作用。已报道的灌浆期Chl含量与TKW一致性位点较多,Chl含量可能对小麦粒重影响较大。
3.3 育种应用价值分析
小麦90K和660K SNP芯片分别有64 643和229 266个SNP位于带注释的基因或启动子间隔中[32],相关性状的显著性位点可直接与基因进行关联。本研究将选用的166份小麦品种同时进行了小麦90K和660K SNP芯片检测,共筛选到326 570个SNP标记用于构建物理图谱和进行关联分析。然后将1个LD内的显著性标记看作1个显著性位点,有效提升了位点的可靠性,也为相关基因发掘奠定了良好的基础。本研究发现,优异等位基因累积可有效提高品种性状的表型值。因此,聚合优异等位基因是提升品种性状表现和进行遗传改良的重要策略。与NDVI-S显著相关的(3B)和(4B)、与NDVI-10显著相关的(3A)和(4B)以及与Chl-10显著相关的(1B)、(2B)、(2D)、(4A)、(5A)和(7A)位点稳定性较好,以上位点与5个多效性位点可在标记转化后用于育种材料的检测。
4 结论
检测到35个与小麦冠层活性相关性状有关的新位点,5个与2个或以上性状有关的多效性位点。其中,与NDVI-S显著相关的(3B)和(4B)、与NDVI-10显著相关的(3A)和(4B),以及与Chl-10显著相关的(1B)、(2B)、(2D)、(4A)、(5A)和(7A)位点稳定性较好。
[1] FREEMAN K W, RAUN W R, JOHNSON G V, MULLEN R W, STONE M L, SOLIE J B. Late-season prediction of wheat grain yield and grain protein. Communications in Soil Science and Plant Analysis, 2003, 34(13/14): 1837-1852.
[2] BABAR M A, REYNOLDS M P, VAN GINKEL M, KLATT A R, RAUN W R, STONE M L. Spectral reflectance indices as a potential indirect selection criteria for wheat yield under irrigation. Crop Science, 2006, 46(2): 578-588.
[3] BABAR M A, REYNOLDS M P, VAN GINKEL M, KLATT A R, RAUN W R, STONE M L. Spectral reflectance to estimate genetic variation for in-season biomass, leaf chlorophyll, and canopy temperature in wheat. Crop Science, 2006, 46(3): 1046-1057.
[4] HAZRATKULOVA S, SHARMA R C, ALIKULOV S, ISLOMOV S, YULDASHEV T, ZIYAEV Z, KHALIKULOV Z, ZIYADULLAEV Z, TUROK J. Analysis of genotypic variation for normalized difference vegetation index and its relationship with grain yield in winter wheat under terminal heat stress. Plant Breeding, 2012, 131(6): 716-721.
[5] XIAO Y G, QIAN Z G, WU K, LIU J D, XIA X C, JI W Q, HE Z H. Genetic gains in grain yield and physiological traits of winter wheat in Shandong province, China, from 1969 to 2006. Crop Science, 2012, 52: 44-56.
[6] GAO F M, MA D Y, YIN G H, RASHEED A, DONG Y, XIAO Y G, XIA X C, WU X X, HE Z H. Genetic progress in grain yield and physiological traits in Chinese wheat cultivars of southern yellow and Huai valley since 1950. Crop Science, 2017, 57(2): 760-773.
[7] HASSAN M A, YANG M J, RASHEED A, YANG G J, REYNOLDS M, XIA X C, XIAO Y G, HE Z H. A rapid monitoring of NDVI across the wheat growth cycle for grain yield prediction using a multi-spectral UAV platform. Plant Science, 2019, 282: 95-103.
[8] 李龙, 彭智, 毛新国, 王景一, 昌小平, 柳玉平, 景蕊莲. 小麦高密度遗传图谱构建及抗旱相关生理性状的遗传解析. 植物遗传资源学报, 2018, 19(3): 531-538.
LI L, PENG Z, MAO X G, WANG J Y, CHANG X P, LIU Y P, JING R L. Genetic map construction and genetic dissection of drought- tolerant related physiological traits in wheat. Journal of Plant Genetic Resources, 2018, 19(3): 531-538. (in Chinese)
[9] AVENSON T J, CRUZ J A, KANAZAWA A, KRAMER D M. Regulating the proton budget of higher plant photosynthesis. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(27): 9709-9713.
[10] EL-FEKI W M, BYRNE P, REID S, HALEY S. Mapping quantitative trait loci for bread making quality and agronomic traits in winter wheat under different soil moisture levels[D]. Fort Collins: Colorado State University, 2010.
[11] GAO F M, WEN W E, LIU J D, RASHEED A, YIN G H, XIA X C, WU X X, HE Z H. Genome-wide linkage mapping of QTL for yield components, plant height and yield-related physiological traits in the Chinese wheat cross Zhou 8425B/Chinese Spring. Frontiers in Plant Science, 2015, 6: 1099.
[12] SHI S K, AZAM F I, LI H H, CHANG X P, LI B Y, JING R L. Mapping QTL for stay-green and agronomic traits in wheat under diverse water regimes. Euphytica, 2017, 213(11): 246.
[13] ZHANG K P, FANG Z J, LIANG Y, TIAN J C. Genetic dissection of chlorophyll content at different growth stages in common wheat. Journal of Genetics, 2009, 88(2): 183-189.
[14] GENC Y, OLDACH K, VERBYLA A P, LOTT G, HASSAN M, TESTER M, WALLWORK H, MCDONALD G K. Sodium exclusion QTL associated with improved seedling growth in bread wheat under salinity stress. Theoretical and Applied Genetics, 2010, 121(5): 877-894.
[15] KUMAR S, SEHGAL S K, KUMAR U, VARA PRASAD P V, JOSHI A K, GILL B S. Genomic characterization of drought tolerance-related traits in spring wheat. Euphytica, 2012, 186(1): 265-276.
[16] JIA H Y, WAN H S, YANG S H, ZHANG Z Z, KONG Z X, XUE S L, ZHANG L X, MA Z Q. Genetic dissection of yield-related traits in a recombinant inbred line population created using a key breeding parent in China’s wheat breeding. Theoretical and Applied Genetics, 2013, 126(8): 2123-2139.
[17] TALUKDER S K, ALI BABAR M, VIJAYALAKSHMI K, POLAND J, PRASAD P V V, BOWDEN R, FRITZ A. Mapping QTL for the traits associated with heat tolerance in wheat (L.). BMC Genetics, 2014, 15(1): 97.
[18] XU Y F, LI S S, LI L H, MA F F, FU X Y, SHI Z L, XU H X, MA P T, AN D G. QTL mapping for yield and photosynthetic related traits under different water regimes in wheat. Molecular Breeding, 2017, 37(3): 34.
[19] BHUSAL N, SHARMA P, SAREEN S, SARIAL A K. Mapping QTLs for chlorophyll content and chlorophyll fluorescence in wheat under heat stress. Biologia Plantarum, 2018, 62(4): 721-731.
[20] HASSAN F S C, SOLOUKI M, ALI FAKHERI B, NEZHAD N M, MASOUDI B. Mapping QTLs for physiological and biochemical traits related to grain yield under control and terminal heat stress conditions in bread wheat (L.). Physiology and Molecular Biology of Plants, 2018, 24(6): 1231-1243.
[21] LIU Y X, WANG R, HU Y G, CHEN J L. Genome-wide linkage mapping of quantitative trait loci for late-season physiological and agronomic traits in spring wheat under irrigated conditions. Agronomy, 2018, 8(5): 60.
[22] 杨斌, 乔玲, 赵佳佳, 武棒棒, 温宏伟, 张树伟, 郑兴卫, 郑军. 小麦旗叶叶绿素含量的QTL定位及验证. 作物学报, 2023, 49(3): 744-754.
YANG B, QIAO L, ZHAO J J, WU B B, WEN H W, ZHANG S W, ZHENG X W, ZHENG J. QTL mapping and validation of chlorophyll content of flag leaves in wheat (L.). Acta Agronomica Sinica, 2023, 49(3): 744-754. (in Chinese)
[23] LI F J, WEN W E, HE Z H, LIU J D, JIN H, CAO S H, GENG H W, YAN J, ZHANG P Z, WAN Y X, XIA X C. Genome-wide linkage mapping of yield related traits in three Chinese bread wheat populations using high-density SNP markers. Theoretical and Applied Genetics, 2018, 131(19): 1903-1924.
[24] LI F J, WEN W E, LIU J D, ZHAI S N, CAO X Y, LIU C, CHENG D G, GUO J, ZI Y, HAN R, WANG X L, LIU A F, SONG J M, LIU J J, LI H S, XIA X C. Genome-wide linkage mapping for canopy activity related traits using three RIL populations in bread wheat. Euphytica, 2021, 217(4): 1-16.
[25] LI F J, WEN W E, LIU J D, ZHANG Y, CAO S H, HE Z H, RASHEED A, JIN H, ZHANG C, YAN J, ZHANG P Z, WAN Y X, XIA X C. Genetic architecture of grain yield in bread wheat based on genome-wide association studies. BMC Plant Biology, 2019, 19(1): 168.
[26] LI H H, YE G Y, WANG J K. A modified algorithm for the improvement of composite interval mapping. Genetics, 2007, 175(1): 361-374.
[27] NYQUIST W E, BAKER R J. Estimation of heritability and prediction of selection response in plant populations. Critical Reviews in Plant Sciences, 1991, 10(3): 235-322.
[28] HOLLAND J, NYQUIST W, CERVANTES-MARTÍNEZ C T. Estimating and interpreting heritability for plant breeding: an update. Plant Breeding Reviews, 2010, 22: 9-112.
[29] INTERNATIONAL WHEAT GENOME SEQUENCING CONSORTIUM (IWGSC). A chromosome-based draft sequence of the hexaploid bread wheat () genome. Science, 2014, 345(6194): 1251788.
[30] PRITCHARD J K, STEPHENS M, ROSENBERG N A, DONNELLY P. Association mapping in structured populations. The American Journal of Human Genetics, 2000, 67(1): 170-181.
[31] BELLUNDAGI A, SINGH G P, PRABHU K V, ARORA A, JAIN N, RAMYA P, SINGH A M, SINGH P K, AHLAWAT A. Early ground cover and other physiological traits as efficient selection criteria for grain yield under moisture deficit stress conditions in wheat (L.). Indian Journal of Plant Physiology, 2013, 18(3): 277-281.
[32] SUN C W, DONG Z D, ZHAO L, REN Y, ZHANG N, CHEN F. The Wheat 660K SNP array demonstrates great potential for marker- assisted selection in polyploid wheat. Plant Biotechnology Journal, 2020, 18(6): 1354-1360.
Genome-wide association studies for canopy activity related traits and its genetic effects on yield-related traits

1Crop Research Institute, Shandong Academy of Agricultural Sciences/National Engineering Research Center of Wheat and Maize/Key Laboratory of Wheat Biology and Genetics and Breeding in Northern Huang-Huai River Plain, Ministry of Agriculture and Rural Affairs/Shandong Technology Innovation Center of Wheat/Jinan Key Laboratory of Wheat Genetic Improvement, Jinan 250100;2College of Agricultral and Biological Engineering (College of Tree Peony), Heze University, Heze 274015, Shandong;3Department of Cell Biology, Zunyi Medical University, Zunyi 563099, Guizhou;4Institute of Crop Sciences, Chinese Academy of Agricultural Sciences, Beijing 100081
【Objective】Canopy activity is an important indicator of wheat growth and development. Identification the loci for canopy activity related traits and their relationships with grain yield (GY) related traits can provide theoretical support for the dissection of genetic structure of yield trait and assisted wheat breeding.【Method】A total of 166 wheat varieties originating from both domestic and international sources were planted in Anyang of Henan province and Suixi of Anhui province in cropping seasons. With the integrated physical map containing 326 570 SNP markers from the wheat 90K and 660K chips, genome-wide association studies for normalized difference vegetation index at seedling stage (NDVI-S) and 10 days after flowering (NDVI-10), and chlorophyll content in flag leaf at 10 days after flowering (Chl-10) were carried out. The results were compared with the previous study for GY related traits using the same material. 【Result】Analysis of variance (ANOVA) showed highly significant effects (<0.01) of genotypes, environments and genotype×environment interactions on NDVI-S, NDVI-10 and Chl-10, with broad-sense heritabilities (2of 0.81, 0.81 and 0.91, respectively. Thirteen, 12 and 15 loci were detected to be significantly correlated with NDVI-S, NDVI-10 and Chl-10, respectively, among which 12, 11 and 12 were new, and five loci were associated with two or more traits. The number of favorable alleles for NDVI-S, NDVI-10 and Chl-10 ranged from 4 to 11, 3 to 11 and 4 to 12, respectively, in the 166 wheat varieties, and the phenotypic values increased with the accumulation of favorable alleles. NDVI-S showed significant (<0.01) and positive correlations with thousand-kernel weight, kernel length and kernel width. Chl-10 was significant positively correlated with GY and flag leaf width (<0.01), whereas significant negatively correlated with spike number per unit area, plant height and uppermost internode length (<0.01). Seven pleiotropic loci were detected co-related with both GY and canopy activity related traits.【Conclusion】NDVI-S can be directly used for selection of yield traits. The stable and pleiotropic loci detected in this study can be used for marker-assisted selection.
; canopy activity; grain yield; normalized difference vegetation index; chlorophyll content; genome- wide association studies

2023-07-17;
2023-09-12
山东省重点研发计划(重大科技创新工程)(2021LZGC009)、泰山产业领军人才工程(LJNY202006)、国家现代农业产业技术体系(CARS-03-6)、2023年山东省农业科学院科技创新工程(CXGC2023A01)、济南市“新高校20条”资助项目(202228067)
李法计,E-mail:lifajily@163.com。通信作者李豪圣,E-mail:lihaosheng810@163.com
(责任编辑 李莉)

