Amycolatopsis sp.的全基因组测序及香兰素合成途径分析
王冠娜,郑义培,郑璞
(江南大学 生物工程学院,工业生物技术教育部重点实验室,江苏 无锡,214122)
香兰素以游离态和葡萄糖苷的形式存在于植物及植物提取物中,如香荚兰豆、丁香油、甜菜根、秘鲁香脂、吐鲁香脂和天冬门等,其中较集中存在于香荚兰豆中[1]。纯净的香兰素具有浓郁的奶香气,有“香料之王”的美誉,被广泛应用于食品、饮料、香料和医药等领域中[2]。目前,市场上香兰素的生产方式主要包括植物提取法、微生物转化法和化学合成法[3]。植物提取法主要是从香荚兰豆中提取香兰素,但香荚兰的种植受气候的影响,其产量有限,价格昂贵,不能满足市场需求。化学合成法生产的香兰素价格低廉,但其生产过程对环境造成严重污染,而且香型单一,安全性也备受质疑,不符合下游应用市场对天然原料的消费趋势,并且欧盟(European Union,EU)和美国食品药品监督管理局(Food and Drug Administration,FDA)不允许将其用于食品[4]。天然底物经微生物转化产生的香兰素被称为“天然香兰素”。以天然阿魏酸为底物,微生物生产的香兰素在价格和品质方面都比化学合成的香兰素占有优势。其中,Amycolatopsissp.ATCC 39116[5]和Streptomycessp.strain V-1[6]用于阿魏酸发酵生产天然香兰素,产量可达10 g/L以上。
全基因组测序是对未知基因组序列的生物体进行高通量DNA测序和生物信息学组装以获得全基因组序列信息[7]。全基因组测序有助于了解菌株代谢特点,认识其遗传信息与生理功能之间的联系[8-10]。本实验室前期筛选到1株拟无枝酸菌(Amycolatopsissp.)CCTCC NO:M2011265,它可以转化阿魏酸合成香兰素[11-13];为深入了解拟无枝酸菌中参与香兰素合成的功能基因及其代谢通路信息,本文通过第二代全基因组测序技术,获得拟无枝酸菌的全基因组信息,然后根据已报道的香兰素合成的基因信息,挖掘拟无枝酸菌(Amycolatopsissp.)CCTCC NO:M2011265潜在参与香兰素生物合成的功能基因。
1 材料与方法
1.1 材料与仪器
1.1.1 材料与试剂
拟无枝酸菌(Amycolatopsissp.)CCTCC NO:M 2011265保存于中国典型培养物中心,E.coliJM109和E.coliJM110均为市售菌株,质粒pULcrRNA由中国上海师范大学生命科学学院王金教授赠予;细菌基因组DNA提取试剂盒(柱式法),宝日医生物技术(北京)有限公司;质粒小量制备试剂盒,上海捷瑞生物工程有限公司;ClonExpress Ⅱ 多片段克隆试剂盒,南京诺唯赞生物科技股份有限公司;引物合成和PCR产物测序,金唯智生物科技有限公司;天然阿魏酸,西安冠宇生物技术有限公司;安普霉素(apramycin,Apr),生工生物工程(上海)有限公司;酵母提取物、胰蛋白胨,英国OXOID有限公司;琼脂粉、牛肉浸膏、酵母浸膏、蔗糖、冰醋酸、甲醇、氯化钠,国药集团有限公司。
1.1.2 仪器与设备
5418离心机、Eporator电转仪,德国艾本德公司;HH-4恒温水浴锅,荣华仪器制造有限公司;G154DWS立式高压灭菌锅,美国致微公司;SW-CJ-1FD超净工作台,苏州安泰空气技术有限公司;Waters2487高效液相色谱仪,美国Waters公司;DYCP-31DN核酸电泳仪,北京六一仪器有限公司;Tanon-3500R凝胶成像仪,上海天能科技有限公司;SPX-150-B生化培养箱,上海博泰实验设备有限公司;HYL-C组合式摇床,太仓市强乐实验设备公司;9700A-PCR仪,南京赛飞生物科技公司。
1.1.3 培养基
斜面培养基、种子培养基、发酵培养基参见文献[12],本氏培养基、YEME培养基参见文献[13]。
1.2 实验方法
1.2.1 菌体的活化与培养
从-80 ℃冰箱取出保存菌种甘油管,放置冰上融化后用接种环沾取少量的菌液,在本氏固体培养基上划线,30 ℃培养7 d后挑取单菌落,接种至本氏液体培养基于30 ℃、220 r/min培养48 h。
1.2.2 基因组DNA提取、测序与功能注释
将斜面保存的拟无枝酸菌接种于新鲜的本氏培养基中,在30 ℃、220 r/min恒温摇床中培养28 h,获得拟无枝酸菌菌液,使用柱式细菌基因组DNA提取试剂盒提取拟无枝酸菌基因组DNA。提取好的拟无枝酸菌基因组DNA经0.8%(质量分数)琼脂糖凝胶电泳检测,用核酸检测仪测量基因组DNA的浓度,并将合格的样品委托上海伯豪生物技术有限公司进行基因组测序。使用Illumina Hiseq platform进行基因组测序、利用FastQC对样本的测序数据质量进行可视化评估,为了保证信息分析质量,对原始数据进行过滤,得到Clean数据。Reads质量的评估标准为:90%以上的碱基Q值>20。序列拼接使用SPAdes-3.5.0软件,利用软件Prokka结合Swiss-Prot库注释,分别使用KOBAS工具、rpsblast工具、和blast2GO算法,将预测的基因分别与KEGG、COG、GO数据库进行比对以注释其功能[14-17]。
1.2.3 香兰素合成与代谢基因筛选
根据已知产香兰素模式菌株Amycolatopsissp.ATCC 39116(GenBank:NZ_AFWY00000000.3)的产香兰素的关键基因,利用NCBI的BLAST工具(https://blast.ncbi.nlm.nih.gov/Blast.cgi)对拟无枝酸菌CCTCC NO:M2011265的全基因组序列进行序列相似性分析,筛选拟无枝酸菌CCTCC NO:M2011265基因组中可能参与香兰素生物合成的功能基因。
1.2.4 过表达质粒的构建
根据Amycolatopsissp.ATCC 39116的ech-fcs和ech2基因设计引物,以拟无枝酸菌CCTCC NO:M 2011265基因组作为模版,分别用引物P1(5′-GTATCTGAAAGGGGATACGCATGAGCACAGCGGTCG-GCAACGGGC-3′)/P2(5′-GCGACGTAGATGACCTGGCCTCAGCCG-AAGCGGCGGCGGACCTCG-3′)和P3(5′-TCCGCCGCCGCTTCGGCTGAGGCCAGGTCATCT-ACGTCGC-3′)/P4(5′-GGGAGATCTACTAGTACGGT-CTTGCCCTGGCTGAACC-3′)扩增获得ech-fcs和ech2片段,通过一步克隆将回收的ech-fcs、ech2片段和线性化pULcrRNA质粒按照2∶1的摩尔比,在37 ℃下反应30 min连接。以上连接产物通过热激法转化至大肠杆菌E.coliJM109感受态细胞中,37 ℃孵育45 min后涂布在对应抗性平板上,获得的转化子使用引物P5(5′-GCTGCTCCTTCGGTCGGACGT-3′)和P6(5′-CACTACTTCTCCGGGTCGA-3′)进行菌落PCR验证,将验证正确的质粒命名为pULwgn03。
1.2.5 过表达菌株的构建
将拟无枝酸菌CCTCC NO:M2011265从斜面接到新鲜的YEME培养基,30 ℃,220 r/min培养36~48 h后以5%的接种量转接至新鲜的YEME培养基,30 ℃,220 r/min过夜培养约12 h,将菌体放于冰上冰浴30 min;随后4 ℃,5 000 r/min,离心5 min收集菌体;倒掉上清液,用冰浴的电击缓冲液洗涤2~3次,然后用1~2 mL电击缓冲液重悬菌液。将菌液按每份100 μL装入1.5 mL离心管中。pULwgn03质粒转入E.coliJM110去甲基化;获得的质粒于98 ℃加热10 min后立即放在冰上冷却,与拟无枝酸菌感受细胞态轻轻混匀,冰上放置5 min,随后加入0.1 cm预冷电转杯中,放置于电转仪中,电转条件为1 800 V、5 ms。电击后立刻加入预热本氏培养基,30 ℃,220 r/min孵育约4 h。菌液在5 000 r/min的条件下离心5 min,去除上清液,将菌体重悬涂布于含有安普霉素的固体本氏培养基,30 ℃培养5~7 d,使用P5/P6引物进行菌落PCR和测序验证。
1.2.6 过表达菌株摇瓶发酵分析
阿魏酸生物转化分为两阶段法:生长阶段和转化阶段。种子液的获得:从斜面保存的过表达菌株接两环于种子培养基中30 ℃,220 r/min培养36 h左右获得种子液。按10%接种量将种子液接种于发酵培养基中,温度30 ℃,220 r/min培养16~24 h,加入终质量浓度为12 g/L的底物阿魏酸,同时温度上升至35 ℃。间隔取样,测定发酵过程中发酵液的OD600、阿魏酸和香兰素的含量。
1.2.7 分析方法
取1 mL发酵液12 000 r/min下离心5 min,稀释10倍后用0.22 μm的微孔滤膜过滤后,进行HPLC分析。色谱柱采用C18柱,进样体积为10 μL,柱温35 ℃,流动相为30%甲醇和0.1%乙酸为流动相,流速为1 mL/min,波长选择280 nm。
2 结果与分析
2.1 基因组DNA的质检
拟无枝酸菌(Amycolatopsissp.)CCTCC NO:M2011265基因组DNA经0.8%琼脂糖凝胶电泳质检(图1),结果为单一且无明显弥散的条带,表明提取基因组DNA的质量较好。如表1的结果显示拟无枝酸菌基因组的总量为0.85 μg,OD260/OD280值为1.80,说明基因组DNA无降解、无污染,达到基因组DNA测序要求,将合格的样品委托上海伯豪生物技术有限公司进行全基因组测序、拼接、注释和生物信息学分析。

表1 拟无酸菌基因组DNA浓度Table 1 Concentration of Amycolatopsis sp.genomic DNA
图1 拟无枝酸菌基因组 DNA 电泳图
2.2 拟无枝酸菌全基因组概况
拟无枝酸菌CCTCC NO:M2011265可以转化阿魏酸生成香兰素,为了更全面地了解拟无枝酸菌合成香兰素基因信息,采用从头测序技术对拟无枝酸菌菌株进行了全基因组测序分析,得到2 726 966 400 bp测序数据量,使用软件SPAdes进行初步组装后将组装结果进行比对评估,组装总共得到64个scaffolds,总长度为8 425 551 bp,其中scaffold平均长度为131 649 bp,最大scaffolds长度为716 347 bp,整个基因组组装大小约为8.43 M,总GC含量为71.89%,N50长度为363 153 bp。上述结果表明拟无枝酸菌基因组的组装质量较好,结果如表2所示。

表2 拟无枝酸菌基因组序列拼接结果Table 2 Results of genome splicing of Amycolatopsis sp.
2.3 基因的预测与功能注释
将上述得到的基因组数据分别使用blast2GO算法、rpsblast工具和KOBAS工具与GO、COG、KEGG数据库进行比对和功能注释[18-19]。结果共有8 185个基因得到注释,占基因总数的97.04%,其中,在COG数据库中得到功能注释的基因较多,为6 910个,占基因总数的84.40%。在GO和KEGG数据库中得到注释的基因分别是5 386和2 824个,分别占基因总数的65.80%和34.50%。
2.3.1 COG注释
COG数据库按照功能一共可以共分为26类,由拟无枝酸菌的COG功能注释统计结果可知,在COG数据库中一共有6 910个基因得到注释,占全部编码基因的百分比为84.40%(图2)。其中Mobilome:prophages,transposons这一分类没有被注释到,一般功能预测基因富集最为显著,达到1 065个,其次是转录类别基因注释为968个,而碳水化合物运输与代谢、氨基酸运输与代谢、能源生产和转换和脂质转运与代谢基因注释也相对较多,分别为555、554、550和447个。

图2 拟无枝酸菌预测基因的 COG 注释图
2.3.2 GO数据库
GO数据库是基因功能描述的分类系统。总基因的65.80%(5 386)在GO数据库得到了注释,注释结果由细胞组分(cellular component,)、分子功能(molecular function)和生物过程(biological process)组成。GO注释蛋白质分类显示,其中“生物过程”类别注释了4 060个基因、“分子功能”类别注释了4 159个基因和细胞组分类别注释了2 327个基因,且大部分基因被注释了多个类别,表明了拟无枝酸菌拥有较为旺盛的初级和次级代谢过程。
2.3.3 KEGG注释
将拟无枝酸菌的氨基酸序列与KEGG数据库进行BLAST比对,共有34.5%(2 824)的基因得到了注释,比对到了1 665个KO号。其中ABC 2型运输系统-ATP结合蛋白(KO1990:ABC-2 type transport system ATP-binding protein)预测到基因最多,其次是ABC 2型运输系统-通透酶蛋白(KO1992:ABC-2 type transport system permease protein)。在KEGG数据库中有占总数18.6%(1 523)的基因得到了注释,其中部分基因被注释到了多条途径,如图3所示,拟无枝酸菌基因组中基因注释最多的15条途径。其中有986个基因被注释到代谢途径(KO01100:metabolic pathways),其次为次不同环境中微生物代谢(KO01120:microbial metabolism in diverse environments),共有476个。

图3 拟无枝酸菌预测基因的部分 KEGG Pathway 注释结果
2.4 香兰素合成与代谢途径相关基因预测
根据GO、COG和KEGG数据库的注释结果,将疑似基因在NCBI数据库进行比对,挖掘与香兰素合成与代谢相关的基因。其中在GO注释结果中挖掘到了1个与香兰素合成的基因z4344,将其氨基酸序列在NCBI数据库比对,确定为阿魏酰辅酶A合成酶(fcs);在KEGG注释结果中挖掘到了1个与香兰素合成的基因z4343,将其氨基酸序列在NCBI数据库比对,确定为烯酰辅酶A醛缩酶(ech)。此外,参考其他细菌中香兰素合成代谢途径,结合3个数据库的注释结果,检索到了2个关键基因z7967和z8076,分别为烯酰辅酶A醛缩酶2(ech2)和香兰素脱氢酶(vdh)。其中,烯酰辅酶A醛缩酶2在GO数据库中被注释为:烯酰辅酶A醛缩酶活性(enoyl-CoA hydratase activity);在COG数据库中被注释为:脂质转运与代谢(enoyl-CoA hydratase activity)。香兰素脱氢酶在KEGG数据库中被注释为:苯甲醛脱氢酶(benzaldehyde dehydrogenase);在GO数据库中被注释为:醛脱氢酶活性(aldehyde dehydrogenase activity);COG数据库中被注释为:能源生产与转换(energy production and conversion)。将这4个酶与NCBI中其他来源序列进行比对,结果如表3所示。
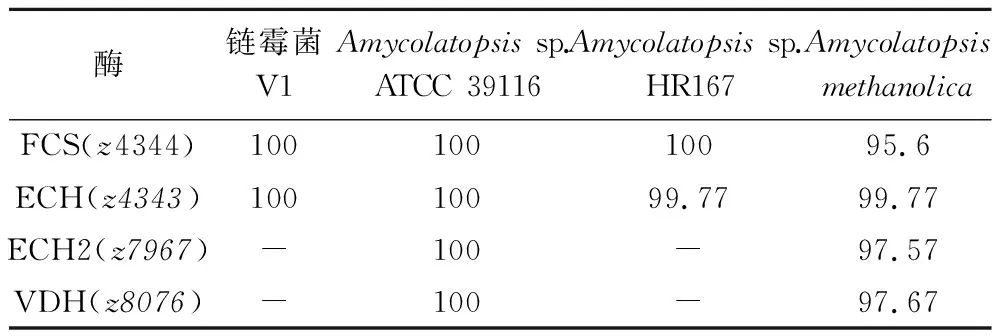
表3 相关的序列比对结果 单位:%
如表4所示香兰素合成与代谢相关酶基本信息,其中fcs基因与ech基因为同一个基因簇。此外,参考其他细菌中香兰素合成代谢途径[20],推测阿魏酸在拟无枝酸菌CCTCC NO:M2011265中是在FCS的作用下生成阿魏酰辅酶A,在ECH和ECH2的共同作用下生成香兰素,最后在VDH的作用下生成香草酸,相关的代谢反应推测如图4所示。
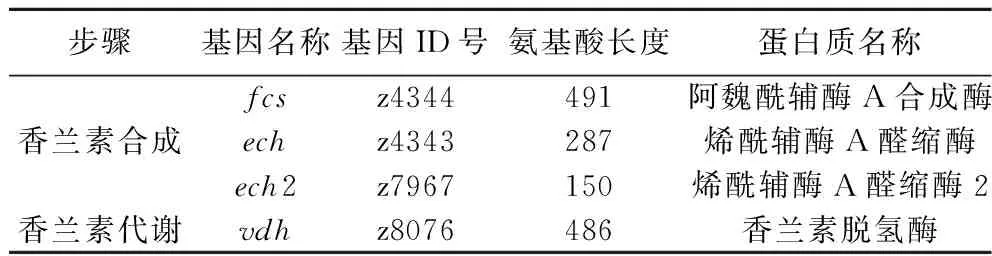
表4 香兰素合成与代谢相关的酶Table 4 Enzymes involved in vanillin synthesis and catabolism

图4 拟无枝酸菌中香兰素合成与分解代谢途径
2.5 过表达(ech-fcs-ech2)菌株转化阿魏酸生成香兰素的分析
2.5.1 过表达菌株的构建
首先以拟无枝酸基因组为模板,分别利用引物P1/P2和P3/P4扩增获得ech-fcs基因片段和ech2基因片段,如图5-a所示,ech-fcs基因片段目的条带约为2 200 bp,与实际大小2 339 bp吻合,ech2基因片段目的条带约为400 bp,与实际大小453 bp吻合,随后将这2个基因片段与线性化pULcrRNA质粒相连,成功构建出过表达质粒pULwgn03。将重组质粒pULwgn03电转入野生型拟无枝酸菌中,涂布在含有安普霉素平板上,生长5~7 d后,随机挑选10个转化子,使用引物P5/P6进行的菌落PCR扩增验证,如图5-b所示,目的条带约为3 000 bp,与实际大小2 792 bp吻合,说明ech-fcs-ech2过表达菌株构建成功。

a-ech-fcs、ech2基因的PCR产物(M-DNA marker;1-ech-fcs基因片段;2-ech2基因片段);b-转化子的PCR鉴定结果(M-DNA marker;1~10-单菌落)图5 目的基因ech-fcs、ech2和转化子的电泳图
2.5.2 过表达菌株的发酵分析
按照1.2.6节发酵方法将过表达菌株与野生型分别接入摇瓶发酵,研究过表达fcs、ech和ech2基因对菌株转化阿魏酸生成香兰素的影响。从图6-a中可以看出过表达菌株与野生型的发酵生长曲线有显著性差异,过表达菌株在48 h后,发酵生长曲线迅速下降,推测是fcs-ech-ech2的组成型表达引起了香兰素的快速合成,短时间内积累高浓度的香兰素对菌体有一定毒害作用,转化至55 h,过表达菌株底物阿魏酸基本上已经消耗殆尽了,转化停止,此时过表达菌株中发酵液香兰素质量浓度达到了11.06 g/L,而野生型菌株的底物阿魏酸残余量为6.41 g/L,香兰素的质量浓度为4.67 g/L(图6-b、图6-c)。过表达菌株代谢阿魏酸和生成香兰素的速率均比野生型快,发酵时间比野生型缩短了一半,野生型在转化94 h时香兰素产量为7.77 g/L(图6-c),过表达菌株比野生型产香兰素的浓度提高了42.30%。上述摇瓶发酵结果证明了ech、fcs、ech2基因与香兰素合成密切相关。

a-OD600;b-阿魏酸的质量浓度;c-香兰素的质量浓度图6 野生型菌株和过表达菌株对阿魏酸的转化
3 结论
本研究运用Illumina二代测序技术对拟无枝酸菌CCTCC NO:M2011265进行了全基因组的测序,获得了64个scaffolds,总长度为8 425 551 bp,GC含量71.89%,包含8 185个预测编码蛋白。同时对组装的基因组进行了基因预测、功能注释和聚类分析。最终获得的这些基础数据有益于从分子水平上认识拟无枝酸菌CCTCC NO:M2011265的生理功能、代谢特性。通过生物信息学分析得到香兰素合成相关基因ech、fcs和ech2基因,其中ech和fcs为一个基因簇,香兰素分解代谢相关基因,vdh基因,与已报道Amycolatopsissp.ATCC 39116香兰素合成与分解代谢的基因一致。构建的过表达ech-fcs-ech2菌株代谢阿魏酸和生成香兰素的速率均比野生型快,发酵时间比野生型缩短了一半。过表达菌株最终产香兰素质量浓度为11.06 g/L,野生型为7.77 g/L,比野生型产香兰素的浓度提高了42.30%,进一步证明了ech、fcs和ech2基因与香兰素生物合成密切相关。

