基于Contig的单面基因组框架填充2-近似算法
柳 楠,卞忠勇,李 洋,朱永琦
(山东建筑大学 计算机科学与技术学院,山东 济南 250101)
0 引 言
近年来,为了高效获得完整的生物基因组序列,基因测序和组装技术的发展受到广泛关注[1-2]。很多时候,使用生物测序手段无法直接获得完整的基因组序列,通过使用计算机技术将多次测序获取的基因组框架进行组装,将缺失基因填充到基因组框架,能够提高基因组序列的完整性[3-4]。
前面两问题中基因组框架的基本单元都是单个基因,对缺失基因插入位置不做限制。但实际应用中,框架多由一系列片段重叠群(contig)组成,缺失基因只允许在contig的两端插入[11]。对于基于contig的单面框架填充的研究[12],Jiang Haitao等人通过将哈密顿路径问题多项式变换到本问题,证明它是NP完全问题,然后使用贪婪策略和最大匹配提出2-近似算法[13],Tan Guanlan等人指出这个算法不具一般性,并通过构造辅助图和最大匹配设计验证了2.57-近似算法[14]。Bulteau L等人基于最大邻接数、最小断点距离提出了k-Mer参数的FPT算法[15],Ma Jingjing等人提出以duo-preservations作为度量,设计实现了近似比为2的一个基本算法,并通过贪婪策略将近似比提高到1.71[16]。
该文研究的问题是基于contig的单面含重复基因组框架填充问题,研究发现该问题的2-近似算法不能避免出现冗余公共邻接,使近似性能比无法达到2,文献[14]提出了一种解决方案,但近似性能比只能达到2.57。该文通过构造以缺失基因、基因位点、断点为节点的辅助图,配合最大匹配、贪婪策略,设计了一个近似算法,证明了算法的近似性能比可达到2,并实现了算法的验证程序。
1 相关定义
给定集合Σ,Σ包含所有用于标识基因的字符。设A是由Σ中元素构成的字符串,A中每个元素可多次出现,称A为序列。用M(A)表示A中的字符集合,由于M(A)可能是多重集,为方便计算,用NM(A)记录每个字符的重数。例如Σ={a,b,1,2},A=ab12a2,M(A)={a,a,b,1,2,2},NM(Α)={a:2,b:1,1:1,2:2}。若x和y在A中相邻,则称xy为A中的一基因对,用P(A)表示A中所有基因对的集合,P(A)也可能是多重集,用NP(A)记录基因对重数。前面例子中P(A)={ab,b1,12,2a,a2},NP(A)={ab:1,b1:1,12:1,2a:2}。易知基因对2a与a2无区别,故后面讨论中xy与yx视为相同基因对。
给定两条序列Α=a1a2…am和B=b1b2…bn,若P(A)中的一基因对aiai+1,在P(B)可以找到bkbk+1与之对应(aiai+1=bkbk+1或aiai+1=bk+1bk),称aiai+1和bkbk+1是公共邻接,用a(A,B)记录A和B全部的公共邻接,易知DA(A,B)=P(A)-a(A,B)和DB(A,B)=P(B)-a(A,B)分别表示A和B中没有匹配到公共邻接的基因对,这些基因对称为A或B中的断点。示例如图1所示。

图1 公共邻接和断点的示例
在基于contig的情况下,两条序列中一条是完整的基因序列G,另一条是待填充的基因框架S=•[C1]•[C2]•…•[Cn]•,S由多个contig序列Ci组成,contig两边的•为可插入的基因位点,称为slot。
下面正式给出基于contig的单面基因组框架填充问题(contig-based one-sided scaffold filling, Contig-One-Sided-SF-max)的定义。
定义1:Contig-One-Sided-SF-max。
输入:一条含重复基因的完整基因序列G=g1g2…gm和一条缺失部分基因的框架S=•[C1]•[C2]•…•[Cn]•,其中gi为单个基因,Ci是contig,且P(S)⊆P(G),m>n。设X=M(G)-M(S)为S中的缺失基因集合,X≠∅。
问题:将X中的基因插入S中的slot得到S',使得|a(G,S')|最大。
输出:填充后的完整序列S'。
2 Contig-One-Sided-SF-max近似算法
给定实例(G,S),研究目标是设计算法将缺失基因插入S得到S',实现公共邻接数|a(G,S')|最大化。初始公共邻接数a(G,S)由已知可直接计算,故算法关注的重点是|a(G,S')-a(G,S)|,即新增公共邻接数量。另外注意到a(G,S')-a(G,S)⊆DG(G,S),因此在算法中判断基因插入位置需要以G中的断点集合DG(G,S)(简称D)为依据,用ND记录D中每个断点的重数。新产生的公共邻接与D中的元素需一一对应,避免出现冗余公共邻接。
新产生的公共邻接根据缺失基因的插入位置的不同可以分为两类:
(1)外邻接:Ci=cistart…ciend是S中的contig,x属于S的缺失基因集合X,若xy可以构成公共邻接,且y=cistart或y=ciend,则称xy为外邻接。
(2)内邻接:除外邻接以外的其他新增公共邻接称为内邻接。
缺失基因串根据其插入S后所产生的公共邻接数量可以分为三类:
n-typeⅠ串:由n个缺失基因组成的字符串(记做Len-n串)插入S中,新增n+1个公共邻接,将该基因串称为n-typeⅠ串。显然n-typeⅠ串只能插入形如ci end]•[ci+1start的位点,该类位点记为slotⅠ。
n-typeⅡ串:Len-n串插入S中,共新增n个公共邻接,就将该基因串称为n-typeⅡ串。显然n-typeⅡ串只能插入形如•[cistart或ciend]•的位点,该类位点记为slotⅡ。
n-typeⅢ串:Len-n串插入S中,共新增n-1个公共邻接,就将该基因串称为n-typeⅢ串。
如图2所示,Len-5串deegg插入S,新增cd,de,ee,eg,gg,g4六个公共邻接,故deegg是5-typeⅠ串,c]•[4为slotⅠ型位点。Len-1串a,a,b插入S,分别得到a2,a3,ab各一个公共邻接,a2和2a记为a2,故a,a,b都是1-typeⅡ串,•[a,2]•,3]•都是slotⅡ型位点。在新增公共邻接中a2,a3,ab,cd,g4为外邻接,de,ee,eg,gg为内邻接。

图2 Contig-One-Sided-SF-max问题示例
2.1 算法核心思想框架
为方便理解,算法的大致轮廓如图3所示。

图3 算法核心思想
2.2 算法初始化
对实例(G,S)进行初始化,得到S中的缺失基因集合X和断点集合D,具体算法如算法1所示。
算法1:Init(G,S)。
输入:完整基因序列G,基因框架S
输出:缺失基因集合X,断点集合D
1X←∅,D←∅;
2 fora∈M(G) do
3 ifNM(G)[a]>NM(S)[a] anda∉X then
4 添加NM(G)[a]-NM(S)[a]个atoX;
5 forab∈P(G) do
6 ifNP(G)[ab]>NP(S)[ab] andab∉Dthen
7 添加NP(G)[ab]-NP(S)[ab]个abtoD;
8 returnXandD
2.3 算法实现
下面对算法每一步做出详细介绍:
步骤一:采用贪婪策略插入1-typeⅠ串。
对于X中的元素a,从左向右扫描框架S,发现有a可插入的slotⅠ型位点,便将a插入S,将a从X中移除一个,更新S中的基因位点并将新增的两个公共邻接从D中删除。具体算法如算法2所示。
算法2:Greedy-one(X,D,S)。
输入:缺失基因集合X,断点集合D,基因框架S=•[C1]•[C2]•…•[Cn]•
输出:更新后的缺失基因集合X,断点集合D,基因框架S
1 fora∈Xdo
2 forCi=cistart…ciend∈Sdo
3 ifi 4 (ciend≠ci+1startor (ciend=ci+1startand 5ND[cienda]≥2)) thenX←X-{a}, 6Ci←cistart…cienda,Ci=Ci∪Ci+1, 7D←D-{cienda,aci+1start}, 8ND[cienda]←ND[cienda]-1, 9ND[aci+1start]←ND[aci+1start]-1; 10 returnXandDandS 对于图2的例子,这一步的操作是将d插入到c]•[4中,与2-近似算法结果一致。 步骤二:采用最大匹配插入1-typeⅡ串。 设SⅡ={s1,s2,…,s2n}为S中slotⅡ位点集合,n等于S中contig的数量,SⅡe={e1,e2,…,e2n},ei为位点si处相邻的基因,构造集合X,D,SⅡ之间辅助图Γ,EXS记录X中元素与SⅡ中元素之间的边,ESD记录SⅡ中元素与D中元素之间的边。 构造Γ后,从中计算出匹配结果M1,M1是多个三元组(a,si,xy)的集合,根据M1来插入1-typeⅡ串和更新断点集合D。具体算法如算法3所示。 算法3:Max-match(X,D,S,SⅡ,SⅡe)。 输入:缺失基因集合X,断点集合D,基因框架S,slotⅡ位点集合SⅡ,SⅡe 输出:更新后的缺失基因集合X,断点集合D,基因框架S 1EXS←∅,ESD←∅,V←∅,E←∅; 2 newSⅡ←∅,newSⅡe←∅; 3D′←D中由X和SⅡe元素组成的断点; 4 fora∈Xdo 5 forsi∈SⅡ do 6 ifasi∈D'thenEXS[a][si]←1; 7 elseEXS[a][si]←0; 8 forsi∈SⅡ do 9 forxy∈D'do 10 ifsi=xandy∈X andEXS[y][si]=1 11 thenESD[y][si][xy]←1; 12 elseESD[y][si][xy]←0; 13V←S∪SⅡ∪D',E←EXS∪ESD; 14 求出Γ(V,E)上匹配结果M; 15 forM[i]∈Mdo 16 将M[i][0]插入到S中的M[i][1]位点, 17 newSⅡ←newSⅡ+M[i][1], 18 newSⅡe←newSⅡe+M[i][0], 19X←X-{M[i][0]},D←D-{M[i][2]}; 20 returnXandDandS 对于图2的例子,2-近似算法在这一步是将缺失基因a,a,b与slotⅡ位点做最大匹配,如图4所示,经过二分匹配后将a插入到b]•,将另一个a插入到•[b,b插入到•[a,三个基因插入后得到三个基因对ab,而断点集合中只有一个断点ab,故经过此步操作只能得到一个新增公共邻接ab。 图4 基于缺失基因、基因位点的匹配算法 文中算法在这步将缺失基因a,a,b与slotⅡ位点、断点ab,3a,2a之间做最大匹配,如图5所示,匹配结果M1={(a,s6,3a),(a,s4,2a),(b,s1,ab)},根据M1,将a插入到3]•,另一个a插入到2]•,b插入到•[a,这样便得到2a,3a,ab三个新增公共邻接。 图5 基于缺失基因、基因位点、断点的匹配算法 步骤三:采用最大匹配,在上一步更新的位点上插入内邻接。 与步骤二相似,这一步仍在缺失基因、位点和断点之间做最大匹配,SⅡ只取上一步更新得到的位点即可,调用Max-match(X,D,S,newSⅡ,newSⅡe)算法完成内邻接的插入。 对于图2的例子,X={e,e,g,g},D={a2,a2,a3,a3,ab,b1,b5,b5,cd,de,d1,d4,ee,eg,gg,g4,61},步骤二中更新的位点为•[b,a]•,a]•,显然D中与缺失基因存在边的所有断点都不能和这些位点构成边,无法构图进行匹配,故跳过此步。 步骤四:采用贪婪策略,在X中匹配内邻接对,将获得的内邻接对作为新contig置入集合S+。 对于X中的元素a,依次扫描其后面的其他元素,若有元素b,满足ab是D中的断点,则将ab作为长度为2的新contig置于S+中,并从D中移除一个断点ab。具体算法如算法4所示。 算法4:Greedy-two(X,D)。 输入:缺失基因集合X,断点集合D 输出:更新后的缺失基因集合X,断点集合D,新contig集合S+ 1i←1,S+←∅; 2 fora∈Xdo 3 forb∈X-{a} do 4 ifab∈Dthen 5Ci←ab,S+←S+∪Ci, 6i←i+1,D←D-{ab}; 7 returnXandDandS+ 对于图2的例子X={e,e,g,g},实际计算中X中元素顺序并不确定,故经过步骤四会有两种可能: (1)先得到内邻接ee,然后又得到内邻接gg,所以S+=•[ee]•[gg]•。 (2)先得到内邻接eg,剩下缺失基因只有e和g,但是D中已经没有断点eg,所以S+=•[eg]•。 步骤五:采用最大匹配在S+中插入1-typeⅡ串。 调用算法Max-match(X,D,S+,S+Ⅱ,S+Ⅱe)得到填充了1-typeⅡ串的S+,下面对步骤四中两种可能的结果继续进行讨论: (1)若步骤四中得到的S+=•[ee]•[gg]•,则X为空,此步无任何操作。 (2)若步骤四中得到的S+=•[eg]•,调用算法Max-match(X,D,S+,S+Ⅱ,S+Ⅱe),得到的匹配结果为M2={(e,s1,ee),(g,s2,gg)},填充后S+=•e[eg]g•。 步骤六:合并S和S+为S',X中的剩余基因,在不破坏现有邻接情况下,可插入S'中任意位点。 步骤七:输出S'。 可以将步骤三~步骤五合并称为匹配内邻接。在2-近似算法中也有相关操作,该算法是将插入1-typeⅠ串后的剩余所有基因(包括二分匹配后插入的基因)构造多重图,在多重图上计算最大匹配,对于图2中的例子,以{a,a,b,e,e,g,g}为顶点构造多图,断点有{a2,a3,b1,b5,b5,de,d1,ee,eg,gg,g4,61}。 如图6所示,通过构造多重图获得匹配结果是两对基因对eg和eg,而断点中只有一个eg,所以2-近似算法匹配内邻接只新增一个公共邻接,最终得到S'=•b[adb]a•a[bc]d[452]a•[6113]a•eg•eg•,总计新增ab,cd,d4,eg共4个公共邻接。在最优解S*=•b[adb]•[bc]deegg[452]a•[6113]a•中共有9个新增公共邻接,近似比为9/4,出现这种情况的原因就是在最大匹配内邻接和外邻接时,都出现了冗余公共邻接现象,从而影响了该算法的近似比。 图6 2-近似算法中多重图最大匹配 对于图2的例子,文中算法结果S'=•b[adb]•[bc]d[452]a•[6113]a•[ee]•[gg]•或S'=•b[adb]•[bc]d[452]a•[6113]a•e[eg]g•,分别新增7个和8个公共邻接,近似比小于2。 2.57-近似算法同样考虑到了避免冗余公共邻接的问题,通过在缺失基因和contig端点基因之间构造辅助图的方式消除冗余边,求出最大匹配来填充基因。但是该算法对于匹配结果与缺失基因和位点之间的对应关系没有做出判断。对于图2的例子,依据2.57-近似算法,可以得到最大匹配结果中的(a,b)这样一组匹配,但是依据(a,b)是将a插入•[b,还是将b插入a]•并没有明断判断,若是前者将导致b无法插入框架,若是后者则a和b都可以插入。由此可见2.57-近似算法虽然做到了避免冗余公共邻接,但是对于外邻接处理不佳,导致近似比大于2。 在本节,通过对比最优解中和文中算法所求近似解中各类型串新增公共邻接数,证明算法近似性能比为2。设OPT为最优解中的新增公共邻接数,OPTΙ,OPTⅡ,OPTⅢ分别为最优解中由typeⅠ,typeⅡ,typeⅢ类型基因串新增公共邻接数,可得: OPT=OPTΙ+OPTⅡ+OPTⅢ (1) 设APP为文中算法所求近似解中新增公共邻接数,APPΙ,APPⅡ,APPⅢ分别为近似解中由typeⅠ,typeⅡ,typeⅢ类型基因串新增公共邻接数,可得: APP=APPΙ+APPⅡ+APPⅢ (2) 文中提到的“新增公共邻接比”指一条或多条基因串在最优解中新增公共邻接数与在文中算法中新增公共邻接数之比。设某最优解中的Len-k串经过步骤四、五得到的内邻接数为nl(k)。 证明:假设k个缺失基因在最优解中可以组成长度为k的连续基因串,则这些缺失基因通过最大匹配可得到⎣k/2」个匹配,即⎣k/2」个内邻接。在步骤四中,使用的是贪婪策略,在最坏情况下,最大匹配中两个匹配对错位匹配,会使得两个基因无法匹配,如:断点有ac,ab,cd,缺失基因有a,b,c,d,最大匹配为{ab,cd},若ac匹配到一起,则b,d无法匹配,称ac为错位匹配。 因此,在步骤四使用贪婪策略可得到m个内邻接,m的取值范围是: k=1和2时显然成立。 综上所述,引理1成立。 证明:步骤一中使用贪婪策略插入1-typeⅠ串,在最坏情况下,一个错位的1-typeⅠ串s1,会导致一个u-typeⅠ串su变为typeⅢ,一个v-typeⅠ串sv和一个w-typeⅠ串sw变为typeⅡ,u,v,w表示各类型串的长度。 用OPT(s),APP(s)分别表示基因s在最优解和近似解中获得的公共邻接数,则s1,su,sv,sw新增公共邻接比为: u=1时,su在最优解中可以得到两个外邻接,在本算法中变成1-typeⅢ串无新增公共邻接,所以 2≤u时,su在步骤四、五得到nl(u)个内邻接,所以 1≤v,w时,sv和sw在步骤二均可获得一个外邻接。2≤v,w≤3时,sv和sw在步骤二获得外邻接后,在步骤三又均获得一个内邻接。 故1≤v,w≤3时,sv,sw新增公共邻接比如下: 4≤v,w时,sv和sw在步骤三结束后都得到两个公共邻接,剩余长度为v-2和w-2的串在步骤四、五获得的公共邻接数分别为nl(v-2),nl(w-2),所以 最优解中类型为r-typeⅠ串且没有受到错位插入影响的基因串sr,在步骤二后均可以获得两个外邻接,若r≥4在步骤三后又可以获得两个内邻接。 易知r≤7时,新增公共邻接比≤2成立。 当r≥8时,新增公共邻接比 综上所述,引理2得证。 证明:最优解中l-typeⅡ串,在步骤二获得一个外邻接,若l≥2在步骤三又可以获得一个内邻接。 易知,1≤l≤3时,新增公共邻接比≤2显然成立。 当l≥4时,新增公共邻接比 综上所述,引理3得证。 证明:由引理1易证得。 证明: (3) 文中算法的时间复杂度主要取决于在X,SⅡ,D集合元素之间求最大匹配,最坏情况下集合的X每个元素,要考虑每条与SⅡ中元素构成的无向边,接着还要考虑每条由相连的SⅡ中元素与D中元素构成的无向边,这需要O(|X||SΙΙ||D|)时间。所以,算法时间复杂度为O(nml),其中n,m,l分别指缺失基因个数、两倍的contig数量以及断点个数。 完成Contig-One-sided-SF-max问题的多项式时间近似算法后,用Python语言实现了该算法,并编写了具有可视化界面的Contig-One-sided-SF-max问题的程序,增强人机交互体验。 程序主界面如图7所示。 用户在程序输入框中输入G和S,点击确定,程序将根据文中设计的算法自动完成基因组框架填充,并给出填充结果和新增公共邻接总数。 程序的执行如图8所示。 图8 程序运行界面 该文重新审视了基于contig的单面含重复基因的基因组框架填充问题。在设计算法时以缺失基因、基因位点、断点的对应关系为依据(而不像以往研究中,只关注于其中两个之间的对应),解决了2-近似算法的冗余公共邻接问题,又解决了2.57-近似算法精确度较低的问题。对于近似比的优化方面,还需要进一步研究,希望能够吸引更多人共同学习来完善这一问题。


2.4 算法的性质









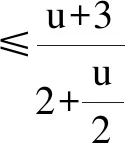










3 算法的程序实现

4 结束语

