茶树OSCA基因家族的鉴定及表达分析
刘丹丹 吴琼 焦小雨 孙明慧 王文杰






关键词:茶树;OSCA基因家族;Ca2+通道;全基因组表达分析
茶树[Camelliasinensis(L.)O.Kuntze]是一种多年生常绿木本植物[1],喜漫射光、温暖潮湿的气候以及弱酸性土壤[2]。低温、干旱、长期辐射、病虫害等非生物胁迫和生物胁迫都会对茶树生长产生不利影响[3-4]。其中干旱胁迫通过影响茶树生长的土壤和水分条件,严重降低茶叶的产量和品质[5]。研究人员对9个不同品种13a树龄的茶树进行40d干旱处理,茶叶产量下降27.27%~68.53%[6]。同时,咖啡因、儿茶素、茶氨酸和游离氨基酸等代谢物也显著较少[1],从而降低茶叶品质。因而研究茶树是如何特异感受干旱胁迫产生信号的分子机理,挖掘相关的抗旱基因,进而通过分子育种技术获得抗性优良品种具有重要的研究意义和实用价值。
钙通透性阳离子通道蛋白(hyperosmdalitygatecalcium-permeablechannels,OSCA),是一种Ca2+机械敏感通道[7],是目前发现的第一个植物高渗胁迫感受蛋白[8]。OSCA基因于2014年在拟南芥中首次报道。研究人员用山梨糖醇模拟干旱条件处理表达了水母发光蛋白的拟南芥植株后,利用Ca2+成像技术发现osca1突变体胞内钙离子相比于野生型释放量显著减少,然而用不具有渗透胁迫的Ca2+诱导剂H2O2处理时,突变体与野生型之间并无明显差异,表明OSCA1是特异性渗透胁迫感受器[8]。OSCA1会在拟南芥的叶、花、根以及保卫细胞等处表达,当进行高渗处理时,突变体的气孔开度不会减小,根系生长受到抑制,也说明了OSCA1对于渗透感受的重要性[8]。拟南芥osca1突变体中OsOSCA1.4的过表达补充了Ca2+信号传导、根生长和气孔运动的缺陷,以响应高渗和盐胁迫[9]。在高渗环境下,AtOSCA1.2通过增加细胞内的Ca2+浓度,增强对Na+和K+的通透性[10]。植物感知生物和非生物胁迫后往往会引起气孔的关闭[11],钙通过质膜的快速流入在这种反应中起着重要作用[12]。THOR等[13]报道了拟南芥Ca2+渗透通道AtOSCA1.3在免疫信号转导过程中控制气孔关闭,然而AtOSCA1.3并不调节脱落酸诱导的气孔关闭。此外,OSCA作为一种Ca2+非选择性陽离子通道蛋白,在植物生长发育过程中发挥着重要作用。有研究指出,OSCA基因家族在植物花中特异表达[14],参与渗透胁迫下的植物花粉的萌发和花粉管的发育[3,15-16]。总之,OSCA家族成员在感受高渗胁迫上是保守的,但在诱导植物产生的抗逆途径上又有区别。
OSCA基因家族对植物生长发育和响应逆境胁迫都至关重要,并已经在拟南芥[8]、烟草[16]、水稻[14]、小麦[17]等物种中做了系统的鉴定与功能分析。然而,目前关于茶树OSCA基因家族相关研究暂未见报道。因此,我们对茶树OSCA家族成员进行了全基因组鉴定与系统发育关系分析,并分析了不同组织和干旱胁迫下的表达情况。本课题的开展为后续深入解析茶树OSCA基因在干旱环境应答中生物学功能和分子机制提供重要参考,有助于进一步研究植物渗透胁迫钙信号通路,为茶树抗性育种提供新的思路。
1材料与方法
1.1材料
干旱胁迫材料:选取安徽省农业科学院茶叶研究所苗圃中生长良好、长势一致的1年生茶树品种舒茶早茶苗,于人工气候室(24±2)℃的环境温度下稳定生长,转移至PEG-6000溶液(浓度为20%)中进行干旱处理,收集0、15、30min和1、2、4、8、12、24、48h后的第二叶。
组织样本:7月份安徽省农业科学院茶叶研究所全国第五轮区试园所在的屯溪区降水量少,茶园土壤干燥,茶农98(CN98)品种已出现明显的干旱胁迫症状,崂山3号(LS3)和中黄1号(ZH1)未出现明显的干旱胁迫症状,此外结合区试园中多年抗旱性观测数据,将此次实验样品LS3和ZH1定义为耐旱型品种,CN98定义为干旱敏感型品种。选取上述3个茶树品种(LS3、ZH1和CN98)的成熟叶第二叶,取样时间为2022年8月3日上午9点。以上每种处理均设置3次重复,样本采集后,置于液氮速冻,转移至–80℃冰箱备用。
1.2方法
1.2.1茶树OSCA基因家族成员鉴定与染色体定位分析从拟南芥基因组数据库(https://www.arabidopsis.org/tools/bulk/sequences/index.jsp)下载拟南芥OSCA基因家族蛋白序列及其功能分类信息。根据拟南芥OSCA基因家族的蛋白序列在茶树基因组数据库(http://tpia.teaplant.org)在线Blast比对茶树原变种(C.sinensisvar.sinensis)舒茶早品种的最新版基因组蛋白序列,根据比对结果下载候选序列蛋白并去冗余。为了进一步验证所鉴定的茶树OSCA基因家族,使用NCBI保守结构域数据库(www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)对所有检索到的序列进行筛选,以确定蛋白质是否含有DUF221结构域,最终得到茶树OSCA基因家族成员12个。为了研究CsOSCA蛋白质的性质,使用在线的ExPASy-ProtParam工具(web.expasy.org/protparam/)预测分子量(MW)和等电点(pI),利用Softberry(http://linux1.softberry.com/all.htm)中的ProtComp程序预测上述候选OSCA蛋白的亚细胞定位。从茶树的基因注释文件(GFF3)中检索CsOSCAs基因的信息,利用Tbtools(v1.09867)软件中的GeneLocationVisualizefromGTF/GFF进行染色体定位可视化绘图。
1.2.2茶树OSCA基因家族蛋白二级结构、保守基序、基因结构分析使用Phyre2(http://www.sbg.bio.ic.ac.uk/~phyre2/html/page.cgi?id=index)在线工具分析CsOSCA蛋白二级结构和跨膜结构域。另外,使用MEME(http://memesuite.org/tools/meme)在线分析CsOSCAs蛋白的保守基序,基序数目设置为20,其他参数设置为默认值。利用Pfam数据库(http://pfam.xfam.org/search#tabview=tab1)搜索CsOSCA基因家族保守Pfam得到的结果文件用TBTools软件进行可视化绘图。在茶树基因注释(GFF3)文件中检索CsOSCAs基因的外显子-内含子结构信息,导入在线程序GSDS2.0(http://gsds.cbi.pku.edu.cn/)比较各自的全长序列结构并生成图表。
1.2.3茶树OSCA基因家族系统进化树分析根据文献信息下载拟南芥[8]和水稻[14]OSCA基因家族蛋白序列,使用MEGA7(https://itol.embl.de/)在线软件中的ClustalW方法对茶树、拟南芥和水稻OSCA蛋白序列进行比对,使用邻接法(neighbor-joiningmethod)构建系统进化树。
1.2.4茶树OSCA基因家族启动子顺式作用元件与反式作用因子预测分析为了分析OSCA基因家族启动子序列中可能存在的顺式元件,使用TBTools软件批量提取OSCA基因家族成员ATG上游的1500bp的启动子序列区域,将上一步获得的启动子序列放进PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)在线网站分析启动子的顺式作用元件,根据返回结果tab文件筛选统计与逆境胁迫相关的顺式元件,使用Tbtools软件的ImpleBioSequenceViewer功能绘图。
CsOSCAs基因啟动子可能结合的反式作用因子预测以拟南芥为参考,在JASPAR(https://jaspar.genereg.net/)数据库的Plantae模块中选择与干旱胁迫相关的转录因子家族AP2/EREDP、bZIP、DOF、MYB和WRKY成员共计181个转录因子,添加到购物车,输入ATG上游的1500bp的启动子序列区域,Relativeprofilescorethreshold选择95%,根据预测结果统计出相对分数大的转录因子。
1.2.5茶树OSCA基因家族组织特异性分析在NCBI中下载茶树在不同组织的RNA-seq数据(登录号:PRJNA79643)。使用TBtools(v1.082)软件中HeatMap对各转录组基因的FPKM值进行归一化处理并进行表达模式可视化分析。
1.2.6茶树RNA的提取和实时荧光定量PCR用TBtools(v1.082)软件提取已下载的茶树基因组数据库中茶树OSCA基因家族的CDS序列,并通过NCBI(https://www.ncbi.nlm.nih.gov/tools/primer-blast/index.cgi)在线网站进行引物设计(表1)。选用茶树β-actin(登录号:KJ946252)作为内参基因。采用多糖多酚植物总RNA提取试剂盒[天根生化科技(北京)有限公司]提取上述处理的总RNA,电泳检测完整性。使用PrimeScriptTMRTMasterMix(perfectrealtime)试剂盒合成cDNA用于实时荧光定量PCR。利用LightCycle®96荧光定量PCR仪[罗氏诊断产品(上海)有限公司]进行qRT-PCR,反应程序为:94℃30s;94℃5s,60℃30s,40个循环;反应体系参照SYBRGreenProTaqHSqPCRKit试剂盒(湖南艾科瑞生物工程有限公司),反应结束后分析荧光值变化曲线及熔解曲线,每个样品均设置3次技术重复。
1.3数据处理
用2‒ΔΔCT算法计算基因相对表达水平,使用SPSS17.0软件对数据进行差异显著性分析,使用GraphPadprism5软件绘图。
2结果与分析
2.1茶树OSCA基因家族成员的鉴定与染色体定位
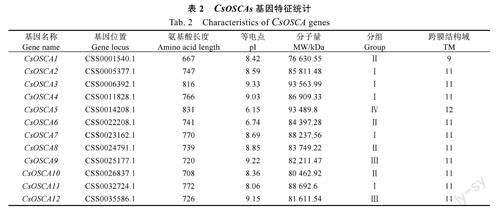
经过与拟南芥OSCA基因家族蛋白序列比对去冗余,蛋白结构域分析验证,最终获得12个OSCA基因家族成员(表2)。CsOSCAs基因编码的氨基酸序列长度为667~831bp。蛋白分子量在76630.55~93563.99kDa之间,等电点在6.15~9.33之间,含有9~12个跨膜结构域。通过与拟南芥基因家族成员进行比对,按照茶树基因组编号顺序将其分别命名为CsOSCA1~CsOSCA12。
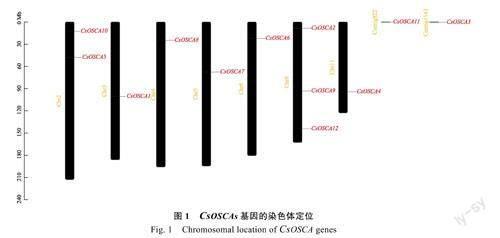
根据茶树基因组信息,茶树OSCA基因家族在染色体上的分布均匀(图1),10个CsOSCAs基因定位于茶树7条染色体上,2个OSCA基因定位于未锚定染色体的contig上,其中8号染色体上有3个OSCA基因,数量最多。
2.2茶树OSCA基因家族蛋白的二级结构、保守基序、基因结构分析
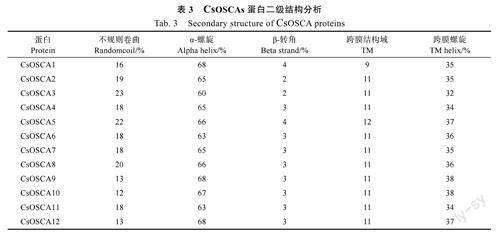
蛋白质二级结构的预测结果显示,茶树OSCA基因编码的蛋白二级结构含有大量的跨膜结构和α-螺旋(表3),α-螺旋的比例在60%~68%之间,跨膜螺旋在32%~38%之间,另外还含有12%~23%的不规则卷曲以及少量的β-转角。
为了进一步分析茶树OSCA家族基因的保守性,分析其保守序列和保守基序。如图2A所示,茶树OSCA基因家族蛋白均含有3个保守结构域,分别是lateexocytosis(RSN1_TM,Pfam:PF13967)、cytosolicdomainof10TMputativephosphatetransporter(DUF4463,PHM7_cyt,Pfam:PF14073)和calciumdependentchannel(DUF221,RSN1_7TM,Pfam:PF02714)。保守基序结果显示,除了CsOSCA5以外,其他CsOSCA序列均高度保守,motif1、motif4、motif9属于PHM7_cyt结构域,motif2、motif3、motif5、motif12构成RSN1_7TM结构域,motif6、motif8、motif11、motif15、motif17构成RSN1_TM。CsOSCA2、CsOSCA3、CsOSCA4、CsOSCA7和CsOSCA11蛋白基序组成模式相似,CsOSCA9和CsOSCA12保守基序组成相似,CsOSCA1、CsOSCA8和CsOSCA10保守基序组成相似(图2B)。
根据茶树OSCA基因家族结构绘制的图谱(图3)可以看出,CsOSCA11基因的序列最长,12个CsOSCAs基因外显子数量范围在1~11个之间,其中CsOSCA2、CsOSCA3、CsOSCA4、CsOSCA7、CsOSCA8和CsOSCA11的外显子数量为11个,CsOSCA6、CsOSCA10的外显子数量为10个,CsOSCA1的外显子数量为9个,CsOSCA9和CsOSCA12外显子数量均为6个,CsOSCA5的外显子数量为1个。
2.3茶树OSCA基因家族蛋白系统进化树分析
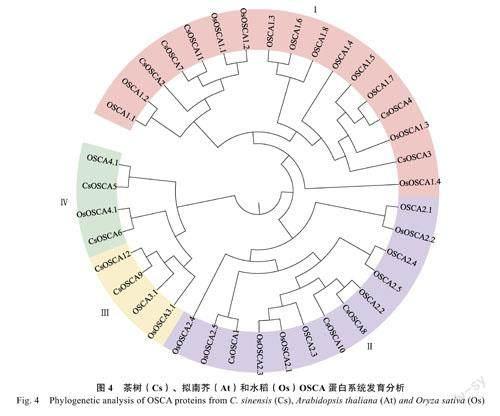
利用拟南芥(15个)、水稻(11个)与茶树(12个)OSCA家族蛋白全长序列构建系统发育树(图4)。根据系统进化关系,这38个OSCA蛋白可分为4个亚族,分别命名为Ⅰ~Ⅳ。茶树、拟南芥和水稻在4个亚族中均有分布。茶树OSCA基因家族中有5个明显的垂直同源基因对,分别是:Ⅰ亚族的CsOSCA4和拟南芥OSCA1.7,Ⅱ亚族的CsOSCA8和拟南芥OSCA2.2、CsOSCA1和水稻OsOSCA2.5,Ⅳ亚族的CsOSCA6和水稻OsOSCA4.1,CsOSCA5和拟南芥OSCA4.1。
2.4茶树OSCA基因家族组织特异性表达
通过分析芽(bud)、花(flower)、果实(fruit)、嫩叶(youngleaf)、成熟叶(matureleaf)、老叶(oldleaf)、根(root)和茎(stem)8个茶树组织转录组数据[18],绘制茶树OSCA家族在茶树不同组织中的表达模式热图(图5)。CsOSCA3、CsOSCA6和CsOSCA7在芽中表达量较高;CsOSCA1、CsOSCA4、CsOSCA9在花中表达量较高,在其他组织中含量很低或不表达;CsOSCA2、CsOSCA6、CsOSCA7在根中的表达量较高;CsOSCA8在老叶中表达量较高;CsOSCA10在成熟叶、老叶和茎中表达量较高;CsOSCA11在茎中表达量较高,在其他组织中表达量较低;CsOSCA12在嫩叶中表达量最高。
2.5CsOSCAs基因在不同抗旱品种間的表达分析
为了研究CsOSCA基因家族成员在不同抗旱品种间的表达情况,选择抗旱较好的LS3、ZH1品种和抗旱较差的CN98品种为实验材料,对茶树OSCA基因家族进行实时荧光定量检测。结果显示,CsOSCA2、CsOSCA3、CsOSCA11、CsOSCA12基因在干旱敏感的品种CN98中的含量显著高于其他2个品种(图6)。
2.6CsOSCAs基因在干旱胁迫下的表达分析
为了明确茶树OSCA基因家族成员对干旱胁迫的响应规律,使用20%PEG-6000模拟干旱胁迫处理1年生舒茶早茶苗,利用qRT-PCR分析CsOSCAs基因的表达情况。如图7所示,20%PEG-6000处理下,1h内CsOSCA3、CsOSCA5、CsOSCA8、CsOSCA10、CsOSCA11、CsOSCA12表达量明显上调,其中CsOSCA3、CsOSCA5、CsOSCA8、CsOSCA10、CsOSCA12基因在处理15min时表达量显著增加,此时CsOSCA10基因相对表达量是对照的3.43倍。CsOSCA2在24h表达量最高,是对照的3.92倍。CsOSCA3和CsOSCA12表达模式相似,在12h表达量较高,是对照的2.68倍和4.82倍,而后表达量降低。CsOSCA5、CsOSCA8和CsOSCA10在PEG-6000处理后一直维持比较高的表达量。
2.7茶树OSCA基因家族启动子顺式元件分析与上游调控转录因子预测
为了研究CsOSCAs基因对各种信号因子的应答作用,对CsOSCAs基因启动子上游1500bp序列进行在线分析,发现CsOSCAs上游启动子存在光周期调控(photoperiodcregulation)、植物激素(hormones)、生物胁迫(bioticstress)和非生物胁迫(abioticstress)相关的元件(图8)。光周期调控相关元件包括3-AF1bindingsite、ACE、AE-box、GA-motif、G-box等19种。响应植物激素相关的元件主要有ABRE、ARE响应脱落酸;CGTCA-motif、TGACG-motif响应茉莉酸甲酯;GARE-motif、P-box、TATC-box响应赤霉素;TCA-element响应水杨酸;TGA-element响应生长素。与生物胁迫相关的元件有:GC-motif参与缺氧特异性诱导;LTR响应低温;MBS干旱诱导;STRE、TCA响应胁迫诱导。与非生物胁迫相关的元件有AT-richelement、AT-richsequence、TC-richrepeats响应防御与胁迫;Wbox、WRE3、WUN-motif响应伤害。在CsOSCAs中含有最多的顺式作用元件是ARE脱落酸响应元件,大部分的基因都存在Box4、G-box、GT1-motif等光响应元件,6个CsOSCA含有干旱诱导响应元件。
启动子区域一般富含丰富的顺式作用元件,通过与反式作用因子结合来调控下游基因的表达。根据干旱胁迫下CsOSCA基因家族成员的响应规律,对干旱胁迫响应强烈的CsOSCA2、CsOSCA3、CsOSCA5、CsOSCA8、CsOSCA10、CsOSCA11、CsOSCA12基因上游启动子序列可能结合的转录因子进行预测,并统计出共同的上游调控转录因子(relativescore>0.95)(表4)。结果显示,DOF家族成员AtDOF2.4、AtDOF5.3、AtDOF5.6和AtWRKY40能同时与这7个基因的启动子结合。
3讨论
本研究利用茶树全基因组测序数据信息全面分析OSCA基因家族,共挖掘12个CsOSCAs基因。根据系统进化关系,茶树OSCA基因家族可划分为4个亚族,与小麦[17]、玉米[19]、烟草[16]、绿豆[20]和梨[3]等物种研究结果一致。拟南芥中脱水早期反应蛋白4(early-responsivetodehydration,ERD4;即AtOSCA3.1)在受到干旱胁迫时会特异性启动转录[21]。本研究中,CsOSCA9和CsOSCA12与AtOSCA3.1同属III亚族,它们是否参与茶树的干旱胁迫响应机制,还需进一步研究。
亚细胞定位发现,茶树OSCA基因家族成员均定位在质膜上,与大豆[22]OSCA基因家族成员相一致。研究表明,OSCAs蛋白序列中存在11个TMs[7,23-25],然而本研究中CsOSCA1和CsOSCA5分别包含9个和12个TMs,推测他们在进化过程中可能具有更大的遗传变异。CsOSCA基因家族同一亚族成员外显子数量相近,而CsOSCA5外显子数量只有一个,在水稻[14]和番茄[26]中也存在这种单个外显子的情况。DUF221结构域在CsOSCA基因家族中高度保守,含有7个钙通道跨膜结构域,命名为RSN1_7TM[10]。在分析CsOSCA基因家族成员蛋白结构时发现,茶树OSCA基因家族成员均包含DUF221结构域,但是CsOSCA5有2段DUF221结构域,保守基序分析也发现CsOSCA5包含的保守基序比其他基因更少。本研究中,CsOSCA5与AtOSCA4.1为垂直同源基因对,且CsOSCA5基因对PEG胁迫响应明显,可能CsOSCA5中的抗逆功能在进化过程中被保留了下来。
本研究中,CsOSCAs基因对PEG-6000诱导的干旱胁迫有响应,这与拟南芥[8]、水稻[14]、玉米[19]、小麦[17]和绿豆[20]等植物中的研究结果一致。在渗透胁迫下,CsOSCA2、CsOSCA3、CsOSCA5、CsOSCA8、CsOSCA10和CsOSCA12基因显著上调,除了CsOSCA2以外,其他5个基因在15min内均显著上调,推测与Ca2+响应速度快有关,其他植物如水稻[14]、番茄[26]、玉米[27]和大豆[22]OSCA基因家族对干旱胁迫的响应也出现在早期。此外,本研究分析了不同耐旱品种间CsOSCA基因家族成员在夏季干旱胁迫下的表达差异,发现CsOSCA2、CsOSCA3、CsOSCA11、CsOSCA12基因在干旱敏感型的茶农98中的表达量显著高于其他2个抗旱品种,推测这些基因可能在茶农98茶树中被诱导表达参与茶树的抗旱反应。CsOSCA2、CsOSCA3、CsOSCA12不仅在PEG处理下诱导表达,在不同品种间表达也存在显著差异,因此CsOSCA2、CsOSCA3、CsOSCA12可能是茶树响应干旱胁迫的关键候选基因。
前人研究结果[14,26]指出OSCA基因对ABA胁迫反应强烈。本研究对12个CsOSCA基因启动子分析显示,其中11个基因启动子含有ARE脱落酸响应元件,这些基因是否响应ABA胁迫以及干旱胁迫中的ABA信号转导值得进一步研究。CsOSCA6、CsOSCA7、CsOSCA9、CsOSCA10、CsOSCA11和CsOSCA12启动子包含干旱诱导响应元件,然而在PEG胁迫下只有CsOSCA10、CsOSCA11和CsOSCA12显著上调。推测CsOSCA10、CsOSCA11和CsOSCA12在茶树响应干旱胁迫过程中发挥着主要作用。转录因子预测结果显示DOF家族成员AtDOF2.4、AtDOF5.3、AtDOF5.6和AtWRKY40可以多数CsOSCAs基因的启动子结合。有研究指出大部分茶树DOFs转录因子家族对干旱胁迫有响应[28-29],推测CsDOFs转录因子可能参与调控CsOSCA基因家族响应干旱胁迫产生的信号感受与传导,目前本课题组正在对其互作关系进行验证。

