加权基因共表达网络分析联合机器学习法筛选缺血性心肌病生物标记及免疫浸润分析
尤红俊 苟棋玲 赵倩倩 董梦雅

摘要目的:利用加权基因共表达网络分析(WGCNA)联合机器学习法筛选缺血性心肌病(ICM)的生物标记,并进行免疫浸润分析。方法:对来自基因表达综合数据库(GEO)的ICM转录谱进行差异分析。对差异表达基因(DEGs)进行基因本体(GO)富集分析和京都基因与基因组百科全书(KEGG)通路富集分析。整合WGCNA和套索回归(LASSO)分析,筛选出ICM的生物标记。绘制受试者工作特征曲线(ROC)评估其诊断效能,并进行免疫浸润分析。结果:ICM左心室组织中有517个DEGs,主要参与细胞外基质构成、含胶原蛋白的细胞外基质等;同时,DEGs参与磷脂酰肌醇3激酶/蛋白激酶B(PI3K/AKT)信号通路、丝裂原活化蛋白激酶(MAPK)信号通路、内质网中的蛋白质加工、酪氨酸激酶/信号传导子及转录激活因子(JAK/STAT)信号通路、糖尿病并发症中的晚期糖基化终产物/晚期糖基化终产物受体(AGE/RAGE)信号通路、细胞凋亡、缺氧诱导因子1(HIF-1)信号通路等。鉴定出的生物标记无孢蛋白(ASPN)和高温需求丝氨酸肽酶1(HTRA1)具备良好的诊断效能。T淋巴细胞介导的免疫反应在ICM的发生发展中发挥重要作用,并与ASPN和HTRA1显著相关。结论:ASPN和HTRA1可作为ICM的生物标记,并与T淋巴细胞显著相关,在ICM的发病机制中发挥重要作用。
关键词缺血性心肌病;加权基因共表达网络分析;机器学习;生物标记;免疫浸润
doi:10.12102/j.issn.1672-1349.2023.23.005
缺血性心肌病(ischemic cardiomyopathy,ICM)是指因冠状动脉病变引起的左心室收缩功能障碍,是心力衰竭常见的病因之一。冠心病的全球大流行造成多达2 600万人受到心力衰竭的影响,全球卫生系统损失超过300亿美元[1-2]。ICM是一种慢性免疫系统激活状态,免疫系统的失调、心肌梗死后的炎症反应及纤维化的过度活跃可导致心肌梗死后心肌的不良重塑,从而导致心力衰竭的发生[3]。炎症和免疫反应的治疗性调节可能为预防梗死后心力衰竭带来希望,因而开发精准的生物标记,并从免疫-炎症角度探索其致病机制,对提高ICM的诊治水平有着积极的临床意义。
加权基因共表达网络分析(weighted gene co-expression network analysis,WGCNA)是一种常用的系统生物信息学方法,不仅用于构建基因网络,还用于检测基因模块和识别模块中的核心基因和/或分子标志物。WGCNA适用于复杂的多样本转录组数据,通過合适的加权系数对基因间的相关系数进行加权运算,使得基因网络近似服从无尺度网络分布,从而具有更好的统计性能,避免了由大量的多重校正导致的假阴性结果[4]。研究表明,WGCNA比未加权的网络具有更稳健的结果[5]。WGCNA不是将单个基因与表型联系起来,而是关注少数模块与性状之间的关系,缓解了微阵列或测序数据分析中固有的多重检验问题,可更准确地筛选出与疾病关联密切的特定模块及模块内基因[6]。
本研究通过对基因表达综合数据库(gene expression omnibus,GEO)中大样本量的ICM病人和健康对照者左心室组织转录谱芯片数据进行生物信息学分析,包括运用R语言筛选出差异表达基因(differentially expressed genes,DEGs),并进行基因本体(Gene Ontology,GO)和京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)通路富集分析,探讨DEGs在ICM发生发展中的作用。同时,进行WGCNA分析,计算基因之间的相关性,构建加权基因共表达网络,与临床表型相结合进一步分析基因与临床表型之间的关系,筛选出ICM候选分子标志物,并整合机器学习法套索回归(least absolute shrinkage and selection operator,LASSO)分析,提高分子标志物鉴定的准确性,再进行免疫浸润分析,探索鉴定的分子标志物与免疫细胞关联性,从而揭示ICM免疫相关分子机制,为临床诊疗提供更多理论依据。
1资料与方法
1.1数据下载及预处理
在GEO数据库[7-8]中搜索“ischemic cardiomyopathy”,获取数据集GSE5406、GSE42955和GSE57338。剔除非ICM病人,保留ICM病人和健康对照的左心室心肌组织样本,样本量及资源贡献见表1。用“sva”包合并GSE5406和GSE42955,作为训练集,GSE57338作为验证集。
表1数据集信息数据集平台样本量(例)贡献者GSE5406(训练集)GPL96ICM(108);健康对照(16)Cappola T P,Margulies K B,Putt M E,et alGSE42955(训练集)GPL6244ICM(12);健康对照(5)Molina-Navarro M M,Roselló-Lletí E,Tarazón E,et alGSE57338(验证集)GPL11532ICM(136);健康对照(95)Liu Y,Morley M P,Brandimarto J,et al
1.2差异表达分析
采用统计软件R4.1.1中limma包进行基因差异表达分析。以|fold change(FC)|>1.2及P<0.05为差异表达基因的筛选条件,并以R语言ggplot2包和pheatmap包绘制火山图和热图可视化DEGs。
1.3DEGs富集分析
利于R语音ClusterProfiler包、org.Hs.eg.db包、enrichplot包等对DEGs进行GO和KEGG分析[9-10]。GO分析分别在生物学过程(biological processes,BP)、细胞组分(cellular component,CC)和分子功能(molecular function,MF)3个方面对基因进行功能注释[11]。并利用R语言ggplot2包将结果可视化。P<0.05被认为差异有统计学意义。
1.4WGCNA
采用R包WGCNA进行WGCNA[5],并使用“pickSoftThreshold”函数分析1~20的参数值。构建权重基因网络,选择最佳软阈值,既保证网络接近无尺度分布,又保证网络平均连通性不会太小,利于网络包含足够信息以挖掘模块。在层次聚类树中,为了保证模块的可靠性,将基因集的最小数目设定为25个。在构建模块之后,设定合并阈值为0.25,经动态分支切割得到若干合并后的模块。
1.5关键模块的鉴定
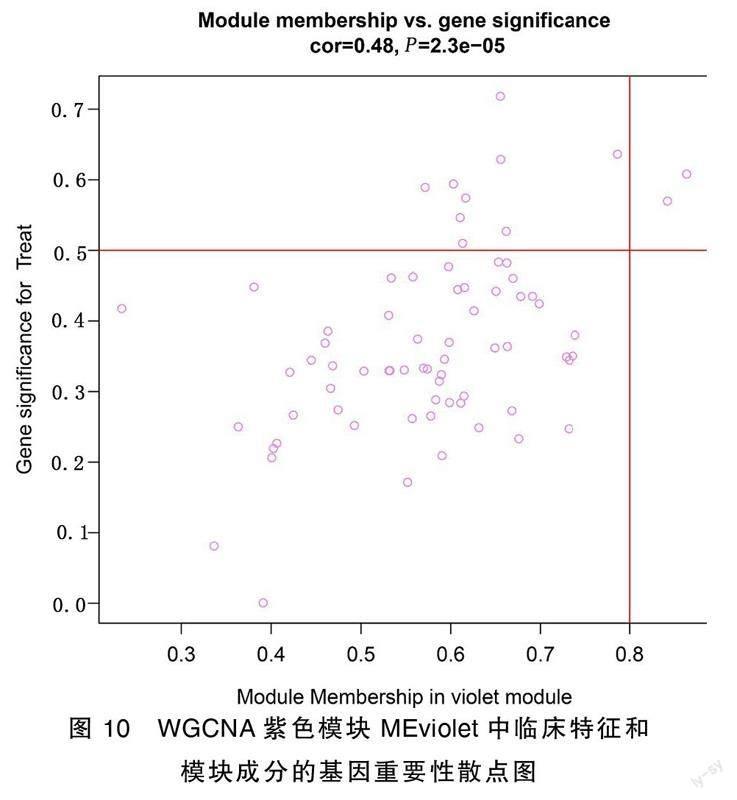
为了鉴定与临床性状显著相关的关键模块,分析临床性状与模块特征基因之间的相关性,并以热图展示。与ICM呈最正相关的模块被确定为关键模块。基因显著性(gene significance,GS)为基因表达与每个临床特征本身的相关性。模块隶属度(module membership,MM)为基因表达与模块之间的关系。为了验证模块-性状的关联,采用Pearson相关分析检验GS与MM的相关性。
1.6核心基因的鉴定
按基因重要性(过滤条件设定为>0.5)和基因与模块相关性(过滤条件设定为>0.8)筛选关键模块内的关键基因,再与DEGs绘制韦恩图,获得交集基因,最后进行LASSO分析获得核心基因。LASSO是一种具有特征选择功能的回归分析模型,属于机器学习方法之一。LASSO回归算法通过R语言中的glmnet包实现,用以筛选区分疾病和正常的生物标记。
1.7核心基因诊断效能评估
比较训练集和验证集中核心基因的表达水平来检测核心基因作为生物标记的准确性和可靠性,并绘制箱线图。利用pROC包在训练集和验证集中分别绘制受试者工作特征曲线(receiver operator characteristic curve,ROC),并计算曲线下面积(area under the curve,AUC)评估核心基因作为生物标记区分疾病或正常的诊断效能。
1.8免疫浸润分析
单样本基因集富集分析(single sample gene set enrichment analysis,ssGSEA)[12]根据表达谱计算每个基因的rank值,统计分析得到每个样本的免疫细胞的相对含量,有助于了解疾病状态下组织中所有免疫细胞的浸润情况,并探究其与疾病转归的关系。采用ssGSEA算法分析ICM中28种免疫细胞在免疫浸润微环境中的比例。并将核心基因与免疫細胞进行相关性分析。采用R语言pheatmap包和vioplot包绘制热图和小提琴图以可视化。
1.9统计学处理
采用R语言进行统计分析,以P<0.05为差异有统计学意义。
2结果
2.1基因差异表达分析
合并GSE5406和GSE42955为训练集后,共筛选出517个DEGs,其中220个上调,297个下调。对DEGs绘制可视化火山图,详见图1。取差异显著前100个基因绘制热图,详见图2。
2.2DEGs富集分析
GO富集分析提示DEGs主要参与对折叠蛋白的应答、对拓扑错误蛋白质的应答、细胞外基质构成、含胶原蛋白的细胞外基质、纤维胶原蛋白三聚体、带状胶原纤维、细胞外基质结构成分赋予抗拉强度等,详见图3。DEGs参与磷脂酰肌醇3激酶/蛋白激酶B(phosphatidylinositol 3 kinase/AKT,PI3K/AKT)信号通路、丝裂原活化蛋白激酶(mitogen-activated protein kinase,MAPK)信号通路、内质网中的蛋白质加工、酪氨酸激酶-信号传导子及转录激活子(Janus kinase/signal transducer and activator of transcription,JAK/STAT)信号通路、糖尿病并发症中的晚期糖基化终产物-晚期糖基化终产物受体(advanced glycation endproducts/receptor for advanced glycation endproducts,AGE/RAGE)信号通路、细胞凋亡、缺氧诱导因子1(hypoxia-inducible factor1,HIF-1)信号通路等,详见图4。
2.3WGCNA鉴定关键模块
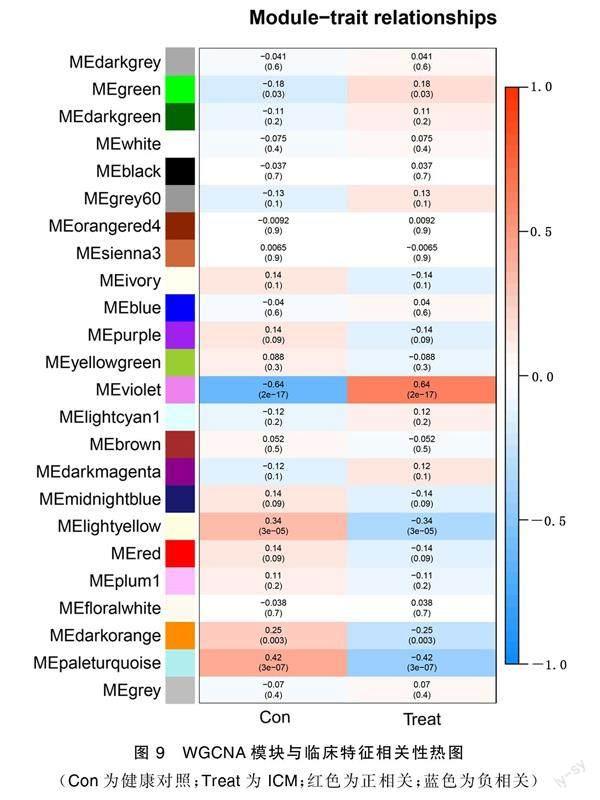
通过WGCNA,选定最佳软阈值为6,既保证网络接近无尺度分布,又保证网络平均连通性不会太小。经动态分支切割得到24个模块。临床性状(ICM患病状态)与模块特征基因之间的相关性分析可见,紫色模块(MEviolet)与ICM呈正相关(相关系数=0.64,P<0.001),被确定为关键模块。详见图5~图10。
2.4核心基因的鉴定
紫色模块(MEviolet)按基因重要性(>0.5)和基因与模块相关性(>0.8)筛选出关键模块内关键基因为无孢蛋白(ASPN)、高温需求丝氨酸肽酶1(high-temperature requirement A serine peptidase 1,HTRA1)和富含亮氨酸重复蛋白的免疫球蛋白超家族(immunoglobulin superfamily containing leucine-rich repeat protein,ISLR),并与DEGs绘制韦恩图,详见图11,获得3个交集基因(ASPN、HTRA1和ISLR)。进行LASSO分析,最终获得2个核心基因(ASPN和HTRA1),详见图12。
2.5核心基因诊断效能评估
在训练集和验证集中比较核心基因ASPN和HTRA1的表达水平,具有一致性,在ICM中均显著上调,绘制箱线图,见图13。利用训练集和验证集分别绘制ROC,AUC提示ASPN和HTRA1作为生物标记区分ICM或正常具备良好的诊断效能(AUC均>0.85)。详见图14。
2.6免疫浸润分析
免疫浸润分析显示,在ICM心肌组织中活化的CD8+T细胞、γδT细胞、辅助型T细胞2(T helper 2 cell,Th2)、效应记忆型CD4+T细胞、中央记忆型CD4+T细胞显著上调;活化的树突状细胞、辅助型T细胞17(T helper 17 cell,Th17)显著下调。详见图15。
上调的核心基因ASPN、HTRA1与免疫细胞相关性分析如图16所示,其中,γδT细胞、中央记忆型CD4+T细胞与ASPN、HTRA1均呈正相关,活化的CD8+T细胞、效应记忆型CD4+T细胞分别与HTRA1呈正相关,活化的树突状细胞与ASPN、HTRA1均呈正相关。
3讨论
本研究结果显示,与健康对照者左心室组织比较,ICM存在517个DEGs。富集分析发现,DEGs的功能主要集中在细胞外基质构成、含胶原蛋白的细胞外基质等;同时,DEGs参与PI3K/AKT信号通路、MAPK信号通路、内质网中的蛋白质加工、JAK/STAT信号通路、AGE/RAGE信号通路、细胞凋亡、HIF-1信号通路等,进一步提示免疫-炎症相关基因在ICM发病机制中的作用。
线粒体未折叠蛋白反应(unfolded protein response,UPR)是关键的进化保守信号通路之一,通过激活蛋白酶、伴侣蛋白和抗氧化酶,清除错误或未折叠的蛋白质等来恢复线粒体功能、维持细胞内稳态[13-14]。UPR通路的适度激活能够保护心肌缺血/再灌注损伤[14]。而UPR通路的过度激活则会促进心肌细胞凋亡,参与ICM在内的多种心血管疾病的发生发展。研究显示,通过调节参与UPR的蛋白质激酶RNA样内质网激酶(protein kinase RNA-like endoplasmic reticulum kinase,PERK)信号通路可减少心肌细胞凋亡[15]。抑制UPR通路可抑制心肌梗死后心律失常的发生[16]。提示UPR信号通路对于维持正常心肌细胞功能至关重要,恢复其正常功能可能是ICM治疗的关键方向之一。心肌梗死后细胞外基质的纤维增生是一种正常的防御反应,目的是快速关闭缺血受损区域,维持心肌组织的完整性,但ICM中细胞外基质反应过度可能导致纤维分隔增多和心脏舒张功能障碍,因此,心肌梗死后细胞外基质反应过程的平衡对于维持心脏正常的结构和功能至关重要[17]。研究表明,在心肌缺血再灌注损伤模型中,通过抑制细胞外基质的产生,可减少心肌细胞凋亡、减少心肌梗死面积[18]。研究显示,ASPN在ICM细胞外基质的重塑中发挥着特异性的作用[19-20]。研究表明,维持正常的细胞外基质结构,能够提高心脏来源的干细胞和祖细胞的生存能力,从而促进心脏修复,是ICM的治疗选择之一[21]。这些结果提示细胞外基质也可能是ICM治疗的靶点之一。ICM发生发展过程中涉及多条信号通路。研究发现,PI3K/AKT信号通路通过促进纤维化、增加氧化应激、诱导细胞焦亡和凋亡等多种途径参与缺血诱导的心肌病变[22-25]。而MAPK信号通路则能够通过增加细胞凋亡、促进炎症反应、增加铁死亡和自噬反应等参与ICM的发生发展[26-29]。JAK/STAT信号通路能够促进巨噬细胞向促炎表型转化、增加心肌细胞凋亡、提高氧化应激水平来参与心肌缺血再灌注损伤[30-33]。虽然AGE/RAGE信号通路参与多种心血管疾病的发生发展,但其在ICM中的研究仍然有限[34]。研究显示,AGE/RAGE信号通路的激活可能通过促进细胞凋亡等途徑来加重心肌细胞缺血损伤[35-36]。这些结果提示ICM的发病机制涉及多个生物过程和多条信号通路,针对这些方向进一步研究,可能探索出ICM新的治疗靶点。
本研究通过WGCNA和LASSO分析等鉴定出在ICM发生发展过程中的2个核心基因(ASPN和HTRA1)。并利用外部数据集验证了其在ICM中具有良好的诊断效能。ASPN是富含亮氨酸的小蛋白聚糖(small leucine rich proteoglycan,SLRP)Ⅰ类家族成员,其主要作用是作为转化生长因子(transforming growth factor,TGF)-β的天然抑制剂,调节与其受体的相互作用[37]。研究显示,ASPN在ICM及扩张型心肌病中表达显著升高,是心力衰竭的生物标志物之一[37-38],这与本研究结果一致。但是,目前对于ASPN在ICM发生发展中的作用机制尚未明确。有学者认为其在心肌缺血再灌注损伤过程中的升高是机体的一种自我保护,是对TGF-β释放和其下游信号通路激活的负反馈,增加ASPN水平能够减轻心肌细胞死亡,降低心肌纤维化和梗死面积,而降低ASPN则损害心肌功能,这些现象可能与ASPN增加线粒体呼吸能力、抑制TGF-β诱导的SMAD2/3信号通路有关[37]。HTRA1是一种丝氨酸蛋白酶,目前缺乏其与ICM或心力衰竭关系及机制的研究。但是HTRA1能够通过结合TGF-β家族的多种蛋白质从而抑制其功能[39]。研究显示,HTRA1-/-小鼠表现为血管内膜增厚、中膜平滑肌异常,最终造成血流减少[40]。本研究结果显示,在ICM中HTRA1水平显著升高,但其机制仍需进一步研究。
本研究进行了免疫细胞浸润分析,发现与健康对照相比,7种细胞在ICM中的含量有显著差异。在心肌遭受缺血这种非免疫介导的损伤后,固有和浸润的免疫细胞就激活了炎症/修复通路,而病理性炎症和组织修复(生理性炎症)之间的动态平衡对心力衰竭的发生发展至关重要[41]。T淋巴细胞介导的免疫反应在冠心病和心力衰竭的进展中发挥核心作用,本研究结果也提示,大部分在ICM中含量显著变化的免疫细胞是T细胞,与之前的报道[42]一致。研究显示,CD8+T细胞、CD4+T细胞、Th2等细胞在ICM的心肌、循环及淋巴组织中数量均升高,而缺乏B细胞和/或T细胞的小鼠则表现出更好的心功能[43-44]。与本研究结果一致。但是目前关于ASPN、HTRA1与这些免疫细胞关系的报道较少,需要在未来的研究中进一步探索。
綜上所述,本研究鉴定的2个核心基因 ASPN和HTRA1通过多种免疫炎症通路参与ICM的发生发展,是ICM防治的潜在靶点。
参考文献:
[1]BRAUNWALD E.The war against heart failure:the Lancet lecture[J].The Lancet,2015,385(9970):812-824.
[2]VIRANI S S,ALONSO A,BENJAMIN E J,et al.Heart disease and stroke statistics-2020 update:a report from the American Heart Association[J].Circulation,2020,141(9):e139-e596.
[3]BANSAL S S,ISMAHIL M A,GOEL M,et al.Response by bansal et al to letter regarding article,"dysfunctional and proinflammatory regulatory T-lymphocytes are essential for adverse cardiac remodeling in ischemic cardiomyopathy"[J].Circulation,2019,139(24):206-221.
[4]LANGFELDER P,HORVATH S.WGCNA:an R package for weighted correlation network analysis[J].BMC Bioinformatics,2008.DOI:10.1186/1471-2105-9-559.
[5]ZHANG B,HORVATH S.A general framework for weighted gene co-expression network analysis[J].Statistical Applications in Genetics and Molecular Biology,2005.DOI:10.2202/1544-6115.1128.
[6]FULLER T F,GHAZALPOUR A,ATEN J E,et al.Weighted gene coexpression network analysis strategies applied to mouse weight[J].Mammalian Genome,2007,18(6):463-472.
[7]EDGAR R,DOMRACHEV M,LASH A E.Gene expression omnibus:NCBI gene expression and hybridization array data repository[J].Nucleic Acids Research,2002,30(1):207-210.
[8]BARRETT T,WILHITE S E,LEDOUX P,et al.NCBI GEO:archive for functional genomics data sets-update[J].Nucleic Acids Research,2013,41(D1):D991-D995.
[9]OGATA H,GOTO S,SATO K,et al.KEGG:Kyoto Encyclopedia of Genes and Genomes[J].Nucleic Acids Research,1999,27(1):29-34.
[10]ASHBURNER M,BALL C A,BLAKE J A,et al.Gene ontology:tool for the unification of biology.The Gene Ontology Consortium[J].Nature Genetics,2000,25(1):25-29.
[11]HUANG D W,SHERMAN B T,LEMPICKI R A.Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources[J].Nature Protocols,2009,4(1):44-57.
[12]HNZELMANN S,CASTELO R,GUINNEY J.GSVA:gene set variation analysis for microarray and RNA-seq data[J].BMC Bioinformatics,2013.DOI:10.1186/1471-2105-14-7.
[13]AZAM T,ZHANG H Y,ZHOU F C,et al.Recent advances on drug development and emerging therapeutic agents through targeting cellular homeostasis for ageing and cardiovascular disease[J].Frontiers in Aging,2022.DOI:10.3389/fragi.2022.888190.
[14]JI H Z,WANG J,MUID D,et al.FUNDC1 activates the mitochondrial unfolded protein response to preserve mitochondrial quality control in cardiac ischemia/reperfusion injury[J].Cellular Signalling,2022.DOI:10.1016/j.cellsig.2022.110249.
[15]MONCEAUX K,GRESSETTE M,KAROUI A,et al.Ferulic acid,pterostilbene,and tyrosol protect the heart from ER-stress-induced injury by activating SIRT1-dependent deacetylation of eIF2α[J].International Journal of Molecular Sciences,2022,23(12):6628.
[16]LIU M,LIU H,PARTHIBAN P,et al.Inhibition of the unfolded protein response reduces arrhythmia risk after myocardial infarction[J].Journal of Clinical Investigation,2021,131(18):e147836.
[17]PIATEK-MATUSZAK P,PASAWSKI R,PASAWSKA U,et al.Assessment of myocardial diastolic dysfunction as a result of myocardial infarction and extracellular matrix regulation disorders in the context of mesenchymal stem cell therapy[J].Journal of Clinical Medicine,2022,11(18):5430.
[18]GE Y,LIU L S,LUO L A,et al.MIR22HG aggravates oxygen-glucose deprivation and reoxygenation-induced cardiomyocyte injury through the miR-9-3p/SH2B3 axis[J].Cardiovascular Therapeutics,2022.DOI:10.1155/2022/7332298.
[19]HUANG C Q,SHARMA A,THAKUR R,et al.Asporin,an extracellular matrix protein,is a beneficial regulator of cardiac remodeling[J].Matrix Biology,2022,110(4):40-59.
[20]ZHAO Y M,GODIER-FURNEMONT A,BAX N A M,et al.Changes in extracellular matrix in failing human non-ischemic and ischemic hearts with mechanical unloading[J].Journal of Molecular and Cellular Cardiology,2022,166(2):137-151.
[21]PARK H J,DE JESUS MORALES K J,BHERI S,et al.Bidirectional relationship between cardiac extracellular matrix and cardiac cells in ischemic heart disease[J].Stem Cells,2021,39(12):1650-1659.
[22]CHEN L,YU Y Y,LIU J,et al.Modular networks and genomic variation during progression from stable angina pectoris through ischemic cardiomyopathy to chronic heart failure[J].Mol Med,2022,28(1):140.
[23]ZHANG B F,LIU G,HUANG B,et al.KDM3A attenuates myocardial ischemic and reperfusion injury by ameliorating cardiac microvascular endothelial cell pyroptosis[J].Oxidative Medicine and Cellular Longevity,2022.DOI:10.1155/2022/4622520.
[24]GUO B B,CAO J X,LIU Y,et al.Cardiac protection of a novel lupane-type triterpenoid from injuries induced by hypoxia-reperfusion[J].International Journal of Molecular Sciences,2022,23(16):9473.
[25]YAN T X,LI X,NIAN T T,et al.Salidroside inhibits ischemia/reperfusion-induced myocardial apoptosis by targeting mir-378a-3p via the Igf1r/PI3K/Akt signaling pathway[J].Transplantation Proceedings,2022,54(7):1970-1983.
[26]WANG Y,GAO H,CAO X H,et al.Role of GADD45A in myocardial ischemia/reperfusion through mediation of the JNK/p38 MAPK and STAT3/VEGF pathways[J].International Journal of Molecular Medicine,2022,50(6):144.
[27]NASEROLESLAMI M,SHARIFI M,NIRI N M,et al.Retraction note:simvastatin-loaded nano-niosomes efficiently downregulates the MAPK-NF-κB pathway during the acute phase of myocardial ischemia-reperfusion injury[J].Molecular Biology Reports,2023,50(6):5533.
[28]ZHANG Q H,PENG Y F,LIU J Y,et al.7-hydroxyflavone alleviates myocardial ischemia/reperfusion injury in rats by regulating inflammation[J].Molecules,2022,27(17):5371.
[29]ZHENG Y,GAO W Q,ZHANG Q,et al.Ferroptosis and autophagy-related genes in the pathogenesis of ischemic cardiomyopathy[J].Frontiers in Cardiovascular Medicine,2022.DOI:10.3389/fcvm.2022.906753.
[30]XU M,LI X Y,SONG L C.Baicalin regulates macrophages polarization and alleviates myocardial ischaemia/reperfusion injury via inhibiting JAK/STAT pathway[J].Pharmaceutical Biology,2020,58(1):655-663.
[31]MA P,LI Y,WANG S S,et al.SOCS3 promotes myocardial cell apoptosis in myocardial ischemia reperfusion rats via JAK/STAT signaling pathway[J].Minerva Cardioangiologica,2020,68(2):164-166.
[32]WY Z,QL Z,MJ X.Effects of propofol on myocardial ischemia reperfusion injury through inhibiting the JAK/STAT pathway[J].European Review for Medical and Pharmacological Sciences,2019,23(14):6339-6345.
[33]FAN S,SUN J,LI R,et al.Lycopene protects myocardial ischemia injury through anti-apoptosis and anti-oxidative stress[J].Eur Rev Med Pharmacol Sci,2019,23(7):3096-3104.
[34]LEE T W,KAO Y H,CHEN Y J,et al.Therapeutic potential of vitamin D in AGE/RAGE-related cardiovascular diseases[J].Cellular and Molecular Life Sciences,2019,76(20):4103-4115.
[35]KHODEER D M,ZAITONE S A,FARAG N E,et al.Cardioprotective effect of pioglitazone in diabetic and non-diabetic rats subjected to acute myocardial infarction involves suppression of AGE-RAGE axis and inhibition of apoptosis[J].Canadian Journal of Physiology and Pharmacology,2016,94(5):463-476.
[36]KANEKO M,BUCCIARELLI L,HWANG Y C,et al.Aldose reductase and AGE-RAGE pathways:key players in myocardial ischemic injury[J].Annals of the New York Academy of Sciences,2005,1043(1):702-709.
[37]NAKAJIMA M,KIZAWA H,SAITOH M,et al.Mechanisms for asporin function and regulation in articular cartilage[J].Journal of Biological Chemistry,2007,282(44):32185-32192.
[38]ZHANG K,WU M,QIN X Y,et al.Asporin is a potential promising biomarker for common heart failure[J].DNA and Cell Biology,2021,40(2):303-315.
[39]LIN X L,YANG T K,LIU X,et al.TGF-β/smad signalling activation by HTRA1 regulates the function of human lens epithelial cells and its mechanism in posterior subcapsular congenital cataract[J].International Journal of Molecular Sciences,2022,23(22):14431.
[40]KATO T,MANABE R I,IGARASHI H,et al.Candesartan prevents arteriopathy progression in cerebral autosomal recessive arteriopathy with subcortical infarcts and leukoencephalopathy model[J].Journal of Clinical Investigation,2021,131(22):e140555.
[41]STRASSHEIM D,DEMPSEY E C,GERASIMOVSKAYA E,et al.Role of inflammatory cell subtypes in heart failure[J].Journal of Immunology Research,2019.DOI:10.1155/2019/2164017.
[42]FUKUNAGA T,SOEJIMA H,IRIE A,et al.Relation between CD4+ T-cell activation and severity of chronic heart failure secondary to ischemic or idiopathic dilated cardiomyopathy[J].The American Journal of Cardiology,2007,100(3):483-488.
[43]CARINA G,ANDR S,SEBASTIAN M,et al.CD8+ T cells with specificity for a model antigen in cardiomyocytes can become activated after transverse aortic constriction but do not accelerate progression to heart failure[J].Frontiers in Immunology,2018.DOI:10.3389/fimmu.2018.02665.
[44]NEVERS T,SALVADOR A M,GRODECKI-PENA A,et al.Left ventricular T-cell recruitment contributes to the pathogenesis of heart failure[J].Circulation Heart Failure,2015,8(4):776-787.
(收稿日期:2023-05-16)
(本文編辑邹丽)
中西医结合心脑血管病杂志2023年23期
