用于多模态MRI脑肿瘤图像分割的融合双重对抗学习CNN-Transformer模型
华楷文,方贤进
(安徽理工大学 计算机科学与工程学院,安徽 淮南 232001)
胶质瘤是人类最为常见的原发性肿瘤之一。目前胶质瘤的影像检查一般使用计算机断层扫描(computed tomography,CT)和核磁共振成像(magnetic resonance imaging,MRI),通过这2种方式可以准确查看到胶质瘤病变的形态以及准确位置。其中,MRI检查在识别脑组织结构上有较高的分辨率,一般作为辅助脑部胶质瘤治疗的首选手段。在临床应用上,经验不同的医生对影像上病变区域进行手动标注和分割时会产生一定差异,这将给最终诊断带来巨大的困难。因此,在MRI图像上是否能准确分割出病变区域对于胶质瘤的辅助治疗十分重要。
随着人工智能和深度学习的快速发展,卷积神经网络(convolutional neural network,CNN)在医学图像分割领域取得了极大成功[1]。在脑肿瘤图像分割领域,Colman等[2]将残差模块引入U-Net模型,将其中104层卷积块替换成残差块,提出具有 104 个卷积层的深度残差 UNet(deep residual UNet with 104 convolutional layers,DR-UNet104)模型。Awasthi等[3]开发了基于注意力机制的U-Net分割模型,该模型用于在MRI影像中识别肿瘤的各个区域。Xu等[4]将注意力模块和深度监督方法结合,提出了用于脑肿瘤图像分割的U-Attention Net框架。Çiçek等[5]在U-Net模型基础上提出了针对3D图像分割的3D U-Net模型,该模型只需将U-Net中的2D卷积块转换成3D卷积块。3D U-Net模型的提出使得3D医学图像的分割性能得到了极大提升。在此基础上,产生了一系列3D U-Net脑肿瘤图像分割模型。如Zhao等[6]在3D U-Net模型基础上提出名为“Categorical Dice”的新型损失函数,通过为不同分割区域设置权重解决了前景和后景图像体素不平衡的问题。何康辉等[7]通过将3D U-Net模型中通道数减少到原来的1/4来降低模型参数量,同时在模型中使用空间-通道压缩和激发(squeeze-and-excitation networks,SE)模块来提升分割精度。Guan等[8]在每个编码器模块中加入了SE网络,并在每个解码器中加入注意力引导过滤器(attention guide filter,AG)模块,利用注意力机制对边缘信息进行提取,设计出了3D注意力引导过滤器和压缩-激发VNet(3D attention guide filter and squeeze-and-excitation VNet,3D AGSE-VNet)模型进行脑肿瘤图像分割。
CNN在对数据特征进行提取时有出色的表现,但由于受感受野大小的限制,无法捕获远距离特征。因此,Vaswani等[9]提出视觉自注意力(vision transformer,ViT)模块,作为基于注意力机制的模块被引入到计算机视觉的各个领域,该模块依靠其出色的远距离特征捕获和建模能力弥补了CNN的不足。随着ViT模块的引入,CNN-Transformer相互结合的模型被迅速提出。在脑肿瘤图像分割方面,Wang等[10]提出使用Transformer进行多模态脑肿瘤图像分割(brain tumor image segmentation using Transformer,TransBTS)模型,将注意力机制与CNN结合起来,构成编码器-解码器架构,利用3D CNN来捕获空间和深度信息,通过ViT模块对远距离特征进行建模,最终得到高分辨率的分割结果。此外,Jia等[11]提出双重Transformer的UNet(bi-Transformer U-Net,BiTr-UNet)模型,通过在编码器-解码器架构中的跳跃连接层上引入多个ViT模块来提取更多数据特征信息,引入的多个ViT模块在一定程度上提升了模型的分割性能,但模型自身参数量将会显著提升,模型复杂度变高。Menze等[12]将20种最先进的肿瘤图像分割算法应用于低级别和高级别神经胶质瘤,发现融合几个算法产生的分割性能始终高于单独的算法,说明脑肿瘤图像分割算法性能还能进一步优化。
上述研究都在一定程度上提高了脑肿瘤图像的分割精度,但这些模型在数据传输过程中由于池化操作会导致一些特征信息丢失,这将使分割模型在训练数据中提取到的特征信息不足,最终应用在测试数据上效果不佳。此外,上述模型都未考虑模型的抗干扰能力,鲁棒性不足。对此,提出融合双重对抗学习的CNN-Transformer(CNN-Transformer model fusing dual adversarial learning,TransFDA)分割模型对脑肿瘤图像进行分割,该模型在分割过程中使用轻量级CNN-Transformer模型,并通过引入双重对抗学习来优化分割模型,以期在不增加模型参数量和复杂度的基础上提高模型的分割精度和鲁棒性。
1 模型的构建
TransFDA基于TransBTS和BiTr-UNet模型,整体训练流程如图1所示。由图1可知,TransFDA的整体架构由3部分子模块构成,分别为基础分割模块、判别器模块、虚拟对抗训练模块。其中,基础分割模块由CNN-Transformer结构组成,在训练过程中,首先将分割模型预测出的结果送入判别器模块中,图1中的黑色线条为基础分割模块正常生成预测标签过程,红色线条为预测标签与训练数据进行对抗过程;红色实线代表2组数据分别进入判别器模块进行特征匹配,生成判别差异损失,并将差异信息反馈给分割模型的过程;红色虚线代表分割模型经判别模块多次对抗优化后预测出越来越完整、准确的肿瘤区域;此过程直至判别模块中生成的损失变化不大、收敛为止,此时分割模型达到了分割最优结果。另外,图1中绿色线条为虚拟对抗训练过程,其中实线部分为在原始图像上增添干扰,然后将生成带干扰的新图像送入分割模型中,预测出带干扰的标签结果过程;虚线为将正常图像预测出的分割结果和干扰图像预测出的结果送入虚拟对抗训练模块,经过判别生成两者之间的虚拟对抗损失的过程。最终,将上述2种损失与分割模型生成的损失结合起来,反馈到分割模型中。上述操作使得基础分割模型在后续训练过程中能够处理之前分割过程中存在的不足,从而学习到图像上更多的数据特征。
1.1 分割模型
基础CNN-Transformer分割模型结构如图2所示,其中浅蓝色矩形代表特征图,矩形周围的数字代表每个特征图的通道个数,紫色矩形代表在跳跃连接层上添加的ViT模块,不同颜色箭头代表不同的操作。由图2可知,首先将4个模态的脑肿瘤图像送入分割模型中,经一系列的特征学习,最终预测分割出图像中病变的3块区域。其中,编码器模块中一系列卷积和下采样操作对进入模型中的高维数据进行降维处理,并在编码器最后一层上使用空洞卷积来捕捉图像的多尺度上下文信息,促使分割模型更好学习脑肿瘤图像中的特征信息。为加强分割模型对图像中重要特征信息的提取,每层卷积块后都引入卷积注意力机制模块(convolutional block attention module,CBAM),通过该模块促使分割模型提取重要的空间和通道信息。另外,编码器中每层提取到的特征都使用批归一化(batch normalization,BN)操作,用整流线性单元(rectified linear unit,ReLU)激活函数进行处理,防止模型在训练过程中出现梯度爆炸或梯度消失等问题,同时还能加快模型训练。此外,解码器同编码器总体架构一致,都是由一系列卷积块和上采样操作组成;解码器架构和编码器架构相互对应,解码器通过上采样操作来恢复编码器中下采样操作压缩的维度,融合层将提取到的特征与解码器中对应的特征进行融合,以此来获得更多的空间特征。最后,在解码器最后一层使用卷积核大小为1×1的卷积和激活函数,将输出的结果映射到期望分割的类别数。分割模型在最后2层跳跃连接处分别引入了1个ViT模块来提取远距离信息。

图2 分割模型结构Fig.2 Segmentation model structure
1.2 判别器模块
判别器模块受马尔可夫判别器启发提出并使用。此模块由3层3×3×3卷积块组成,卷积块之后添加BN与ReLU激活函数层,加快训练速度。常用的判别器模块在得到预测标签后,通常使用预测标签与真实标签进行判别,而此判别器模块受无监督与半监督模型启发,不使用真实标签,而是将分割模型预测到的结果与原始图像送入到判别器模块中,在判别器处理过程中,整个脑部都被认作存在肿瘤的区域。首先,将整个脑部与预测得到的标签一同送入判别器模块中进行判别,由于刚开始分割模型能力有限,预测到的分割标签上肿瘤区域较小,判别器很容易就对其判断为假,并生成损失,同时将信息反馈给基础分割模型,此时基础分割模型对整个脑部中之前未分割的区域加强识别分割;随着多次训练的叠加,基础分割模型与判别器模块反复相互优化,直至判别模块中的损失达到拟合,不再发生过多变化为止,此时模型会分割出最优结果。
1.3 虚拟对抗训练
在训练过程中训练数据数量有限,分割模型鲁棒性存在不足,对外界可能出现的各种干扰应对能力较差。为提高模型抵抗干扰数据的能力,引入虚拟对抗训练。首先,找到使得输出偏差最大的扰动方向,在此方向上对输入的原始数据加入扰动,该扰动通过分割模型的损失与正则化后的原始数据梯度组合而成。然后,将原始数据与添加扰动后的新数据送入分割模型,经过处理后分别得到各自的分割结果标签,其中添加扰动的数据分割出的标签不同于正常分割结果,会产生异常标签。同上述判别器模块一致,此模块中同样不使用真实标签,将添加扰动后分割得到的标签与正常数据分割后得到的标签进行判别,将得到这2个标签之间的损失,并将其反馈给分割模型;分割模型收到多次信息反馈优化后,添加干扰的数据分割出的结果与正常数据经分割模型预测出的标签越来越一致。在整个对抗过程中,随着训练轮次的叠加,模型鲁棒性逐渐加强,外界干扰对分割模型的影响越来越低,模型将变得更加稳健。
2 实验与结果
2.1 实验数据集
分割模型训练和验证过程中使用国际医学图像计算和计算机辅助干预协会(medical image computing and computer assisted intervention society,MICCAI)提供的Brats2020多模态脑肿瘤数据集[12]。此数据集将369张病例作为训练集,125张病例作为验证集,训练集中包含高级别胶质瘤(high-grade glioma,HGG)和低级别胶质瘤(low-grade glioma,LGG)信息,验证集中案例级别未知。由于Brats2020公开数据集中未提供测试集数据,为进一步验证模型性能,从Brats2021数据集中随机收集125例未出现在Brats2020数据集中的案例作为测试集。每个案例的4个模态和真实标签如图3所示。由图3可知,4个模态分别为液体衰减反转恢复序列(fluid attenuated inversion recovety,FLAIR)、T1、T1ce、T2,训练数据集中提供了真实分割标签,包括背景、非增强肿瘤区域(non-enhancing tumor,NET)、浮肿区域(edema,ED)和增强肿瘤区域(enhancing tumor,ET)。标签图像中的绿色部分代表ED,此区域对应标签2;红色代表NET,此区域对应标签1;黄色代表ET,此区域对应标签4;背景区域对应标签0。低级别胶质瘤相较高级别胶质瘤会缺少一些病变区域,若模型分割到这些区域时分割系数会显示为0,因此对低级别胶质瘤进行分割时难度会有所上升。最终任务是对脑肿瘤图像中的肿瘤整体区域(whole tumor,WT)、肿瘤核心区域(tumor core,TC)和增强肿瘤区域(enhancing tumor,ET)进行分割。其中WT区域包含所有的标签,即标签图像中红色、绿色、黄色3个区域的合并部分,TC区域为红色、黄色区域的合并部分,ET为标签中黄色区域。
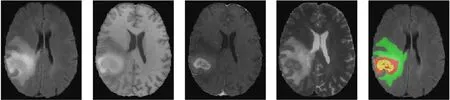
(a) FLAIR图像 (b) T1图像 (c) T1ce图像 (d) T2图像 (e) 真实标签图3 不同模态的脑肿瘤和标签Fig.3 Different modality brain tumor images and labels
2.2 数据集预处理
数据集中所有病例包含的4个模态数据大小都为240×240×155,4个模态共享分割标签。首先,将4张不同模态脑肿瘤图像进行合并处理,将其合并成1张4D(H×W×D×C,C=4)图像,并与分割标签一同保存为pkl文件。这是由于此格式的文件读写速度较快,有利于加快训练速度。在对实验数据进行观测时发现,原始图像的周围存在着大规模的黑边,因此将实验数据统一裁剪成128×128×128尺寸大小的图片,并对其在[-0.1,0.1]范围内随机变换强度,在[0.9,1.1]的范围内缩放。
2.3 实验环境与配置
实验使用Ubuntu16.04操作系统,在2张带有24GB显存的NVIDIA GeForce RTX 3090显卡上进行模型的训练、验证、测试。训练过程中使用初始化学习率为0.0002的Adam优化器。实验基于Pytorch框架实现,批处理大小为8,共迭代1000次。
2.4 评价指标
为了对所提模型进行性能评估,采用95%豪斯多夫距离(Hausdorff distance,HD)和戴斯相似系数(Dice similarity coefficient,DSC)作为分割模型的评价指标。
HD指标在医学图像分割中是指2张掩码边界像素距离集合中的最大值,即真实分割标签与预测出的标签2组点集之间距离的度量,具体计算公式如下所示:
(1)
其中,y′和y分别表示模型预测标签和真实标签,a和b为2个体素集中的体素点,H为HD指标的计算结果,值越小,说明真实分割标签与预测标签两组点集之间距离越近,相似度就越高。
DSC指标为语义分割中的重要评价指标之一,可以衡量2个样本之间的相似程度;数值越接近1,表明模型分割精度越好,具体计算公式如下所示:
(2)
其中,PDSC为DSC指标的计算结果,Dtrue代表分割图像的真实标签,Dpre代表模型分割出的预测标签。
2.5 损失函数
分割模型中使用广义戴斯损失(generalized Dice loss,GDL)函数。GDL函数是在戴斯损失函数基础上优化而来,后者是针对特定类别分割的损失,在需要分割出多个病灶的场景下并不适用,而GDL函数可将多场景下分割的病灶损失整合为1个指标,将其作为分割结果进行评估,具体计算方式如下所示:
(3)
其中,PGDL为GDL函数计算结果,Rln表示第l个类别图像在第n个位置的真实分割结果,Pln表示相应的预测概率,Wl表示第l个类别的权重。
在判别器模块中,为了使最终分割结果更加接近于真实标签结果,使用和大多数生成式对抗模型中相同的策略,最大化判别器判别能力,最小化分割模型中分割结果与真实图像之间的差异性,具体可表示为:
(4)
其中,θs代表基础分割模块的参数,θd代表判别模块的参数,X为输入的数据集合,包括输入图像和生成的预测标签。
判别器模块对预测出的标签与整个脑部之间的差异大小进行判别。判别差异损失定义为
(5)
其中,Ladv(θs;θd;X)为判别器模块生成的判别差异损失,x为输入图像,E(x,y′)为输入图像和生成预测标签的集合,H、W为图片高度与宽度,c、d分别为图片高度与宽度上的像素点,ψ∈(0,1)H×W的大小表示判别器的功能;当从分割模型中生成分割掩码时,η=0,当从输入图片中提取样本时,η=1。
虚拟对抗损失用来判别预测出的分割标签与添加扰动后生成的标签之间的差异,最小化虚拟对抗损失,即是最大化模型克服添加扰动数据的能力,使模型的鲁棒性得以加强。具体定义如下所示:
(6)
其中,Lvat(θs;X;radv)为虚拟对抗训练生成的虚拟对抗损失,radv是对原始图像上施加的扰动信息,DKL为KL(Kullback-Leibler)散度,F代表分割模型。
整个模型最终损失由上述3部分损失函数线性累加组成,具体定义如下所示:
L(θ;X;Y)=PGDL(θs;Y)+λLadv(θs;θd;X)+ωLvat(θs;X;radv),
(7)
其中,L(θ;X;Y)为最终模型的总损失,θ代表模型中所有模块参数,Y为真实标签结果与预测标签结果集合,λ、ω分别为2组对抗模块对应的超参数。为保证最终分割模型的稳定性,经过多次的参数测试后,最终设置为λ=0.002、ω=0.003,其他参数值的设置可能会出现异常结果。模型训练过程中损失函数的数值变化情况如图4所示。由图4可知,随着训练轮次的增加,损失函数值逐渐下降,在训练到900轮左右时,损失函数的变化趋势已经不大,呈现出收敛状态。
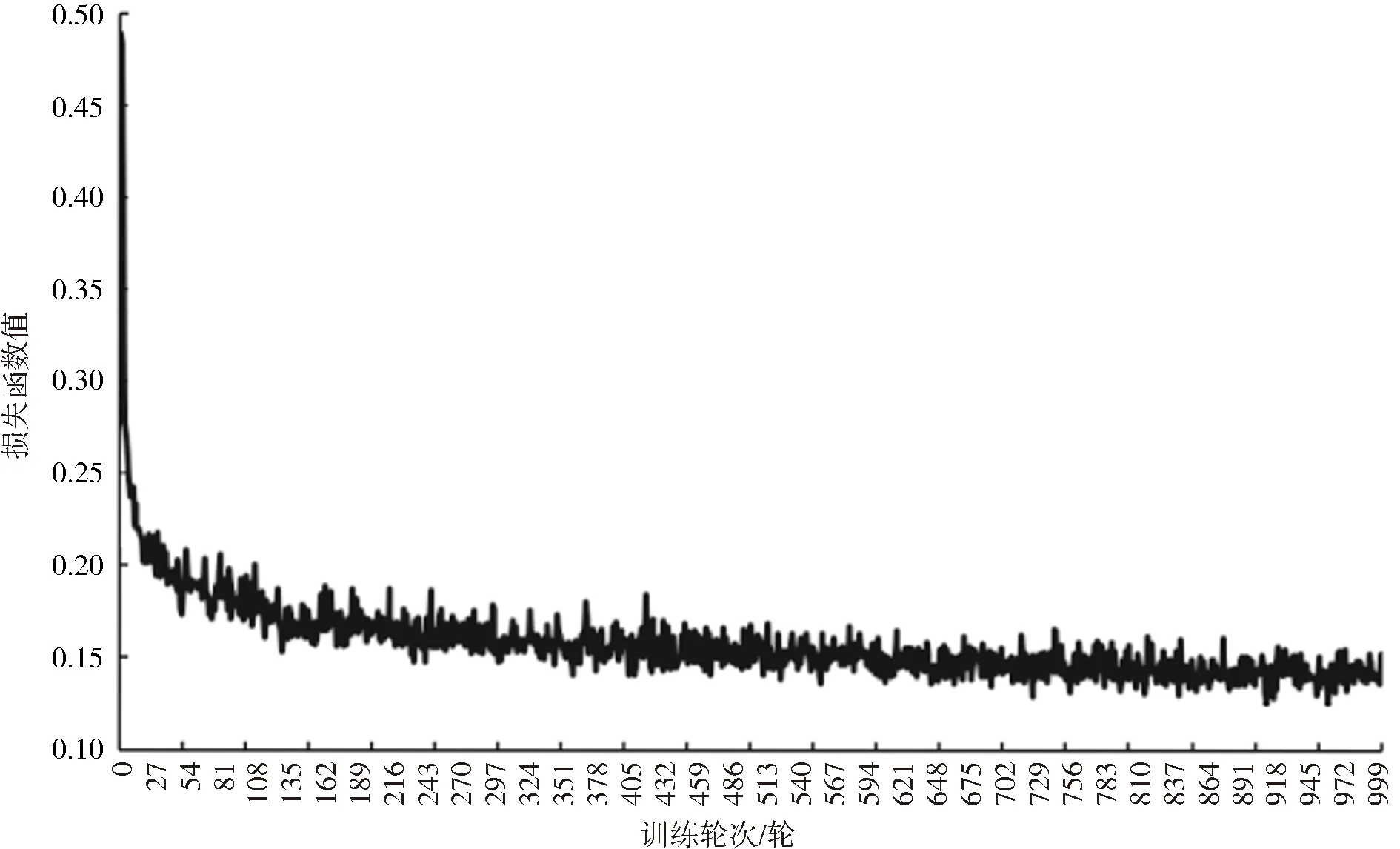
图4 损失函数变化趋势Fig.4 Change trend of loss function
2.6 TransFDA实验结果
TransFDA在Brats2020验证集上的分割结果如表1所示。由表1可知,对ET、WT、TC的PDSC平均值分别达到0.7530、0.8922、0.7909,对ET、WT、TC的H平均值分别达到27.51、6.32、15.67;其中,WT的分割结果较ET与TC更优,这是由于WT在脑肿瘤中所占比例较大,ET与TC病变区域较小,另外这2个区域数据标签之间存在较大的不确定性,尤其是对低级别脑肿瘤数据进行分割时,模型获取信息不足,导致最终结果较差。

表1 Brats2020验证集上的分割结果Tab.1 Segmentation results on the Brats2020 validation dataset
2.7 对比实验
为验证TransFDA的性能优势,将其分别与不同的2D、3D脑肿瘤分割模型和参加BraTS2020挑战赛的其他团队所提模型进行对比,结果如表2、表3所示,表格中结果来源于各种模型原论文和挑战赛官方网站上提供的实验数据。由表2、表3可知,所提模型对ET、WT、TC的PDSC数值均高于其他脑肿瘤分割模型,所提模型对ET、WT、TC的H也取得了较为优秀的结果。

表2 对比实验结果Tab.2 Comparison of experimental results

表3 与Brats2020挑战赛其他团队在验证集上的对比实验结果Tab.3 Comparison of experimental results with other teams in the Brats2020 Challenge on the validation dataset
此外,TransFDA与经典CNN-Transformer分割模型参数的对比结果如表4所示。由表4可知,相较于经典的CNN-Transformer分割模型,TransFDA在模型参数量、计算量、模型大小上有着明显的优势。

表4 与经典CNN-Transformer分割模型的参数对比Tab.4 Comparison of parameters with classic CNN-Transformer segmentation models
2.8 消融实验
在Brats2020验证集和Brats2021测试集上进行了5组消融实验,以此来验证提出的模块是否能够优化模型的分割性能,所有消融实验均使用相同的实验环境和超参数,验证集上的实验结果如表5所示。由表5可知,通过对比前3个模型实验数据发现,在验证集上基础分割模型相较于TransBTS和BiTr-UNet对WT、TC的分割精度有明显提升,特别是对TC,相较于TransBTS,PDSC数值提升了0.0230,H数值降低了6.68。对比第3个模型和第4个模型实验数据可以看出在添加新型判别器模块后,模型对ET、WT分割精度进一步得到了提升,但对TC分割精度有所下降,通过对Brats2020实验数据的分析,最终判断是因为分割模型对TC的过分割以及模型缺乏抗干扰能力所致。因此,再引入虚拟对抗模块(即完整的TransFDA)来进一步加强模型分割能力。对比第4个模型和第5个模型实验数据可以看出添加虚拟对抗训练模块后,模型对3个区域分割结果都得到了有效的提升,其中对ET、WT的分割结果达到了最优,相较于基础模型,PDSC数值分别提升0.0141、0.0123,H数值分别降低了3.29、1.88。另外,对TC的分割性能相较于基础分割模型有所不足,通过对所有数据参数进行分析,推测是因为双重对抗模块对Brats2020验证集中的一些数据存在过分割;但综合所有参数指标的比较结果,最终表明TransFDA性能仍能达到最优。
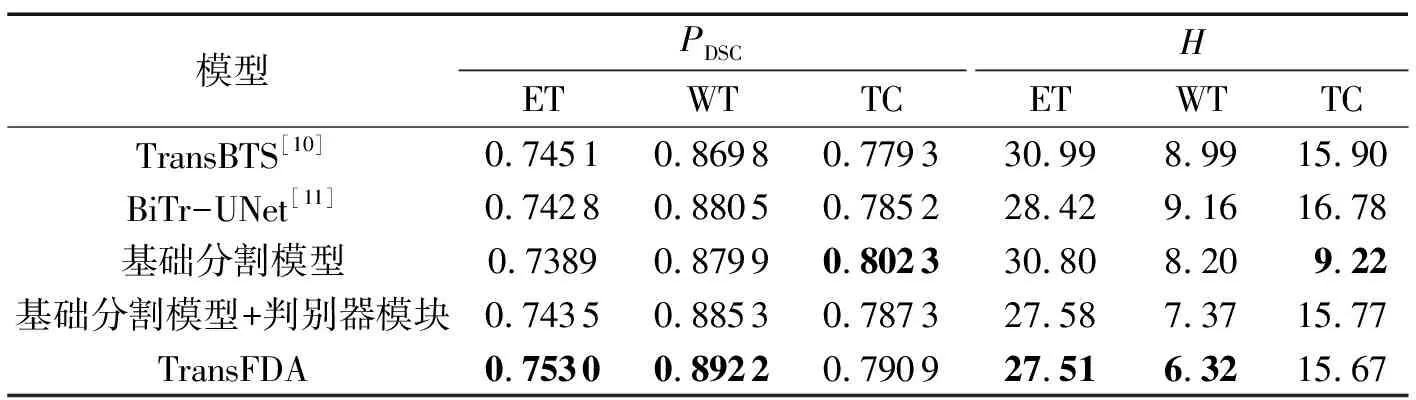
表5 在验证集上的消融实验Tab.5 Ablation experiment on validation set
测试集上的实验结果如表6所示。由表6可知,在基础分割模型上引入双重对抗学习后,TransFDA对ET的H数值有轻微提升,表示图像真实标签和模型预测生成标签的相似度降低,其余所有指标相较于基础分割模型都有着明显的性能提升;对于ET、WT、TC的PDSC平均值分别达到0.8438、0.9155、0.8900,对ET、WT、TC的H平均值分别达到2.00、5.00、2.83;相较于表格中其他CNN-Transformer分割模型,PDSC数值都有着一定的提升,但比较H数值,发现模型的性能有一定的欠缺。
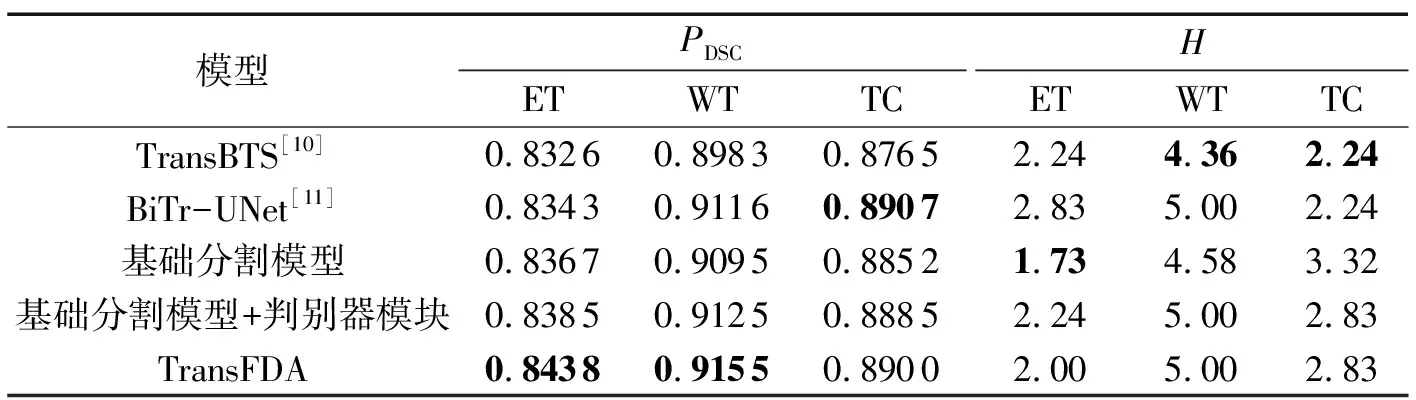
表6 在测试集上消融实验Tab.6 Ablation experiment on the test set
2.9 定性与定量实验
为更加直观地分析模型中各模块的分割效果,分别挑选了高级别胶质瘤和低级别胶质瘤数据进行可视化处理,通过可视化结果能够更好地观测各模块之间的差异。MRI脑肿瘤水平面、矢状面、冠状面分割结果如图5、图6所示。由图5、图6可知,在分割区域上可以明显看出完整的TransFDA分割出的区域较其他模型更加丰富。
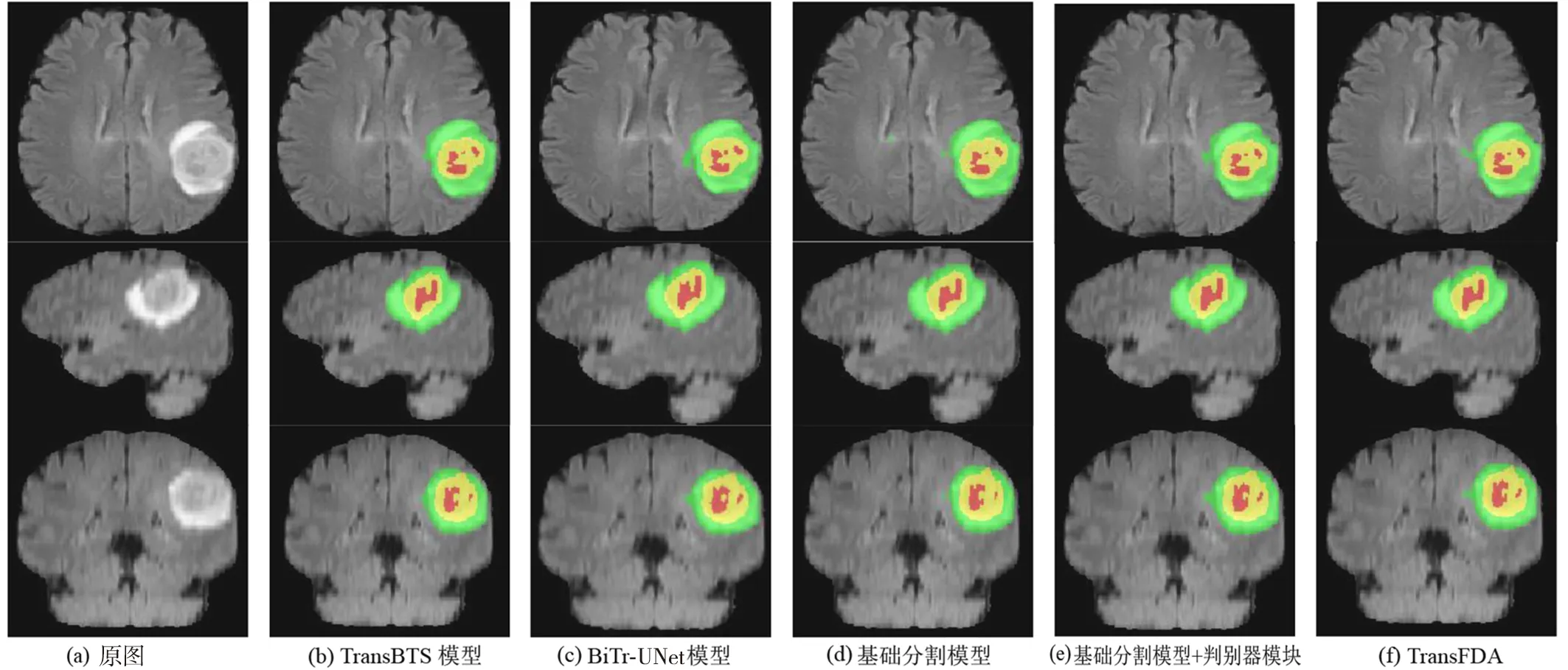
图5 MRI高级别脑肿瘤(validation_058)分割结果在水平面、矢状面、冠状面的对比Fig.5 MRI high-grade brain tumor (validation_058) segmentation results in the horizontal,sagittal,and coronal planes compared
由于Brats2020验证集数据不提供真实标签,因此将各模型的指标结果绘成表格,结果如表7、表8所示。由表7可知,对于高级别胶质瘤的分割,TransFDA对ET、WT、TC的分割性能提升显著,而高级别胶质瘤较易分割;对于难分割的低级别胶质瘤,由表8可知,DSC和HD指标也有一定提升,且不同模型性能差异较为明显,表示所提TransFDA对于此类型数据的分割效果较好。

表7 不同模块在Brats2020_validation_058上的实验结果Tab.7 Experimental results of different modules on Brats2020_validation_058

表8 不同模块在Brats2020_validation_072上的实验结果Tab.8 Experimental results of different modules on Brats2020_validation_072
同时,在Brats2021测试集上进行可视化实验,测试集数据提供真实标签,可视化结果如图7所示。由图7可知,TransFDA分割出来的标签和真实标签相似度极高,在分割区域的细节处理上更加完善,对脑肿瘤边缘区域的分割相较于其他模型更加细致。以上实验结果表明,对于较易分割的高级别脑肿瘤数据,大多数模型分割性能差异表现不大,但对于较难分割的低级别脑肿瘤数据,不同模型的性能差异较为明显。通过以上定性与定量结果分析可知,TransFDA模型的综合性能表现最优。

图7 MRI高级别脑肿瘤测试集分割结果在水平面、矢状面、冠状面的对比Fig.7 MRI high-grade brain tumor test set segmentation results in the horizontal,sagittal,and coronal planes compared
3 结论
提出融合双重对抗学习的CNN-Transformer分割模型TransFDA,所提TransFDA是对传统CNN-Transformer模型进一步优化改进而来,加入的新型判别器模块能有效加强基础分割模块对于脑肿瘤图像上特征信息的提取,显著提升模型分割性能;另外,针对基础分割模型中存在的过分割和抗干扰能力不足问题,引入虚拟对抗训练模块,可增强分割模型的鲁棒性;最后,在Brats2020验证集上进行性能测试,对ET、WT、TC的PDSC平均值分别达到0.7530、0.8922、0.7909,对ET、WT、TC的H平均值分别达到27.51、6.32、15.67。实验结果表明,TransFDA可以有效提升分割精度,同时与经典的基于CNN和CNN-Transformer的2D、3D分割模型,以及Brats2020挑战赛上其他团队所提模型相比较,在脑肿瘤分割精度上都有着一定的优势。通过引入Brats2021上部分数据集来进一步证明在测试集上TransFDA的性能优势,发现对ET、WT、TC的PDSC平均值分别达到0.8438、0.9155、0.8900,对ET、WT、TC的H平均值分别达到2.00、5.00、2.83。虽然所提TransFDA在脑肿瘤分割上有优秀表现,但仍然存在一些问题,比如对于低级别胶质瘤的分割,在增强肿瘤与肿瘤核心区域的分割精度较为不足,特别对肿瘤边界的分割较为毛糙,不够平滑,并存在一些过分割问题,后续将针对这些问题展开深入研究。

