一株高效降解血液蛋白的枯草芽孢杆菌NWMCC0137全基因组测序及分析
刘高强 李新鹏 刘文钊 周魏 彭乔烽 周雪雁 默罕默德•索布瑞•塔克瑞夫 丁功涛







摘要: NWMCC0137是從屠宰场下水道污泥中分离获得的1株高效分解血液蛋白并具有良好生物防控特性的枯草芽孢杆菌(Bacillus subtilis)。为探究枯草芽孢杆菌NWMCC0137(B.subtilis NWMCC0137)高效水解血液蛋白机制及挖掘次级代谢产物编码基因资源,利用DNBSEQ与PacBio测序平台对其进行全基因组测序并进行序列组装和基因预测、功能注释、比较基因组分析。结果表明B.subtilis NWMCC0137的基因组长度为4 215 585 bp,G+C含量为44.23%;编码基因总长度为3 711 885 bp,编码4 384个基因,编码序列占整个基因组长度的88.05%,非编码RNA中包含85个tRNA基因。在KEGG数据库中B.subtilis NWMCC0137基因组被注释到7种胞外蛋白酶编码基因,其中与丝氨酸蛋白酶相关的编码基因有8个。通过antiSMASH预测发现,菌株NWMCC0137基因组中包含7种与已知次生代谢产物合成基因簇相似度为100%的基因簇,包括产孢致死因子、聚酮类化合物、丰原素、枯草芽孢杆菌素168、儿茶酚型嗜铁素、芽孢杆菌素、杆菌溶素合成基因簇。进化分析结果表明,NWMCC0137为枯草芽孢杆菌,并且与枯草芽孢杆菌168具有很高的共线性。B.subtilis NWMCC0137基因组序列已提交至GenBank数据库,登录号为CP103066。本研究从基因层面揭示了菌株NWMCC0137的遗传信息,为该菌株应用于屠宰场血液蛋白降解及生物防控提供了参考。
关键词: 枯草芽孢杆菌;全基因组;基因功能注释;蛋白酶
中图分类号: Q934.124 文献标识码: A 文章编号: 1000-4440(2023)07-1460-12
Whole genome ; sequencing and analysis of Bacillus subtilis NWMCC0137, an efficient blood protein degrading strain
LIU Gao-qiang1,2, LI Xin-peng1,3, LIU Wen-zhao1,2, ZHOU Wei1,2, PENG Qiao-feng1,2, ZHOU Xue-yan1,2, MOHAMMED Sobri-takriff4, DING Gong-tao1
(1.China-Malaysia National Joint Laboratory, Biomedical Research Center, Northwest Minzu University, Lanzhou 730030, China;2.College of Life Sciences and Engineering, Northwest Minzu University, Lanzhou 730030, China;3.College of Chemical Engineering, Northwest Minzu University, Lanzhou 730030, China;4.Department of Chemical and Process Engineering, Faculty of Engineering and Built Environment University Kebangsaan Malaysia, Bangi 43600, Malaysia)
Abstract: NWMCC0137 is a Bacillus subtilis strain isolated from sewage sludge in slaughterhouses, which efficiently decomposes blood proteins and has good biological control characteristics. In order to explore the mechanism of efficient hydrolysis of blood protein by Bacillus subtilis NWMCC0137 and explore the gene resources encoded by secondary metabolites, DNBSEQ and PacBio sequencing platforms were used to sequence the whole genome, and sequence assembly, gene prediction, functional annotation and comparative genome analysis were carried out. The results showed that the genome length of Bacillus subtilis NWMCC0137 was 4 215 585 bp, and the G+C content was 44.23%. The total length of the coding gene was 3 711 885 bp, encoding 4 384 genes, accounting for 88.05% of the entire genome length. The non-coding RNA contained 85 tRNA genes. In KEGG database, Bacillus subtilis NWMCC0137 genome was annotated to seven extracellular protease coding genes, including eight coding genes related to serine protease. According to the prediction of antiSMASH, the genome of strain NWMCC0137 contained seven gene clusters with 100% similarity to the known secondary metabolite synthesis gene clusters, including sporulation killing factor, bacillaene, fengycin, sublancin 168, bacillibactin, subtilosin A and bacilysin gene clusters. The evolutionary analysis results indicated that NWMCC0137 was a Bacillus subtilis and had high collinearity with Bacillus subtilis 168. The genome sequence of Bacillus subtilis NWMCC0137 had been submitted to the GenBank database with accession number CP103066. This study revealed the genetic information of strain NWMCC0137 at the genetic level, providing a reference for its application in degrading blood proteins and biological prevention and control.
Key words: Bacillus subtilis;whole genome;gene function annotation;protease
血液中蛋白质含量为20%,动物屠宰后血液中的蛋白质资源[1]只有少部分能被利用,大多数血液未能被妥善处理。这不仅造成资源浪费,还会引起水土污染甚至是动物疾病[2]。提高动物屠宰血液利用率和废弃血液的资源化利用,是目前亟待解决的问题。目前国内外废弃血液利用方式主要有饲料化、能源化、食品化、药品化、肥料化等[3]。在饲料化和肥料化利用中,酶解和微生物发酵是重要的处理方式。酶解法对环境影响小,可以有效抑制毒物的生成[4];与酶解法相比,微生物发酵的处理方式具有成本低、产酶种类丰富、操作简单等优势[5-6]。
目前微生物发酵所用的菌种有芽孢杆菌、酵母菌、乳酸菌[7]等。庞伟[8]利用枯草芽孢杆菌对血粉发酵,检测结果显示,发酵后蛋白质最大水解度为32.30%,发酵产物中氨基酸含量是原来的4.44倍,可溶性蛋白质含量提高了53.77%。陶艳华等[9]和Yao等[10]均采用短小芽孢杆菌发酵,发酵后血红蛋白降解率分别达到53.64%和85.00%。崔玮琪等[11]筛选得到菌株X-17,以猪血作为唯一氮源发酵,经优化后血液蛋白水解度达到18.52%,可溶性蛋白质含量达到2.75 mg/ml。有研究结果表明,菌株对蛋白质的降解率受血液占比影响较大,血液在发酵底物中的占比低于10%时,菌株对血液蛋白降解率较高[12],血液在发酵底物中的占比高于10%时,降解率明显降低。Zhang等[13]使用重组酵母菌发酵血液,血液在发酵底物中的占比为3.2%时,蛋白质降解率为49.0%。而马子豪等[14]发现,血液在发酵底物中的占比为30%时,蛋白质降解率只有9.86%。本试验前期使用枯草芽孢杆菌NWMCC0137,在动物屠宰血液占发酵底物90.0%情况下,血液中蛋白质水解率仍有17.6%,多肽含量为15.16 mg/ml,具有较高的水解效率,表明菌株NWMCC0137在动物屠宰血液再利用方面有着较好的潜力。
枯草芽孢杆菌(Bacillus subtilis)是一种重要的工业微生物[15]。研究结果表明,枯草芽孢杆菌能产生至少8种胞外或细胞壁相关蛋白酶[16]、纤维素酶、木聚糖酶[17]以及聚酮类化合物、细菌素等多种抗菌物质[18],还具有促进养殖动物生长、降解环境废弃物、净化水质等作用[19]。为探究菌株NWMCC0137对血液中蛋白质降解的作用机制,通过DNBSEQ平台与第三代测序技术PacBio测序平台对其进行全基因组测序,在获得基因序列的基础上,利用GO、COG、KEGG、CAZy等数据库进行基因功能注释。并通过比较基因组学更好地了解菌株NWMCC0137分类地位及其与其他枯草芽孢杆菌的差异性,揭示其高效降解屠宰动物血液蛋白的作用机制,为后期的生产应用提供理论依据。
1 材料与方法
1.1 试验材料
1.1.1 菌株 本试验使用的菌株为B.subtilis NWMCC0137,由课题组前期分离所得。保存于西北民族大学生物医学研究中心中国-马来西亚国家联合实验室。
1.1.2 相关试剂 LB培养基:琼脂粉18.0 g/L,胰蛋白胨10.0 g/L,酵母粉5.0 g/L,NaCl 10.0 g/L,pH值7.4(所用培养基中试剂均为分析纯);细菌基因组DNA提取试剂盒(TaKaRa MiniBEST Universal Genomic DNA Extraction Kit Ver.5.0,TaKaRa公司产品)。
1.2 方法
1.2.1 菌株基因組的提取及测序 将菌株在LB固体培养基复苏培养24 h,取单菌落接入LB液体培养基,于30 ℃,150 r/min条件下培养至对数生长期,使用细菌基因组DNA提取试剂盒提取全基因组DNA。提取的DNA经检测符合条件后送至深圳华大基因股份有限公司通过第二代DNBSEQ平台与第三代PacBio平台进行基因组测序。
1.2.2 基因组组分与功能分析 使用SMRT Link V5.0.1软件(https://www.pacb.com/support/software-downloads/)进行数据组装。使用Glimmer(V3.02)软件(http://www.cbcb.umd.edu/software/glimmer/)[20]预测编码基因。利用RNAmmer(V1.2)软件(http://www.cbs.dtu.dk/services/RNAmmer/)预测rRNA[21],利用tRNAscan-SE(V1.3.1)软件预测tRNA[22],并与Rfam(V9.1)数据库(http://rfam.sanger.ac.uk/)比对得到sRNA[23];使用CRISPR CasFinder软件(V4.2.19)识别CRISPRs,得到DRs和Spacers;使用Tandem Repeat Finder(V4.04)软件[24]预测串联重复序列,根据重复单元长度及数目筛选出其中的微卫星以及小卫星序列,软件的参数设置为:2778010502000-d-h。Tandem Repeat Finder查找算法是一个两阶段查找算法,由检测阶段和分析阶段两部分组成,算法首先会基于重复片段的概率模型的统计信息对序列进行检测并得到可能满足条件的候选重复片段,之后通过对候选结果的分析和筛选得到最终的查找结果。并且Tandem Repeat Finder查找算法不需要预先知道任何如模式组成、模式长度、模式重复次数等关于模式的已知信息,运行速度快、耗时少、重复序列准确,能够满足数据分析的要求。
基因功能注释使用Diamond软件与Gene Ontology(GO)、Cluster of Orthologous Groups of proteins(COG)、Kyoto Encyclopedia of Genes and Genomes(KEGG)、Carbohydrate-Active Enzymes Database(CAZy)等数据库对比得到对应的功能注释信息。
1.2.3 比较基因组分析 利用Ortho Finder(v2.2.6,Genome Biology 2019,https://github.com/davidemms/OrthoFinder)和参考基因组(https://www.ncbi.nlm.nih.gov/)找出多个同源基因,通过Ortho Finder软件默认参数构建系统发育树,其中进化树可视化采用的是Mega最大简约法模型,参数为默认。使用MUMmer软件(V3.22)进行共线性分析。通过antiSMASH version(V6.1.1,secondarymetabolites.org)对菌株NWMCC0137进行次级代谢产物预测。
2 结果与分析
2.1 B.subtilis NWMCC0137全基因组概况
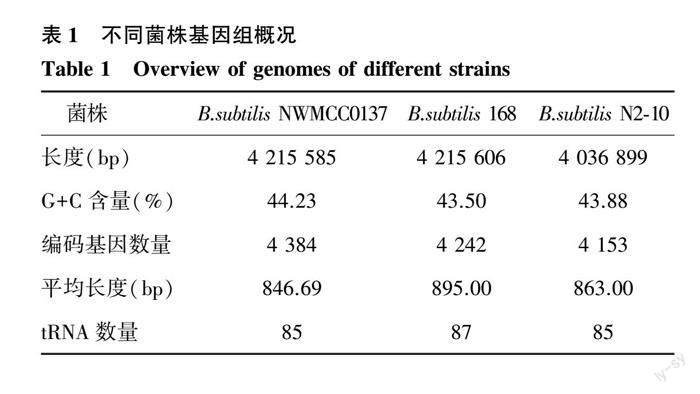
B.subtilis NWMCC0137基因组长度为4 215 585 bp,G+C含量为44.23%,无质粒。基因组共有4 384个编码基因,编码基因总长度为3 711 885 bp,平均长度846.69 bp,占基因组的88.05%。串联重复序列(Tandem repeat,TR)55个,重复序列单元大小为12~253 bp,总长度4 141 bp,占基因组的0.0982%。小卫星DNA(Minisatellite DNA)42个,单元大小为15~60 bp,总长度2 458 bp,占基因组的0.0583%。将NWMCC0137基因组完整测序数据提交至GenBank数据库,登录号为CP103066。将B. subtilis NWMCC0137与另外2株枯草芽孢杆菌进行差异性比较分析结果(表1)表明,NWMCC0137与B. subtilis 168的基因组长度和G+C含量占比都很接近,但NWMCC0137的编码基因数却比B.subtilis 168多142个。B.subtilis N2-10[25](GenBank登录号CP098417)的基因组长度小于NWMCC0137,菌株NWMCC0137的G+C含量占比也大于B.subtilis N2-10。
2.2 B.subtilis NWMCC0137基因组组分分析
非编码RNA(ncRNA)是一类可直接在RNA水平对生命活动发挥作用的RNA分子。NWMCC0137基因组含tRNA、rRNA、sRNA 3种非编码RNA,其中tRNA 85个,5s rRNA、16s rRNA及23s rRNA均为10个,sRNA 34个。B.subtilis NWMCC0137的基因组圈图如图1所示。由于DNA单链中G与C含量不一定相同,单链中G与C含量的偏移称为GC-Skew,因此第8圈的GC-Skew值是用来衡量在DNA单链中碱基G和C相对含量的不同。GC-Skew+表示G含量大于C,GC-Skew-表示G含量小于C。DNA复制中的前导链(Leading strand)富含G和T,而滞后链(Lagging strand)中的A和C偏多,表现出碱基组成上的不对称性。因此从复制起点延伸的前导链中是GC-Skew+,而在滞后链中为GC-Skew-。
2.3 B.subtilis NWMCC0137基因功能分析
将全基因组数据与GO、KEGG和COG等数据库进行比对注释。B.subtilis NWMCC0137基因组中被注释到GO数据库的基因数目为2 555个(58.28%),被注释到COG的基因数目为3 207个(73.15%),KEEG数据库中被注释到有具体Pathway的基因数目为2 609个(59.51%),NR数据库中被注释到的基因数目为4 381个(99.93%),CAZy数据库中被注释到的基因数目为152个(3.46%),VFDB数据库中被注释到的基因数目为206个(4.69%)。
2.3.1 GO數据库注释 B.subtilis NWMCC0137基因功能GO数据库注释结果如图2所示,基因被注释到与生物过程相关的类别最多,有19种,前3种分别是细胞过程(1 407个基因)、代谢过程(1 347个基因)和定位(387个基因)。基因被注释到与分子功能相关的类别有12种,基因数量最多的前两类是催化活性和结合。细胞组分中与细胞组织相关的功能注释基因最多,为716个。GO数据库中还注释到抗逆性相关的基因,如孢子形成(GO:0043934)、萌发(GO:0030435);此外,菌株还含有嗜铁素生物合成基因(GO:0019290)。GO数据库注释结果表明,该菌株不仅在代谢过程中结合、转运和催化等方面具有较强能力,还具有抗逆、抗真菌等特性。
2.3.2 COG数据库注释 根据B.subtilis NWMCC0137基因功能的COG数据库注释结果(图3),其中参与氨基酸转运与代谢的基因有329个;关于碳水化合物的运输和代谢的基因,有327个;与转录相关的有330个基因。同时还发现有232个基因与细胞壁、细胞膜的生物反应有关,说明菌株具有较强的生成细胞膜的能力。还有102个基因与防御机制有关,这些基因保障了菌株在复杂环境中的生存能力,使菌株能够在下水道污泥中存活。在COG数据库注释结果中,菌株NWMCC0137的基因功能主要被注释到氨基酸、碳水化合物、无机离子的转运与代谢3个途径。
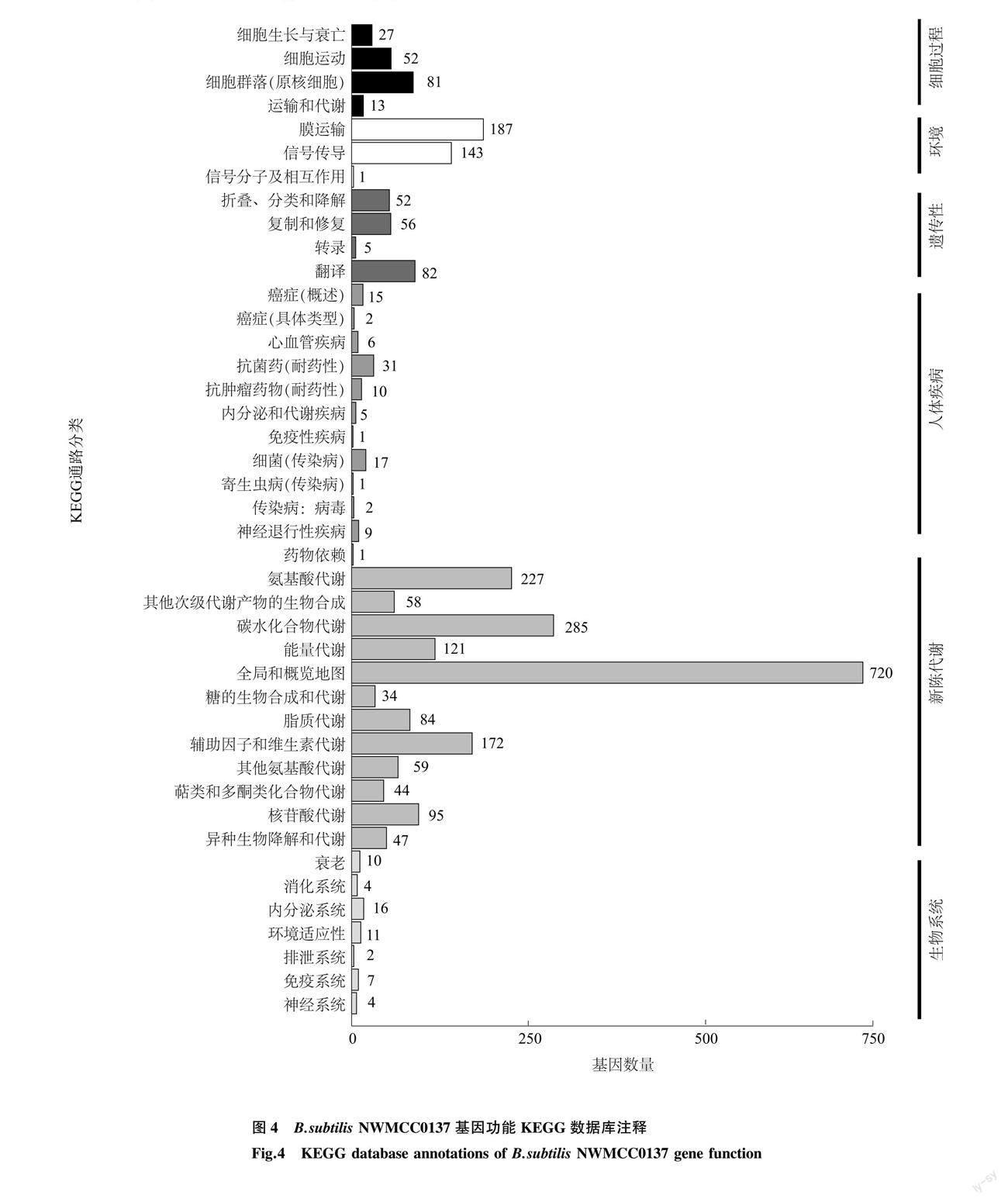
2.3.3 KEGG数据库注释 B.subtilis NWMCC0137基因组与KEGG数据库比对,共有2 609个基因分别在6大功能42个通路上得到功能注释(图4)。在新陈代谢方面被功能注释的基因最多,有1 710个基因。12个代谢通路中,与碳水化合物代谢相关的基因有285个,与氨基酸代谢相关的基因有227个。还有331个基因在环境信息处理上得到功能注释,其中与膜运输相关的基因有187个,与信号传导相关的基因有143个。在KEGG数据库中,被注释到的数量最多的是与碳水化合物代谢相关的基因,其次是与氨基酸代谢相关的基因,还注释到44个与萜类和聚酮类化合物代谢相关的基因。这些基因控制着表面活性素生物合成以及丰原素合成。其中与表面活性素(Surfactin family)相关的基因有3个,分别为srfAA(GL000364)、srfAB(GL000365)、srfAC (GL000366);与丰原素(Fengycin family )相关的基因有6个,分别为ppsA(GL001976)、ppsB(GL001975)、ppsC (GL001973)、ppsD(GL001972、GL001974)、ppsE(GL001971)。
根据前期研究结果,菌株NWMCC0137所产的中性蛋白酶具有半胱氨酸蛋白酶和丝氨酸蛋白酶的性质,可裂解大分子蛋白质中的肽键,使之成为小分子寡肽[26],在水解反应过程中发挥着重要作用。通过NWMCC0137基因组与KEGG数据库比对分析可得,NWMCC0137基因组中包括枯草杆菌蛋白酶AprE(GL001852,EC 3.4.21.62)、中性金属蛋白酶NprE(GL001588,EC 3.4.24.28)、丝氨酸蛋白酶Epr(GL004096,EC 3.4.21.-)和Vpr(GL004062,EC 3.4.21.-)、杆状肽酶F(Bpr, GL001652,EC 3.4.21.-)、中性蛋白酶B(NprB, GL001176,EC 3.4.24.-)、细胞壁相关蛋白酶 (WprA,GL001142,EC 3.4.21.-)编码基因。其中编码丝氨酸蛋白酶的基因有8个(表2),分别是丝氨酸蛋白酶Do(Serine protease Do)、主要细胞内丝氨酸蛋白酶(Major intracellular serine protease)、丝氨酸蛋白酶AprX(Serine protease AprX)、膜结合丝氨酸蛋白酶ClpP类(Membrane-bound serine protease)、丝氨酸蛋白酶(Serine protease)、微量细胞外丝氨酸蛋白酶Vpr(Minor extracellular serine protease Vpr)。推断这些基因是枯草芽孢杆菌NWMCC0137高效降解屠宰动物血液蛋白的基础。
2.3.4 碳水化合物相关酶(CAZy)基因数据库注释 将B.subtilis NWMCC0137的基因组序列与碳水化合物相关酶数据库进行注释比对,注释结果显示,编码CAZy的基因有152个,占基因总数的3.46%。其中,NWMCC0137基因组中有68个糖苷水解酶(GHs)编码基因、43个碳水化合物酶结合模块(Carbohydrate-binding modules,CBMs)编码基因、31个编码糖基转移酶(GTs)基因、13个碳水化合物酯酶(CEs)编码基因、7个多糖裂解酶(PLs)编码基因、1个辅助活性酶(AAs)编码基因。在碳水化合物酶结合模块中α-淀粉酶(EC 3.2.1.1)与分支酶(EC 2.4.1.18)具有最高的覆盖度。在糖苷水解酶中,覆盖度较高的有分支酶、几丁质酶(EC 3.2.1.14)、β-1,4-木聚糖内切酶(EC 3.2.1.8)、双功能胞壁酰胺酶DD内肽酶(EC 3.2.1.17)、G型溶菌酶(EC 3.2.1.17)。这些酶在菌株生长代谢过程中发挥着重要作用。菌株NWMCC0137基因组中还含有内切葡聚糖酶(EC 3.2.1.4)、β-葡萄糖苷酶(EC 3.2.1.21)等酶的编码基因,从基因层面进一步了解了菌株NWMCC0137在碳水化合物代谢中的潜力。
2.4 系统发生分析与共线性分析
根据全基因组序列,使用TreeBeST软件对B.subtilis 168、B.subtilis NWMCC0137、B.subtilis ATCC 11774、B.subtilis CGMCC2108、B.subtilis ATCC 21228、B. tequilensis EA-CB0015、B. velezensis JS25R构建系统发育进化树(图5)。从图中可以看出B.subtilis NWMCC0137与枯草芽孢杆菌168聚合在同一分支,说明这两者之间的进化距离最为接近。
为研究B.subtilis NWMCC0137分别与B.subtilis 168、B.subtilis ATCC11774基因组的整体相似性,采用MUMmer 软件对它们进行共线性分析(图6)。通过共线性分析,可以得到测序基因组与参考基因组之间遗传基因变化情况,以及菌株基因组在进化过程中发生的结构性变异情况。枯草芽孢杆菌B.subtilis NWMCC0137分别与B.subtilis 168、B.subtilis ATCC11774均具有良好的共线性,存在着大量同源性基因,说明菌株之间的进化关系较近。而B.subtilis NWMCC0137与B.subtilis ATCC11774之间的遗传差异较为明显。
2.5 次级代谢产物合成基因注释结果
通过antiSMASH 对菌株NWMCC0137进行次级代谢产物合成基因注释(表3)。该菌株共有13种次级代谢产物合成基因簇被注释,其中包括非核糖體多肽类(NRPS)、萜烯(Terpene)、塞克肽类(Sactipeptide)、Transatpks、第三类聚酮合酶(T3PKS)、甘氨酸(Glycocin)、内酯(Beta-lactone)、表肽(Epipeptide)、兰提肽(Ranthipeptide)和CDPS基因簇。在已鉴定的基因簇类型中,基因簇相似性为100%的有产孢致死因子(Sporulation killing factor)、聚酮类化合物(Bacillaene)、丰原素(Fengycin)、枯草芽孢杆菌素168 (Sublancin 168)、儿茶酚型嗜铁素(Bacillibactin)、芽孢杆菌素(Subtilosin A)、杆菌溶素(Bacilysin)合成基因簇。有3类基因簇未能注释到明确的代谢产物。非核糖体多肽类(NRPS)基因簇与表面活性素(Surfactin)基因簇相似度为83%,表肽(Epipeptide)基因簇与泰蓝抑素A(Thailanstatin A)基因簇相似度仅为10%,表明菌株NWMCC0137可能合成新的代谢产物。
3 讨论
屠宰场废弃动物血液不能得到价值化利用,导致大量蛋白质资源被浪费。课题前期搜集到的B.subtilis NWMCC0137是产蛋白酶的优势菌株,能够有效将血液中的大分子蛋白质分解为小分子多肽及氨基酸。由于传统方法难以挖掘该菌产中性蛋白酶关键基因,因此对该菌进行全基因组测序以挖掘其基因资源。
B.subtilis NWMCC0137基因组在GO数据库中被注释到与生物过程、分子功能有关的基因占比多,这些基因对菌株的生物学过程影响较大。B.subtilis NWMCC0137基因组在COG数据库中被注释到与氨基酸、碳水化合物转运与代谢有关的基因较多。B.subtilis NWMCC0137基因组在KEGG数据库中被注释到与代谢途径有关的基因最多,其中碳水化合物代谢与氨基酸代谢是被注释到基因最多的两种代谢途径。3个数据库的注释结果一致。KEGG数据库中还注释到B.subtilis NWMCC0137基因组中的7种胞外蛋白酶编码基因,最主要的胞外蛋白酶是由AprE编码的枯草杆菌蛋白酶和NprE编码的中性金属蛋白酶[27],枯草杆菌蛋白酶可将环境中的不溶性蛋白质降解为多种寡肽和氨基酸,以此来生产含氮化合物供细菌本体利用。此外丝氨酸蛋白酶Epr和Vpr、杆状肽酶F、中性蛋白酶B、细胞壁相关蛋白酶WprA编码基因,这些基因也是NWMCC0137能够高效降解血液中蛋白质的基础。而且前期研究结果表明,菌株NWMCC0137所产蛋白酶具有金属蛋白酶、半胱氨酸蛋白酶和丝氨酸蛋白酶的特性。菌株代谢产生丝氨酸蛋白酶表明AprX基因也是影响芽孢杆菌属产胞外蛋白酶的重要基因,因此NprE与AprX均是影响菌株NWMCC0137产酶的关键基因。贾仲昕等[28]在细菌生长的后期阶段对蛋白酶基因的表达与产酶能力进行了检测,结果表明除枯草杆菌蛋白酶AprE和中性金属蛋白酶NprE编码基因外,AprX也是影响芽孢杆菌属细菌产胞外蛋白酶能力的重要基因[29]。前人研究结果验证了本研究结果。此外,蛋白酶是一种诱导酶,在合成过程中受多方面因素的影响。在CAZy数据库注释结果中,菌株NWMCC0137含有许多与α-淀粉酶、β-1,4-木聚糖内切酶、G型溶菌酶、几丁质酶、β-葡萄糖苷酶、内切葡聚糖酶相关的编码基因。菌株NWMCC0137可产生β-葡萄糖苷酶,内切葡聚糖酶、β-1,4-木聚糖内切酶等纤维素酶。动物饲料中含有大量的纤维素,纤维素无法被动物高效利用,枯草芽孢杆菌可作为添加剂应用于饲料中[30]。将枯草芽孢杆菌混入饲料后,在微生物的分解作用下纤维素能够被动物机体快速吸收,转变为动物生长发育过程中的供能物质,枯草芽孢杆菌产生的碳水化合物分解酶在这一过程中有着不可或缺的作用。G型溶菌酶不但具有广谱抗菌作用,在抗病毒与抗肿瘤活性中也有廣泛的应用[31]。此外还注释到与几丁质酶等细胞壁降解酶相关的编码基因,推测菌株NWMCC0137可通过降解病原真菌的细胞壁来抑制病原微生物的生命活动,从而完成抑菌作用。
在抗菌物质合成方面,NWMCC0137菌株有13个合成基因簇,包括10个已知基因簇和3个未知基因簇,菌体在生长过程中可以产生枯草菌素、丰原素、杆菌溶素、表面活性素等活性物质,在致病菌的防治中具有很大潜力。Bacillaene是一类能抑制蛋白质合成的聚酮类化合物,因此对细菌和真菌均有良好的抑制作用。丰原素是一种具有很强抗真菌活性的环状脂肽类物质[32]。枯草芽孢杆菌素168是一类具有广谱抗菌作用的抗菌肽,在寄生虫防治方面具有突出作用[33]。儿茶酚型嗜铁素是一种对铁离子有高亲和性的螯合剂,而铁离子是病原菌生长过程中必不可少的元素,因此在儿茶酚型嗜铁素的作用下致病菌的生长受到抑制[34]。还有研究结果表明,儿茶酚型嗜铁素具有控制微生物间竞争的作用[35]。杆菌溶素是一类双肽抗菌物质,对真菌和细菌都表现出强大的抑菌活性[36]。表面活性素是由芽孢杆菌属细菌产生的一种环脂肽类小分子活性物质,不仅具有良好的抗菌、抗病毒活性[37],还展现出了灭蚊活性[38]。抗菌物质的存在也为菌株NWMCC0137在生物防治中的应用提供了依据。
通过GO、COG、KEGG数据库注释,在B.subtilis NWMCC0137基因组中发现,与氨基酸转运和代谢、碳水化合物运输和代谢、转录代谢途径有关的基因占比较多。在KEGG数据库注释中,还发现了7种蛋白酶编码基因,而在CAZy数据库注释中,发现与碳水化合物降解、生物合成等途径相关的编码基因也十分丰富。推测这些酶的存在是菌株NWMCC0137能够高效水解屠宰动物血液蛋白的内在因素。表面活性素、丰原素、儿茶酚型嗜铁素和杆菌溶素等次级代谢产物对致病菌具有较好的防控效果,在水解过程中能够抑制致病微生物的生长从而减少营养物质的损失。
4 结论
本研究采用第二代DNBSEQ平台与第三代PacBio平台高通量测序技术对NWMCC0137进行全基因组测序,全基因组长度为4 215 585 bp,G+C含量为44.23%,同时将完整的基因组数据与GO、KEGG、COG和CAZy等数据库进行功能注释,从分子生物学的角度探究NWMCC0137的生物学特性以及基因组功能特性。在KEGG数据库中B.subtilis NWMCC0137基因组被注释到7种蛋白酶基因以及与萜类和聚酮类化合物代谢相关的基因。在CAZy数据库中B.subtilis NWMCC0137基因组被注释到α-淀粉酶、几丁质酶、分支酶、β-1,4-木聚糖内切酶、G型溶菌酶、β-葡萄糖苷酶、内切葡聚糖酶等多种酶的编码基因,这些基因在菌株生长过程中发挥着重要作用。通过antiSMASH分析,菌株NWMCC0137含有多个次级代谢产物合成基因簇,因此对抑制致病菌的生长具有较好的效果。本研究从基因层面揭示了菌株NWMCC0137的遗传信息,为该菌株用于降解屠宰动物血液蛋白及生物防控提供了数据支持。
参考文献:
[1] BAH C S, CARNE A, MCCONNELL M A, et al. Production of bioactive peptide hydrolysates from deer,sheep,pig and cattle red blood cell fractions using plant and fungal protease preparations[J]. Food Chemistry,2016,202:458-466.
[2] LYNCH S A, MULLEN A M, O′NEILL E E, et al. Harnessing the potential of blood proteins as functional ingredients:A review of the state of the art in blood processing[J]. Comprehensive Reviews in Food Science and Food Safety,2017,16(2):330-344.
[3] 马咸莹,马石霞,周雪雁,等. 一株降解牛血液蛋白细菌的筛选及酶学性质研究[J]. 微生物学杂志,2021,41(1):43-51.
[4] BHAT Z F, KUMAR S, BHAT H F. Bioactive peptides of animal origin:a review[J]. Journal of Food Science and Technology,2015,52(9):5377-5392.
[5] WANG L, ZHANG B R, HAN J, et al. Optimization of hydrolysis condition of blood meal by Bacillus subtilis with response surface methodology[J]. International Biodeterioration & Biodegradation,2015,104:112-117.
[6] ZHENG Y B, ZHANG H, WANG D B, et al. Strain development and optimized fermentation conditions for blood meal using Aspergillus niger and Aspergillus oryzae[J]. Journal Microbiological Methods,2014,101:70-80.
[7] 郭秀云,张开臣,孟庆丰,等. 饲用血粉加工工艺及其产品质量研究进展[J]. 中国家禽,2019,41(19):50-54.
[8] 庞 伟. 猪血多肽的发酵法制备及其ACE抑制活性研究[D]. 合肥:合肥工业大学,2009.
[9] 陶艳华,姚大偉,王 政,等. 血红蛋白降解菌的分离筛选与鉴定[J]. 食品与生物技术学报,2009,28(6):854-857.
[10]YAO D W, QU J, CHANG P W, et al. Production and characterization of alkaline protease from hemoglobin-degrading Bacillus pumilus NJM4 to produce fermented blood meal[J]. Journal of Food Science and Technology,2012,49(5):626-631.
[11]崔玮琪,王红丽,王 莘. 发酵猪血菌株X-17发酵条件优化[J]. 吉林农业大学学报,2015,37(4):488-492.
[12]马咸莹. 分解牛血液蛋白菌株的筛选及其产酶特性研究[D]. 兰州:西北民族大学,2021.
[13]ZHANG H, ZHANG B R, ZHENG Y B, et al. Neutral protease expression and optimized conditions for the degradation of blood cells using recombinant Pichia pastoris[J]. International Biodeterioration and Biodegradation,2014, 93:235-240.
[14]马子豪,向 军,纪晓岚,等. 1株降解牦牛血液菌株的分离鉴定[J]. 现代畜牧兽医,2022,8(8):1-5.
[15]EARL A M, LOSICK R, KOLTER R. Ecology and genomics of Bacillus subtilis[J]. Trends in Microbiology,2008,16(6):269-275.
[16]CHATTERJEE J, GIRI S, MAITY S, et al. Production and characterization of thermostable alkaline protease of Bacillus subtilis (ATCC 6633) from optimized solid-state fermentation[J]. Biotechnology and Applied Biochemistry,2015,62(5):709-718.
[17]MORIKAWA M. Beneficial biofilm formation by industrial bacteria Bacillus subtilis and related species[J]. Journal of Bioscience and Bioengineering,2006,101(1):1-8.
[18]袁 萍,江明锋,官久强,等. 基于枯草芽孢杆菌多组学数据全基因组规模代谢模型与蛋白质降解模拟[J]. 东北农业大学学报,2020,51(11):61-69.
[19]GUO X H, LI D F, LU W Q, et al. Screening of Bacillus strains as potential probiotics and subsequent confirmation of the in vivo effectiveness of Bacillus subtilis MA139 in pigs[J]. Antonie Van Leeuwenhoek,2006,90(2):139-146.
[20]DELCHER A L, BRATKE K A, POWERS E C,et al. Identifying bacterial genes and endosymbiont DNA with Glimmer[J]. Bioinformatics,2007,23(6):673-679.
[21]LAGESEN K, HALLIN P, RDLAND E A, et al. RNAmmer:consistent and rapid annotation of ribosomal RNA genes[J]. Nucleic Acids Research,2007,35(9):3100-3108.
[22]LOWE T M, EDDY S R. TRNAscan-SE:a program for improved detection of transfer RNA genes in genomic sequence[J]. Nucleic Acids Research,1997,25(5):955-964.
[23]GARDNER P P, DAUB J, TATE J G, et al. Rfam:updates to the RNA families database[J]. Nucleic Acids Research,2009,37(suppl 1):136-140.
[24]BENSON G. Tandem repeats finder:a program to analyze DNA sequences[J]. Nucleic Acids Research,1999,27(2):573-580.
[25]孙悦龙,刘 浩,朱宝成,等. 枯草芽孢杆菌N2-10的基因组测序和比较基因组分析[J]. 微生物学通报,2023,50(1):131-147.
[26]熊 科,邓 蕾,柳佳芸,等. 蛋白酶水解底物特异性机制研究进展[J]. 食品与发酵工业,2019,45(19):292-298.
[27]CONTESINI F J, MELO R R D, SATO H H. An overview of Bacillus proteases:from production to application[J]. Critical Reviews in Biotechnology,2017,38(3):321-334.
[28]TOYMENTSEVA A A, MASCHER T, SHARIPOVA M R. Regulatory characteristics of Bacillus pumilus protease promoters[J]. Current Microbiology,2017,74:550-559.
[29]贾仲昕,赵佳男,季 芳,等. 芽孢杆菌蛋白酶基因的比较基因组学分析[J]. 广东农业科学,2022,49(5):110-117.
[30]王宏浩,张高瑜,逯梦凡,等. 枯草芽孢杆菌ZX-11培养条件及抑菌性能研究[J]. 中国酿造,2022,41(7):138-143.
[31]赵春晖,徐 玮,冯 斌,等. 七鳃鳗g型溶菌酶的分子特征和抗菌活性[J]. 中国生物化学与分子生物学报,2015,31(5):495-504.
[32]GIMENEZ D, PHELAN A, MURPHY C D, et al. Fengycin a analogues with enhanced chemical stability and antifungal properties[J]. Organic Letters,2021,23(12):4672-4676.
[33]劉扬科,赵天效,卢晓颖,等. 枯草芽孢杆菌YT168-6产抗菌肽sublancin的发酵工艺优化及培养基筛选的研究[J]. 中国畜牧兽医,2021,48(9):3232-3241.
[34]戚家明,孙杉杉,张东旭,等. 芽孢杆菌BS-6基于全基因组数据的分类鉴定及拮抗能力分析[J]. 生物技术通报,2019,35(10):111-118.
[35]DIMOPOULOU A, THEOLOGIDIS I, BENAKI D, et al. Direct antibiotic activity of bacillibactin broadens the biocontrol range of Bacillus amyloliquefaciens MBI600[J]. mSphere,2021,6(4):e0037621.
[36]NANNAN C, VU H Q, GILLIS A, et al. Bacilysin within the Bacillus subtilis group:Gene prevalence versus antagonistic activity against gram-negative foodborne pathogens[J]. Journal of Biotechnology,2021,327:28-35.
[37]SANTOS V S V, SILVEIRA E, PEREIRA B B. Toxicity and applications of surfactin for health and environmental biotechnology[J]. Journal of Toxicology and Environmental Health,Part B:Critical Reviews,2018,21(6/7/8):382-399.
[38]GEETHA I, MANONMANI A M. Surfactin:a novel mosquitocidal biosurfactant produced by Bacillus subtilis ssp. subtilis (VCRC B471) and influence of abiotic factors on its pupicidal efficacy[J]. Letters in Applied Microbiology,2010,51(4):406-412.
(责任编辑:成纾寒)

