基于SSR分子标记的154份桃品种遗传多样性分析
殷纪伟 韩贝贝 马莹雪 武星廷 徐振江 姜建福 陈昌文 韩瑞玺


摘要:利用简单重复序列(SSR)分子标记对桃种质资源的遗传多样性和群体结构进行分析,构建桃品种的分子数据库,为桃品种的鉴定提供技术依据。利用10个SSR标记对154份桃品种进行指纹采集,通过聚类分析和群体结构分析研究其遗传多样性。结果表明,10对SSR引物共检测出140个等位变异,变异范围为8~27;共获得304个基因型,变化范围为17~59;引物多态性信息含量(PIC)变化范围为0.510 9~0.824 2,申请品种保护和登记的品种遗传多样性较已知品种低。根据Neis遗传距离进行非加权组平均法(UPGMA)聚类分析,供试品种可划分为5个大类,各品种间的遗传距离在0.029 3~1.000 0之间,其中2对品种未区分开。对供试品种进行群体结构分析,发现可以划分为4个亚群,大部分材料的亲缘关系比较单一。方差分析结果表明,10%的遗传变异来自群体间,72%的遗传变异来自群体内部,群体内的变异大于群体间。154份桃品种的遗传多样性水平适中,群体间的遗传分化程度处于中等水平,同一育种单位的品种遗传距离较近。研究结果可为不同类型桃育种创新、分子指纹数据库的构建及品种鉴定提供参考依据。
关键词:桃;SSR;遗传多样性;群体结构
中图分类号:S662.102文献标志码:A
文章编号:1002-1302(2023)16-0018-08
收稿日期:2022-10-11
基金項目:物种品种资源保护项目(编号:h20210472);农产品质量安全标准体系建设(编号:2130109)。
作者简介:殷纪伟(1998—),男,安徽宣城人,硕士研究生,从事分子生物学研究。E-mail:jiwyin@163.com。
通信作者:韩瑞玺,博士,高级农艺师,主要研究方向为植物新品种保护和DNA分子技术应用。E-mail:wudifeixue007@163.com。
桃(Prunus persica L.)是蔷薇科(Rosaceae)李属(Prunus)的一种重要落叶果树,是全球第三大重要的温带水果[1]。中国拥有世界上最长的桃栽培历史[2],种质资源比较丰富,其种植面积、产量均居世界首位[3]。利用DNA分子标记技术对桃进行品种鉴定及种质资源的遗传多样性研究,对我国桃种质资源利用和育种创新具有重大意义。随着基因组学的不断发展,基于DNA的遗传标记在植物分子研究中被广泛应用,简单重复序列(SSR)分子标记技术由于具有可重复性好、共显性遗传等优点,在DNA指纹分析和遗传多样性分析方面有重要的应用价值[4]。目前,SSR分子标记技术已经被用于鉴定葡萄[5]、柑橘[6]、苹果[7]、猕猴桃[8]、樱桃[9]等果树品种或种质资源。近年来SSR分子标记技术被广泛应用于各项研究中,如品种鉴定、遗传图谱构建、遗传多样性研究等。Cheng等利用7对SSR引物构建了32个桃地方品种的指纹图谱,并评估了品种的遗传多样性和亲缘关系[10];葛志刚等利用SSR标记对22个蟠桃品种及另外9种类型桃品种的遗传多样性的分析结果显示,蟠桃的遗传多样性较高[11];Xie等采用34个SSR标记分析了浙江地区的94份桃种质资源的多样性,发现引进品种的种质多样性高于地方品种[12];李雄伟等利用16个SSR标记对国内外669份桃的遗传多样性进行测试并构建了相应的分子指纹图谱[13];凌士鹏等对不同来源的桃品种进行SSR分析,发现聚类结果和品种的地理分布之间存在一定的相关性,相同地理来源的品种较为集中地聚在一起[14];根据桃果实性状,可以将桃划分为多个类型,魏姗姗等开发了18个SSR标记对桃果实的形状进行分析,发现扁球形桃类群的遗传多样性较球形丰富,对桃有无毛性状的分析结果表明,有毛类群的遗传多样性和基因杂合度高于无毛类群[15];刘伟等以60份山东省地方桃种质资源为研究材料,利用SSR标记构建了分子身份证,相关研究可为山东省地方桃品种资源鉴定和种质资源利用与保护奠定基础[16]。通过对桃种质资源的遗传多样性进行评价,可为今后桃种质资源创新和利用提供参考依据。但是,目前对于新育成的桃品种,尚未有人分析并及时构建DNA分子数据库。本研究首次以近5年申请品种权保护的桃品种为材料,并且通过果实类型进行聚类分析,前人在此方面的研究报道较少。随着国内桃新品种权申请量逐年增加,通过比较申请品种权保护品种和已知品种间的遗传多样性,不同育种单位和地区育种水平的相关研究较少。本研究构建了154份桃品种的DNA指纹数据库,并对申请品种保护的品种和已知品种间进行遗传多样性比较分析,可为加快我国桃种质资源的创新利用和桃品种权保护提供参考,为桃品种鉴定和辅助桃新品种特异性、一致性和稳定性测试(简称DUS测试)提供理论依据。
1 材料与方法
1.1 材料与试剂
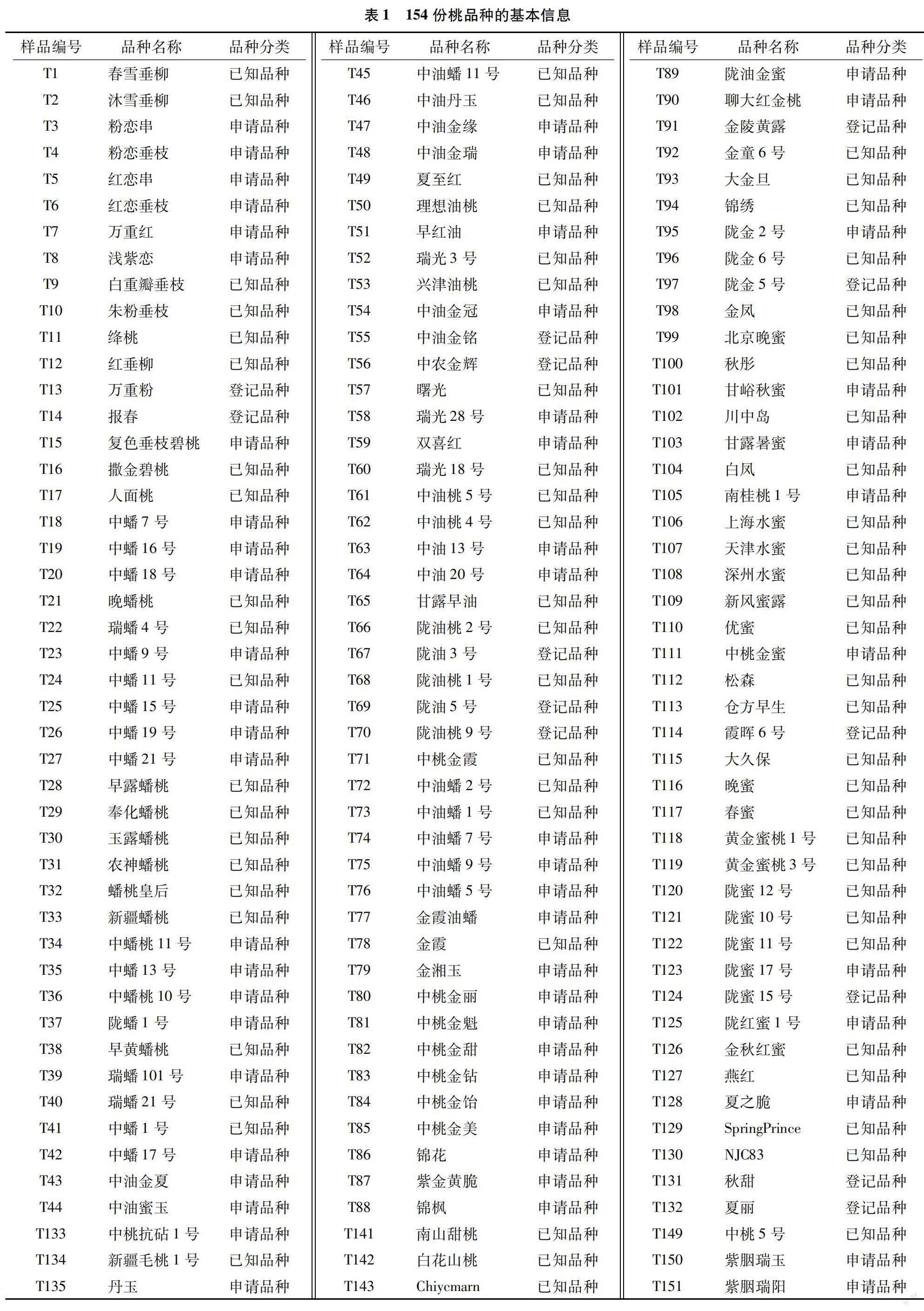
本试验于2022年在农业农村部科技发展中心植物新品种测试中心进行,试验材料包括申请新品种权保护的品种63份、登记品种13份、已知品种78份,共154份,由农业农村部植物新品种测试中心提供,供试品种的基本信息见表1。
DNA聚合酶、PCR Buffer等购于南京诺维赞生物科技股份有限公司;甲酰胺、LIZ500购于ABI公司。主要设备仪器:Nano Drop 1000微量分光光度仪、ETC-811定性PCR仪、Applied BiosystemsTM3730基因分析仪、组织研磨仪等。
1.2 SSR引物
本研究中用于检测的SSR引物选自《桃品种鉴定 SSR分子标记法》中规定的10对核心引物(表2),普通引物、荧光引物均由生工生物工程(上海)股份有限公司合成,根据等位变异大小在荧光引物5′端添加6-羧基荧光素(FAM)、六氯荧光素(HEX)、羧基四甲基罗丹明(TAMRA)荧光基团。
1.3 DNA提取和PCR扩增
采用改良的十六烷基三甲基溴化铵(CTAB)法[17]提取新鲜叶片的基因组DNA,并用2%琼脂糖凝胶电泳进行DNA的完整性检测,用紫外分光光度计Nano drop1000测定DNA的质量和浓度,并将其稀释至50 ng/μL,于-20 ℃保存。PCR扩增反应总体积为10 μL,包含1 μL 50 ng/μL DNA模板、1 μL 10×PCR Buffer、各 0.3 μL 10 μmol/L上下游引物、0.1 μL Taq DNA聚合酶、2.5 mmol/L dNTP,加 6.5 μL ddH2O补足至10 μL。PCR 扩增参考《桃品种鉴定 SSR分子标记法》中的扩增程序,具体如下:95 ℃ 5 min;95 ℃ 30 s,56 ℃ 30 s,72 ℃ 30 s,共35個循环;4 ℃保存20 min。
1.4 PCR产物检测
分别吸取1.2 μL不同荧光标记扩增产物,混匀后加超纯水稀释100倍,从稀释混合液中分别吸取1 μL加入96孔板中。每孔另外加入8.9 μL去离子甲酰胺、0.1 μL ABI GeneScan 500LIZ分子量内标,离心30 s后于94 ℃变性5 min,冷却后上机检测,具体操作步骤和流程参考仪器操作说明。
1.5 数据分析方法
用SSR Analyser (V1.2.6,http://172.16.2.68:8080/ssr3/no/index)对电泳的原始数据进行处理。用Powermarker V3.25计算不同品种间的遗传距离,并构建基于Neis遗传距离的非加权组平均法(UPGMA)聚类图。利用Ntsys 2.11进行遗传相似度分析,用Structure 2.3.4软件进行群体结构分析,假定群体数目(K值)为1~10并且逐一进行测试,每个K值重复估算20次,迭代次数5 000次,马尔可夫链蒙特卡洛方法(Markov chain monte carlo,MCMC)值为50 000次。使用lnP(D)的平均值进行种群估计,并通过Structure Harvester程序估算最佳种群数量的K值,对群体间及群体内分子变异作方差检验(AMOVA),分析群体内、群体间的遗传变异情况。
2 结果与分析
2.1 SSR标记多态性和遗传多样性分析
根据SSR标记的等位变异范围,进行多重电泳组合,本研究将10对引物分为4组来提高电泳效率,图1为品种SpringPrince的毛细管电泳结果。用SSR Analyser软件处理毛细管电泳的原始数据,从表3可以看出,10对SSR引物在154份材料中共检测出140个等位变异位点,变异范围为8~27,平均值为14;共获得304个基因型组合,变化范围为 17~59个,平均值为30.4个。多态性信息含量能够反映一个群体的遗传变异程度,本研究中,PIC值的变化范围为0.510 9~0.824 2,平均值为0.698 4,其中PIC值最高的标记是SSR125(0.824 2),最低的标记是SSR107(0.510 9),各标记的PIC值均高于0.5,说明该标准所选引物多态性较好,适用于桃品种的鉴定,能够有效反映桃品种的遗传多样性信息。供试材料的基因多样性变化范围为0.530 8~0.839 6,平均值为0.728 2,表明供试材料的遗传多样性丰富,遗传背景较为复杂。
154份桃品种中共包括63份申请保护品种、13份登记品种和78份已知品种,利用GenAlEx6.503对申请和登记的品种与已知品种进行群体遗传多样性分析(表4)。已知品种的等位基因数、有效等位基因数分别为12、4.534,Shannons信息指数(I)为1.762,期望杂合度(He)为0.752,均高于申请品种保护和登记品种,说明已知品种的群体遗传多样性比申请品种保护和登记的品种更丰富。
2.2 聚类分析
本研究通过计算各品种间的遗传距离,用UPGMA法对154份桃品种进行聚类分析,发现该聚类结果和品种类型有一定的相关性(图2)。154份桃种质被划分为5个主要类群,其中类群Ⅰ全部为观赏类桃品种,类群Ⅱ主要是蜜桃品种,类群Ⅲ主要为蟠桃品种,类群Ⅳ主要为黄桃品种,类群Ⅴ主要为油桃品种。观赏类桃与可食用桃品种间的遗传信息差异较大,单独聚为一类。而其他几个类群中存在品种类型相互交叉的现象,如油蟠桃品种在类群Ⅲ、类群Ⅴ中均有分布,类群Ⅱ中除了蜜桃品种外,还有些黄桃品种。在154份材料中,共有2对品种未区分开,分别是甘峪秋蜜(T101)和中油桃4号(T62),川中岛(T102)和中油桃5号(T61),由于该标准选用的是SSR随机标记,可能未覆盖相应的等位变异差异区域,因此从分子水平上表现出相似性。除此之外,中蟠9号(T23)和中油蟠9号(T75)、中蟠19号(T26)和中蟠1号(T41)、中桃金霞(T71)和金霞(T78)的遗传距离最小,均为0.029 3。进一步分析发现,同一育种单位的品种聚在一起,遗传距离较近,如北京市林业果树科学研究院选育的瑞蟠系列品种瑞蟠4号(T22)、瑞蟠21号(T40)和瑞蟠101号(T39),其中瑞蟠21号是由瑞蟠4号为父本杂交选育的极晚熟品种, 瑞蟠101号则是由瑞蟠21号为父本杂交选育的晚熟黄肉品种。此外遗传距离较近的还有甘肃省农业科学院林果花卉研究所的陇系列品种陇金6号(T96)、陇红蜜1号(T125)、陇蜜17号(T123)、陇蜜15号(T124)、陇蜜12号(T120)等,其中陇蜜15号和陇蜜12号都是由陇蜜9号为母本杂交选育而来。同一育种单位的品种由于遗传背景相似而聚在一起,说明该聚类结果符合实际育种现状。为了探讨利用该分子数据库在桃特异性、一致性、稳定性(DUS)测试中筛选近似品种的可行性,随机选取6个申请品种与对应的近似品种进行分析。从表5、图2可以看出,申请品种中油金夏(T43)、中油蜜玉(T44)、金京红(T153)等对近似品种的选择比较合理,二者的遗传距离较近,但是中蟠7号(T18)、中蟠9号(T23)、中桃金饴(T84)在分子水平上有更加近似的品种。
2.3 群体结构分析
为了揭示154份桃品种的群体结构,基于贝叶斯的方法分析了不同类型桃品种的群体遗传结构。结果表明,当K=4时,似然值最大,如图3-A、图3-B 所示。遗传多样性分析结果显示,将154份桃品种划分为4个亚群较为合适,分别命名为Ⅰ、Ⅱ、Ⅲ、Ⅳ(图3-C、图4)。亚群Ⅰ共有23份材料,主要是蟠桃和蜜桃品种;亚群Ⅱ共有15份材料,主要为观赏类桃品种;亚群Ⅲ有70份材料,大部分为油桃品种;亚群Ⅳ有46份材料,以蜜桃品种为主。从群体结构来看,大部分材料的亲缘关系比较单一,还有部分材料的亲缘关系比较复杂。分子变异方差分析(AMOVA)结果表明,90%的变异来自于个体间,个体间的遗传变异大于群体间的遗传变异,同时群体内的遗传变异远高于群体间的遗传变异(表6)。群体间遗传分化的程度可以通过遗传分化指数来判定[18],当0≤Fst<0.05时,表示遗传分化水平极低;当0.05≤Fst<0.15时,表明遗传分化处于中等水平;当0.15≤Fst<0.25时,表示遗传分化较大;当Fst≥0.25时,表示群体间有很大的遗传分化。本研究中,Fst=0.099,表明该群体间遗传分化程度中等。
3 讨论
SSR分子标记技术具有较高的稳定性和准确性,已被广泛应用于各类种质资源的鉴定和遗传多样性分析中。近年来,关于桃种质资源的SSR引物的研究也越来越多,Jouy等选出16对具有高多态性的标记用于品种鉴定和保护[19]。关利平等从已发表的桃基因组SSR引物中筛选出10对核心引物用于指纹库的构建[20]。本研究所用10对引物的平均PIC值为0.698 4,多态性含量均大于0.5,高于叶宇芸等研究中所用的20对SSR引物[21]。在本研究中,10对引物等位基因平均值和有效等位基因数(Na=11.05,Ne=4.168)均显著高于凌士鹏等的研究结果[14,22-23],说明本研究选用的10个SSR标记多态性较为丰富,可用于桃品种的鉴定和遗传多样性分析。相较于李雄伟等构建的分子数据库[13],本研究所用引物较少,但采用了精度更高的ABI 3730基因分析仪采集指纹,并用SSR Analyser软件处理,更利于数据的整合和可视化。从本研究中的聚类分析结果来看,报春(T14)等观赏桃类单独聚在一起,说明观赏桃与鲜食桃的遗传差异较大,其起源可能存在较大差异,与周平等的研究结果[24]相似。而油蟠类型的品种在前人研究中也很少提到,该聚类结果显示,油蟠桃类品种在油桃、蟠桃群体中均有分布,说明油桃、蟠桃这2类群体间的杂交可以创制更多新种质。各亚群既有独立进化,又有部分品种出现高度遗传混合,亚群内品种间遗传相似度较高,尤其是对于同一育种单位、同一地区的品种。因此,将不同类型、不同地域的桃品种间进行杂交育种,有利于桃种质资源的创新。AMONA分析结果显示,群体间的遗传变异小于群体内,与王淋等的研究结果[3]一致,这种现象在核桃[25]、油桐[26]等木本植物中也有發现,这也与多年生木本植物的遗传变异规律[27-28]相符。
DNA指纹数据库的构建在品种管理和品种保护方面具有重要的应用价值。本研究利用10个SSR标记构建了154份桃的DNA指纹数据库,一方面可用于品种真实性的鉴定、品种管理和维权等,另一方面还可辅助DUS测试中近似品种的筛选。植物新品种保护条例规定,授权品种必须满足特异性(distinctness)、一致性(uniformity)和稳定性(stability)的要求。近似品种选择是否合理会直接影响测试品种特异性的审查结果,而申请者了解的品种有限,原则上近似品种应从世界范围内的已知品种中选择。通过采集申请品种和目前市场上已知品种的指纹信息,构建桃分子指纹库,再利用SSR分子标记技术辅助筛选近似品种,结合桃已知品种表型数据库,能够进一步提高近似品种选择的准确性,加快品种授权的速度,并且降低重复授权的风险。另外,还可利用DNA指纹数据库的信息,对申请品种和已知品种进行聚类分析,根据品种间的遗传距离对品种进行分类和区分,这种方法也能够较好地辅助近似品种的选择。目前,DNA指纹数据库已被应用于梨[29]、枇杷[30]等果树的近似品种的筛选和特异性判定中。
我国桃种质资源丰富,DNA分子标记技术较传统表型分析具有较高的稳定性和准确性,被广泛应用在桃种质资源鉴定和遗传多样性分析方面,但是采用的标记类型比较单一,后续可加强对桃品种研究方面新型分子标记的开发,如单核苷酸多态性(SNP)、表达序列标签微卫星(EST-SSR)、多核苷酸多态性(MNP)等,同时建立桃品种的DNA指纹库,利用分子标记构建品种特异性指纹,为桃品种鉴定和品种权保护服务。
4 结论
本研究利用10对SSR核心引物构建了154份桃品种的DNA指纹数据库,能够进行品种鉴定并辅助DUS测试中近似品种的筛选。分析申请品种与已知品种的遗传多样性和群体结构,发现已知品种的遗传多样性更加丰富,同一育种单位的品种遗传距离较近,群体间的遗传分化程度呈中等水平,可为我国桃种质资源的创新提供一定参考价值。
参考文献:
[1]Zeballos J L,Abidi W,Giménez S R,et al. Mapping QTLs associated with fruit quality traits in peach[Prunus persica (L.) Batsch]using SNP maps[J]. Tree Genetics and Genomes,2016,12(3):1-17.
[2]Cheng Z P,Huang H W. SSR fingerprinting Chinese peach cultivars and landraces (Prunus persica) and analysis of their genetic relationships[J]. Scientia Horticulturae,2009,120(2):188-193.
[3]王 淋,敖 敦,包文泉,等. 基于SSR分子标记的桃品种鉴别及指纹图谱构建[J]. 中南林业科技大学学报,2021,41(6):131-138.
[4]Guan L,Cao K,Li Y,et al. Detection and application of genome-wide variations in peach for association and genetic relationship analysis[J]. BMC Genetics,2019,20(1):1-13.
[5]王富强,李贝贝,樊秀彩,等. 葡萄品种SSR分子鉴定体系的建立及应用[J]. 果树学报,2020,37(9):1281-1293.
[6]李 益,马先锋,唐 浩,等. 柑橘品种鉴定的SSR标记开发和指纹图谱库构建[J]. 中国农业科学,2018,51(15):2969-2979.
[7]王 娟,宋尚伟. SSR标记在苹果种质资源及遗传育种研究中的应用[J]. 河南农业科学,2009(5):16-19.
[8]高建有,李洁维,罗 庆,等. 基于SSR分子标记的63份猕猴桃种质资源的遗传多样性[J]. 分子植物育种,2022,20(17):5711-5723.
[9]付 涛,王志龙,林 立,等. 樱属植物种质资源系统鉴定方法的研究[J]. 园艺学报,2015,42(12):2455-2468.
[10]Cheng Z P,Huang H W. SSR fingerprinting Chinese peach cultivars and landraces (Prunus persica) and analysis of their genetic relationships[J]. Scientia Horticulturae,2009,120(2):188-193.
[11]葛志刚,俞明亮,马瑞娟,等. 蟠桃种质SSR标记的遗传多样性分析[J]. 果树学报,2009,26(3):300-305.
[12]Xie R J,Li X W,Chai M L,et al. Evaluation of the genetic diversity of Asian peach accessions using a selected set of SSR markers[J]. Scientia Horticulturae,2010,125(4):622-629.
[13]李雄偉,孟宪桥,贾惠娟,等. 桃品种特异性荧光SSR分子标记数据库构建[J]. 果树学报,2013,30(6):924-932.
[14]凌士鹏,孙 萍,林贤锐,等. 基于SSR标记的桃种质资源遗传多样性研究[J]. 江西农业学报,2018,30(11):14-18.
[15]魏姗姗,杨敏生,梁海永. 桃品种遗传多样性SSR分析[J]. 耕作与栽培,2022,42(1):1-5.
[16]刘 伟,李 淼,李桂祥,等. 应用SSR荧光标记法构建山东地方桃种质资源分子身份证[J]. 山东农业科学,2022,54(2):6-13.
[17]Matthes N,Westphal K,Haldemann C,et al. Validation of a modified CTAB method for DNA extraction from protein-rich maize feedstuffs[J]. Journal of Consumer Protection and Food Safety,2020,15(4):331-340.
[18]Wright S. The genetical structure of populations[J]. Ann Eugen,1949,15(1):323-354.
[19]Jouy C,Gandelin M H,Guitouni C,et al. Management of peach tree reference collections:ongoing research & development program relevant to the community plant variety rights protection system[J]. Acta Horticulturae,2012,962(1):51-56.
[20]关利平,王玲玲,曹 珂,等. 桃品种鉴定的SSR核心引物筛选及其应用[J]. 中国果树,2021,6(1):33-38.
[21]叶宇芸,王清明,马建伟,等. 基于SSR标记的桃品种遗传多样性分析及遗传相似系数比较[J]. 华南农业大学学报,2017,38(3):39-45.
[22]Ali F,Behzad G,Nazari A H,et al. Assessment of the genetic diversity of almond Prunus dulcis) using microsatellite markers and morphological traits[J]. Iranian Journal of Biotechnology,2008,6(2):98-106.
[23]史红丽,韩明玉,赵彩平. 桃遗传多样性的SARP和SSR标记分析[J]. 华北农学报,2009,24(6):187-192.
[24]周 平,郭 瑞,张小丹,等. SSR分析50份桃种质资源遗传多样性[J]. 福建农业学报,2017,32(1):47-50.
[25]Massimo V,Cristina C,Steluta R,et al. Genetic diversity of walnut (Juglans regia L.) in the Eastern Italian Alps[J]. Forests,2017,8(3):81.
[26]Zhang L L,Liu X L,Peng J H. Genetic diversity and geographic differentiation of tung tree,Vernicia fordii (Euphorbiaceae),a potential biodiesel plant species with low invasion risk[J]. Agronomy,2019,9(7):402.
[27]Yeh F C,Chong D K,Yang R C. RAPD variationwithin and among natural populations of trembling aspen (Populus tremuloides Michx.) from Alberta[J]. Journal of Heredity,1995,86(6):454-460.
[28]Gillies A M,Navarro C,Lowe A J,et.al. Genetic diversity in Mesoamerican populations of mahogany (Swietenia macrophylla),assessed using RAPDs[J]. Heredity,1999,83(6):722-732.
[29]王 斐,张艳杰,欧春青,等. 梨品种SSR分子鉴定体系的建立及应用[J]. 分子植物育种,2021,19(22):7499-7509.
[30]胡文舜,邓朝军,许奇志,等. 19个枇杷杂交新品种(系)的SSR鑒定和指纹图谱构建[J]. 热带亚热带植物学报,2020,28(2):153-162.

