2021~2022年荣成海域多相赤潮的分子鉴定*
刘奎艳 王洪淑 刘 峰 陈楠生①
2021~2022年荣成海域多相赤潮的分子鉴定*
刘奎艳1, 2, 3, 4王洪淑1, 2, 3刘 峰1, 2, 3陈楠生1, 2, 3, 4①
(1. 中国科学院海洋研究所 海洋生态与环境科学重点实验室 山东青岛 266071; 2. 崂山实验室 山东青岛 266237; 3. 中国科学院海洋大科学研究中心 山东青岛 266071; 4. 中国科学院大学 北京 100049)
山东半岛濒临渤海和黄海, 海岸带复杂, 海岸线长, 近岸海域赤潮事件频发。然而, 由于部分赤潮物种的形态难以识别, 或易降解不稳定, 加上形态鉴定专业要求高, 导致致灾赤潮物种未得到准确鉴定。2021年11月至2022年4月间山东荣成海域海带养殖区暴发严重赤潮事件, 面积达1 440 km2, 导致海带白化、溃烂, 造成严重经济损失。对该海域水样固定样本的观察鉴定到两种致灾赤潮物种, 红色赤潮藻()和多纹膝沟藻(), 二者均为山东近海常见的致灾赤潮物种。该研究通过对赤潮样本中活体单细胞的显微观察和单细胞测序分析, 根据藻细胞形态特征与分子标记序列相似性鉴定到三种优势致灾赤潮物种, 包括红色赤潮藻、多纹膝沟藻和灰白下沟藻()。其中灰白下沟藻由于在固定过程中不稳定而较少得到鉴定。对该赤潮海域水样开展宏条形码分析, 除了发现上述三种致灾赤潮物种外, 还发现了另外一个致灾赤潮物种纺锤环沟藻()。纺锤环沟藻也由于在固定过程中不稳定而较少得到鉴定。由此可见, 宏条形码分析方法更加准确系统地鉴定了该赤潮事件的致灾赤潮物种。由于宏条形码分析流程比较便于规范化, 并且该方法可以获得比仅利用细胞形态特征进行鉴定, 或兼用基于形态特征和分子标记方法进行鉴定更加全面的结果, 研究表明宏条形码分析可以准确系统地鉴定组成较为复杂的多相赤潮事件的致灾赤潮物种。
赤潮; 甲藻; 宏条形码分析; 红色赤潮藻; 多纹膝沟藻
随着人类社会的进步, 人类活动对生态环境造成越来越严重的影响(周名江等, 2001; Anderson, 2008, 2012), 赤潮成为一种频发海洋生态灾害(俞志明等, 2019)。20世纪以来, 赤潮事件暴发呈增强趋势, 持续时间延长、发生规模扩大、频率增加(Hallegraeff, 2010; Zhang, 2012, 2022; McCabe, 2016), 赤潮暴发演替过程更加复杂化(俞秀霞, 2012; León- Muñoz, 2018)。赤潮已经对全球的经济持续发展、人类食品安全、海洋生态系统平衡稳定带来了严重的威胁(周名江等, 2001; Yu, 2017)。
山东省濒临渤海和黄海, 海岸线居全国第三(周健等, 2020), 属于温带季风气候, 近海水文情况复杂(周斌等, 2013), 且近年来包括养殖业、旅游业等人类活动影响增加, 加剧了水体富营养化水平(宋建杰, 2011)。在气候、海流和人类活动的多重影响下, 山东近海赤潮的规模不断扩大、暴发频率逐渐上升, 在生态环境、人类健康和经济发展多方面带来了严重负面影响。山东近海1952~2021年间共记录赤潮事件140次, 其中原因种得到鉴定的共有114次, 以单相型赤潮为主, 偶有双相型(陈楠生等, 2023)。得到鉴定的主要致灾赤潮物种包括夜光藻()、红色赤潮藻()、中肋骨条藻()、海洋卡盾藻()、球形棕囊藻()、赤潮异弯藻()、多纹膝沟藻()、具刺膝沟藻()、米氏凯伦藻()和大洋角管藻()(陈楠生等, 2023)。目前, 致灾赤潮物种的鉴定主要根据其形态学特征, 对于细胞尺寸较小、形态特征高度相似的隐存种、形态特征不稳定或可塑性高以及固定过程中细胞不稳定性的赤潮物种往往很难获得准确鉴定(黄长江等, 2000b; 甄毓等, 2006; Liu, 2020)。因此, 上述致灾赤潮物种的鉴定结果有可能并不准确或者并不全面。
基于分子标记的物种鉴定大大地提高了分辨率(Lavrinienko, 2021)。基于通用分子标记扩增子高通量测序的宏条形码分析技术也能够用于系统分析浮游植物组成(Margulies, 2005; Stoeck, 2010; 陈楠生, 2020), 可以实现对致灾赤潮物种鉴定的标准化、规范化, 具有较高的可操作性和可重复性, 规范化的宏条形码分析流程可能用于在全面系统、准确地鉴定致灾赤潮物种, 解析赤潮物种组成。
2021年11月至2022年4月在山东省荣成市的海带养殖区海域发现海水颜色异常, 赤潮面积高达1 440 km2, 养殖区内海带苗生长停滞, 部分海带出现白化、溃烂, 海带受灾面积超过93 km2(李晓东等, 2023)。本研究对此次赤潮暴发区域的表层水体样本进行致灾赤潮物种的鉴定调查, 首先通过对活体样本中浮游植物进行形态学观察初步鉴定, 再利用分子生物学手段包括单细胞扩增、环境DNA克隆文库测序以获得优势致灾物种的分子标记序列从而进行鉴定。与此同时, 利用宏条形码分析方法解析该赤潮样本内的浮游植物组成以及相对丰度。比较两种方法的鉴定结果, 证明本次赤潮事件是一次以红色赤潮藻、多纹膝沟藻、纺锤环沟藻()和灰白下沟藻()为主导的多相赤潮事件, 从而表明宏条形码分析方法可以用于准确鉴定赤潮事件的致灾赤潮物种。
1 材料与方法
1.1 样品采集及处理
2022年1月3日在山东省荣成市沿岸的赤潮暴发区域(37°22′6″N, 122°37′49″E)(图1)采集到2.5 L表层海水, 借助倒置显微镜(Nikon Ts2, 日本)观察活体水样并进行拍照(ECHO RVL-100-G, 美国)。水样经过200 μm筛绢(安平县丝网制品有限公司, 中国)过滤去除海水中的大型浮游生物及杂质。将3个平行(各平行体积0.8 L)的水样分别收集至0.22 μm聚碳酸酯膜(Millipore, 美国)上放入液氮保存直至DNA提取。
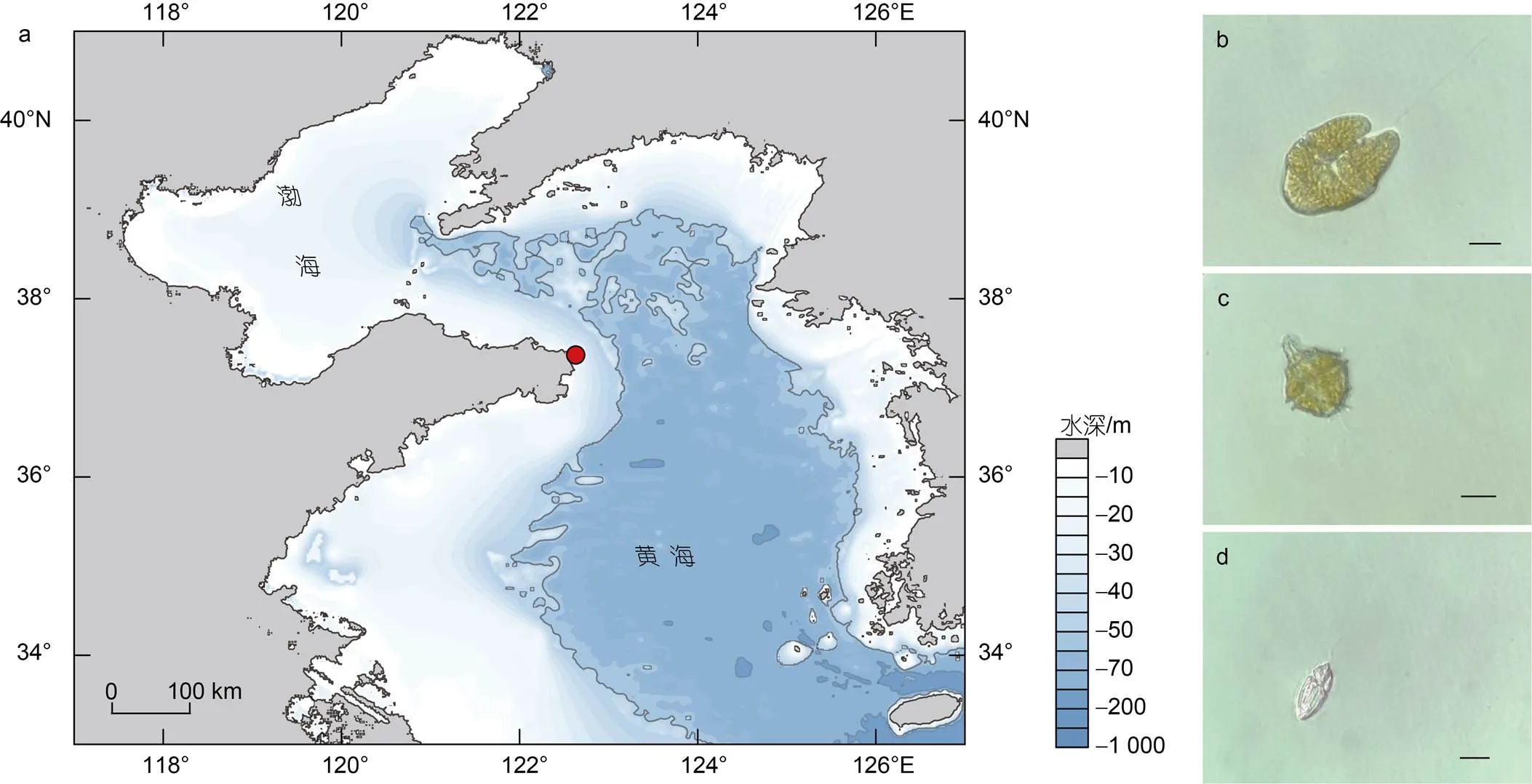
图1 赤潮样品采集位置及浮游植物优势种
注: a: 采集位置(红点); b: 镜检并分离的红色赤潮藻; c: 镜检并分离的多纹膝沟藻; d: 镜检并分离的灰白下沟藻(图b~d比例尺=10 µm)
1.2 单细胞PCR
借助毛细管单细胞分离法在倒置显微镜(Nikon Ts2, 日本)下从活体水样中挑取占主导的赤潮物种单细胞, 作为模板扩增其18S rDNA全长序列。PCR流程按照Plant Direct PCR Kit试剂盒(诺唯赞, 中国)中加热裂解法的说明操作进行, 将单细胞在10 μL Plant DirectLysis Buffer A中, 95 °C加热5 min, 加热后短暂离心, 取其2 μL上清作为25 μL扩增体系的模板。扩增引物为28F(5′-CGAATT CAA CCT GGT TGA TCC TGC CAG T-3′)和42R(5′-CCGGAT CCT GAT CCT TCT GCA GGT TCA CCT AC-3′)(Saldarriaga, 2003)。扩增条件: 98 °C预变性5 min; 然后95 °C变性10 s, 57 °C退火15 s, 72 °C延伸2 min, 共35个循环; 最后72 °C延伸5 min, 经1%琼脂糖凝胶电泳检测PCR产物。扩增产物纯化及sanger测序由生工生物工程(上海)股份有限公司完成。测序获得序列经ContigExpress软件(ThermoFisher, 美国)组装后, 共获得6条序列CNT00031-CNT00036 (登录号: OQ152428-OQ152432、OQ162335, 已提交至NCBI的GenBank数据库)。
1.3 环境样品DNA提取扩增、测序
环境滤膜样品的DNA提取使用HP Plant DNA kit试剂盒(Omega, 美国), 流程参照(Liu, 2020)。环境DNA样品用于两项分析: (1) 用于18S rDNA全长序列的扩增、克隆测序: 扩增引物为28F和42R (上述1.2单细胞PCR流程提及)。扩增条件: 94 °C预变性4 min; 然后94 °C变性1 min, 57 °C退火30 s, 72 °C延伸2 min, 共32个循环; 最后72 °C延伸10 min, 经1%琼脂糖凝胶电泳检测PCR产物。使用Hieff Clone®ZeroTOPO-TA Cloning Kit 试剂盒(上海翊圣生物科技有限公司, 中国)将纯化后产物与pESI-T载体连接, 随后转化至DH5 α感受态细胞。从中挑选出30个克隆进行第一代DNA测序(即Sanger测序), 测序引物为M13F(5′-TGTAAAACGACGGCCAGT-3′)和 M13R(5′-CAGGAAACAGCTATGACC-3′)。以上扩增产物的纯化、克隆测序由上海生工生物工程完成。测序获得序列经ContigExpress软件(ThermoFisher, 美国)组装后, 共获得30条序列CNT00001-CNT00030 (登录号: OQ152393、OQ152394、OQ152408-OQ152427、OQ162327-OQ162334, 已提交至NCBI的GenBank数据库); (2) 用于宏条形码分析的18S rDNA V4区扩增、测序: 流程参照(Stoeck, 2010; Liu, 2021), 原始数据已提交至NCBI, BioProject编号为PRJNA936563。
1.4 系统进化分析
对本研究所获得的共36条18S rDNA全长序列(包括6条单细胞PCR产物序列和30条环境样本扩增克隆序列)借助NCBI的BLAST (http://www.ncbi. nlm.nih.gov/BLAST/)默认参数进行序列相似性比对。以从NCBI获得的来自世界各海域的19条红色赤潮藻、6条多纹膝沟藻和1条底刺膝沟藻()的18S rDNA全长序列为参考序列(Gunderson, 2001; Hee, 2004; Jeong, 2005; Takano, 2006; Ki, 2007; Chen, 2015, 2018; Kim, 2023), 结合本研究中获得的红色赤潮藻的17条序列和多纹膝沟藻的10条序列(Genbank登录号: OQ152393、OQ152394, OQ152408-OQ152432), 以三角异帽藻()为外类群(Saunders, 1997), 借助IQtree软件(Nguyen, 2015)分别选择了最适合的核苷酸置换模型TN+F+I, 自展值为1 000, 重构自展一致树(Bootstrap consensus tree)。
1.5 宏条形码分析
宏条形码测序结果的分析借助R软件包DADA2 (Callahan, 2016), 分析流程参考(Liu, 2021), 生成的扩增子序列变异(amplicon sequence variants, ASVs)序列基于Protist Ribosomal Reference (PR2) 数据库(Guillou, 2013)进行初步的分类学信息注释。进一步地, 以相对丰度>0.01%的ASV为重点研究对象, 基于NCBI NT数据库借助BLAST将各ASV序列注释至最高PID结果所对应的物种信息(PID阈值为99%, coverage阈值为95%)。对注释为浮游植物门类的ASV进行后续分析, 使用R软件包ggplot2 (Wickham, 2009)基于ASV数目和ASV相对丰度分别绘制柱状图和饼图。另外借助POPART 1.7软件(Leigh, 2015)对注释为红色赤潮藻和多纹膝沟藻的单倍型分别构建了TCS网络图以展示多样性。
2 结果
2.1 致灾赤潮物种的形态和分子鉴定
光学显微镜下对活体水样镜检观察, 发现该样本中主要存在3种可运动的藻类细胞(图1b, 1c, 1d)。依据参考文献报道描述的典型形态特征(Daugbjerg, 2000; Kim, 2006; 刘瑞玉, 2008; Gómez, 2016), 将3个物种依次鉴定为疑似红色赤潮藻、膝沟藻和疑似灰白下沟藻。为了验证形态观察结果, 对3种藻细胞进行单细胞分离并对其进行18S rDNA全长序列的单细胞PCR, 最终共成功获得6个18S rDNA全长序列的测序结果(图1b物种登录号: OQ152428- OQ152430; 图1c物种登录号: OQ152431和OQ152432; 图1d物种登录号: OQ162335)。其中, 图1b物种的3个18S rDNA全长序列皆以100%的相似度比对至红色赤潮藻(登录号: OK638976); 图1c物种的两个18S rDNA全长序列分别以99.83%和99.72%的相似度比对至多纹膝沟藻(登录号: AJ833631) (Jeong, 2005); 图1d物种的1个18S rDNA全长序列以99.93%的相似度比对至灰白下沟藻(登录号: KP790161)(Reñé, 2015)(附表)。单细胞分子标记扩增结果证实了对活体水体样本的形态学鉴定结果, 即该赤潮是一次以红色赤潮藻、多纹膝沟藻和灰白下沟藻为主导的多相赤潮。
进一步借助从赤潮位点的环境DNA样本所获得的共30个18S rDNA全长克隆测序结果(附表), 分别比对到红色赤潮藻(13条, 相似度99.67%~99.94%)、多纹膝沟藻(7条, 99.61%~99.72%)、纺锤环沟藻(1条, 99.71%)、其他浮游植物(4条)、其他海洋生物(5条)。红色赤潮藻和多纹膝沟藻的检出和高占比, 证明了这两个物种的确作为赤潮原因种引起了荣成海域的赤潮暴发。除此之外, 对赤潮海域环境样本针对18S rDNA分子标记进行克隆测序也检出了纺锤环沟藻, 表明分子分析的准确性和可靠性。
2.2 赤潮原因种的系统发育分析
以三角异帽藻为外类群, 分别针对红色赤潮藻(图2a)和多纹膝沟藻(图2b)重建18S rDNA全长序列系统发育树。发现所有红色赤潮藻株系主要形成3个分支: 首先, 亚洲海域的2个株系, 韩国GSW0207株系(Kim, 2004)和中国涠洲岛S2-2019-Ak株系聚为一支; 其次, 来自美国不同海域的6个株系以97的较高自展支持值聚为一支; 最后, 本次荣成的克隆测序和单细胞分离株系得到的红色赤潮藻序列皆与绝大部分的亚洲海域株系, 包括中国、日本、韩国株系, 和欧洲海域的2个株系, 法国和挪威株系, 聚为一个大支。所有多纹膝沟藻株系, 除了CNT00007序列之外, 以67的自展支持值与韩国海域的6个株系聚为一支。
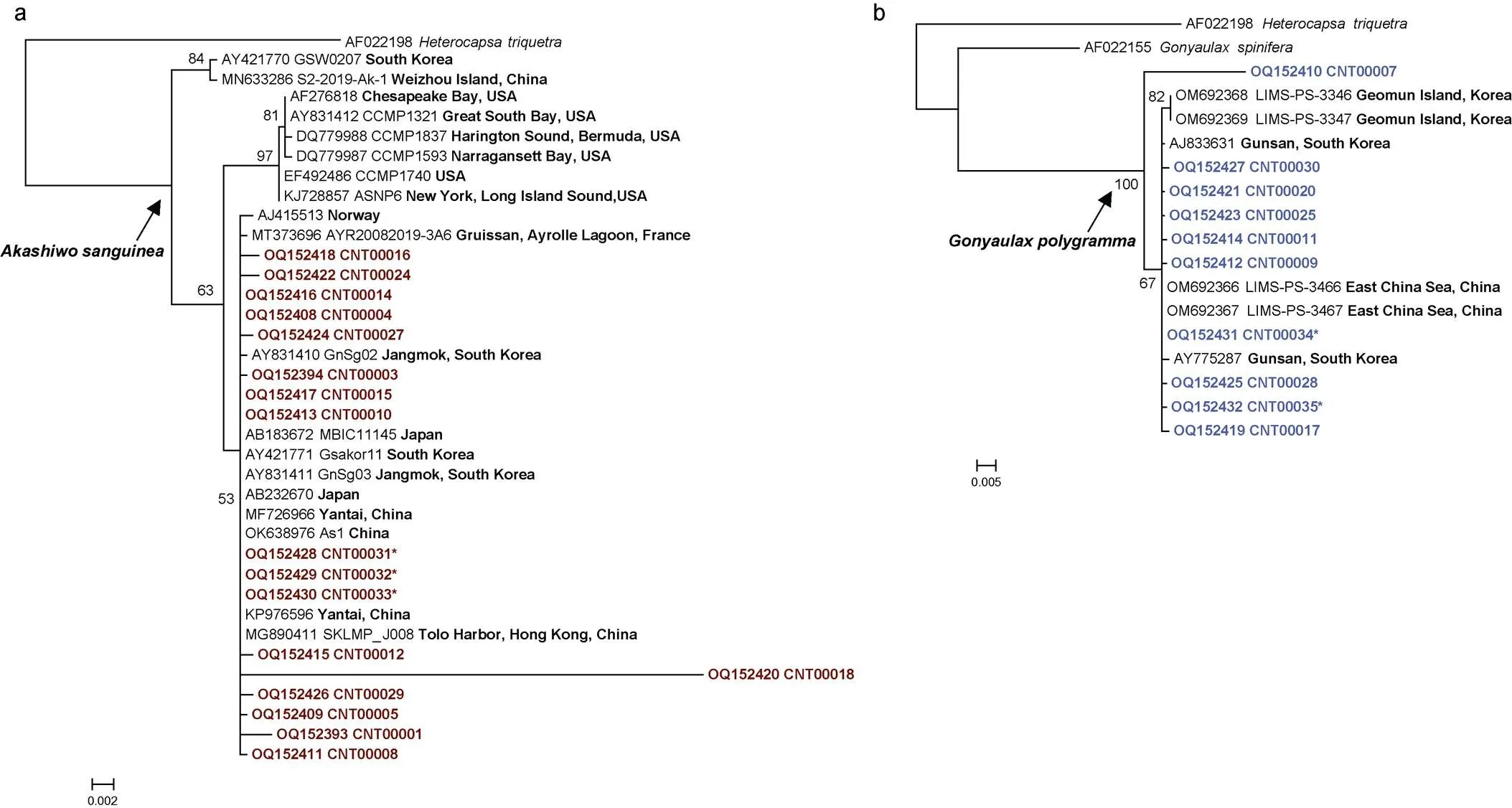
图2 基于18S rDNA全长序列的赤潮优势种系统发育树(以三角异帽藻为外类群)
注: a: 红色赤潮藻; b: 多纹膝沟藻; 加粗字体表示株系来源地区, 红字表示本研究红色赤潮藻测序结果序列, 蓝字表示本研究多纹膝沟藻测序结果序列, *表示单细胞扩增测序结果
2.3 赤潮物种组成的宏条形码分析
为了评估宏条形码分析方法在鉴定致灾赤潮物种中的应用价值, 我们对赤潮水体开展了宏条形码分析。对水体中浮游植物DNA的18S rDNA V4区扩增和高通量测序结果进行了DADA2分析, 共获得了1 016个ASVs, 经过过滤获得的666个ASVs, 包括405个浮游植物ASVs (图3a)。在405个浮游植物ASVs中, 超过一半(268个)注释为甲藻门, 相对丰度为94.6% (图3b)。这些浮游植物中相对丰度最高的十种物种依次为红色赤潮藻、多纹膝沟藻、纺锤环沟藻、灰白下沟藻、球形异帽藻()、剧毒卡罗藻()、日本下睫虫()、夜光藻、相关亚历山大藻()和。其中红色赤潮藻在浮游植物中的相对丰度最高, 多纹膝沟藻(ASV2)在浮游植物中的相对丰度第二(包括两个高度相似但不同的ASV, 即ASV2和ASV5)(图3c)。该方法鉴定出的致灾赤潮物种与兼用基于形态学和分子标记的鉴定结果基本相符, 且宏条形码分析揭示出更多的物种, 包括纺锤环沟藻, 即该次赤潮是一次以红色赤潮藻、多纹膝沟藻、纺锤环沟藻和灰白下沟藻为主导的多相赤潮事件。

图3 宏条形码分析流程及赤潮浮游植物组成
3 讨论
随着生态环境的复杂化, 混合多相赤潮呈现愈来愈频繁发生的趋势。红色赤潮藻近几年更易形成多相赤潮, 2011年在厦门同安湾海域发生了范围105 km2的红色赤潮藻、中肋骨条藻和角毛藻的三相赤潮(俞秀霞, 2012), 2016年惠州市大亚湾海域发生了范围74 km2的红色赤潮藻、中肋骨条藻和夜光藻的三相赤潮(2016 年广东省海洋灾害公报), 另在2021年在山东东营以北渤海海域发生了范围高达3 800 km2的红色赤潮藻和多纹膝沟藻的双相赤潮(自然资源部北海局, 2022)。这种由非单一物种引发的赤潮, 反映出水体内物种结构和物种多样性的高度复杂化, 这也为鉴定致灾赤潮物种, 探究物种之间互相作用、赤潮暴发机制带来一定的难度。
本研究以此次赤潮暴发区域的表层水体样本为例, 对常规基于形态特征和分子标记的鉴定方法与宏条形码分析方法两者进行评价比较, 为后续的赤潮鉴定提供了更多的可行思路。首先借助形态学初步判断海水中浮游生物的种类和密度, 依据其具备的形态特征对赤潮优势种进行鉴定, 这也是传统上公认的赤潮致灾物种鉴定方式, 在直观性和定量化的操作上被认同并一直发挥着不可替代的作用(黄长江等, 2000a; 李雪梅等, 2011)。然而, 形态特征的差别细微、具有较强形态可塑性或固定过程中细胞不稳定性则要求鉴定者需要更丰富的经验和更精细的观察工具例如扫描电镜或透射电镜(齐雨藻等, 1977; 赖红艳等, 2007; 李雪梅等, 2011), 为监测应用带来偏差和复杂。更有甚者, 当物种没有明确的分类形态特征时, 基于形态学的鉴定方式则很难开展下去(甄毓等, 2006)。本研究通过对赤潮样本中的活细胞进行形态鉴定, 发现该赤潮事件是一个多相赤潮事件, 优势致灾赤潮物种包括红色赤潮藻、多纹膝沟藻和灰白下沟藻, 比最近报道的基于对固定样本观察的鉴定结果(李晓东等, 2023)多了一个物种灰白下沟藻。灰白下沟藻没有被鉴定出, 是由于该藻具有固定后细胞不稳定的特点(黄长江等, 2000b)。本研究利用宏条形码分析除了鉴定出上面三种致灾赤潮物种外, 还鉴定出纺锤环沟藻细胞, 表明宏条形码分析更加准确可靠。基于形态和单细胞测序分析均未鉴定到纺锤环沟藻细胞, 可能是由于该藻细胞较小, 且无色素体(顾海峰等, 2014)而容易被错过。
分子生物学技术在微生物的分类鉴定上被广泛、有效地应用, 18S rDNA基因具有稳定的保守区和可变区结构, 因此使得作为分子标记具备通用性和高分辨率(Lavrinienko, 2021), 且针对浮游植物已建立了相对丰富的18S rDNA序列数据库, 已被推荐作为物种鉴定和系统发育分析的分子标记(Sorhannus, 2004; Luddington, 2012)。本研究中采用了3类分子生物学技术, 包括单细胞DNA扩增、克隆文库测序和宏条形码分析技术对赤潮生物样品进行解析。单细胞DNA扩增可以短时间内获得目标微藻细胞的全长18S rDNA基因序列, 进而确定其物种信息。然而该方法在目标微藻的选择和分离上具有局限性, 不能展现群落的物种组成结构和物种多样性。
基于18S rDNA的克隆文库测序则可以同时获得环境DNA中多个物种的分子标记序列, 进行系统发育分析和生物进化分析, 开始对群落的物种多样性甚至遗传多样性进行解读(Medlin, 1988; Giovannoni, 1990; Lin, 2020), 然而由于测序通量低和挑取阳性克隆的随机性等弊端(张玉等, 2018), 系统性取样难以接近饱和, 不能做到准确地表征环境样品中的生物多样性(Edgcomb, 2002; Stoeck, 2007)。本研究利用此方法鉴定出了红色赤潮藻、多纹膝沟藻和纺锤环沟藻这三种致灾赤潮物种, 灰白下沟藻没有得到鉴定, 其随机性导致低估甚至忽略了致灾赤潮物种, 不能全面准确地对本次赤潮进行解析。基于高通量测序的宏条形码分析技术, 则提供了前所未有的数据获得量(Margulies, 2005), 这意味着不仅可以更深度全面并准确地分析环境样品中物种组成, 还可以依据各序列丰度所占比例相对定量的判断, 便于跟踪各物种的丰度变化趋势。此外, 宏条形码分析技术更易实现鉴定流程的标准化、规范化, 具有较高的可操作性和可重复性(陈楠生, 2020)。本研究通过比较宏条形码分析方法与兼用基于形态特征和分子标记的鉴定方法, 获得了一致的鉴定结果, 甚至在具有特殊性质的藻细胞鉴定上有更佳表现, 如难以固定或固定后细胞立即破碎、藻细胞难以分离观察等。本研究中纺锤环沟藻和灰白下沟藻细胞经过固定后立即破碎(黄长江等, 2000b), 并且分离单细胞较困难, 然而通过宏条形码分析方法表明二者作为优势物种, 是赤潮暴发时的重要组成, 由此表明宏条形码分析方法可以用于准确系统鉴定包括组成较为复杂的多相赤潮事件在内的致灾赤潮物种。
4 结论
2021年冬季出现在山东省荣成海域的赤潮物种鉴定研究通过对基于形态特征和分子标记的鉴定方法与宏条形码分析方法两者进行评价比较, 获得了一致的赤潮鉴定结果, 甚至后者在具有特殊性质的藻细胞鉴定上有更佳表现, 即一次以红色赤潮藻、多纹膝沟藻、纺锤环沟藻和灰白下沟藻为主导的多相赤潮事件, 由此表明宏条形码分析方法可以用于准确系统鉴定包括组成较为复杂的多相赤潮事件在内的致灾赤潮物种。
刘瑞玉, 2008. 中国海洋生物名录[M]. 北京: 科学出版社.
齐雨藻, 张子安, 1977. 应用扫描电子显微镜的硅藻分类研究[J]. 中山大学学报(自然科学版)(4): 14-29.
自然资源部北海局, 2022, 《2021年北海区海洋灾害公报》[R/OL]. 19-21(2022-05). http://ncs.mnr.gov.cn/n1/n128/ n298/220609161508806020.html.
李晓东, 苏丽, 李晓捷, 等, 2023. 2021—2022年山东荣成海带产区大规模溃烂灾害综合调查分析[J]. 中国农业科技导报, 25(1): 206-222.
李雪梅, 宫春光, 安鑫龙, 2011. 赤潮微藻的分类鉴定方法[J]. 河北渔业(12): 58-59, 63.
宋建杰, 2011. 山东近海生物资源可持续利用对策研究[D]. 青岛: 中国海洋大学.
张玉, 米铁柱, 甄毓, 等, 2018. 海洋沉积物中硫酸盐还原菌和硫氧化菌群落分析方法的比较[J]. 环境科学, 39(1): 438-449.
陈楠生, 2020. 有害藻华的宏条形码分析: 机会与挑战[J]. 海洋科学, 44(7): 116-134.
陈楠生, 丁翔翔, 崔宗梅, 2023. 山东近海赤潮物种多样性研究进展[J]. 海洋与湖沼, 54(5): 1258-1273.
周名江, 朱明远, 张经, 2001. 中国赤潮的发生趋势和研究进展[J]. 生命科学, 13(2): 54-59, 53.
周健, 王玮, 吴志宏, 等, 2020. 山东沿海赤潮灾害基本特征及防控对策建议[J]. 海洋环境科学, 39(4): 537-543.
周斌, 马龙, 周雅卓, 等, 2013. 山东近海潮流能电场建设环境影响分析[J]. 海洋开发与管理, 30(4): 72-76.
俞志明, 陈楠生, 2019. 国内外赤潮的发展趋势与研究热点[J]. 海洋与湖沼, 50(3): 474-486.
俞秀霞, 2012. 厦门同安湾海域一次三相型赤潮及其成因分析[J]. 福建水产, 34(3): 203-207.
顾海峰, 罗肇河, 刘丽丽, 等, 2014. 中国近海甲藻环沟藻属2个新记录种: 纺锤环沟藻和莫氏环沟藻[J]. 生物多样性, 22(3): 401-406.
黄长江, 董巧香, 2000a. 1998年春季珠江口海域大规模赤潮原因生物的形态分类和生物学特征I[J]. 海洋与湖沼, 31(2): 197-204.
黄长江, 董巧香, 2000b. 1998年春季珠江口海域大规模赤潮原因生物的形态分类和生物学特征Ⅱ[J]. 海洋与湖沼, 31(3): 233-238.
赖红艳, 侯建军, 王振河, 2007. 用扫描电镜观察裸甲藻的方法研究[J]. 湖北民族学院学报(自然科学版), 25(3): 338-342.
甄毓, 于志刚, 米铁柱, 2006. 分子生物学在微藻分类研究中的应用[J]. 中国海洋大学学报, 36(6): 875-878.
ANDERSON D M, BURKHOLDER J M, COCHLAN W P,, 2008. Harmful algal blooms and eutrophication: Examining linkages from selected coastal regions of the United States [J]. Harmful Algae, 8(1): 39-53.
ANDERSON D M, CEMBELLA A D, HALLEGRAEFF G M, 2012. Progress in understanding harmful algal blooms: paradigm shifts and new technologies for research, monitoring, and management [J]. Annual Review of Marine Science, 4: 143-176.
CALLAHAN B J, MCMURDIE P J, ROSEN M J,, 2016. DADA2: High-resolution sample inference from Illumina amplicon data [J]. Nature Methods, 13(7): 581-583.
CHEN T T, LIU Y, SONG S Q,, 2015. The effects of major environmental factors and nutrient limitation on growth and encystment of planktonic dinoflagellate[J]. Harmful Algae, 46: 62-70.
CHEN T T, LIU Y, SONG S Q,, 2018. Characterization of the parasitic dinoflagellatesp. infectingin coastal waters of China [J]. Journal of Eukaryotic Microbiology, 65(4): 448-457.
DAUGBJERG N, HANSEN G, LARSEN J,, 2000. Phylogeny of some of the major genera of dinoflagellates based on ultrastructure and partial LSU rDNA sequence data, including the erection of three new genera of unarmoured dinoflagellates [J]. Phycologia, 39(4): 302-317.
EDGCOMB V P, KYSELA D T, TESKE A,, 2002. Benthic eukaryotic diversity in the Guaymas Basin hydrothermal vent environment [J]. Proceedings of the National Academy of Sciences of the United States of America, 99(11): 7658-7662.
GIOVANNONI S J, BRITSCHGI T B, MOYER C L,, 1990. Genetic diversity in Sargasso Sea bacterioplankton [J]. Nature, 345(6270): 60-63.
GÓMEZ F, TAKAYAMA H, MOREIRA D,, 2016. Unarmoured dinoflagellates with a small hyposome:andgen. nov. for(Gymnodiniales, Dinophyceae) [J]. European Journal of Phycology, 51(2): 226-241.
GUILLOU L, BACHAR D, AUDIC S,, 2013. The Protist Ribosomal Reference database (PR2): a catalog of unicellular eukaryote Small Sub-Unit rRNA sequences with curated taxonomy [J]. Nucleic Acids Research, 41(D1): D597-D604.
GUNDERSON J H, GOSS S H, COATS D W, 2001. FISH probes for the detection of the parasitic dinoflagellatesp. Infecting the dinoflagellatein chesapeake bay [J]. Journal of Eukaryotic Microbiology, 48(6): 670-675.
HALLEGRAEFF G M, 2010. Ocean climate change, phytoplankton community responses, and harmful algal blooms: a formidable predictive challenge [J]. Journal of Phycology, 46(2): 220-235.
HEE K S, KIM K Y, KIM C H,, 2004. Phylogenetic Analysis of Harmful Algal Bloom (HAB)-Causing Dinoflagellates Along the Korean Coasts, Based on SSU rRNA Gene [J]. Journal of Microbiology and Biotechnology, 14(5): 959-966.
JEONG H J, YOO Y D, SEONG K A,, 2005. Feeding by the mixotrophic red-tide dinoflagellate: mechanisms, prey species, effects of prey concentration, and grazing impact [J]. Aquatic Microbial Ecology, 38(3): 249-257.
KI J S, HAN M S, 2007. Cryptic long internal repeat sequences in the ribosomal DNA ITS1 gene of the dinoflagellate(Dinophyceae): a 101 nucleotide six-repeat track with a palindrome-like structure [J]. Genes & Genetic Systems, 82(2): 161-166.
KIM K Y, KIM Y S, HWANG C H,, 2006. Phylogenetic analysis of dinoflagellatestein responsible for harmful algal blooms based on the partial LSU rDNA sequence data [J]. Algae, 21(3): 283-286.
KIM S H, KIM K Y, LEE W S,, 2004. Phylogenetic analysis of harmful algal bloom (HAB)-causing dinoflagellates along the Korean Coasts, based on sSU rRNA gene [J]. Journal of Microbiology and Biotechnology, 14(5): 959-966.
KIM H J, LI Z, GU H F,, 2023.sp.. and two allied species (Gonyaulacales, Dinophyceae) from Korean coastal waters and East China Sea: morphology, phylogeny and growth response to changes in temperature and salinity [J]. Phycologia, 62(1): 48-67.
LAVRINIENKO A, JERNFORS T, KOSKIMÄKI J J,, 2021. Does intraspecific variation in rDNA copy number affect analysis of microbial communities? [J]. Trends in Microbiology, 29(1): 19-27.
LEIGH J W, BRYANT D, 2015. POPART: full‐feature software for haplotype network construction [J]. Methods in Ecology and Evolution, 6(9): 1110-1116.
LEÓN-MUÑOZ J, URBINA M A, GARREAUD R,, 2018. Hydroclimatic conditions trigger record harmful algal bloom in western Patagonia (summer 2016) [J]. Scientific Reports, 8(1): 1330.
LIN S H, HU Z X, DENG Y Y,, 2020. An assessment on the intrapopulational and intraindividual genetic diversity in LSU rDNA in the harmful algal blooms-forming dinoflagellate(=)based on clonal cultures and bloom samples from Jiaozhou Bay, China [J]. Harmful Algae, 96: 101821.
LIU S Y, GIBSON K, CUI Z M,, 2020. Metabarcoding analysis of harmful algal species in Jiaozhou Bay [J]. Harmful Algae, 92: 101772.
LIU S Y, ZHANG M J, ZHAO Y F,, 2021. Biodiversity and spatial-temporal dynamics ofspecies in Jiaozhou Bay, China [J]. International Journal of Environmental Research and Public Health, 18(21): 11637.
LUDDINGTON I A, KACZMARSKA I, LOVEJOY C, 2012. Distance and character-based evaluation of the V4 region of the 18S rRNA gene for the identification of diatoms (Bacillariophyceae) [J]. PLoS One, 7(9): e45664.
MARGULIES M, EGHOLM M, ALTMAN W E,, 2005. Genome sequencing in microfabricated high-density picolitre reactors [J]. Nature, 437(7057): 376-380.
MCCABE R M, HICKEY B M, KUDELA R M,, 2016. An unprecedented coastwide toxic algal bloom linked to anomalous ocean conditions [J]. Geophysical Research Letters, 43(19): 10366-10376.
MEDLIN L, ELWOOD H J, STICKEL S,, 1988. The characterization of enzymatically amplified eukaryotic 16S-like rRNA-coding regions [J]. Gene, 71(2): 491-499.
NGUYEN L T, SCHMIDT H A, VON HAESELER A,, 2015. IQ-TREE: a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies [J]. Molecular Biology and Evolution, 32(1): 268-274.
REÑÉ A, CAMP J, GARCÉS E, 2015. Diversity and phylogeny of gymnodiniales (Dinophyceae) from the NW Mediterranean Sea revealed by a morphological and molecular approach [J]. Protist, 166(2): 234-263.
SALDARRIAGA J F, MCEWAN M L, FAST N M,, 2003. Multiple protein phylogenies show thatandare early branches of the dinoflagellate lineage [J]. International Journal of Systematic and Evolutionary Microbiology, 53(Pt 1): 355-365.
SAUNDERS G W, HILL D R A, SEXTON J P,, 1997. Small-subunit ribosomal RNA sequences from selected dinoflagellates: testing classical evolutionary hypotheses with molecular systematic methods [M] // BHATTACHARYA D. Origins of Algae and their Plastids. Vienna: Springer: 237-259.
SORHANNUS U, 2004. Diatom phylogenetics inferred based on direct optimization of nuclear-encoded SSU rRNA sequences [J]. Cladistics, 20(5): 487-497.
STOECK T, BASS D, NEBEL M,, 2010. Multiple marker parallel tag environmental DNA sequencing reveals a highly complex eukaryotic community in marine anoxic water [J]. Molecular Ecology, 19(S1): 21-31.
STOECK T, KASPER J, BUNGE J,, 2007. Protistan diversity in the Arctic: a case of paleoclimate shaping modern biodiversity? [J]. PLoS One, 2(8): e728.
TAKANO Y, HORIGUCHI T, 2006. Acquiring scanning electron microscopical, light microscopical and multiple gene sequence data from a single dinoflagellate cell [J]. Journal of Phycology, 42(1): 251-256.
WICKHAM H, 2009. Ggplot2: Elegant Graphics for Data Analysis [M]. New York: Springer.
YU Z M, SONG X X, CAO X H,, 2017. Mitigation of harmful algal blooms using modified clays: Theory, mechanisms, and applications [J]. Harmful Algae, 69: 48-64.
ZHANG Q C, QIU L M, YU R C,, 2012. Emergence of brown tides caused byHargraves et Sieburth in China [J]. Harmful Algae, 19: 117-124.
ZHANG Q C, WANG Y F, SONG M J,, 2022. First record of abloom in Haizhou Bay in response to dissolved organic nitrogen and phosphorus [J]. Marine Pollution Bulletin, 178: 113572.
附表 株系克隆测序注释比对结果
Supplementary Table The annotations for clone sequencing strains
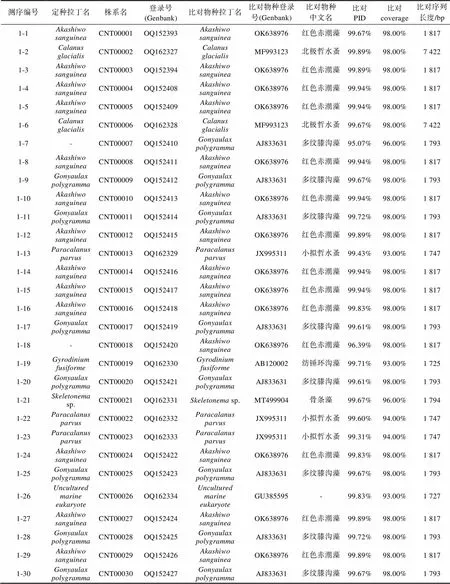
测序编号定种拉丁名株系名登录号(Genbank)比对物种拉丁名比对物种登录号(Genbank)比对物种中文名比对PID比对coverage比对序列长度/bp 1-1Akashiwo sanguineaCNT00001OQ152393Akashiwo sanguineaOK638976红色赤潮藻99.67%98.00%1 817 1-2Calanus glacialisCNT00002OQ162327Calanus glacialisMF993123北极哲水蚤99.89%98.00%7 422 1-3Akashiwo sanguineaCNT00003OQ152394Akashiwo sanguineaOK638976红色赤潮藻99.89%98.00%1 817 1-4Akashiwo sanguineaCNT00004OQ152408Akashiwo sanguineaOK638976红色赤潮藻99.94%98.00%1 817 1-5Akashiwo sanguineaCNT00005OQ152409Akashiwo sanguineaOK638976红色赤潮藻99.94%98.00%1 817 1-6Calanus glacialisCNT00006OQ162328Calanus glacialisMF993123北极哲水蚤99.67%98.00%7 422 1-7-CNT00007OQ152410Gonyaulax polygrammaAJ833631多纹膝沟藻95.07%96.00%1 793 1-8Akashiwo sanguineaCNT00008OQ152411Akashiwo sanguineaOK638976红色赤潮藻99.94%98.00%1 817 1-9Gonyaulax polygrammaCNT00009OQ152412Gonyaulax polygrammaAJ833631多纹膝沟藻99.67%98.00%1 793 1-10Akashiwo sanguineaCNT00010OQ152413Akashiwo sanguineaOK638976红色赤潮藻99.94%98.00%1 817 1-11Gonyaulax polygrammaCNT00011OQ152414Gonyaulax polygrammaAJ833631多纹膝沟藻99.72%98.00%1 793 1-12Akashiwo sanguineaCNT00012OQ152415Akashiwo sanguineaOK638976红色赤潮藻99.89%98.00%1 817 1-13Paracalanus parvusCNT00013OQ162329Paracalanus parvusJX995311小拟哲水蚤99.43%93.00%1 747 1-14Akashiwo sanguineaCNT00014OQ152416Akashiwo sanguineaOK638976红色赤潮藻99.94%98.00%1 817 1-15Akashiwo sanguineaCNT00015OQ152417Akashiwo sanguineaOK638976红色赤潮藻99.94%98.00%1 817 1-16Akashiwo sanguineaCNT00016OQ152418Akashiwo sanguineaOK638976红色赤潮藻99.83%98.00%1 817 1-17Gonyaulax polygrammaCNT00017OQ152419Gonyaulax polygrammaAJ833631多纹膝沟藻99.61%98.00%1 793 1-18-CNT00018OQ152420Akashiwo sanguineaOK638976红色赤潮藻96.39%98.00%1 817 1-19Gyrodinium fusiformeCNT00019OQ162330Gyrodinium fusiformeAB120002纺锤环沟藻99.71%93.00%1 725 1-20Gonyaulax polygrammaCNT00020OQ152421Gonyaulax polygrammaAJ833631多纹膝沟藻99.61%98.00%1 793 1-21Skeletonema sp.CNT00021OQ162331Skeletonema sp.MT499904骨条藻99.67%96.00%1 794 1-22Paracalanus parvusCNT00022OQ162332Paracalanus parvusJX995311小拟哲水蚤99.60%94.00%1 747 1-23Paracalanus parvusCNT00023OQ162333Paracalanus parvusJX995311小拟哲水蚤99.31%94.00%1 747 1-24Akashiwo sanguineaCNT00024OQ152422Akashiwo sanguineaOK638976红色赤潮藻99.83%98.00%1 817 1-25Gonyaulax polygrammaCNT00025OQ152423Gonyaulax polygrammaAJ833631多纹膝沟藻99.67%98.00%1 793 1-26Uncultured marine eukaryoteCNT00026OQ162334Uncultured marine eukaryoteGU385595-99.83%93.00%1 727 1-27Akashiwo sanguineaCNT00027OQ152424Akashiwo sanguineaOK638976红色赤潮藻99.89%98.00%1 817 1-28Gonyaulax polygrammaCNT00028OQ152425Gonyaulax polygrammaAJ833631多纹膝沟藻99.72%98.00%1 793 1-29Akashiwo sanguineaCNT00029OQ152426Akashiwo sanguineaOK638976红色赤潮藻99.89%98.00%1 817 1-30Gonyaulax polygrammaCNT00030OQ152427Gonyaulax polygrammaAJ833631多纹膝沟藻99.67%98.00%1 793
续表
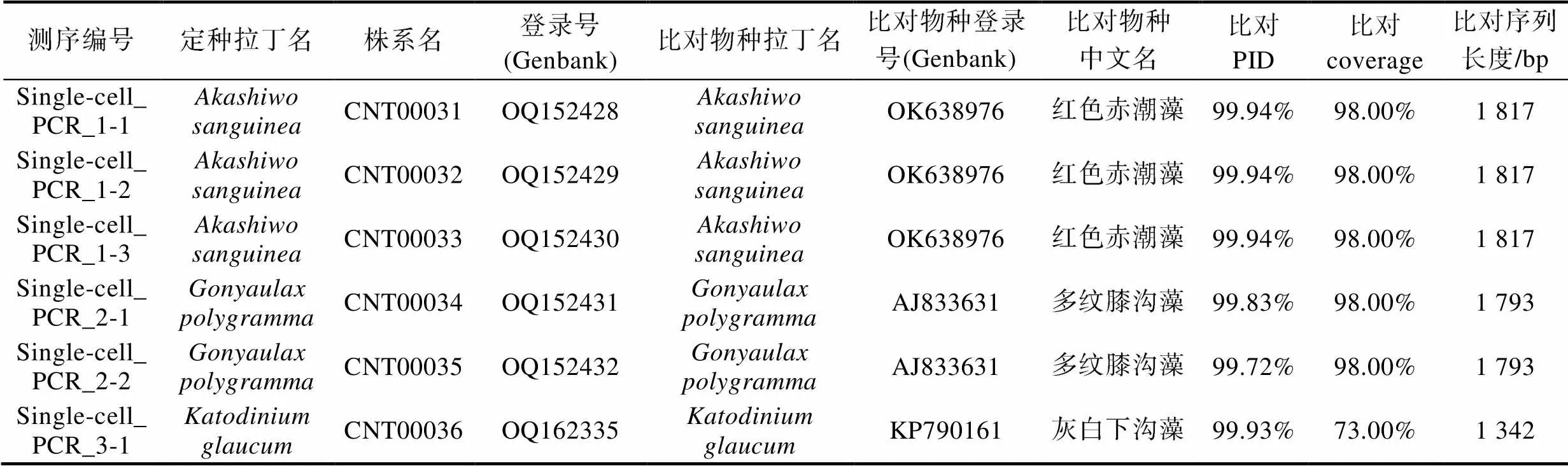
测序编号定种拉丁名株系名登录号(Genbank)比对物种拉丁名比对物种登录号(Genbank)比对物种中文名比对PID比对coverage比对序列长度/bp Single-cell_PCR_1-1Akashiwo sanguineaCNT00031OQ152428Akashiwo sanguineaOK638976红色赤潮藻99.94%98.00%1 817 Single-cell_PCR_1-2Akashiwo sanguineaCNT00032OQ152429Akashiwo sanguineaOK638976红色赤潮藻99.94%98.00%1 817 Single-cell_PCR_1-3Akashiwo sanguineaCNT00033OQ152430Akashiwo sanguineaOK638976红色赤潮藻99.94%98.00%1 817 Single-cell_PCR_2-1Gonyaulax polygrammaCNT00034OQ152431Gonyaulax polygrammaAJ833631多纹膝沟藻99.83%98.00%1 793 Single-cell_PCR_2-2Gonyaulax polygrammaCNT00035OQ152432Gonyaulax polygrammaAJ833631多纹膝沟藻99.72%98.00%1 793 Single-cell_PCR_3-1Katodinium glaucumCNT00036OQ162335Katodinium glaucum KP790161灰白下沟藻99.93%73.00%1 342
Molecular analysis of a polyphasic red tide in Rongcheng OFFSHORE, Shandong PENINSULA IN 2021~2022
LIU Kui-Yan1, 2, 3, 4, WANG Hong-Shu1, 2, 3, LIU Feng1, 2, 3, CHEN Nan-Sheng1, 2, 3, 4
(1. CAS Key Laboratory of Marine Ecology and Environmental science, Institute of Oceanology, Chinese Academy of Sciences, Qingdao 266071, China; 2. Laoshan Laboratory, Qingdao 266237, China; 3. Center for Ocean Mega-Science, Chinese Academy of Sciences, Qingdao 266071, China; 4. University of Chinese Academy of Sciences, Beijing 100049, China)
Shandong Peninsula is surrounded by the Bohai Sea and Yellow Sea, features complex coastal zoning and long coastline, and suffers from frequent red tide outbreaks in the recent decade. Some red tide species are small in cell size or have inconspicuous morphological characteristics, and are often fragile and unstable during fixation, making it a challenge work for experts to identify the causative red tide species based only on morphological features. Therefore, causative species of many red tides have not been accurately identified, especially for those polyphasic red tide events. From November 2021 to April 2022, a devastating red tide event of 1 440 km2occurred in Rongcheng offshore in a kelpaquaculture region, resulting in the bleaching or rotting of the kelp and serious economic losses. Considering both morphological features of live algal cells in water samples and molecular marker sequences amplified from single cells, three dominant causative red tide species were identified, including,, and, which was different from a recent report in which only the former two were identified in this region.is easily missed out as it is fragile and unstable during fixation. Metabarcoding analysis of water samples identified another causative species,, being also fragile and unstable during fixation. This study showed that the metabarcoding analysis is standardized and more accurate in species identification, and can accurately and systematically identify red tide causative species especially for complex polyphasic red tide events.
harmful algal bloom (HAB); Dinoflagellata; metabarcoding analysis;;
Q948
10.11693/hyhz20230300062
* 崂山实验室科技创新项目, LSKJ202203700号; 国家自然科学基金项目, 42176162号; 国家重点研发计划, 2022YFC3105200号; 中国科学院战略性先导科技专项(B类), XDB42000000号; 荣成市海洋与渔业监测减灾中心委托项目, 2022-2023。刘奎艳, 博士研究生, E-mail: liukuiyan@qdio.ac.cn
陈楠生, 博士生导师, 研究员, E-mail: chenn@qdio.ac.cn
2023-03-16,
2023-04-17
——环沟直放抱卵虾

