云算GPM 混合图谱分析系统在混合样本检验中的应用
陈安琪
(1.复旦大学 基础医学院 法医系,上海 200032; 2.司法鉴定科学研究院 上海市法医学重点实验室上海市司法鉴定专业技术服务平台 司法部司法鉴定重点实验室,上海 200063)
混合样本鉴定是法医物证领域最为常见的难题之一[1-2]。 源于犯罪现场或涉及刑事纠纷(例如性侵、人身攻击与谋杀等)的生物学检材,其样本均存在DNA 混合的情况[3-4]。 该类样本在进行基因分型时,可出现两个或多个同性或异性贡献者的DNA图谱,给其中贡献者的个体识别带来了极大的挑战。 为克服混合样本的鉴定困难,提高法医工作者对混合样本的检测能力,越来越多的分子标记借助二代测序(next-generation sequencing,NGS)技术手段来解决混合样本的鉴定问题[5-6]。 虽然这些方法能够在一定程度上解决混合样本的拆分问题,但其应用依旧面临两大弊端:一方面,基于NGS 的方案将产出数量巨大的原始数据,其分析需要依赖强大的生信团队,故并不适用于普通法医学实验室;另一方面,这些依赖新技术的新型分子标记没有可供比对的人群数据库,意味着在缺乏嫌疑人基因分型数据的情况下将很难锁定罪犯,无法对案件侦破提供更具指向性的线索。
基于毛细管电泳(capillary electrophoresis,CE)的短串联重复序列(short tandem repeat,STR)分型方法(CE-STR)是当今世界范围内公认的法医物证鉴定方法[7],STR 具有高度多态性和显著的个体间差异,许多国家都建立了基于STR 的人群数据库[8]。 因此,开发一款以法医常用STR 分子标记为目标的窗口化分析系统极具价值。 云算GPM 混合图谱分析系统是一项基于全连续法概率分型的软件[9],相较于其他常用概率模型(如:二进制法、半连续法),其更为全面地考量了影响STR 准确检出的各种因素(如:峰值变异性、混合比例及stutter 峰等),通过分析图谱中的全部信息,以概率的形式给出可能的分型结果。 全连续法概率模型是目前领先的混合图谱拆分方法,该分析系统或许是一个可靠的混合样本分析系统。 为探究该分析系统对混合样本的拆分能力,本研究应用Power-Plex21R○基因分型试剂盒对13 例2~3 人混合样本进行了STR 分型,观察了其在预设阈值条件下的分型情况,探究了云算GPM 混合图谱分析系统对模拟混合样本的基因型拆分情况。
1 材料与方法
1.1 材料
本研究所用DNA 样本源于5 名已知基因型的无关个体,模拟混合样本按以下比例(表1)进行混合。
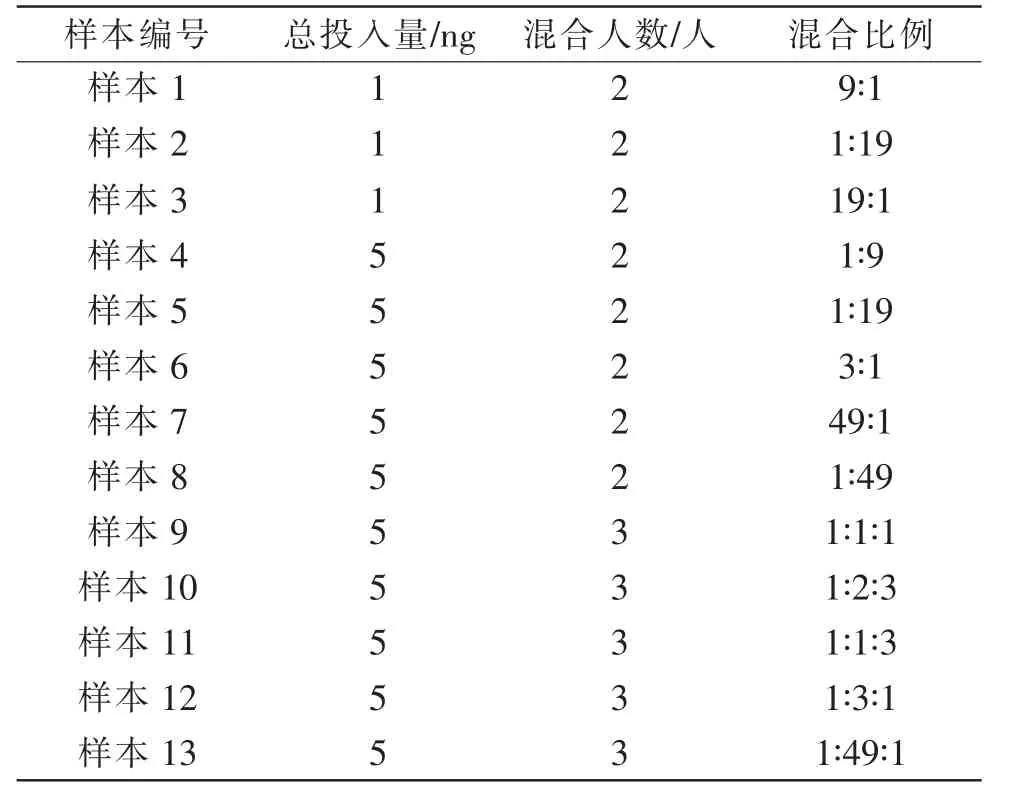
表1 混合样本概况
1.2 方法
1.2.1 STR 分型
采用Power-Plex21R○试剂盒(美国Promega 公司)对13 例混合DNA 样本进行复合扩增,扩增体系及程序严格遵照试剂盒说明书进行。 PCR 扩增产物用3130XL 型遗传分析仪(美国Applied Biosystems 公司)进行毛细管电泳分型检测,STR 基因座分型结果用GeneMapper ID-X 软件(美国Applied Biosystems公司)在试剂盒预设的默认阈值下进行分析。
1.2.2 基于云算GPM 分析系统的混合图谱分析
将毛细管电泳输出的“FSA”格式原始文件导入云算GPM 混合图谱分析系统(北京瑞源文德科技有限公司),并在软件内部完成STR 分型分析。为减少人工判读误差对后续软件分析的影响,本研究以混合样本理论上的分型结果作为参考,对混合图谱上的等位基因应标尽标,继而进行下游的混合图谱拆分分析。
2 结果
2.1 混合样本的基因型检出情况
本研究利用Power-Plex21R○试剂盒检测了13例混合样本的基因分型情况。 在系统预设的默认阈值条件下,其基因型检出情况与预期结果存在较大差异。 如图1 所示,绝大多数基因座的分型结果均不符合预期。 在基因座水平上,Amel 的基因型一致性最高,高达100%(13/13),TH01 次之(92.31%,12/13),D3S1358 位列第三(69.23 %,9/13)。 D13S317 与D16S539 是检出一致性最低的基因座,仅为7.68%(1/13)。 在样本水平上,多数样本仅有1/4~1/5 的等位基因分型结果与预期相符, 样本9 的分型结果一致性最高,约为76.19%(16/21)。 此外,样本12、样本10 和样本6 的分型一致性同样相对较高,分别为52.38%(11/21)、47.62%(10/21)和38.10%(8/21)。
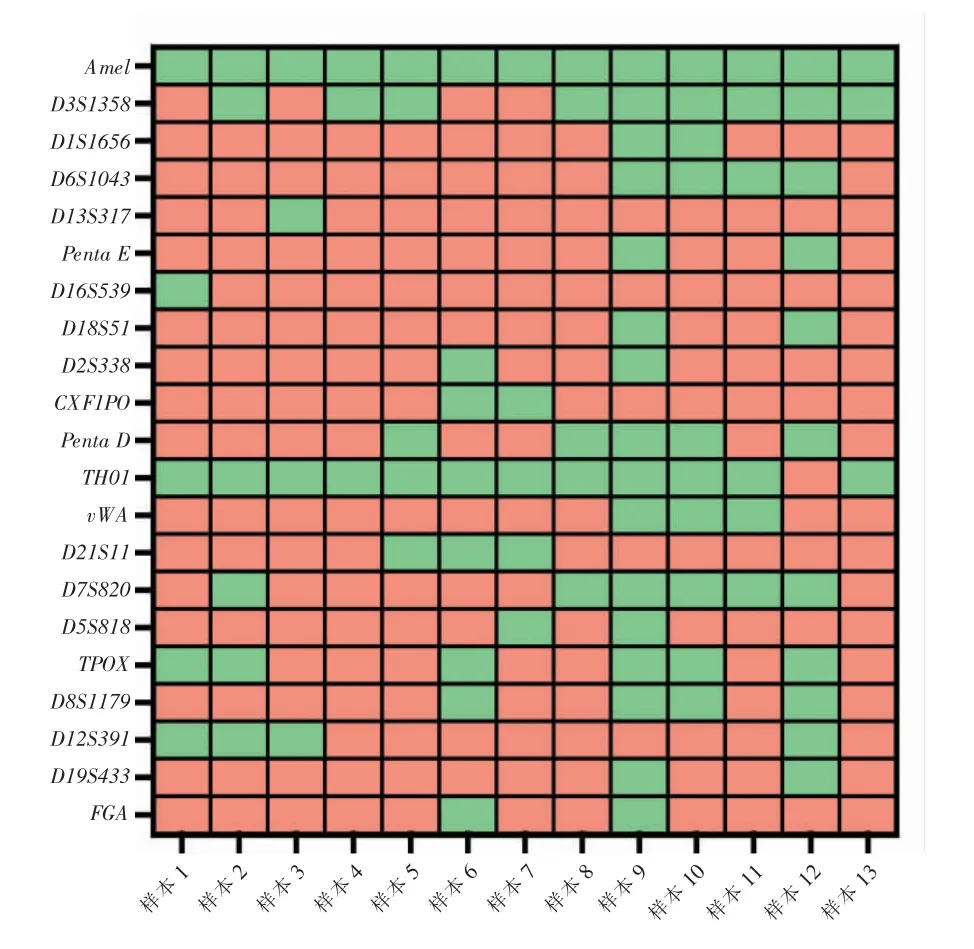
图1 混合样本基因型的预期分型与实际检出情况的比较
2.2 基于云算GPM 的混合图谱拆分
云算GPM 分析系统可对混合图谱的基因型进行拆分,结果如图2 所示(红色表示分型结果完全不符合预期;橙色表示分型结果有部分符合预期;绿色表示分型结果与预期完全相同)。 13 例混合图谱均可被成功拆分,除样本1 的主要贡献者与次要贡献者的基因型拆分结果完全正确外,其他样本的基因型结果均存在一定的误差。 其中,主要贡献者的检出情况较为准确,仅样本3 存在一个基因座(D12S391)的分型错误,约6.54%(18/273)的基因座分型存在拆分结果错误的情况,绝大多数基因座(93.04%,254/273)的分型结果完全正确[图2(a)]。在次要贡献者的等位基因检出方面,其总体分型准确率远不如主要贡献者。 15 个次要贡献者共产生315 个基因座分型结果,约50.48%(159/315)的次要贡献者的基因型完全正确,约41.59%(131/315)的基因型仅有部分满足预期,约7.94%(25/315)的分型结果是完全错误的[图2(b)]。
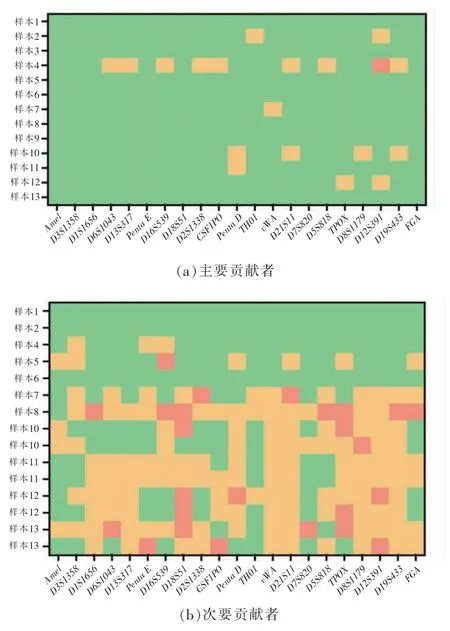
图2 基于云算GPM 混合图谱分析系统的混合样本基因型拆分结果
总体来看,虽然各基因座的分型正确的个数均占多数,但不同基因座水平间的拆分结果准确性仍存在差异。 D7S820 是基因型拆分准确率最高的基因座(89.29%,25/28),Amel 次之(85.71%,24/28),TH01 位列第三(85.71%,24/28)。 除以上3 个基因座之外,还有其他9 个基因座(D3S1358、D1S1656、D6S1043、D13S317、Penta E、D2S1338、CSF1PO、D5S818和FGA)的准确率均在70%以上。 其中,Amel、D3S1358、D13S317、TH01 和vWA 没有分型完全错误的情况发生。与此同时,基因型拆分结果较差的基因座分别为D18S51、TPOX 和D12S391(图3)。
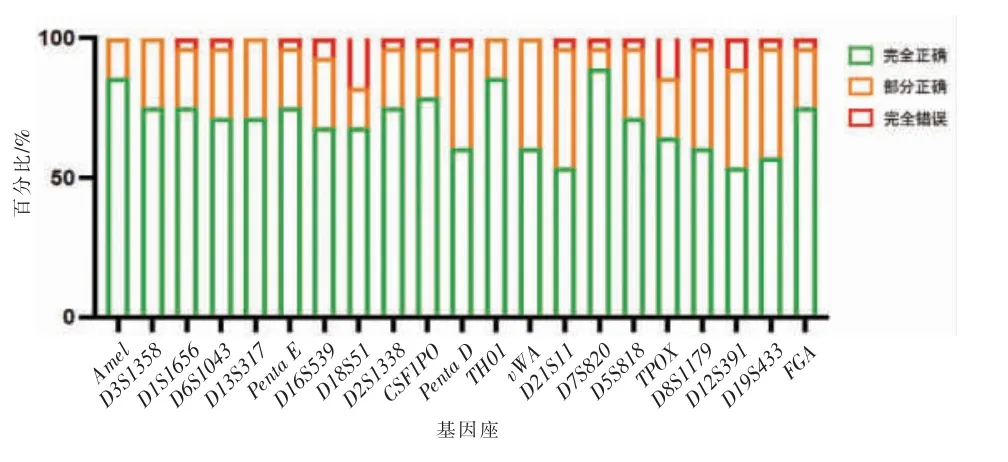
图3 混合图谱基因型拆分结果在基因座水平的表现
2.3 weight 值与分型结果的可靠性分析
为确保拆分结果的准确性,云算GPM 分析系统采用预设weight 值(权重)衡量所得基因型的可靠性。 本研究结果中,weight 值在90%以上的基因座有267 个,占总数的41.01%(267/651),其中,分型完全正确的基因座、部分正确的基因座和完全错误的基因座所占比例分别为99.63%(266/267)、0.37%(1/267)和0%(0/267)。值得注意的是,并非所有正确分型的基因座weight 值均大于90%。基因座分型完全正确、部分正确和完全错误这3 种情况所对应的weight 平均值分别为82.36 %±24.68 %、40.22%±16.56%和35.12%±18.24%[图4(a)]。为进一步分析各分型结果下的weight 值分布情况,本研究对各分型结果下的基因座数进行了基于该频率的拟合分析[图4(b)]。 结果显示,3 种分型结果在weight 值低于90 %时,均存在一定的交叠,虽然weight 值越低,其归属于错误分型的可能性越高,但无法完全根据某一weight 值作出结果是完全正确、部分正确或是完全错误的推断。
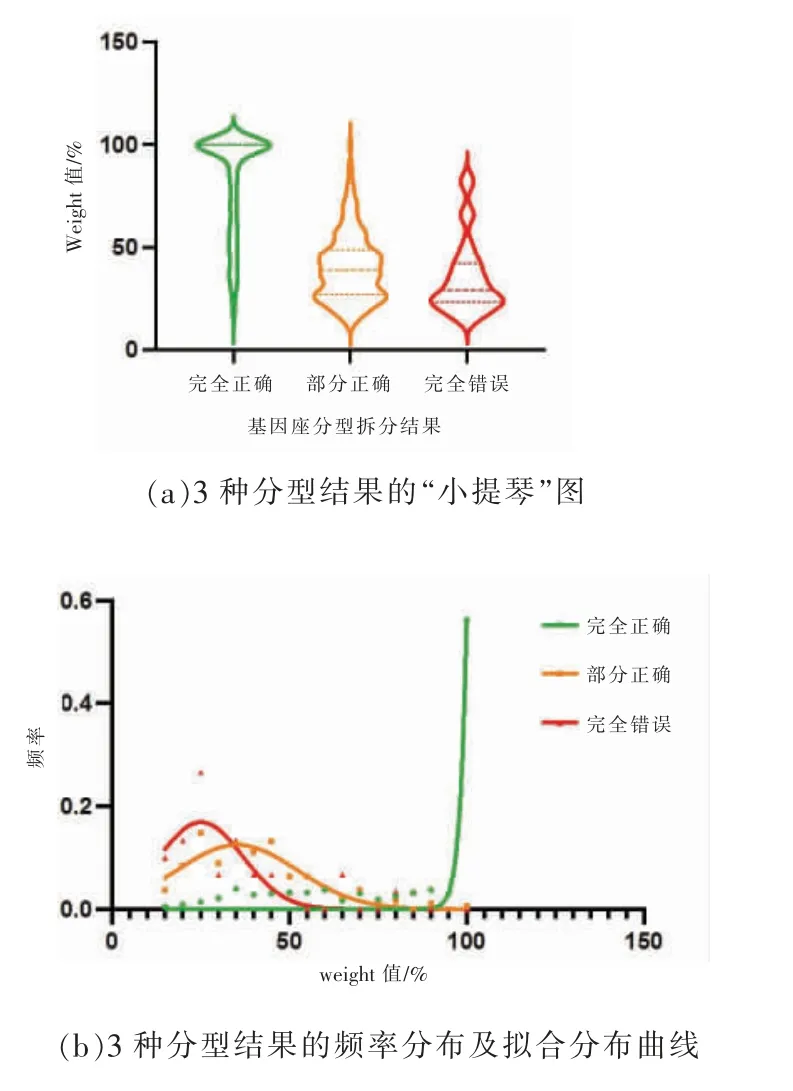
图4 混合图谱基因型拆分结果与weight 值的关系
3 讨论
混合样本的鉴定一直以来都是司法鉴定的重点与难点,其基因分型的成功拆分将为后续的案件侦破提供有效证据与线索[2]。 虽然用于混合样本基因型检测的方案层出不穷,但基于STR 分子标记的窗口化分析系统却较为少见。 STRmix 和云算GPM分析系统是目前较为成熟的混合样本拆分系统,李甫等[9]曾对这两种分析系统进行了比较,认为两者均可用于混合样本的拆分,但其结果存在一定差异。 上述研究的样本来源多限于真实案件样本,缺乏对已知组分的模拟混合DNA 研究,因而难以确定检测系统的正确性。 为进一步探究云算GPM 混合图谱分析系统在混合样本检验中的表现,本研究模拟了13 例2~3 人DNA 混合样本,并用经过广泛验证的Power-Plex21R○试剂盒[10-14]对其进行检测。为确保分型数据的有效性,避免因DNA 不足而导致的等位基因丢失,本研究中所有次要贡献者的投入量均高于其最低检出限(50 pg[11])。 由于该试剂盒的主要检测对象为单一来源样本,因此其系统内置的分析阈值在混合样本的结果输出上或许并不准确。 不出所料,在Power-Plex21R○试剂盒预设的默认阈值条件下,绝大多数的基因型均与预期不同(图1)。 在对这21 个基因座的分型分析中发现,不同等位基因间的分型一致性存在差异。 所有Amel的基因型均符合预期,TH01 与D3S1358 的分型一致性同样较高,分别为92.31 %和69.23 %,而D13S317 与D16S539 的一致性却仅为7.68%。 基于人类遗传基本规律,正常人的Amel 分型结果仅可能是XX、XY 的一种,故而推测本研究中STR 分型的一致性差异或与基因座的遗传多态性相关。 有研究[15]表明,D13S317 与D16S539 的多态信息量(polymorphism information content,PIC)分别为0.794 3 和0.791 7,约是TH01(PIC 为0.603 6)与D3S1358(PIC为0.649 1)的1.22~1.32 倍,提示TH01 与D3S1358的高一致性极有可能是由该基因座有限的基因型组合所致。 对于多数混合样本而言,仅有约20%的基因座分型结果一致,然而样本9、样本12、样本10和样本6 中分型一致的基因座数却远高于平均水平。 由于以上4 例样本的投入量均为5 ng,因此可排除因投入量差异而带来的分型差异。 在混合比例方面,由于以上3 个混合样本的混合比例相对均衡,故而其主要贡献者与次要贡献者的信号也相对均衡,不易被占比高的组分所掩盖,这可能是造成其在默认阈值下一致性相对较高的原因。 用于CESTR 分型检测的试剂盒有很多,绝大多数的CESTR 检测均是服务于单一来源DNA,而非混合样本。使用默认阈值下的分析结果,必然会存在次要等位基因被覆盖的偏差。 因此,混合图谱的基因型认定依旧非常依赖法医工作者的个人经验。
由于云算GPM 分析系统主要依赖于研究人员对混合图谱的基因型认定,而混合DNA 的等位基因认定对于结果拆分的正确性起着至关重要的作用。 为排除人工误差,并最大限度地测试云算GPM分析系统的去卷积能力,本研究根据预期的DNA混合图谱结果对电泳图谱进行注释,以期探究其对主要贡献者和次要贡献者的基因型识别情况。 结果显示,混合图谱的拆分结果并不能确保100%的准确性,在13 例混合样本中,除样本1 的拆分结果完全正确外,其他混合样本或多或少均存在差错(图3)。主要贡献者的总体分型准确率(93.04%)高于次要贡献者(50.48%),该结果与常识相符,主要贡献者因其投入量高的原因,信号也往往更强,能提供更为确切的信息[16-17]。
在法医实践中,拆分所得的基因型往往有同罪犯数据库比对的需求,因此,用于比对的基因座分型正确性对于嫌疑目标的筛选至关重要。基因座在群体遗传学中的多态性及其在特定检测试剂盒中的性能表现,造成了其拆分的难易度存在差异。本研究中,各基因座拆分后的准确率显示出了明显的差异,基因型拆分准确性最高的基因座是D7S820,其准确率(89.29%)约为最低者(D21S11 和D12S391,53.57%)的1.67 倍(图3),这提示某些基因座或许不适用于混合样本的检测。 如法医工作人员有进行数据库比对的必要,则可优先选择weight 值较高的基因型,避免因基因型的拆分差错而导致嫌疑人筛查错误。 就如何判断已拆分基因分型的正确与否,云算GPM 混合图谱分析系统内置的weight 值可用于判断结果的可靠性,一般而言,weight 值大于90%被认为是分型结果可靠的指标[18]。 本研究中,各分型结果的weight 值存在差异,分型完全正确的基因座weight 值最高,部分正确与完全错误的基因座weight 值均较低,且这两者间的差异也相对较小[图4(a)]。 与此同时,基于weight值的频率分布图也显示绝大多数weight 值大于90%的分型结果是完全正确的[图4(b)]。 以上结果表明,区分基因座分型结果完全正确者相对较易,weight 值大于90%是基因分型正确的既不充分也不必要条件。
4 结论
服务于单一来源样本的STR 分型检测试剂盒对混合样本的分型帮助有限,现有的混合样本拆分软件也不能解决混合图谱中的基因型认定问题,混合样本中的等位基因识别仍旧高度依赖法医工作者的个人经验。 基因分型的准确与否与该贡献者的占比存在相关性,weight 值大于90%是一个相对可靠的分型结果评价指标。 由于多数样本并未达到100%的分型一致性, 因此在有参考数据库的情况下,选择weight 值较高的基因座进行嫌疑人的筛选或许是最佳选择。 综上,本研究测试了云算GPM 混合图谱分析系统对混合样本基因型拆分的结果,探讨了该系统在混合样本基因型拆分上的可靠性,可为云算GPM 混合图谱分析系统的使用及未来优化提供参考数据。

