边界加权的甲状腺癌病理图像细胞核分割方法
韩 冰,高 路,高新波,2,陈玮铭
(1.西安电子科技大学 电子工程学院,陕西 西安 710071;2.重庆邮电大学 计算机科学与技术学院 图像认知重庆市重点实验室,重庆 400065)
1 引 言
甲状腺癌是实体癌中发病率增速最快的恶性肿瘤之一[1],但其治愈率也很高。如果能及早诊断,大多数甲状腺癌都能被很好地治愈。病理学诊断是医生诊断疾病的黄金标准,受到权威机构的广泛认可。病理图像中,细胞核形态的差异是目前肿瘤诊断的主要依据。细胞核的平均大小、密度、形状等特征关系到肿瘤的临床诊断和后续治疗。因此,对病理图像中细胞核的精确分割是进一步分析的基础。
卷积神经网络(Convolutional Neural Networks,CNN)[2]因其优秀的特征表示能力被广泛应用于图像分割等领域中,尤其是应用于自然图像时表现优异。相比于自然图像,医学图像含有更多的噪声,目标边界也比较模糊。因此,针对医学图像分割,需要设计合适的方法。2015年,RONNEBERGER等[3]提出了针对医学图像分割U-Net网络模型,在上采样的过程中通过跳转链接逐步融合浅层特征,并在多个医学图像数据库上取得了很好的分割效果,是医学图像分割领域的经典之作,很多算法都是在U-Net的基础上进行改进的。UNet++[4]在跳转链接中添加了密集的卷积模块,但是这种方法无法获得足够的全局信息。UNet 3+[5]重新设计了跳转链接以融合底层和高层之间不同尺度的特征。ZHANG等[6]结合残差模块原理设计了ResUnet。ResUnet++[7]在ResUnet的基础上整合了SE模块[8]、特征金字塔[9]和注意力模块。使用注意力机制来改进U-Net也在一些其它的工作[10-12]中取得了不错的效果。FU等[13]提出M-Net用于视杯视盘的分割,该网络接受多尺度输入,并通过深层监督训练网络。QIN等[14]提出了由两层UNet嵌套组成的U2-Net,通过残差结构获取不同尺度的上下文信息。UTNet[15]首次将Transformer[16]模块融入到U-Net中以提升传统U-Net网络的分割效果。CGNet[17]通过设计上下文信息引导模块同时学习图像的局部特征和全局特征,获得了较好的检测效果。
细胞核分割是病理图像自动分析的关键步骤,是医学图像分割任务中最为基础的组成部分。与其它医学图像相比,病理图像背景复杂,细胞核目标小而密集,且形状多变。上述方法常用于分割病灶或器官等图像中单一且占比较大的目标,直接用于细胞核分割时,效果不好。因此,一些适用于细胞核分割的模型相继被提出。
CHEN等[18]提出了DCAN,这是一种通过利用实例和轮廓的互补信息来分割细胞核的深度轮廓感知网络。ZENG等[19]提出了RIC-Unet,在U-Net的基础上添加了一条解码器分支用来预测轮廓以完善分割结果。ODA等[20]提出了BESNet来进行细胞核分割,与RIC-Unet相同,添加了一条解码器分支用来预测轮廓,区别是BESNet中的两个解码器通过串联的方式进行特征互补。ZHOU等[21]提出了基于轮廓感知与信息融合的细胞核分割网络CIA-Net,与BESNet一样有一个预测轮廓的解码分支,不同之处是该方法使用一个信息聚合模块在上采样的同时不断聚合和交换两个解码器的特征,并使用深度监督来加强网络对多层次信息的学习。NAYLOR等[22]提出将分割标签转化为距离热图,并以此训练细胞核分割模型。
在图像中,细胞核的边界只占目标区域的一小部分。因此,当分割算法能够分割出目标的大部分区域时,损失已经很低,这时网络已经很难再优化。换句话说,训练得到的网络可以得到较好的客观结果,但仍然不能有效判别目标的边界。所以分割任务中有一个普遍存在的现象,即距离边界越近,分割精度越低,这一现象在文献[23]中已被证明。
甲状腺癌细胞核不同于其它类型的细胞,一般会出现沟壑,或有核内假包涵体等。由于这些沟壑和包涵体的存在,容易导致分割结果出现空洞等现象。
针对上述问题,设计了一个边界加权模块为细胞核边界附近的像素赋予不同的权重,使网络在训练时就能够更多关注细胞核边界。另一方面,为了避免提出的网络过分关注边界而忽视细胞核主体部分,导致一些染色较浅的细胞核分割失败,提出了前景增强分割网络;该网络在上采样的过程中通过前景增强模块不断增强前景并抑制背景的作用,从而实现甲状腺病理图像细胞核的精确分割。
文中的主要贡献如下:
(1) 提出前景增强分割网络,在U-Net的基础上,在上采样的过程中通过前景增强模块不断增强前景及抑制背景的作用。
(2) 提出边界加权模块,使网络在训练时就能够更多关注细胞核边界,提升边界处的分割效果。
(3) 针对癌变细胞核相对正常细胞核难分割的情况,提出新的损失函数。
(4) 初步构建了甲状腺乳头状癌细胞核分割数据库,在自建数据库和公共数据库上的实验证明了所提方法的有效性。
2 边界加权的细胞核精准分割算法

图1 基于边界加权的细胞核分割算法流程图
2.1 前景增强分割网络
众所周知,在基于编码器-解码器结构的分割网络中,编码器主要是将输入图像映射成高维空间中的特征,解码器对该高维特征进行解码,每个像素位置的解码结果有两种:前景或背景,前景即为目标。分割网络的编码和解码能力是通过损失函数回传梯度来训练的,而网络本身的结构也是决定训练效果的关键。
为了避免在训练过程中,算法过分关注边界而忽视细胞核主体,从而导致一些染色较浅的细胞核分割失败,文中以U-Net为基础网络设计了前景增强分割网络,网络结构如图2所示。CB为拼接块(Concatenate Block,CB)。编码阶段和U-Net分割网络相同,使用5层上采样拼接块(Down-sample and Concatenate Block,DCB)编码层。每个编码层有两个卷积层,每个卷积层后有一个批归一化层(Batch Normalization,BN)和一个ReLU激活函数。编码层之间用池化层连接,以降低输入特征的空间尺寸,增大深层卷积的感受野。
在解码阶段,每个解码层是常规的上采样卷积块,即图2中的UCB(Upsample and Concatenate Block)。由编码器得到的特征图在经过一系列上采样卷积块的解码后,能够区分图像中的细胞核和背景。但当细胞核和背景颜色相近时,细胞核很难从背景中有效分割。
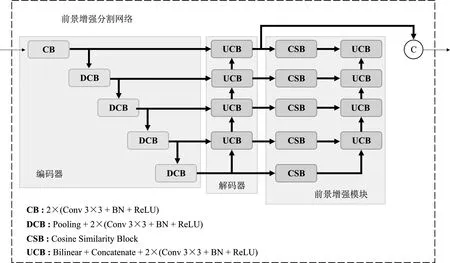
图2 前景增强分割网络结构图
为了增大细胞核与背景之间的特征距离,使网络能够分割出染色较浅的细胞核,设计了前景增强模块(Foreground Enhancement Module,FEM)。如图2所示,它由余弦相似度计算块(Cosine Similarity Block,CSB)和上采样卷积块组成。相似度计算模块的结构如图3所示。
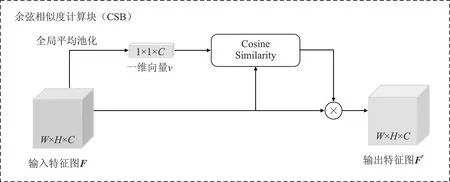
图3 余弦相似度计算模块结构图
相似度计算模块输入尺寸为W×H×C的特征图F,W、H和C分别是特征图的宽、高和通道数。特征图F经过全局平均池化后得到与F通道数相同的一维全局特征向量v1×1×C,通过计算特征向量v和特征图F之间的相似性,可以得到特征图中对应的像素位置与全局特征向量之间的相似性。在训练过程中不断优化迭代该相似性,可将全局特征向量v更靠近细胞核特征向量而远离背景向量,从而达到增强前景的目的。相似度图S的尺寸为W×H。余弦相似度计算如式(1):
(1)
其中,Sv,F(i,j)∈[-1,1]表示v和F在位置(i,j)处的相似度,F(i,j)表示特征图F在位置(i,j)处的特征向量。利用式(2)计算相似度图S与特征图F的最终特征图F*:
(2)
其中,c∈{1,2,…,C}表示特征图通道的索引。
2.2 边界加权模块
为了使分割网络更多地关注细胞核的边界部分,加强网络在细胞核边界附近的分割能力,设计了边界加权模块,用于在训练过程中感知预测边界与真实边界的距离,从而更好地优化分割网络。边界加权模块结构如图1所示。
使用边缘提取算法得到分割边界图Pb,笔者希望通过边界加权后,离边界近的位置赋予较高权重,离边界远的位置赋予较低权重,即边界以外位置元素的像素值是单调递减的。 基于这个思想,设计了边界加权模块中的边界加权核KBW,如图4(a)所示。 边界加权模块的加权核KBW尺寸为n×n,使用图4(a)所示加权核对边界图Pb进行卷积后,边界图中的所有像素都会分配一个权重,距离边界越远,权重越低,距离大于(n-1)/2的像素其权重为0。 Tanh激活函数的作用是将所有权重的值映射到0到1之间,便于后续损失函数的计算。 最终得到输入图像的边界权重图。
边界加权模块的工作方式可以用式(3)表示:
FBW(P)=tanh(KBW*P) ,
(3)
其中,P表示边界加权模块的输入,*表示卷积操作,KBW表示加权核。
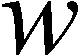
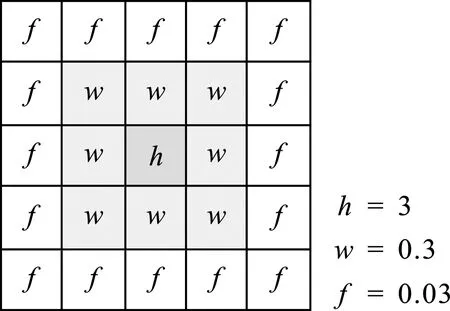
(a) 加权核KBW
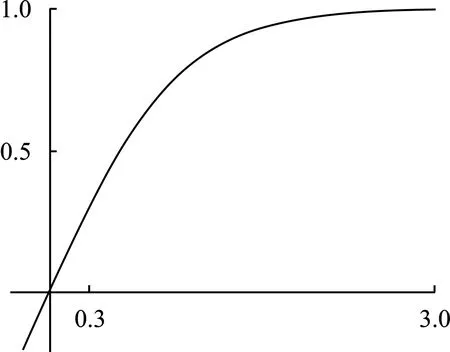
(b) tanh函数图
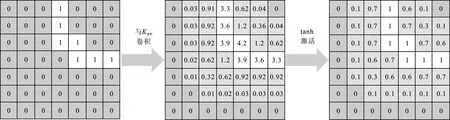
(c) 加权操作示意图
2.3 损失函数
在细胞核分割任务中,需要判断每个像素属于细胞核的概率,因此可将其视为逐像素二分类任务。二元交叉熵(Binary Cross Entropy,BCE)损失函数是二分类中最常用的损失函数。该损失函数的定义如式(4)所示:
(4)
其中,gi表示训练样本标签g的第i个像素的值,若为正样本,则gi取值为1,若为负样本,则取值为0;p表示样本预测为正的概率,pi表示p的第i个像素的值;N表示g中的像素数。
针对细胞核的主体部分和边界部分,在二元交叉熵损失函数的基础上,构建了两个损失函数项,一个是预测结果与真值图之间的损失L1,通过添加权重因子的方式使训练倾向于难分割的部分;另一个是预测结果的边界图与真值图的边界图之间的损失L2。 两个损失的计算如式(5)和式(6)所示:
(5)
L2=LBCE(FBW(P),FBW(G)) ,
(6)
其中,P代表预测结果,G代表手工标注的真值图,FBW(·)代表边界加权模块。则总的损失函数可定义为
(7)
其中,α和β表示两个损失函数的融合系数。通过设定不同的α和β,可以调节目标的主体信息和边界信息在监督训练中的比重。
3 实验结果与分析
3.1 甲状腺癌病理图像分割数据库
在甲状腺癌病理图像分类数据库VIP-TCHis[24]的基础上,构建了细胞核分割数据库VIP-TCHis-Seg。构建数据库所用的病理图像均来自西安西京医院病理科,共收集了55例甲状腺乳头状癌患者的病理图像。每个病理图像有数百万像素,病理医师在显微镜下首先标记出癌变区域,然后在20倍放大下对影像进行剪切,裁切后的图像大小为512×512,数据库统计结果如表1所示,共包含224幅经过剪切后得到的病理图像,用Labelme软件[25]人工标记17 013个细胞核。其中,含正常细胞的病理图像116幅,8 093个细胞核;含甲状腺乳头状癌细胞的病理图像108幅,8 920个细胞核。
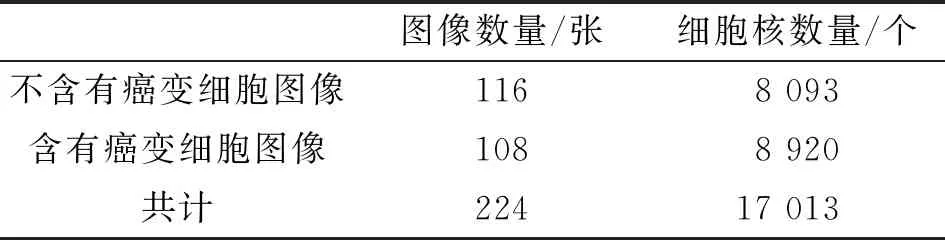
表1 VIP-TCHis-Seg数据库统计
在VIP-TCHis-Seg数据库上构建了6组实验来证明所提方法的有效性,用目前细胞核分割领域常用的相似系数(Dice)和像素准确率(PA)来评估文中的方法。实验在64位Ubuntu 20.04操作系统上使用PyTorch(版本1.1.0)深度学习框架进行。所使用的计算机硬件配置为:Intel(R) Core(TM)i7 CPU @ 3.5 GHz,128 GB内存和一块显存为12 GB的NVIDIA TITAN X GPU。所有实验都在同样的配置下进行并使用相同的训练参数。

图5 VIP-TCHis-Seg数据库样例
3.2 参数设置实验
3.2.1 加权核尺寸k对分割结果的影响
在边界加权模块中,使用不同尺寸的加权核将得到不同宽度的边界图。设加权核KWB的尺寸为k∈{1,3,5,…,},k取奇数可以保证使用加权核对边界图进行卷积后,边界线两侧被加权的像素数量相同,即边界像素两侧距离为d以内的像素会被加权,d可以用k表示,即d=(k-1)/2。k越大,表示距离边界线越远,在训练过程中就能捕获更多的边界分割错误的情况,并以此引导网络优化的方向。但同时k越大,运算复杂度也越大。

表2 使用不同尺寸的加权核的实验结果对比 %
为了验证加权核尺寸k对分割结果的影响,对不同尺寸的边界加权核下的模型进行训练,分割结果如表2所示。
从表2中可以看出,随着加权核尺寸的增大,相似系数Dice从78.49%上升至85.26%再下降到76.86%,像素准确率PA从89.11%上升至95.89%再下降至88.27%。两个评价指标都是在k=5时得到最优值。当k取值较小时,网络不能有效判别边界处的错误分割,因此两个指标结果较低;然而当k取值持续增大,由于细胞核尺寸小、数量多,会出现同一像素被多次加权的情况,这使得边界加权模块不仅未能优化网络,还造成了负面效果,即分割精度降低。
图6给出了不同尺寸加权核实验对比结果。从图中可以看出,当两个细胞核边界距离较小时,k的取值过大会导致边界处分割失败。因此,在后续实验中,令k=5。

图6 使用不同尺寸的加权核的实验结果对比
3.2.2 损失函数中α和β的取值对分割结果的影响
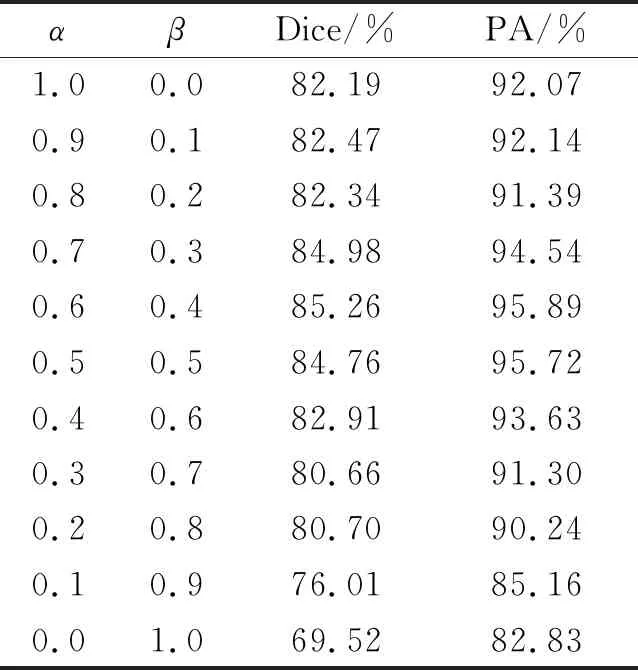
表3 不同融合系数对比实验
损失函数中α和β表示分割的主体部分和边界部分在训练中的作用。设置不同α和不同β以验证其对分割结果的影响。其中,α∈[0,1],β∈[0,1],且α+β=1。实验结果如表3所示。
从表3可以看出,当α=1,β=0时,即损失函数中只有L1起作用,相似系数Dice和像素准确率PA分别为82.19%和92.07%。 随着α减小、β增大,即损失函数中L2的作用不断增大,即网络更加关注边界处的分割,相似系数和像素准确率也不断提升。 当损失函数中α和β分别取0.6和0.4时,网络分割效果最好,相似系数和像素准确率均达到最高值,分别为85.26%和95.89%。 随着α继续减小和β继续增大,相似系数和像素准确率开始下降。 当α=0,β=1.0时,即损失函数中只有L2起作用,相似系数和像素准确率达到最低,分别为69.52%和82.83%。 这说明分割的主体部分在训练中起较大作用,边界部分同样影响着训练的效果。因此,在后续实验中,损失函数L1和L2的融合系数取α=0.6,β=0.4。
图7给出不同融合系数实验对比结果。 当α较大β较小时,网络较多关注细胞核主体部分,虽然能分割出大部分区域,但由于甲状腺癌细胞核会出现沟壑现象,即细胞核内会有浅色区域而非一个完整的颜色一致的细胞,对于细胞核内染色与背景相似的细胞核分割时会出现空洞现象。 而当α较小β较大时,网络的关注点集中于边界部分,从而导致将细胞核内部的沟壑也容易作为边界被分割出来,并出现大面积空洞。 因此,在后续实验中,损失函数中L1和L2的融合系数取α=0.6,β=0.4。

图7 使用不同融合系数的实验结果对比
3.3 边界加权模块与前景增强模块效果实验
为了验证提出的边界加权模块的有效性与前景增强模块是否能够提高网络对细胞核的分割能力,对是否添加边界加权模块和前景增强模块的分割效果进行了对比,实验结果如表4所示。

表4 使用边界加权模块和前景增强模块与否的实验结果对比 %
由表4可知,添加了边界加权模块后,相似系数Dice提高了约1.55%,像素准确率PA提高了约0.70%,说明了边界加权模块的有效性。添加了前景增强模块后,相似系数Dice提高了约5.01%,像素准确率PA提高了约5.28%。由此可知,所提出的前景增强模块能够提高网络对细胞核的分割能力。
图8展示了未使用边界加权模块和使用边界加权模块的分割效果对比。

图8 边界加权模块对分割效果的影响
由图8(c)可以看出,未使用边界加权模块时,细胞核边界处的分割结果不清晰,而图8(d)展示了文中方法可以分割出清晰的边缘,验证了边界加权模块的有效性。
图9为是否添加前景增强模块的主观对比实验结果。可以看出,第1行的图像中,细胞核饱满,边缘清晰,染色较深,与背景对比明显,无论是否添加前景增强模块,都能够得到较好的分割结果。而对于染色较浅,或细胞核内部有区域颜色与背景颜色相同的细胞核,未添加前景增强模块的算法存在分割失败的情况,而添加了前景增强模块的算法能够正确地分割这些较难分割的细胞核。从客观评价指标和主观观测结果上都验证了前景增强模块的有效性。

图9 前景增强模块对分割效果的影响
3.4 不同损失函数的消融实验
针对细胞核的主体部分和边界部分,在二元交叉熵损失函数的基础上构建了一种新的损失函数用于细胞核的精确分割。分别使用文中所提出的损失函数和二元交叉熵损失函数作为网络总体损失函数进行训练。实验结果如表5所示。
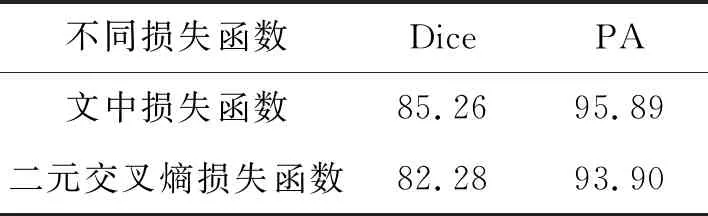
表5 不同损失函数消融实验 %
由表5可以看出,在使用文中所提出的损失函数进行训练后,网络分割效果最好,相似系数Dice和像素准确率PA均为最佳,分别为85.26%和95.89%。相较于使用二元交叉熵损失函数进行训练,使用文中提出的损失函数在相似系数和像素准确率上分别提高了约2.98%和1.99%。实验结果验证了所提损失函数的有效性。
3.5 不同算法的分割效果对比
为了验证所提出方法的有效性,将文中方法与6种分割模型进行对比实验。其中,U-Net[3]是生物医学图像分割中最经典的全卷积架构,也是文中方法的基础模型。U-Net 3+[5]在U-Net的基础上引入了全尺度跳跃连接和深度监督。CGNet[17]设计了上下文信息引导模块,可以同时学习局部特征和全局特征。DCAN[18]是首先使用边界信息来辅助细胞核分割的网络。RIC-Unet[19]和BESNet[20]都使用两个解码路径(边界解码路径和主译码路径)来增强边界学习。CIA-Net[21]在两个特定任务(语义分割和边界检测)的解码器之间采用多级信息聚合模块。UTNet[15]首次将Transformer[16]模块融入到U-Net中以提升传统U-Net网络的分割效果。DCAN,RIC-Unet,BESNet和CIA-Net都使用了目标边界信息来帮助训练分割网络。实验结果如表6所示,所有算法的训练环境和迭代次数、批处理大小、学习率等训练参数设置相同,迭代次数为300轮、批处理大小为2、学习率为0.000 1。
从表6可以看出,所提出的方法在VIP-TCHis-Seg数据库上无论是相似系数Dice还是像素准确率PA,相较于对比算法都取得了最好的结果。其中,相似系数Dice比排名第二位的UTNet算法提高约1.62%;像素准确率比排名第二位的U-Net 3+算法提高了约1.98%。
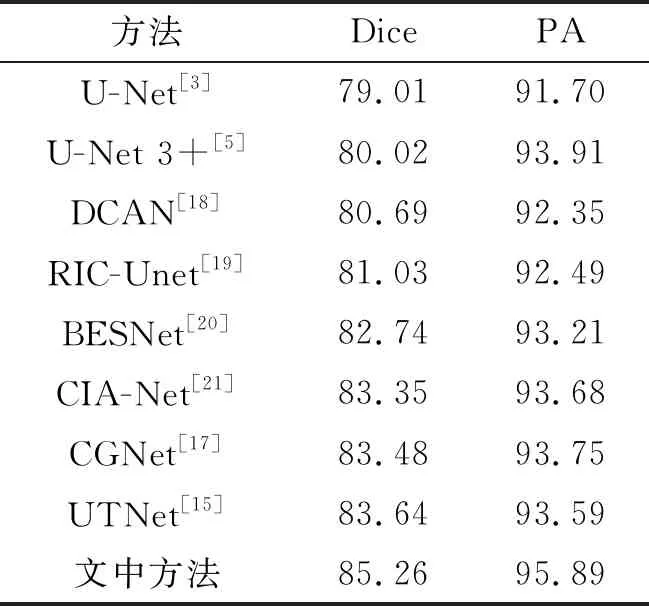
表6 不同方法在自建细胞核数据库上的对比实验 %
各算法在VIP-TCHis-Seg数据库上的主观对比结果如图10所示。可以看出,对于第1行和第2行细胞核比较清晰的图像,所有算法均能够较为准确地分割出细胞核,在边缘等细节处也处理得较好。对于第三行背景不够清晰明确以及细胞核有重叠等较为复杂的图像,U-Net、DCAN和RIC-Unet算法在细胞核部位都出现了较多空洞,如图10(c)、10(d)和10(e)所示;BESNet、CIA-Net和UTNet算法虽然对于细胞核主体部分分割较好,没有出现空洞现象,但是可以看出,3种算法对于边界处的分割不是很成功,如图10(f)、10(g)和10(h)所示。而文中的方法能够分割出所有的细胞核,且分割结果中不存在空洞现象,明显优于其它算法。但是也会错误地将一些染色较深的背景区域划归为细胞核。分割失败案例如图10第4行所示,图中细胞核染色较浅,边界不清晰,当内部染色与背景相同时,需要通过肉眼仔细观察才能分辨出细胞核,这样的细胞核很难成功分割。

图10 自建数据库上不同算法的分割效果对比
3.6 在MoNuSeg数据集上的对比实验
在MoNuSeg细胞核分割数据集[26]上验证文中方法的有效性,该数据集来自于MICCAI 2018多器官细胞核分割挑战赛(Multi-organ nuclei segmentation challenge),包括从18个研究所收集的7个不同器官(乳房、肝脏、肾脏、前列腺、膀胱、结肠和胃)采集的H&E染色组织图像。数据集包括一个训练集和一个测试集。训练集包含30张图像,测试集包含14张图像,图像尺寸均为1 000×1 000像素。在该数据库上的实验结果如表7所示,所有算法的训练环境和迭代次数、批处理大小、学习率等训练参数设置相同。

表7 不同方法在MoNuSeg细胞核分割数据集上的对比实验 %
从表7可以看出,所提方法在MoNuSeg数据库取得了最优的结果。其中,相似系数Dice比排名第二位的CIA-Net算法提高了约0.42%;像素准确率PA比排名第二位的CIA-Net算法提高了约0.53%。
各算法在MoNuSeg细胞核分割数据集上的主观对比结果如图11所示。在第1行和第2行的分割结果中,其它算法均有将背景区域识别为细胞核的情况,而文中算法并没有发生这种错误。在第3行中,原图背景染色较深且细胞核距离较近,其它算法的分割结果中均有不同程度的粘连,而文中算法不仅正确分割所有细胞核且边缘清晰,不存在粘连情况。第4行给出在这个数据库上分割失败的案例,图像中存在与背景难以区分的细胞核,即使人眼观察仍较难区分。

图11 MoNuSeg细胞核分割数据集上不同算法的分割效果对比
4 结束语
针对甲状腺癌病理图像细胞核分割任务中细胞核边界及其附近位置难以分割的问题提出一种基于边界加权的细胞核分割方法。首先设计了一个边界加权模块,该模块能够使网络在训练时关注细胞核边界特征,另一方面提出了前景增强分割模块,避免网络过分关注边界而忽视细胞核主体部分,导致一些染色较浅的细胞核分割失败。在已有甲状腺病理分类数据库的基础上,构建了甲状腺细胞核分割数据库VIP-TCHis-Seg。文中方法与其它的分割算法在自建数据库VIP-TCHis-Seg和MoNuSeg数据库上均取得了最好的结果,证明了算法的有效性。
在实际应用中,细胞核标注需要极大的成本,同时还有漏标与样本不平衡的情况,因此使用弱标注数据进行弱监督训练是未来的工作方向。

