基于单片段代换系的水稻苗高QTL定位和上位性效应分析
张龙廷,吴 静,熊喜娟,董景芳,张少红,赵均良,刘自强,杨梯丰
(1 广东省农业科学院 水稻研究所/广东省水稻育种新技术重点实验室/广东省水稻工程实验室/农业农村部华南优质稻遗传育种重点实验室(省部共建),广东 广州 510640;2 华南农业大学 农学院,广东 广州 510642)
水稻是全球一半以上人口的主食来源,是我国最重要的粮食作物之一。近年来,随着年轻劳动力逐渐流向城市,我国农村人口出现老龄化、过疏化现象。水稻育苗移栽技术消耗大量的人工和物力,成本较高,已经无法适应我国农村劳动力短缺的现状。水稻直播技术相比传统育苗移栽技术减少了育秧环节,可以实现水稻生产的轻型化与机械化,减少劳动力与成本消耗,提高资源利用率[1];但是水稻直播往往伴随着草害的发生,并且难以防除[2]。选用前期生长迅速的直播水稻品种,在营养和光能竞争中优于杂草,将能有效抑制杂草生长,从而达到杂草防控的目的[3-7]。苗高是衡量水稻前期生长速率最重要的指标之一,因此是直播稻育种的重要性状。
随着分子标记技术的发展,关于苗高的QTL研究已经取得了一定进展[8-9]。最早,Redoña等[10]利用F2群体分别在第1、3、5、9号染色体上定位到4个苗高相关QTL,贡献率为7.4%~17.1%。随着对苗高遗传基础研究的重视,利用双亲遗传群体开展苗高QTL定位的研究越来越多[4-6,11-22]。随着测序技术的发展,全基因组关联分析成为定位苗高QTL的有效工具[23-26]。2021年,Yang等[27]利用由籼稻YZX与粳稻02428构建的重组自交系群体,分别在第3、4号染色体上检测到2个苗高相关QTLqSH-3和qSH-4,贡献率分别为6.98%和5.92%。Zeng等[28]利用200份籼稻品种进行全基因组关联分析,定位到32个苗高相关SNP位点,分别位于第1、3、4、5、8、9、10、11、12号染色体上。综观上述研究,大多数研究应用的是重组自交系、分离群体等初级作图群体,这些遗传材料背景复杂,往往会使得QTL的定位不准确;而且大多数的研究只有单次试验的研究结果,因此大多数苗高QTL的稳定性不明确。此外,苗高QTL之间上位性效应的研究也很少。
本研究前期在进行水稻苗高的全基因组关联分析中,发现在3种不同栽培环境下,分别在第3号染色体的32 121 054、32 457 552和33 812 040 bp处检测到与苗高显著相关的SNP;并且在繁种覆盖第3号染色体长臂端的单片段代换系(Single segment substitution line,SSSL)群体时,也观察到来源于IR65598-112-2 SSSL株系的苗高存在差异。因此,本研究选择了4份来源于IR65598-112-2、覆盖水稻第3号染色体长臂端的SSSLs进行苗高QTL的定位,并分析其可能存在的上位性效应,为下一步开展苗高QTL精细定位和克隆打下基础。
1 材料与方法
1.1 试验材料
本研究采用以IR65598-112-2为供体、优良品种‘华粳籼74’为受体构建的覆盖第3号染色体长臂端的4份SSSLs为材料。它们的基本信息如表1所示。用于检测代换片段的分子标记序列见表2。

表1 试验SSSLs的信息Table 1 Information of testing SSSLs

表2 用于检测代换片段的分子标记Table 2 Molecular markers for screening of substitution segments
1.2 苗高的测定
挑选色泽鲜亮、饱满无损的种子,报纸封装后于49 ℃烘箱放置4 d打破休眠,3%(φ)次氯酸钠溶液消毒,然后浸水1 d,将种子平铺在垫有2层经高压灭菌处理滤纸的培养皿底部,加入6 mL无菌水,催芽2 d后用镊子将萌发的种子芽朝上放入96孔黑色培养盒的孔中,培养盒灌满水,移至人工气候箱继续黑暗催芽1 d,调整气候箱强光照、黑暗各12 h,温度30 ℃,相对湿度75%。在气候箱中生长2~3 d,然后换用0.05 %(φ)Yoshida营养液培养,此后每隔7 d换1次0.05 %(φ)Yoshida营养液,自装入培养盒生长14 d后,使用直尺测量水稻幼苗高度(根茎连接处至苗尖端长度)并记录。该试验独立进行3次,每次试验3次重复,每次重复测定20株。
1.3 分子标记
DNA的提取、PCR扩增及带型检测参照杨梯丰等[29]的方法并适当修改。具体程序为:取1~2 cm长嫩叶置于深孔板中,加入高压灭菌处理的钢珠,移液枪吸取800 μL抽提液(6.057 g Tris、17.53 g NaCl、102.68 g蔗糖,定容至1 L,调节pH=8.0)注入样孔,利用Geno Grinder 2010高通量组织研磨仪磨碎,95 ℃水浴30 min,离心机2 000 r/min离心13 min,上清液即为DNA模板。PCR扩增的15 μL反应体系包含0.15 μmol/L正反向引物,200 μmol/L dNTP,1× PCR反应缓冲液(50 mmol/L KCl、10 mmol/L Tris-HCl、1.5 mmol/L MgCl2、0.1 mg/L明胶、pH 8.3),50~100 ng DNA模板,1UTaq酶,ddH2O补齐。PCR反应在S1000型DNA扩增仪(美国BIO-RAD公司)中进行,PCR仪反应程序为94 ℃预变性5 min;94 ℃变性30 s、55℃退火30 s、72 ℃延伸40 s,35次循环;72 ℃终延伸8 min。PCR扩增产物用聚丙烯酰胺凝胶电泳分离,经Goldview染色后用凝胶成像系统(美国BIO-RAD公司)读取带型。
1.4 代换作图
QTL代换作图参考Wissuwa等[30]的方法进行。原理如下:利用分子标记将SSSL的区间表示出来,若有1个QTL存在于几个或多个SSSLs中,则此QTL处在这些SSSLs重叠区域之间;若检测到的QTL只在1个SSSL内,但其不在这个SSSL和其他SSSLs的部分重叠区域,则此QTL处于非重叠区间。
1.5 加性效应分析
利用EXCEL的TTEST函数对试验用SSSLs的苗高数据进行t测验,P< 0.01为差异极显著,P< 0.05为差异显著,筛选与‘华粳籼74’的苗高有显著差异的SSSLs。参考Eshed等[31]的方法计算QTL加性效应及表型贡献率:
1.6 上位性分析
2个QTL之间上位性效应的估算参照赵芳明等[32]的方法。对“包含双QTL SSSL的表型平均值+受体亲本‘华粳籼74’的表型平均值”与“QTL1的表型平均值+QTL2的表型平均值”进行t测验判定差异显著性,当P< 0.05时,认为QTL间存在上位性效应。
1.7 基因组重测序和序列差异分析
样品基因组DNA用Illumina-Hiseq 4000(百迈客,北京)进行测序。对测序得到的原始reads(双端序列)进行质量评估并过滤得到Clean reads,以‘日本晴’IRGSP1.0为参考基因组,用Hisat2工具将测序样品的Clean reads比对到参考基因组上,然后用SNPEFF软件进行SNP和InDel (≤50 bp)检测和变异注释。
2 结果与分析
2.1 苗高QTL的鉴定及其效应
本研究利用SSSLs进行了3次独立的苗高鉴定试验,结果如图1所示。SSSL K1的苗高在试验1中相比于‘华粳籼74’极显著变矮(P< 0.01),在试验2中无显著差异,在试验3中显著矮于‘华粳籼74’(P< 0.05)(图1A);SSSL K11在3次独立试验中均极显著矮于‘华粳籼74’(P< 0.01)(图1B)。
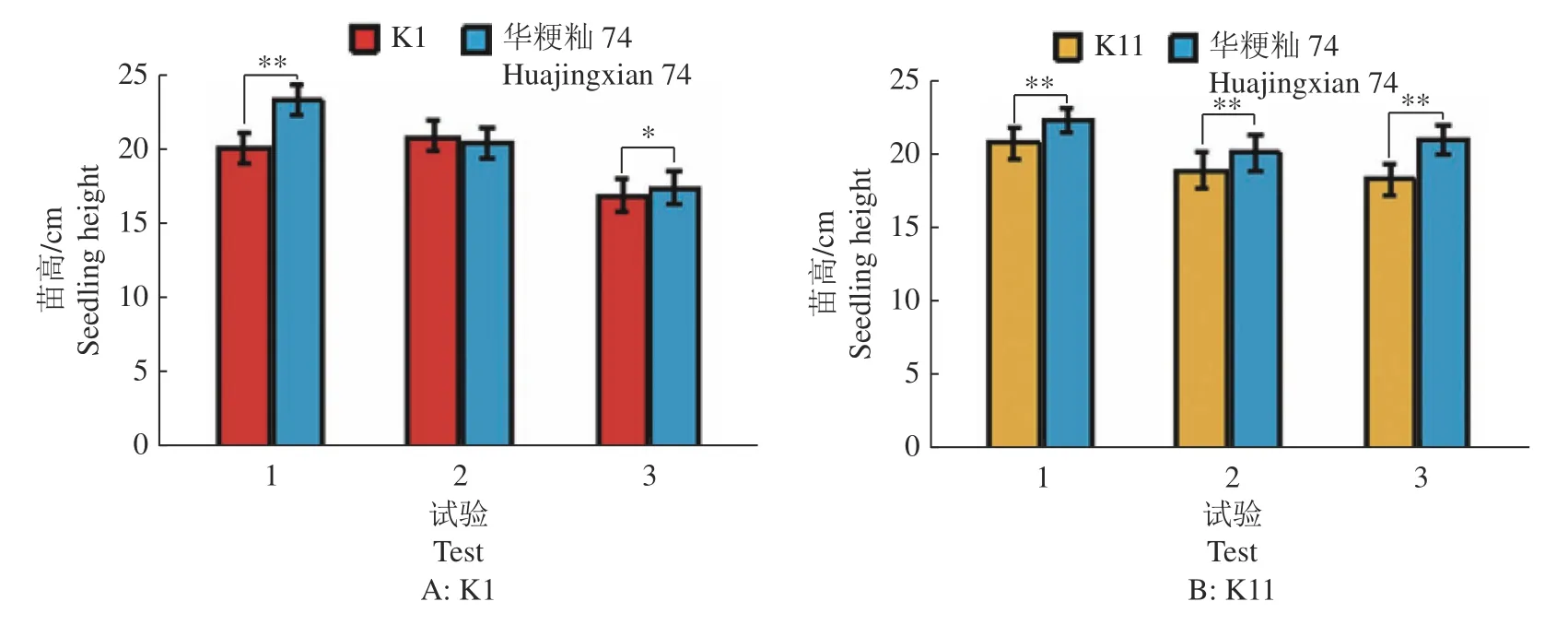
图1 K1、K11与‘华粳籼74’的苗高差异Fig.1 Differences in seedling height between K1,K11 and ‘Huajingxian 74’
通过分子标记对SSSL K1和K11的染色体代换片段进行验证,发现K1的代换片段区间为第3号染色体的31.76—33.08 Mb,它携带1个苗高QTL,该QTL命名为qSH3-1,长度为1.32 Mb;K11的代换片段区间为第3号染色体的33.16—34.81 Mb,它也携带1个苗高QTL,该QTL命名为qSH3-2,长度为1.65 Mb(图2)。
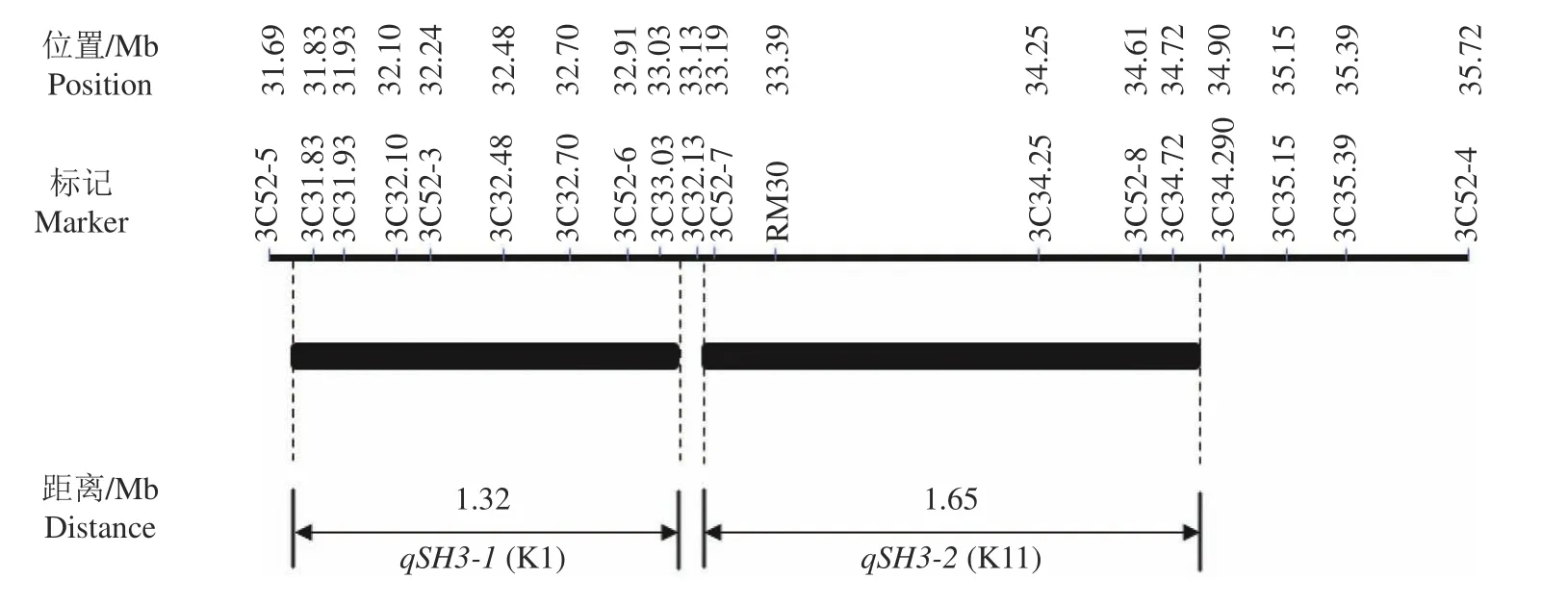
图2 qSH3-1和qSH3-2位置示意图Fig.2 Position schematic diagram of qSH3-1 and qSH3-2
对2个QTL进行加性效应分析,结果如表3所示。qSH3-1在试验1和3的加性效应分别为-1.57和-0.27 cm,在试验2中未检测到加性效应;qSH3-2在3次试验中都检测到加性效应,分别是-0.75、-0.62、-1.33 cm。通过3次试验整体估算,qSH3-1和qSH3-2加性效应分别为-0.86和-1.09 cm,加性效应表型贡献率分别为-4.14%和-5.15%。结合图1、2和表3可知,qSH3-1和qSH3-2是2个相邻的苗高QTL,而qSH3-2稳定性更好。

表3 苗高QTL的加性效应与加性效应表型贡献率1)Table 3 Additive effect and additive effect phenotypic contribution rate of QTL in seedling height
2.2 qSH3-1代换作图
为了进一步缩小苗高QTLqSH3-1的区间,用K1和K6进行代换作图,虽然K1和K6有部分重叠区间,但是苗高检测结果表明:K6的苗高与‘华粳籼74’没有显著差异,而K1与‘华粳籼74’存在显著性差异(图3)。可见,K1与K6的非重叠区域就是qSH3-1的区间,该区间为第3号染色体的32.59—33.08 Mb,长度为0.49 Mb(图4)。

图3 K1、K6和‘华粳籼74’的苗高差异Fig.3 Differences in seedling height of K1,K6 and‘Huajingxian 74’
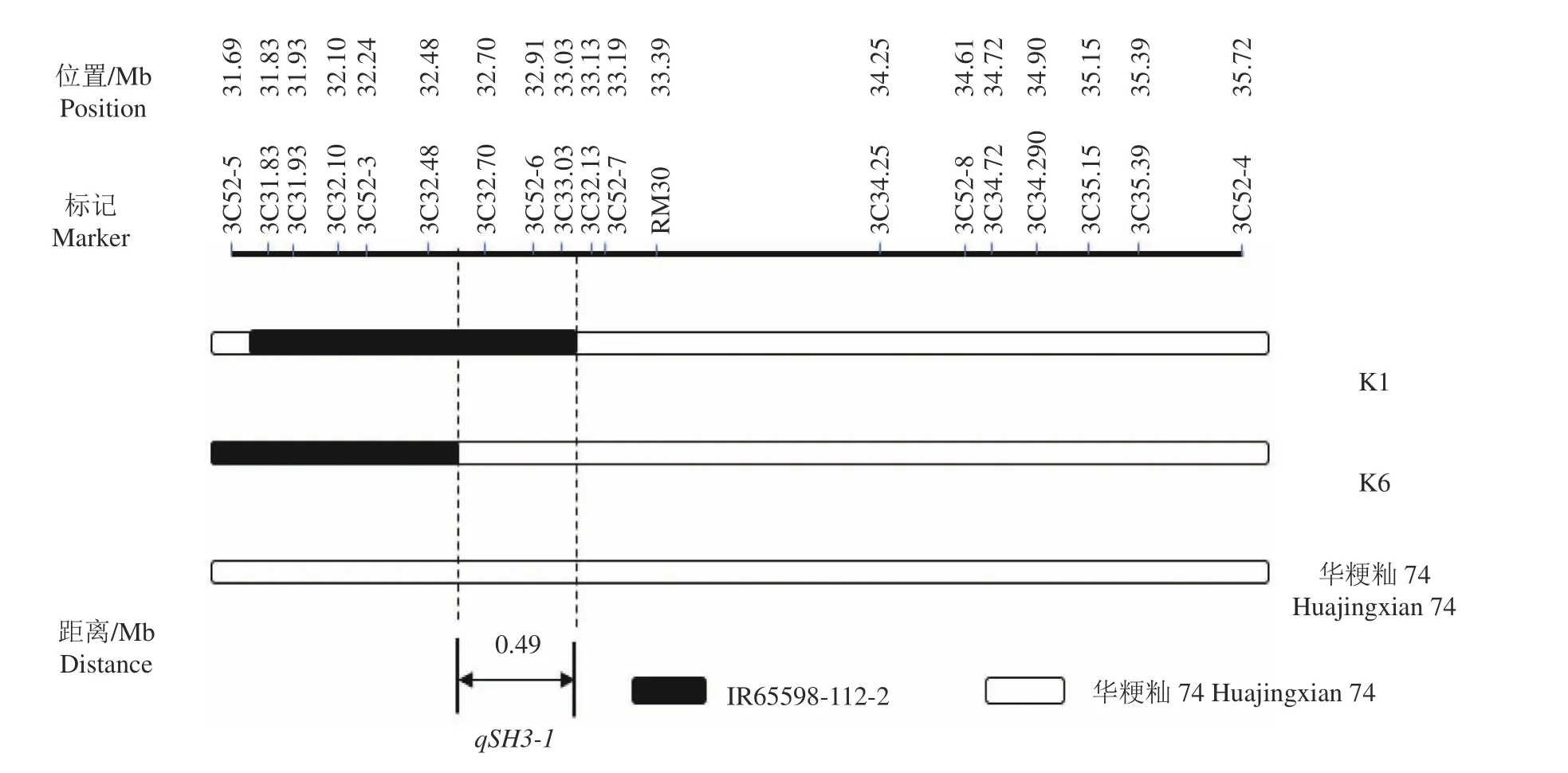
图4 qSH3-1的代换作图Fig.4 Substitution mapping of qSH3-1
2.3 2个QTL区间的候选预测
根据SSSL K13(代换区间为第3号染色体的31.69—34.81 Mb)以及‘华粳籼74’的基因组序列信息,对在qSH3-1(32.59—33.08 Mb)区间内的基因组序列进行差异分析,该区间共有90个预测基因,有变异的基因为79个,其中15个CDS变异的基因,64个启动子变异的基因。同时在CDS区和启动子有变异的基因共有11个;这11个基因的CDS区域有4种变异类型,其中发生移码等较大变异的有4个基因,分别是Os03g0788000、Os03g0790000、Os03g0793100和Os03g0793700(表4)。

表4 qSH3-1和qSH3-2区间内CDS和启动子区域都发生变异的基因Table 4 Variation genes in both CDS and promoter regions in qSH3-1 and qSH3-2 intervals
在qSH3-2(33.16—34.81 Mb)区间,共有270个预测基因,有变异的基因为200个,其中有41个基因在CDS区域发生变异,159个基因在启动子上发生变异。在2个区域都发生变异的基因有31个;这31个基因的CDS区域有7种变异类型,其中发生移码、起始密码子缺失、终止密码子缺失等较大变异的有6个基因,分别是Os03g0814500、Os03g0824300、Os03g0824400、Os03g0826600、Os03g0815100和Os03g0821250(表4)。
2.4 2个QTL的上位性效应分析
分别携带苗高QTLqSH3-1和qSH3-2的SSSL K1和K11的苗高与‘华粳籼74’差异显著,而同时包含苗高QTLqSH3-1和qSH3-2的SSSL K13的苗高与‘华粳籼74’无显著差异(图5)。可见,qSH3-1和qSH3-2单独存在时,水稻苗高会与‘华粳籼74’表现出差异,当qSH3-1和qSH3-2同时存在时,苗高与‘华粳籼74’没有显著差异;因此,qSH3-1和qSH3-2存在显著的上位性效应,为2.07 cm(图5、6)。
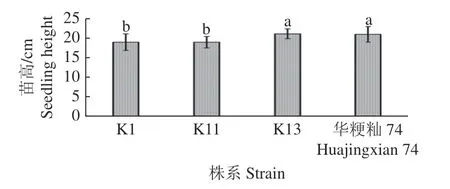
图5 K1、K11、K13和‘华粳籼74’的苗高差异Fig.5 Differences in seedling height of K1,K11,K13 and‘Huajingxian 74’
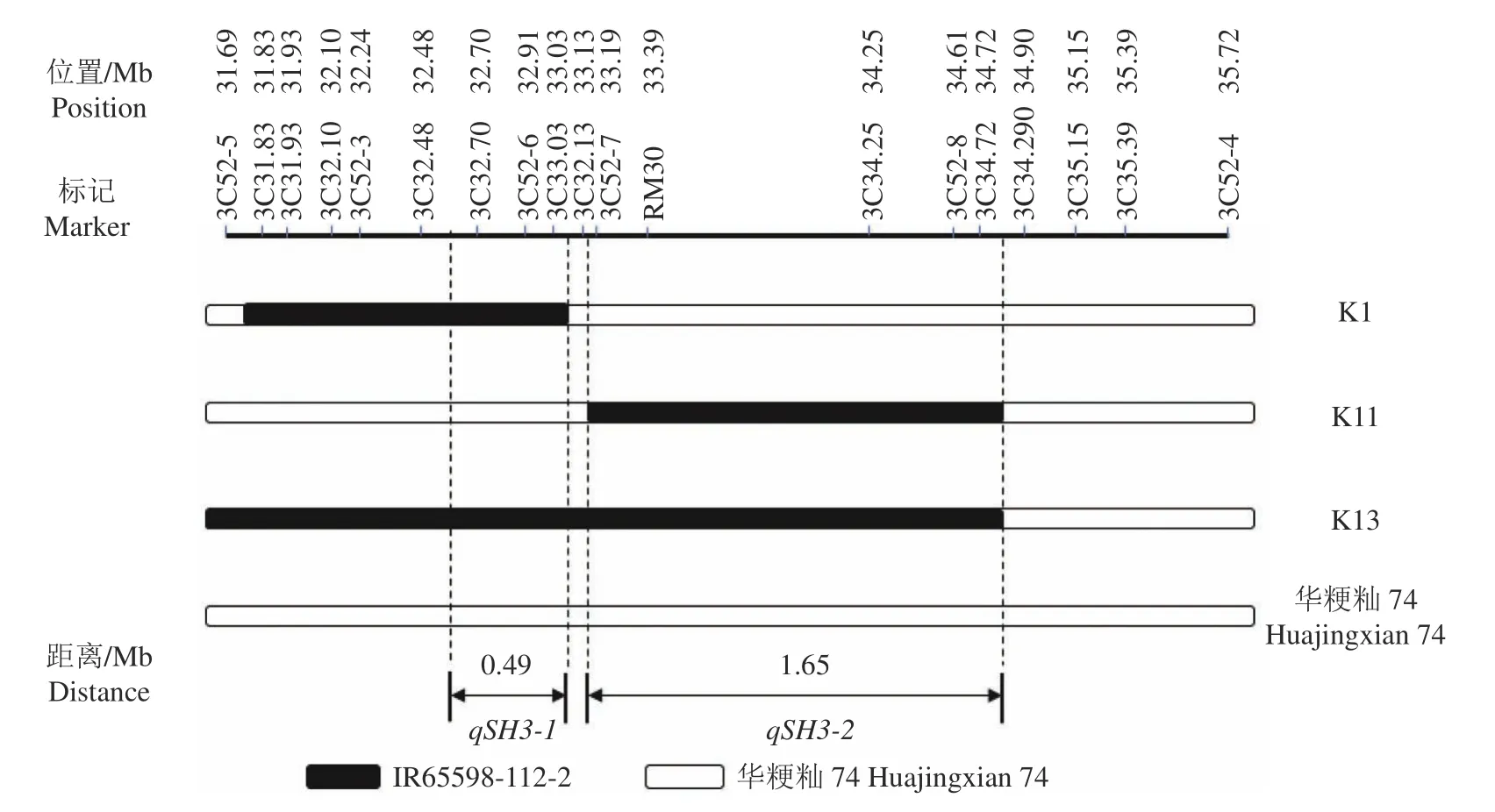
图6 qSH3-1和qSH3-2上位性效应Fig.6 Epistatic effects between qSH3-1 and qSH3-2
3 讨论与结论
3.1 与已有苗高QTL位置的比较
在直播稻中,苗高对于抑制杂草生长非常重要,是水稻直播育种的重要性状之一。剖析水稻苗高QTL是开展分子育种的关键。本研究基于单片段代换系在第3号染色体的32.59—33.08 Mb和33.16—34.81 Mb区间内分别定位到2个相邻的苗高QTLqSH3-1和qSH3-2。水稻第3号染色体控制与苗高相关QTL的研究已经有不少报道[4,6,10,13,16-19,22,25,27-28],在第3号染色体长臂端还克隆了1个苗高基因OsGA20ox1[17,33]。通过定位区间的比较,发现qSH3-1与Yang等[27]定位的qSH-3相邻;qSH3-2包含在qSV3e区间内[24],而且与qPh3和OsGA20ox1相邻[17,19,33]。qSV3e是通过全基因组关联分析得到的QTL区间,可见该QTL在广泛的遗传背景中都有显著的控制苗高的效应。本研究定位的稳定QTLqSH3-2也在该区间内,但是区间范围更小,可能具有广泛的育种应用价值。
3.2 qSH3-2是一个稳定表达的QTL
水稻苗高是多基因控制的复杂性状[10,14,27],不同QTL的表达受环境因素的影响也不同[6,10,14-15,18,21-22],要在生产上产生应用效果,需找到稳定表达的QTL。不少研究通过单次试验鉴定相关QTL[4-5,11-13,16-17,19-20,23-28],但单次试验无法检验QTL效应的稳定性。分子育种中首选稳定表达遗传的QTL,避免不确定性和偶然性,因此开展多次试验准确鉴定稳定可靠的QTL很有必要。在本研究中,3次独立表型试验结果显示,携带苗高QTLqSH3-2的K11与‘华粳籼74’存在显著差异,说明qSH3-2是一个稳定表达的QTL,具有潜在育种应用价值。
3.3 2个苗高QTL之间可能存在上位性
QTL聚合育种除了要定位能稳定表达的QTL,还需要了解用于聚合的QTL之间的互作关系。上位性效应常用来度量QTL之间的互作,被估算为基因型效应与单QTL累加效应的偏差。带有单个QTL的近等基因系是上位性分析的理想材料[34]。SSSL与近等基因系类似,如果片段聚合后的基因型效应显著地偏离各单片段系加性效应之和,则表明片段之间存在上位性。
在本研究中,qSH3-1和qSH3-2的加性效应分别是-0.86和-1.09 cm;而K13包含了qSH3-1和qSH3-2,如果这2个QTL之间没有上位性,K13的加性效应理论值为-1.95 cm;但是,K13的苗高与‘华粳籼74’无显著差异,未检测到加性效应。由于遗传背景一致,因此推测这2个紧密连锁的苗高QTL之间存在显著上位性效应。通过遗传背景一致的SSSL开展聚合育种来培育聚合系,并调查聚合系与亲本的累加加性效应偏差,是检验QTL的上位性最直接有效的手段。因此,在后续研究中,将通过K1和K11的聚合系来进一步确证这2个苗高QTL之间的互作效应。
致谢:感谢华南农业大学张桂权教授提供单片段代换系材料。

