高致病性猪繁殖与呼吸综合征病毒云南株的分离鉴定及遗传变异分析
王金萍,付嘉佳,李富祥,何于雯,宋建领,高华峰
(1.云南省热带亚热带动物病毒病重点实验室,云南昆明 650024;2.云南农业大学,云南昆明 650201)
猪繁殖与呼吸综合征(porcine reproductive and respiratory syndrome,PRRS)又称猪蓝耳病,是由猪繁殖与呼吸综合征病毒(porcine reproductive and respiratory syndrome virus,PRRSV)引起的一种急性接触性传染病,主要表现为妊娠母猪繁殖障碍以及仔猪呼吸道症状[1]。我国于1996年首次从疑似感染猪群中检出PRRSV抗体,并完成了病毒分离[2]。目前,PRRS仍然是制约我国养猪业健康发展的主要疫病之一[3]。
PRRSV属于尼多病毒目动脉炎病毒科动脉炎病毒属成员,其基因组全长约15 kb,有8个开放阅读框(ORF)[4-5]。研究[6]表明,复制酶基因ORF1a编码的聚合蛋白经裂解后产生6个非结构蛋白(NSP1α、NSP1β以及NSP2~5),其中NSP2基因含有B细胞表位免疫的优势基因,该区段的部分基因缺失与毒力增强有重要关系。Ruedas等[7]研究表明,NSP2高变异区90个碱基缺失是毒力增强的关键因素。ORF2—ORF7基因编码病毒结构蛋白,由OFR3基因编码的GP3蛋白为高度糖基化蛋白,是PRRSV各毒株间保守性最差的蛋白之一,其羧基端有1个与病毒血清型有关的非中和抗原表位[8];由ORF5基因编码的囊膜糖蛋白GP5是重要的抗原蛋白,可诱导机体产生中和抗体。在所有结构蛋白中,GP5变异程度最大,其变异可导致不同致病性毒株不断出现及疫苗保护率降低[9-10]。为了解云南省边境地区PRRSV遗传特征及相关基因变异情况,本研究对2018年从云南省孟连县一养殖场分离的PRRSV进行了全基因组、NSP2高变区、GP3及GP5基因核苷酸及推导氨基酸序列遗传进化分析,以期及时掌握边境地区PRRSV遗传变异情况,并为制定有针对性的防控策略提供参考。
1 材料与方法
1.1 材料
1.1.1 病料采集 2018年3月云南省孟连县某养猪场部分猪群发病,病猪表现高热,体温高达41 ℃,腹部、耳及后躯发红,发病率及病死率均在20%以上。从该养殖场采集发病猪只肺脏组织用于病原检测及分离培养,同时采集血清用于抗体检测。
1.1.2 培养基、细胞及毒株 Marc-145细胞、PRRSV阳性血清、PRRSV对照毒株YN-1株(云南省分离的强毒株),由云南省热带亚热带动物病毒病重点实验室保存;MEM培养基、小牛血清,为Gibico公司产品。
1.1.3 主要试剂 一步法RT-PCR试剂、病毒提取试剂、胶回收试剂盒及DNA Marker等分子生物学试剂,购自大连宝生物公司;FITC标记的兔抗猪IgG,购自北京索莱宝公司;引物合成及病毒全基因组测序,由昆明硕擎生物技术有限公司完成。
1.2 PRRSV毒株分离
待Marc-145细胞长成单层后,弃上清,接种1 mL经0.45 μm滤膜过滤的病料样品组织液,37 ℃吸附1 h;弃去吸附后的病料样品,添加含1%小牛血清的MEM维持液,置于CO2培养箱中培养;3~5 d后产生细胞病变(CPE),收集细胞,反复冻融3次后接种Marc-145细胞连续传代3代,待出现稳定的CPE后,收集病毒并置于-70 ℃保存备用。
1.3 PRRSV检测及鉴定
以病毒基因组提取试剂盒分别提取组织样品以及分离培养物上清和沉淀的总RNA,参考文献[11]合成3条引物(P1:5'-TGTCCCYGCYCCGCGCAGGAAGG-3';P2:5'-CGCGTAGAACTGTGACAACAACG-3';P3:5'-GACARGGAGCTGCTGCTTGATGACAC-3')进行检测及病原初步鉴定。其中:P1、P3用于扩增经典毒株及高致病性毒株,扩增片段长度分别为529、439 bp;P2、P3用于扩增高致病性毒株,扩增产物大小为218 bp。RT-PCR反应体系50 μL:酶混合液2 μL,2×Buffer 25 μL,无RNase H2O 16 μL,模板5 μL,上下游引物各1 μL。扩增条件:50 ℃ 反转录30 min;94 ℃ 预变性2 min;94 ℃变性40 s,60 ℃退火30 s,72 ℃延伸30 s,35个循环;72 ℃终延伸7 min。反应结束后,将扩增产物进行1.2%琼脂糖凝胶电泳并以胶回收试剂盒纯化,扩增产物送昆明硕擎生物技术有限公司测序,将所得序列与GenBank中RRRSV序列进行比对验证。

图1 病毒分离培养特性及IFA结果
1.4 PRRSV分离株毒价测定
将出现规律性病变的PRRSV分离株用1%MEM培养基进行10倍倍比稀释,并接种到长成单层Marc-145细胞的96孔细胞培养板中,每个稀释度接种8个孔,每孔接种100 μL,共接种8列,后4列为细胞对照,然后置于5% CO2培养箱中37 ℃培养5 d,逐日观察病变情况并记录。按Reed-Muench法计算病毒TCID50。
1.5 间接免疫荧光试验(IFA)
将病毒接种至长成单层的Marc-145细胞,同时设置不接种病毒的细胞作为空白对照;置于37 ℃ 5% CO2培养箱中培养4 d,待出现CPE后弃去上清,用PBS洗涤细胞3次;加入1 mL 1:10稀释的PRRSV阳性血清,37 ℃孵育30 min;用PBS洗涤3次,加入1:50稀释的FITC兔抗猪IgG,37 ℃孵育30 min;用PBS洗涤3次,置于荧光显微镜下观察。
1.6 病毒全基因组测序及数据处理
提取病毒核酸,完成建库后通过Illumina Novaseq 6 000 PE150进行测序。采用Illumina测序技术完成毒株的基因组扫描测序,全基因组数据获取及组装均由北京擎科生物有限公司完成。
1.7 PRRSV分离株生物信息学分析
将完成测序及拼接后的PRRSV分离株与GenBank收录的34株近年来主要在我国流行的毒株,利用MEGA 11软件构建NJ进化树,分析不同毒株之间的遗传进化特征;构建基于PRRSV非结构蛋白基因NSP2高变区、结构蛋白GP3及GP5编码基因的系统发育进化树,用DNAstar软件分析比对PRRSV全基因组、NSP2高变区、结构蛋白GP3及GP5编码基因序列核苷酸及推导氨基酸的同源性。所选取的PRRSV毒株信息见表1。

表1 我国分离的34株PRRSV基因组序列信息
2 结果
2.1 病毒分离培养特性及毒价测定
将研磨处理后的肺部组织病料接种于Marc-145细胞进行病毒分离培养。在病料接种3~4 d后细胞开始出现聚集,同时变圆、脱落,经连续传代3代后,病毒能稳定适应细胞,并产生典型的CPE(图1-A、B)。将形成稳定CPE的培养物接种Marc-145细胞,培养5 d后完成毒价测定。经检测,其在96孔细胞板的毒价为10-4.88TCID50/0.1 mL。IFA结果(图1-C、D)显示,该病毒能与PRRSV阳性血清发生反应,且细胞膜的荧光强度较细胞核更强。结果表明,该病毒的中和抗原主要表达于细胞膜。
2.2 RT-PCR鉴定
对连续培养3代的病毒分离物,分别提取病毒(本试验分离株及PRRSV对照毒株YN-1)培养上清及细胞沉淀总RNA,经2对鉴定引物扩增NSP2基因高变区后获得了与预期相符的条带,分别为439和218 bp,阴性对照未见扩增(图2)。与经典毒株相比,本试验分离株和YN-1株均为缺失90 bp的强毒株。分离株扩增产物经胶回收完成测序验证,所得序列经Blast分析比对,最后确定为高致病性PRRSV,将其命名为云南孟连分离株(YNML-2018株)。
2.3 YNML-2018株遗传进化分析
通过全序列测定获得YNML-2018株全基因组序列,发现完成全序列拼接后的病毒全基因组大小为15 357 bp。采用DNAstar和MEGA 11软件分别完成YNML-2018株与我国主要流行PRRSV毒株全基因组序列、NSP2高变区、GP3及GP5蛋白编码基因核苷酸及推导氨基酸序列比对分析。结果(表2)显示:分离株YNML-2018全序列核苷酸与近年来我国流行的PRRSV毒株的同源性为95.3%~99.3%,其中同源性最低的为湖北省2012年分离的高致病性毒株HZ-31(95.3%),最高的为广西分离株GX1003(99.3%);YNML-2018株与近年来云南省地方分离毒株同源性较高,变异程度较低,核苷酸同源性为98.9%~99.0%,但与早期国内经典株S1有较大变异,核苷酸同源性仅为89.2%。YNML-2018株NSP2基因高变区长度为1 340 bp,与国内早期流行毒株相比,其基因组有不同程度的碱基缺失,分别是在第2 778~2 780 nt处缺失了3个碱基(TTT),编码1个氨基酸(苯丙氨酸),以及在第2 947~3 033 nt处缺失了87个碱基,编码27个氨基酸(图3),这与近年来的报道一致。NSP2高变区氨基酸分析结果(表2)显示:YNML-2018株与近年来国内流行的高致病性PRRSV毒株氨基酸同源性为92.1%~97.8%,同源性最低的为湖北省2012分离毒株HZ-31(92.1%),最高的为河北分离株HEB-2013(97.8%);与早期经典株S1相比出现较大变异,氨基酸同源性仅为69.5%。GP3、GP5蛋白氨基酸序列分析结果(表2)显示:YNML-2018株结构蛋白GP3氨基酸序列与我国近期流行的高致病性PRRSV毒株同源性为84.3%~99.6%,其中较低的为湖北省分离的高致病性毒株HZ-31(84.3%),最高的为云南分离株YNPL2016(99.6%);结构蛋白GP5氨基酸序列与我国近期流行的高致病性PRRSV毒株同源性为83.1%~99.0%,其中较低的为湖北高致病性毒株HZ-31(83.1%),最高的为河北分离株TS01(99.0%),但与广东省重组毒株QYYZ相比出现较大变异,氨基酸同源性仅为81.1%。

表2 PRRSV YNML-2018株与国内流行株基因核苷酸及推导氨基酸同源性分析结果 单位:%
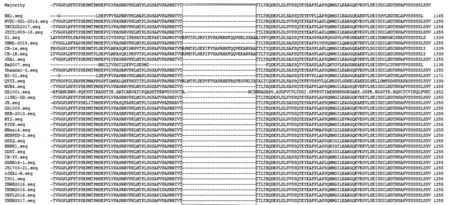
图3 分离株YNML-2018 NSP2高变区氨基酸同源性比较结果

图4 基于NSP2高变区、GP3及GP5蛋白编码基因序列构建的系统发育树
2.4 分离株YNML-2018部分基因系统发育树
基于NSP2基因1 340 bp高变异区、GP3及GP5蛋白编码基因ORF序列构建系统发育树,分析遗传进化特征。基于NSP2高变区序列构建的系统发育树显示,YNML-2018分离株与近年来我国分离的高致病性毒株HEB-2013、Shaanxi-2遗传距离最近,与经典株CH-1a及疫苗株CH-1R遗传距离较远,与S1株遗传距离最远(图4-A)。基于GP3蛋白编码基因构建的系统发育树显示,YNML-2018分离株与Shaanxi-2及近年来云南省流行株YNSM2016遗传距离较近,与S1及HN1株遗传距离最远(图4-B);基于GP5蛋白编码基因构建的系统发育树显示,YNML-2018分离株与GXBB16-1及云南省流行株YNPL2016遗传距离较近,与S1及HN1株遗传距离较远,与重组毒株QYYZ遗传距离最远(图4-C)。以上遗传进化分析表明,云南省边境地区PRRSV分离株YNML-2018与2006年首次报道的高致病性代表 毒株JXA1等国内流行株具有较近的遗传距离,与云南省报道的流行毒株存在一定变异,但变异程度较低,仍与国内流行毒株高度同源。
3 讨论
PRRSV基因组为单股正链RNA,全长约15 kb,含8个ORF。不同毒株基因组存在较大差异,其中NSP2蛋白的氨基酸序列在美洲株和欧洲株之间的同源性仅为32%[5]。鉴于PRRSV的高变异性,针对病原学的研究需要增强。借助成熟的二代测序技术,获得病原基因组信息,以此分析优势流行毒株病原生物学特征,可为PRRS的综合防治及疫苗选择提供科学依据。本研究中,针对云南省边境地区发生的典型病例完成了病原分离鉴定,并对其基因组全序列进行遗传进化分析,明确了云南省边境地区高致病性PRRSV病原学生物特征。
从2006年将以高热为典型特性并伴有较高死亡率的病原确定为NSP2高变区基因缺失的PRRSV以来,NSP2序列越来越成为研究热点。研究[10,12]表明,从1995年首次完成病毒分离至2006年期间,我国流行的主要是以CH1a毒株为代表的经典株;2006年江西省、湖南省等暴发由高致病性毒株(JXA1毒株)引发的疫情以来,以基因亚群V为主导的高致病性毒株成为我国及云南省周边国家的优势流行株,而NSP2序列高变区的基因缺失是这类毒株的重要特征。云南省分离株YNML-2018序列分析显示,该毒株NSP2基因高变区存在90个碱基缺失,表明其是国内近年流行的高致病性毒株;分离株全基因组核苷酸序列分析显示,其与我国近年来流行的高致病性PRRSV毒株序列同源性较高(92.1%~97.8%),表明尽管存在部分位点突变,但本次分离鉴定的YNML-2018毒株仍为近年来我国主要流行的高致病性毒株。
PRRSV基因组中,由ORF5基因编码的病毒囊膜糖蛋白GP5可诱导机体产生中和抗体。在所有结构蛋白基因中ORF5变异程度最大,推测这是由于缺乏RNA依赖性RNA聚合酶(RdRp),从而缺乏校对功能,导致具有不同致病性的毒株不断重组及进化,使其具有较多表型及基因型[13]。GP5变异所产生的中和抗原变异有助于指导疫苗选用。分离株YNML-2018结构蛋白GP5氨基酸分析显示,其与近年我国流行的高致病性PRRSV毒株同源性为83.1%~99.0%,与云南省流行株相比,虽有部分点突变发生,但仍为基因亚群V流行毒株,表明现阶段广泛使用的疫苗仍对其有效。

