1 株牦牛乳源产细菌素融合魏斯氏菌ZW21全基因组测序及序列分析
郑 雪,梁 琪,* ,宋雪梅,张 炎
(1.甘肃农业大学食品科学与工程学院,甘肃 兰州 730070;2.甘肃省功能乳品工程实验室,甘肃 兰州 730070)
甘南地区位于甘肃省西南部,具有独特的地理环境和生物多样性[1-2]。牦牛是甘南藏区牧民的主要经济来源和生活支柱,传统发酵牦牛乳是以牦牛乳为原料经传统方法制作而成的特色牦牛乳制品,在长期发酵过程中经自然驯化使传统牦牛发酵乳中具有丰富的乳酸菌[3-4],从中筛选得到的融合魏斯氏菌(Weissella confusa)ZW21具有较强的抑菌、抗氧化等功能活性。
融合魏斯氏菌属乳杆菌目(Lactobacillales)、明串珠菌科(Leuconostocaceae)、魏斯氏菌属(Weissella),是一类有益乳酸菌[5]。其生物学特性为一种兼性厌氧的革兰氏阳性菌,过氧化氢酶阴性,菌体形态呈杆状或球状[6]。魏斯氏菌属的微生物已被国际乳品联合会列入发酵食品用菌种名单[7],德国科学基金会食品安全委员会也验证了其在传统食品发酵中的使用[8]。该菌属微生物多存在于酸奶、泡菜等各种发酵食品中[9],也定植于人体和动物的肠道中[10-11],具有避免肠道感染、促进消化吸收、降低胆固醇以及癌症发病率等作用[12]。研究表明,W.confusa可产生胞外多糖,具有抗氧化和抗炎等功能特性[13]。此外,W.confusa还具有产细菌素的潜力,Tenea等[14]从W.confusaCys2-2中分离得到的细菌素对沙门氏菌等革兰氏阴性致病菌均具有明显的抑制作用;Malik等[15]通过全基因组测序从W.confusaMBF8-1发现产细菌素的质粒pWcMBF8-1,具有编码3 种抗菌肽相关基因。
全基因组测序技术可通过对生物体进行测序得到其全基因组序列,进而分析物种间的相互作用和物种进化关系,从分子层面对生物体进行解读,从而挖掘功能基因,为筛选优良菌株奠定基础[16]。本课题组前期从传统牦牛发酵乳中分离获得功能活性突出的W.confusaZW21,拟从基因组学层面挖掘该菌的功能基因和代谢通路,以揭示该菌株的关键基因。本研究采用Illumina测序平台对W.confusaZW21进行全基因组测序分析,并利用基因功能(Gene Ontology,GO)、京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)、蛋白质直系同源簇(Clusters of Orthologous Groups,COG)、非冗余蛋白(Non-Redundant Protein,NR)、碳水化合物活性酶(Carbohydrate-Active Enzymes,CAZy)、转运蛋白分类数据库(Transporter Classification Database,TCBD)、Swiss-Prot和Pfam数据库进行基因功能注释,从基因水平揭示W.confusaZW21的关键功能基因,旨在为其抗菌机理提供理论依据。
1 材料与方法
1.1 材料与试剂
W.confusaZW21由甘肃省功能乳品工程实验室前期从甘南牦牛发酵乳中分离并保存。
MRS培养基、MRS琼脂 北京索莱宝科技有限公司;NEBNext®Ultra™ DNA文库制备试剂盒 美国NEB公司。
1.2 仪器与设备
NanoDrop超微量核酸蛋白测定仪 上海光谱仪器有限公司;Qubit 2.0核酸蛋白定量仪 美国Invitrogen公司;Covaris超声波细胞/核酸破碎仪 美国Covaris公司;2100生物分析仪 美国Agilent公司;Illumina Nova Seq PE150全基因组测序系统 美国Illumina公司。
1.3 方法
1.3.1 基因组DNA的提取
-80 ℃冷冻保存的W.confusaZW21采用平板划线法在MRS固体培养基进行活化后,挑取单菌落至MRS液体培养基中纯化,37 ℃培养12 h后离心去除上清液送至北京擎科生物科技有限公司进行后续检测以及文库构建。
采用十二烷基硫酸钠法提取W.confusaZW21基因组DNA。提取的基因组DNA利用超微量核酸蛋白测定仪、核酸蛋白定量仪和0.35%琼脂糖凝胶电泳进行纯度、浓度和完整性检测。
1.3.2 文库构建及测序
选取提取的1 μg DNA作为DNA样本制备材料。使用NEBNext®Ultra™ DNA文库制备试剂盒生成测序文库,并将索引代码添加到每个样品的属性序列。检测合格的DNA样品用超声波破碎仪随机打断成长度约为350 bp的片段,经末端修复、加A尾、加测序接头、纯化、聚合酶链式反应(polymerase chain reaction,PCR)扩增等步骤完成整个文库制备。文库构建完成后,首先使用核酸蛋白定量仪进行初步定量,稀释文库至2 ng/μL,随后使用生物分析仪对文库的Insert Size进行检测,Insert Size符合预期后,使用实时PCR(real-time PCR)方法对文库的有效浓度进行准确定量(文库有效浓度大于3 nmol/L),以保证文库质量。库检合格后,把不同文库按照有效浓度及目标下机数据量的需求pooling后使用Illumina Nova Seq PE150全基因组测序系统进行W.confusaZW21全基因组测序。
1.3.3 功能原件分析及全基因组图谱绘制
过滤除去低质量和长度过短的reads后,对过滤后的reads进行从头组装,并对组装后的draft基因组进行纠错,之后使用SOAPde novo、Abyss、SPAdes拼接工具对测序结果进行组装并使用CISA软件整合3 个软件的组装结果。
对测序结果进行编码基因、非编码RNA(noncoding RNA,ncRNA)、基因岛、重复序列等预测。其中使用基因组工具Prokka对开放阅读框(open reading frame,ORF)进行预测及过滤;核糖体RNA(ribosomal RNA,rRNA)、转运RNA(transfer ribonucleic acid,tRNA)、细菌非编码小RNA(small non-coding RNA,sRNA)分别使用rRNAmmer、tRNAscan-SE、Rfam软件进行预测;转座子使用Transposon PSI软件进行预测;基因岛通过Island Viewer 4(http://www.pathogenomics.sfu.ca/islandviewer/)网站进行预测;重复序列采用Repeat Masker软件(verison 4.0.5)进行预测。
将基因组序列、基因组预测及RNA预测信息整合成gbk(GeneBank)文件,随后采用cgView(https://proksee.ca/)绘制全基因组图谱。
1.3.4 生物信息学分析
采用GO(http://geneontology.org/)、KEGG(https://www.kegg.jp/)、COG(https://www.ncbi.nlm.nih.gov/research/cog/)、NR(ftp://ftp.ncbi.nlm.nih.gov/blast/db/)、TCDB(http://www.tcdb.org/)、CAZy(http://www.cazy.org/)、Pfam(http://pfam.xfam.org/)和Swiss-Prot(http://www.ebi.ac.uk/uniprot)数据库对W.confusaZW21编码基因进行功能注释。
1.3.5 系统发育树构建
将W.confusaZW21测序的16S rDNA序列在NCBI网站中进行BLAST同源性分析,选取相似性99%以上的序列利用MEGA 7.0软件使用邻接法构建系统发育树。
2 结果与分析
2.1 W. confusa ZW21基因组组装及基因组组分
ZW21染色体基因组大小为2.44 M b,总长度为2224179 bp,GC含量为45.66%。如表1所示,W.confusaZW21全基因组中编码基因总长度为1947771 bp,有2175 个编码基因,平均长度为896 bp,占基因组的87.57%;转座子30 个,总长度为1908 bp,平均长度64 bp,占基因组的0.086%;长散在重复序列40 个,总长度为2415 bp,平均长度61 bp,占基因组的0.109%;短分散重复序列18 个,总长度为1122 bp,平均长度62 bp,占基因组的0.050%;tRNA 78 个,总长度为5916 bp;基因岛 5 个,总长度为48800 bp。
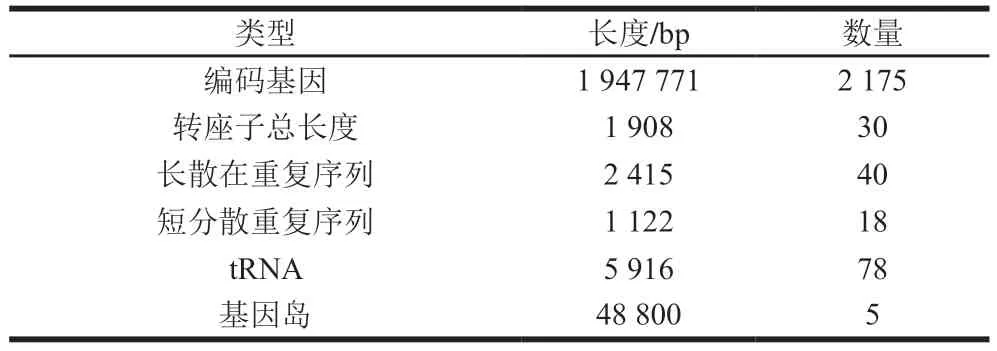
表1 W. confusa ZW21基因组Table 1 Genome statistics of W. confusa ZW21
由图1可知,W.confusaZW21的基因组最内圈(第1圈)是基因组序列位置坐标,其后由内而外第2~3圈分别为基因组GC Skew值分布、基因组GC含量,最外部2 圈为ncRNA及编码序列(coding DNA sequence,CDS),其中内圈为正链、外圈为反链。
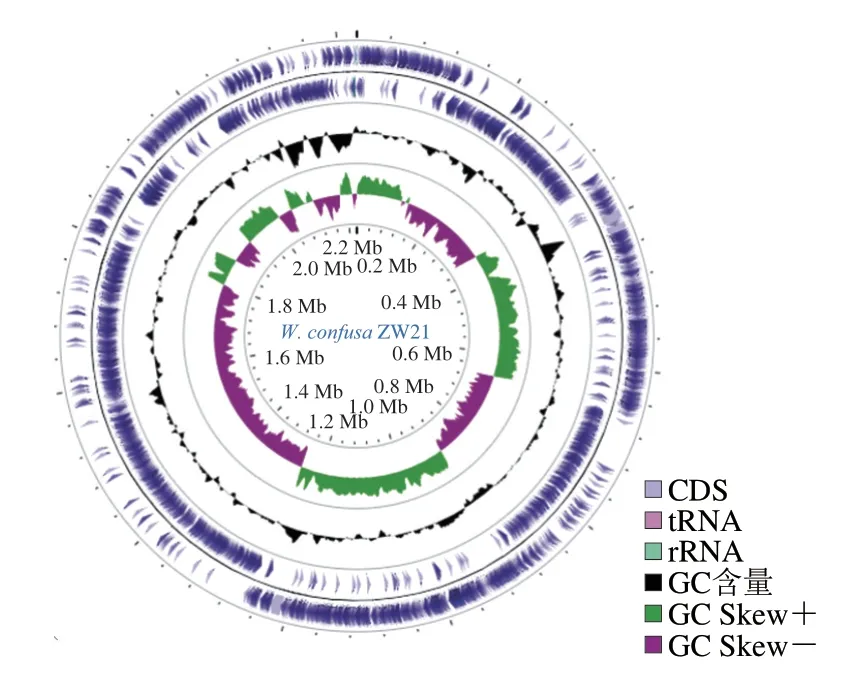
图1 W. confusa ZW21全基因组图谱Fig.1 Whole genome map of W. confusa ZW21
2.2 W. confusa ZW21基因注释
对预测得到的编码基因在GO、KEGG、COG、NR、TCDB和CAZy数据库进行功能注释,如表2所示,W.confusaZW21在GO数据库中共注释到1445 个基因,在COG数据库中共注释到1377 个基因,在KEGG数据库中共注释到1817 个基因,在NR数据库中共注释到2106 个基因;在TCDB中共注释到219 个基因;在CAZy数据库中共注释到77 个基因。

表2 W. confusa ZW21基因功能统计分析Table 2 Statistical analysis of gene functions of W. confusa ZW21
2.2.1 GO数据库注释
GO数据库分为细胞组分(cellular component,CC)、分子功能(molecular function,MF)和生物过程(biological process,BP)3 类。如图2所示,W.confusaZW21在GO分类中共有1445 个基因被注释,其中CC中富集基因数量最多的前3 个功能条目分别为大分子复合物、细胞和细胞部分;MF中转运活性、催化活性和结合是基因数量最多的功能条目;BP中得到最多注释的前3 个功能条目为代谢过程、细胞过程和定位。其中,与抗氧化活性相关的基因ID分别为GM000507、GM000577、GM001988,涉及免疫系统相关基因ID分别为GM000354、GM000169、GM000122、GM000963、GM001770。
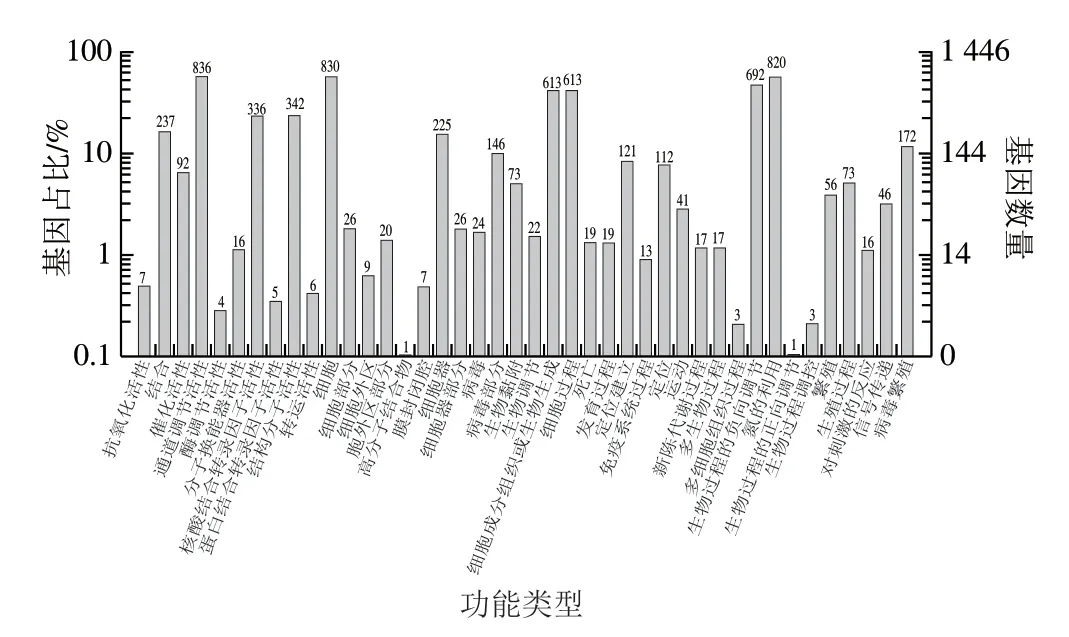
图2 W. confusa ZW21基因功能注释GO功能分类Fig.2 GO functional classification of W. confusa ZW21 gene functional annotation
2.2.2 COG数据库注释
COG数据库是根据细菌、藻类和真核生物完整基因组的编码蛋白系统进化关系分类构建而成。通过比对可以将蛋白序列注释到COG数据库中,进而推测该序列的功能。如图3所示,W.confusaZW21共有1377 个基因被注释,分布于23 个COG条目中。参与复制、重组和修复的基因有93 个,参与翻译、核糖体结构与生物合成的基因有183 个,与转录相关的基因有83 个,参与碳水化合物运输和代谢的基因有129 个,参与氨基酸转运和代谢的基因有113 个。

图3 W. confusa ZW21基因功能注释COG功能分类Fig.3 COG functional classification of W. confusa ZW21 gene functional annotation
注释结果表明W.confusaZW21具有参与合成MFS(major facilitator superfamily)转运蛋白功能的序列(Atg22家族),基因编号为GM 000025;以及合成膜蛋白的功能序列(DUF1440家族),基因编号为GM000592。
2.2.3 KEGG数据库注释
KEGG数据库将生物通路划分为8 类,将W.confusaZW21的氨基酸序列与KEGG 数据库进行比对,将目标物种的基因与其相对应的功能注释信息结合。W.confusaZW21菌株在KEGG数据库中共有1817 个基因分别注释到细胞过程、环境信息处理、遗传信息处理、人类疾病、新陈代谢、组织系统6 大功能的35 个通路。如图4所示,W.confusaZW21在代谢途径和遗传信息处理得到较多的基因功能注释。其中,817 个基因注释到代谢通路,在12 个代谢通路中,碳水化合物代谢相关的基因为118 个,占代谢通路注释基因的14.44%;131 个基因在环境信息处理层面得到注释,其中与信号传导相关的基因为33 个,与膜转运相关的基因为98 个。

图4 W. confusa ZW21基因功能注释KEGG代谢通路图Fig.4 KEGG metabolic pathways of W. confusa ZW21 gene functional annotation
表3所示为W.confusaZW21在KEGG数据库中注释的关于免疫相关基因及其通路信息。W.confusaZW21基因组中包含参与抗生素、生物降解等代谢过程的基因,以及可调控免疫和炎症通路(包括过氧化物酶体增殖物激活受体(peroxisome proliferator activated receptor,PPAR)和核苷酸结合寡聚化结构域样受体蛋白受体(nucleotide-binding oligomerization domain-like receptor,NLR))的组织系统通路相关基因。此外,W.confusaZW21基因组还含有可调节金黄色葡萄球菌感染的通路map05150,涉及5 种相关基因(GM000756、GM001068、GM001069、GM001070和GM001071),在感染疾病通路水平上参与拮抗金黄色葡萄球菌感染。W.confusaZW21中基因GM001319参与原核生物糖代谢调控,参与新霉素、卡那霉素和庆大霉素等抗生素的生物合成;基因GM001149、GM001154、GM001155、GM00115参与单内酰胺类抗生素的生物合成;基因GM000115、GM001122、GM001123参与灵菌红素生物合成;基因GM000057、GM001133、GM001319参与链霉素生物合成。W.confusaZW21中GM000458和GM001545参与PPAR信号通路的调控。W.confusaZW21中1 个基因(GM002091)参与NLR信号通路的调控。此外,W.confusaZW21还存在编码抗叶酸的基因,包括GM001745、GM000317、GM001606、GM000868。

表3 W. confusa ZW21基因组抗菌和免疫调控通路及其相关基因Table 3 Genes related to antibacterial activity and immune regulatory pathways in the genome of W. confusa ZW21
2.2.4 NR数据库注释及系统发育分析
N R 数据库注释结果中包含有物种信息,可用于物种分类。如图5A所示,有1739 个基因注释为W.confusa。基于NCBI数据库已知的W.confusa全基因组和W.confusaZW21全基因组,采用MAGE 7.0软件采用邻接法构建系统发育树。如图5B所示,W.confusaZW21与W.confusaN17的亲缘关系最近,因此,将其鉴定为1 株融合魏斯氏菌。
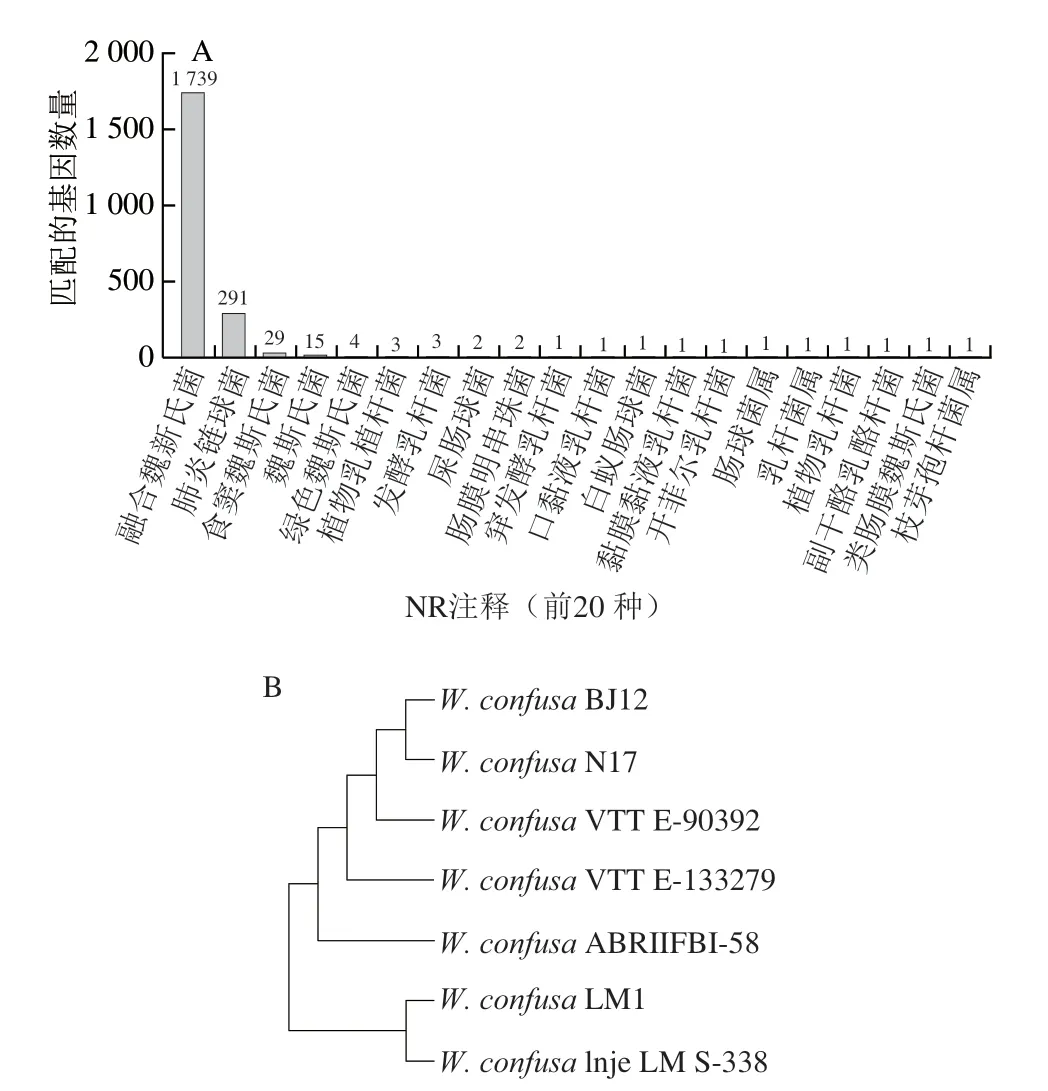
图5 W. confusa ZW21 NR注释(A)及系统发育树(B)Fig.5 NR annotation (A) and phylogenetic tree (B) of W. confusa ZW21
对W.confusaZW21潜在的细菌素基因进行挖掘,如表4所示,W.confusaZW21含有8 个编码细菌素相关基因。其中,2 个编码大肠菌素V基因(GM000070、GM001948),1 个编码含双甘氨酸前导肽的II类细菌素基因(GM001064),1 个编码ABC型抗菌肽转运系统的基因(GM001172),1 个编码杆菌肽抗性蛋白BacA的基因(GM001752),1 个编码乳球菌素家族的基因(GM000397),1 个编码羊毛硫抗生素转运系统ATP结合蛋白nisF的基因(GM002164),以及1 个编码ABC型转运蛋白ATP结合蛋白EcsA的基因(GM000146),证明该菌具有合成并分泌细菌素的潜力。

表4 W. confusa ZW21潜在的细菌素基因Table 4 Potential bacteriocin related genes of W. confusa ZW21
2.2.5W.confusaZW21大肠菌素序列分析
将W.confusaZW21大肠菌素的核苷酸序列翻译成氨基酸序列并在NCBI数据库中进行比对,如图6所示,W.confusaZW21与W.confusaLBAE C39-2的大肠菌素生产蛋白序列(WP_003609935.1)完全相同。

图6 W. confusa ZW21菌株大肠菌素核苷酸序列翻译(A)及氨基酸序列比对(B)Fig.6 Nucleotide sequence translation (A) and amino acid sequence alignment (B) of colicin from W. confusa ZW21
2.2.6 TCDB数据库注释
TCDB数据库是转运蛋白分类数据库,将W.confusaZW21的氨基酸序列与TCDB数据库进行比对,结果如图7所示。ZW21存在初级活性转运蛋白123 个、电化学电位驱动的转运蛋白61 个。

图7 W. confusa ZW21 TCDB注释Fig.7 TCDB annotation of W. confusa ZW21
2.2.7 CAZy数据库注释
如图8所示,W.confusaZW21共有77 个基因被注释,其中碳水化合物结合结构域基因17 个、糖酯酶基因2 个、糖苷水解酶基因35 个、糖基转移酶相关基因23 个。

图8 W. confusa ZW21 CAZy注释Fig.8 CAZy annotation of W. confusa ZW21
3 讨论
细菌素是由包括乳酸菌在内的一些微生物产生的一类强效抗菌肽,一般由核糖体合成,主要对亲缘关系较近的细菌及部分食源性致病菌发挥抗菌活性。因其高效、广谱、安全的特性,目前被认为是抗生素的有效替代品[17-18]。本研究通过对1 株从发酵牦牛乳中分离得到的W.confusaZW21进行全基因组测序和生物信息学分析,解析了W.confusaZW21的基因组结构和具体功能。通过NR、Pfam、Swiss-Prot等数据库在W.confusaZW21基因组中共注释得到包括大肠菌素V和乳球菌素家族在内的5 种已知的细菌素相关基因。大肠菌素主要通过破坏敏感靶细胞的膜电位抑制细菌生长,对革兰氏阴性细菌具有良好的抑制作用[19]。W.confusaZW21全基因组中包含编码大肠菌素VcvpA基因(GM000070、GM001948)、赋予该菌株大肠菌素V抗性的杆菌肽抗性蛋白BacA的基因(GM001752)[20]、编码参与大肠菌素分泌的含双甘氨酸前导肽的II类细菌素基因(GM001064)。由于大肠菌素V符合革兰氏阳性菌中II类细菌素的定义[21],早在1997年Van Belkum等[22]研究提出双甘氨酸型前导肽是II类细菌素分泌所必需,这表明W.confusaZW21具有分泌大肠菌素V的能力。Goh等[23]从W.confusaA3中发现了产大肠菌素V的基因,Amari等[24]在W.confusaLBAE C39-2中也发现大肠菌素V的基因,本研究将W.confusaZW21翻译得到的蛋白序列与W.confusaA3和W.confusaLBAE C39-2产生的V族大肠菌素进行对比后发现,3 株菌株的产大肠菌素V的序列完全相同。此外,本研究在W.confusaZW21基因组中发现编码乳球菌素家族(GM000397)、羊毛硫抗生素转运系统ATP结合蛋白nisF(GM002164)及ABC型转运蛋白ATP结合蛋白EcsA(GM000146)的基因,表明该菌株可能具有产生乳球菌素的潜力。乳酸菌细菌素是由乳酸菌在代谢过程中通过核糖体合成机制产生的一类具有抑菌活性的多肽或前体多肽,具有显著抑制革兰氏阳性细菌的能力[25]。其抗菌机制是穿过病原菌的细胞膜,增加其渗透性,导致离子流失以及膜电位消耗,最终导致病原菌的死亡[26]。此外,在该菌株中还发现了编码ABC型抗菌肽转运系统的基因(GM001172),可能参与上述两种细菌素的分泌。Horn等[27]发现1 株具有分泌乳球菌素和大肠菌素V能力的乳酸乳球菌。本研究结果也显示W.confusaZW21具有联产大肠菌素V和乳球菌素的能力,表明其具有对革兰氏阴性菌和阳性菌的广泛抑制能力。
课题组前期研究表明W.confusaZW21具有抗氧化活性,这与W.confusaZW21的抗氧化基因密切相关。通过GO注释分析发现,W.confusa有3 个基因参与抗氧化过程,5 个基因参与免疫过程。COG注释结果中发现参与合成胆汁耐受基因(MFS型转运蛋白功能Atg22序列)和耐酸基因(DUF1440家族序列),与W.confusaZW21的耐酸、耐胆盐特性相吻合[28]。此外,KEGG数据库的注释结果表明,W.confusaZW21基因组注释到调控免疫和炎症的PPAR和NLR通路,其中PPAR信号通路与能量代谢、细胞分化、增殖、凋亡和炎症反应密切相关[29];NLR可以通过识别细菌细胞壁成分、微生物毒素等外源物质进而参与拮抗入侵细菌,且被证明与炎症反应密切相关[30]。W.confusaZW21还含有编码抗叶酸相关基因,抗叶酸是第一类抗代谢药物,已被证明在治疗癌症和炎症性疾病中有效[31]。已有研究证实,W.confusa的存在可减轻炎症反应,具有成为抗感染强效治疗剂的潜力[32-33]和预防癌细胞增殖的功能[34]。
4 结论
本研究通过对1 株牦牛发酵乳源W.confusaZW21进行全基因组测序,从基因层面对该菌株的抗菌机理进行解析。结果表明,W.confusaZW21基因组大小为2.44 Mb,有2175 个编码基因;W.confusaZW21含有8 种细菌素相关基因,具有合成大肠菌素V及乳球菌素的潜力,其产生的细菌素分别对革兰氏阴性和阳性菌具有抑制作用;除合成基因外,该菌株还具有参与细菌素分泌及赋予菌株细菌素抗性的基因,证明该株W.confusaZW21具有分泌细菌素且不被自身所分泌的细菌素杀灭的能力。但目前极少有关于W.confusa产大肠菌素的报道,因此其产生及分泌的机制仍不清楚。本研究对W.confusaZW21的全基因组测序分析丰富了W.confusa的生物信息学数据库,后续可通过代谢组学的方法深入研究该菌株分泌大肠菌素及乳球菌素的代谢通路,探索其产生机制及其对致病菌的抑菌机理,并进一步对W.confusaZW21所产细菌素的安全性进行评估,为推进该菌株在食品保鲜及防腐方面的应用研究提供理论依据。

