基于DeepMeSHⅡ模型的生物医学文献多标签分类
张 子 寒
(复旦大学计算机科学技术学院 上海 200433)
0 引 言
随着互联网的飞速发展,信息量出现了指数级增长的势头。如果不对信息进行及时的分类存储,将造成大量的无效信息,只能以噪声的形式存在于数据库中。因此,如何整合与处理大量信息成为亟待解决的问题,而通过使用机器学习方法进行文本多标签分类来减少人力的投入,则成为当前NLP研究中的一大热点。
文本多标签分类就是对一段文本信息进行自动分类的过程。与单分类问题相比,各个文本有可能被划分为多个类别而非单个类别,因此多标签分类问题更为符合当前实际问题中数据分类以及索引的需求。在生物医学领域中,科研人员以不同语言发表了大量的论文,取得了丰硕的成果。如何对于文献进行整理和数据分类,使研究成果充分为后人所用是目前该领域面临的紧迫问题。通过进行多标签分类对文档进行检索,对于生物文本挖掘和信息检索有着重要的应用[1-3]。然而,完整的人工索引是昂贵的和劳力密集型的——为了准确有效地索引期刊文章,标注人员必须仔细阅读论文确定文章的主题内容,才能给出准确的标签。本文研究的自动医学多标签分类方法可以节省大量的人工资源。
PubMed数据库[4]建立在国立生物医学信息中心(the US National Center for Biotechnology Information,NCBI)平台上,它是一个免费的文献搜索工具,可以提供生物医学方向的论文和摘要等搜索功能。目前PubMed拥有主要来自MEDLINE的三千多万条生物医学文献的信息。为了方便检索和编撰,医学主题词MeSH(Medical Subject Headings)是美国国立医学图书馆对于生物医学文献统一使用的标签集,对MEDLINE/PubMed数据库标注时就选择利用MeSH这一主题词来对藏书进行索引。美国国立医学图书馆每年投入大量时间与金钱,雇用专业的主题词标注人员对数据库中的论文进行标注,据估计索引一篇生物文献的平均成本约为9.4美元[5]。MeSH词汇表数量很大,为了涵盖生物医学领域的所有方面知识,截至目前已经有近30 000个常用的MeSH标签。
为了鼓励全球研究人员设计新的有效MeSH索引模型和推进这个研究领域,BioASQ(Biomedical Semantic Indexing and Question Answering)[6]由欧盟资助支持,从2013年起每年举办一次[7]。BioASQ是关于生物医学语义索引和问题回答的挑战赛,每年都有生物医学专家作为合作伙伴和第三方的支持者参与该比赛。通常每年的BioASQ比赛分为两个任务,分别是taskA(MeSH语义索引)和taskB(问答)。在MeSH语义索引任务中,主办方提供给参赛者文献的期刊名、标题、摘要、时间等信息,BioASQ的参与模型需要在人类标注者标注之前使用相关的MeSH术语对新的MEDLINE文章进行注释。人类标注者对这些新文章进行手动注释后,这些数据被用来作为ground truth来评估参与者的表现。在本文实验中,研究团队参加了该生物医学主题词预测比赛,并在官方的数据集中进行了模型的分析。
MTI(Medical Text Indexer)[8]是NLM的官方标注模型,主要基于生物医学文章的标题和摘要,结合MetaMap Indexing(MMI)和PubMed相关的文献(PubMed-Related Citations,PRC)[9]进行MeSH标注预测。MetaLabeler[10]首次将MeSH多标签分类问题看作多个二分类问题,并分别使用二元分类器进行预测,曾赢得了BioASQ比赛并为后续模型提供了关键思路。MeSHLabeler[11]在MetaLabeler基础上,集成了KNN等多个分类器的结果,将所有打分存入排序MeSH候选表,并得到最终预测结果。DeepMeSH[12]模型在MeSHLabeler的基础上,加入了文本的深度学习语义表示(Doc2Vec representation)[13]。MeSHProbeNet[14]方法在2019年BioASQ比赛中效果仅次于本文提出的DeepMeSHⅡ,它使用了Bi-GRU[15-16]的方法来对序列进行向量表示,并且在表示计算中加入了自注意力机制[17-18]。
目前方法不足之处在于还有未被挖掘的信息,目前模型仅针对文本本身进行预测,并不能有效地运用文字外的信息。另外,目前还没有模型能够对深度学习和传统机器模型进行融合,而DeepMeSH集成模型中的模块还不够完善,没有效果显著的深度学习模型,排序学习基础模型选择也有待调整。
本文研究的主要贡献在于:(1) 将端到端的深度机器学习模型AttentionXML-base模型融合入DeepMeSH中,提出DeepMeSHⅡ模型;(2) 在特征表示的学习过程中添加MTIFL类期刊的特征表示,通过验证集中的大量实验设计不同的集成方案;(3) 本文提出的DeepMeSHⅡ模型参加了2019年BioASQ比赛,取得了第一名的成绩。
1 相关模型基础
1.1 多标签分类问题
多标签分类的重点是预测文本所属的类别。假设类别数为N,每个样本可以表示成(x,y)的形式,其中x∈X是特征空间X∈Rn中的一个向量表示(在深度学习中,输入为文本信息,特征由端到端自动学习),而y∈Y={-1,1}N表示其标签,其中yk∈{-1,1}表示样本x属于(或不属于)第k类。
机器学习方法是利用决策函数f=(f1,f2,…,fN):X→Y进行标签分数,在实验中需要区分正负样本,最终决定每个样本的预测标签。
1.2 评估方法
本文的标签具有层次结构,评估预测结果有基于最近公共祖先的F-measure评估(Lowest Common Ancestor F-measure,LCA-F)以及基于标签的评估Micro F-measure(MiF),以MiF值为主要排名依据,评估指标如下:
(1) 精确度Accuracy(Acc):
(1)
(2) 实例平均精确率Example Based Precision(EBP):
(2)
(3) 实例平均召回率Example Based Recall(EBR):
(3)
(4) 实例平均F值Example Based F-Measure(EBF):
(4)
(5) 宏平均精确率Macro Precision(MaP):
(5)
(6) 宏平均召回率Macro Recall(MaR):
(6)
(7) 宏平均F值Macro F-Measure(MaF):
(7)
(8) 微平均精确率Micro Precision(MiP):
(8)
(9) 微平均召回率Micro Recall(MiR):
(9)
(10) 微平均F值Micro F-Measure(MiF):
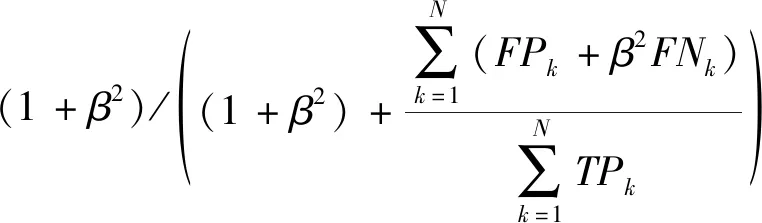
(10)
式中:T表示数据集文本个数;N表示标签个数,precision,i、recall,i和Fscore,i表示对于第i个样本的准确率、召回率和F值,precision,k、recall,i和Fscore,k表示对于第k个标签的准确率、召回率和F值。
实例平均指的是对所有文献的Precision/Recall进行平均;宏平均指的是对于所有标签的指标进行平均;微平均是将所有的正样本、真实样本统计后总体计算的精确率、召回率、F值。比赛中主要关注的参考指标有MiP、MiR和MiF,在评估中β取1。
1.3 排序学习与DeepMeSH
排序学习(Learning to Rank,LTR)是一种监督学习的排序方法,兴起于信息检索领域,目前已被广泛应用到文本挖掘的很多领域。排序学习的核心仍是机器学习,首先确定损失函数后,以最小化损失函数为目标进行优化,得到排序模型的参数。常用的排序学习方法分为pointwise、pairwise和listwise三类,分别将排序结果以三种不同的视角对排序结果进行近似拟合,因此也设计出了三种不同类型的损失函数,本文实验中选择了XGBoost中的排序学习实现。
随着BioASQ比赛的进行与机器学习模型的涌现,出现了基于排序学习的集成模型,其中具有代表性的有MeSHLabeler[11]和DeepMeSH[12]。DeepMeSH模型将多标签文本分类问题看作多个类别上的二分类问题,使用传统机器学习与深度学习对文本进行了不同的特征表示,并使用多种机器学习分类器对其进行二分类预测,得到每个标签的预测概率作为打分,并使用排序学习模型对这些打分进行整合与最终的预测。
1.4 AttentionXML模型
AttentionXML是2019年发表于NeurIPS的工作[19],该模型主要解决大规模文本分类问题,在小型数据集上不使用标签概率树(PLT树)[20],同样有着优秀的表现。在医学主题词分类的项目中,由于标签和数据规模很小,直接使用AttentionXML里的单个分类网络即可,本文称之为AttentionXML-base网络,模型如图1所示。
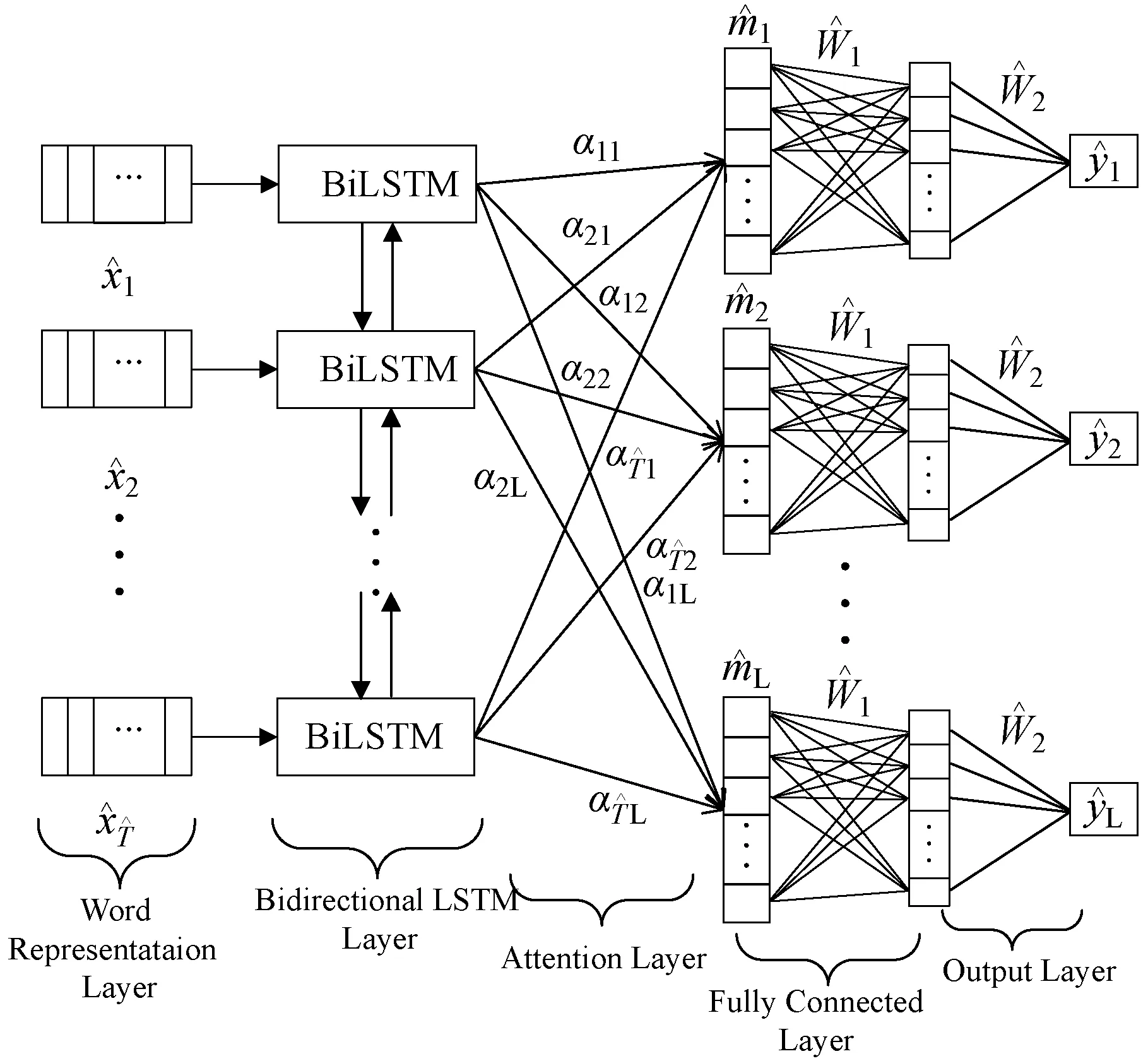
图1 深度网络AttentionXML-base模型
AttentionXML-base模型包含五层:
1) 单词表示层(Word Embedding Layer),用300维的glove模型对单词进行词向量表示,每个句子的最大长度根据数据情况设置为默认值350,空余部分使用Padding填充为0。
2) BiLSTM[21]层,LSTM可以有效地防止梯度消失及梯度爆炸问题[22]。模型中使用双向的LSTM对句子进行表示学习,用式(11)作为LSTM层的表示。
(11)

(12)
(13)
4) 全连接层。AttentionXML-base选择使用一层的全连接层作为分类器的学习使用。该层输出的维度为(N,L,K),其中:N为batch size;L表示标签个数;K表示全连接层输出向量维度。
5) 输出层。在全连接层中,网络为每个标签映射得到了一个K维的特征向量,在输出层中,使用K×1的全连接得到该标签的最终预测得分,得到预测值后使用Binary Cross Entropy Loss作为损失函数进行网络的学习。
模型中使用了dropout[23]机制防止模型过拟合,使用Adam优化器[24]进行梯度下降。
2 DeepMeSHⅡ模型与其优化
2.1 DeepMeSHⅡ模型
随着深度模型AttentionXML-base的提出,本文对其进行了大量的调参实验,使其适用于该生物医学文献分类任务。在DeepMeSH模型的基础上,添加深度模型分支并提出DeepMeSHⅡ模型。在深度模型分支中,模型将输入的文本信息转化为one-hot编码后传入AttentionXML-base模型中进行预测,并参与排序模型的学习。
DeepMeSHⅡ模型在预测文献标签时,通过传统机器学习的方法设计文档的特征,利用SVM、KNN等分类器及模式匹配的方法得到各个标签的打分,通过深度学习多标签分类模型AttentionXML-base得到各标签的得分。结合NLM官方提供的MTI打分,对数据集中进行LTR排序学习,得到每个标签的打分。结合标签个数的回归学习,最终得到预测结果,预测流程如图2所示。
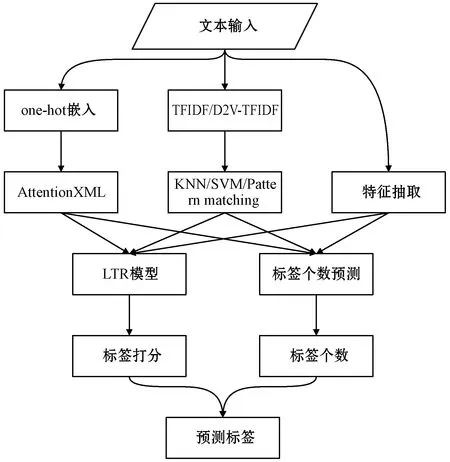
图2 DeepMeSHⅡ模型流程
在实验中,将文献的标题与摘要进行拼接,形成了待预测的文本内容。对于传统文本机器学习分类器,使用常用的TFIDF[25]方法对文本内容进行特征化,使用Doc2vec[26]的方法对文本进行深度学习特征向量表示,并结合TFIDF得到D2V-TFIDF特征表示。在输入TFIDF特征和Doc2vec-TFIDF特征后,使用KNN和SVM两种基础分类器分别对每个标签进行打分。进行KNN分类时,将文献个数为100万的数据集作为KNN的样本空间,取k为200进行k近邻的预测;使用SVM分类器时,对每一个标签采用单独的SVM模型预测,最终得到每个标签的打分。对于深度机器学习分类器,使用上文提到的AttentionXML-base模型进行预测,该深度模型是端到端的,不再以TFIDF等特征作为模型输入,而是直接以one-hot形式将文本信息输入后对其进行预测,通过集中预训练得到的glove模型,作为初始化的词向量表示。
在得到各标签的多组分值后,将分值作为特征,添加已设计好的基于期刊统计信息的特征表示,以及MTI的官方标签打分、PRA相关文献打分等,使用XGBoost排序学习框架[27]进行训练预测,得到标签的最终打分。得到打分后,结合XGBoost模型对每个文献进行标签个数的回归学习,得到每个文献预测的标签数量,并确定最终预测标签。
2.2 训练数据设置
在BioASQ比赛中,通过不断优化模型,DeepMeSHⅡ最终模型的训练集配置如表1所示。共筛选了(BioASQ比赛开始前)最新300万篇MEDLINE文献作为深度模型与传统分类器的训练数据集。在比赛最终阶段(batch 3)时,比赛第一阶段(batch 1)的文献中具有人工标注的28 990篇,第二阶段(batch 2)被标注的有26 718篇文献,这些数据被用于学习LTR排序学习模型。此外,比赛最终阶段(batch 3)中的所有数据以及前两阶段中未被人工标注的数据均作为测试数据进行模型预测。
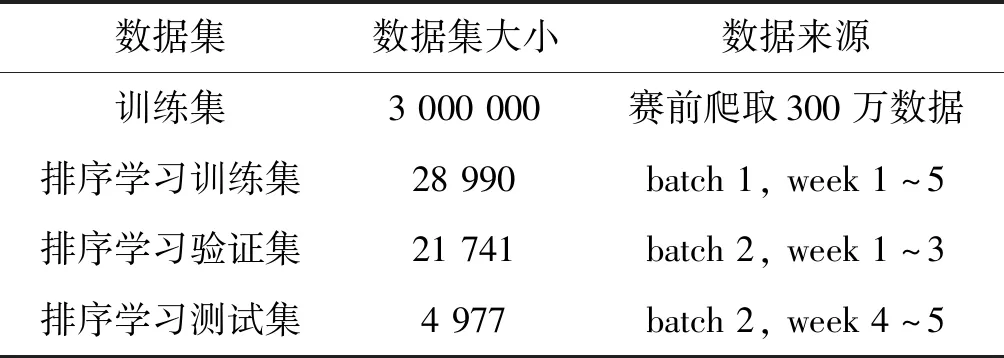
表1 DeepMeSHⅡ模型数据集大小及来源
2.3 改进的特征表示
在PubMed和MEDLINE的数据标注中,并非所有数据均为专业人士直接阅读文献并自行标注所得。MTI First Line是美国国家医学图书馆自动化标准索引方法。对于一部分的期刊,NLM首先使用MTIFL的方法对文献进行标注,接着由专业标注人员添加遗漏的标注、删除错误的标注,并提供出版物类型。这一类标注被官方称作“MTIFL Completion”。这类期刊共有583个,可以在NLM的网站中获得该类期刊的编号,本文称之为“MTIFL类期刊”。
经过实验发现,各方法在MTIFL期刊中的预测效果要明显优于非MTIFL期刊的预测效果,表2展示的batch2中第4周的数据中,DeepMeSHⅡ模型与两种MTI官方模型在MTIFL类期刊中表现均远远高于非MTIFL类期刊。因此,将MTIFL类期刊作为特征加入到排序学习的特征表示中,添加一维的特征表示,令MTIFL类期刊特征为1,非MTIFL类期刊特征为0。加入期刊信息后,平均MIF值获得了0.01的显著提升。
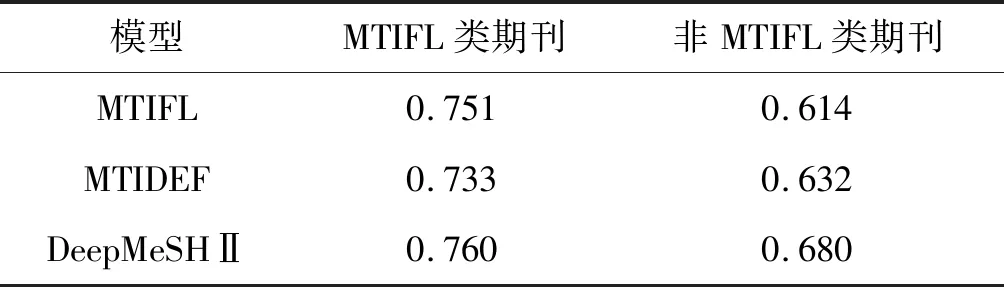
表2 三种模型在两类期刊中MiF指标对比
2.4 DeepMeSHⅡ模型的更多优化
参考BERT模型[28]中Multi-Head的思路,DeepMeSHⅡ同时考虑了多个深度模型的集成。DeepMeSHⅡ将深度模型的分支数设置为2,即同时进行两个参数不同的深度模型的打分预测,并将预测结果传入排序学习模块,以提高模型的鲁棒性与准确率。在期刊信息的挖掘中,DeepMeSHⅡ针对MTIR类期刊设计了不同的排序学习方案,通过提高MTI打分的权重来提升模型的预测效果。
3 实验结果与分析
表3中展示了DeepMeSHⅡ模型在batch3第4周数据中的实验结果,可以看出通过将深度模型融入DeepMeSH模型,实现了深度学习模型与传统机器学习模型的结合,其预测效果有明显的提升。这说明AttentionXML深度学习网络可以学习出传统机器学习难以习得的非线性关系,并以此在DeepMeSH的基础上大幅提高预测精度。然而我们在比赛中发现,无论是MeSHProbeNet或是AttentionXML的单个神经网络模型,相较DeepMeSHⅡ都有一定的差距,这也证明了神经网络模型也有其局限性,与传统模型结合后方可以各取所长。另外在加入MTIFL的期刊特征信息后,预测效果有了进一步的显著提升,这说明了该分类问题中隐藏着未被挖掘的信息,使用机器学习模型自动地进行学习拟合效果有限,人为地对特征进行干预是非常有效的方法。
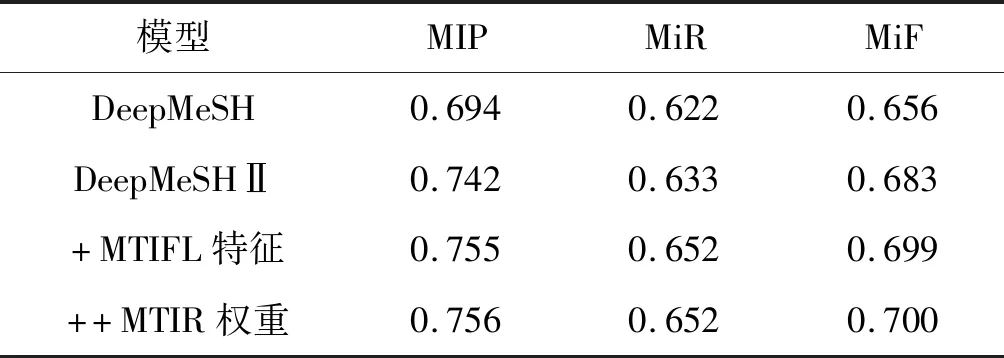
表3 DeepMeSHⅡ模型实验结果
此外,DeepMeSHⅡ对MTIR类期刊在排序学习时进行了单独的加权,使其偏好使用MTI的打分。实际上,该操作使MTIR类期刊中预测结果的MiF指标从0.80提升至了0.92,但由于此类期刊占比过小,对整体模型的提高不够明显。
在2.4节中提到DeepMeSHⅡ使用了双分支的深度学习模型,表4中对该设置进行了对比实验。实验发现相比单分支的深度学习模块,双分支的深度学习模块整体预测效果会有0.002的提升。而分支数继续增加后对实验结果基本不再有影响,因此DeepMeSHⅡ最终将深度学习分支数固定为2。
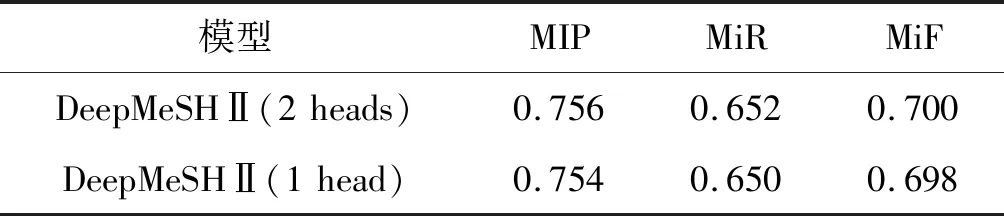
表4 DeepMeSHⅡ模型深度学习分支数对比实验
在MTIFL类期刊信息挖掘的实验中,本文尝试了更多的对比实验:在不使用MTIFL特征时,将MTIFL类期刊与非MTIFL类期刊作为两个不同的任务分别训练模型并预测。在对比实验中我们发现模型效果在MTIFL类期刊中的预测精度没有提高,在非MTIFL类期刊中有精度的降低,整体精度反而略有降低。通过该实验分析得到,使用MTIFL期刊信息作为特征可以联合并有区分地训练两种期刊模型,相较划分数据分别训练不易受到数据量的限制。
在2019年的BioASQ task7A医学主题词自动标注比赛(ECML/PKDD 2019 competition:BioASQ 2019 task7A)中,DeepMeSHⅡ模型获得了第一名的成绩,也成为了当前生物医学文本自动标注方向的最新进展。图3所示为在BioASQ 7A比赛的最后一组测试数据中官方公布的各评估指标结果,其中第二列MiF为最终排名指标,可以看出DeepMeSHⅡ模型的五组预测结果均远远高出了第二名的预测结果。
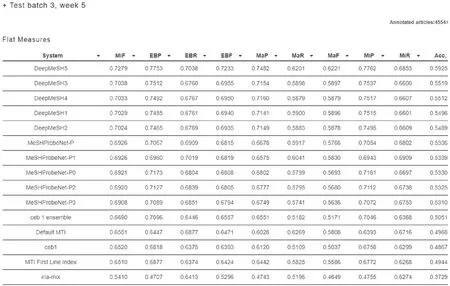
图3 比赛最终周的官方指标排名
4 结 语
本文提出的模型DeepMeSHⅡ达到了领域内SOTA的结果,证明了深度模型与传统机器模型进行集成的可行性与期刊信息挖掘的合理性。但美中不足的是DeepMeSHⅡ作为一个排序学习模型,训练过程过于繁琐。
在接下来的实验中,我们将致力于在不损失模型精度的情况下,尽量降低模型的时间复杂度,尝试减少耗时较多的模块或发掘能够逼近集成效果的端到端模型。

