基于基因组survey 数据的疣吻沙蚕微卫星特征分析及多态标记开发
杨 尉,司圆圆,许瑞雯,陈兴汉
阳江职业技术学院,广东 阳江 529566
疣吻沙蚕 (Tylorrhynchusheterochaetus) 又名疵吻沙蚕,属环节动物门、多毛纲、叶须虫目、沙蚕科,俗称禾虫、流蜞,在中国东南沿海河口地区的泥沙质浅滩或稻田中广泛分布[1-2]。作为一种经济价值极高的多毛类,其味道鲜美且营养丰富,是中国广东、广西、福建、香港、澳门及东南亚各地独具特色的水产品,素有“水中冬虫夏草”之美誉[2-3]。更重要的是,疣吻沙蚕与水稻具有天然的共生关系,发展疣吻沙蚕稻田综合种养模式可显著增加水稻种植的经济效益[4]。“水稻+疣吻沙蚕”生态综合种养技术正日趋成熟,自2021 年起连续3 年入选广东省农业主推技术,在助力乡村振兴、保障国家粮食安全等方面应用潜力巨大。然而,疣吻沙蚕具有生境狭窄、种群地理隔离、群体恢复力低等物种特性,受栖息地破坏、过度捕捞、环境污染等因素的影响其自然种群呈逐年衰退的趋势,在有些地区甚至已经绝迹[4-5]。在突破疣吻沙蚕全人工繁殖技术的基础上,近年已实现其苗种的规模化培育,增养殖业的发展以及技术的革新培育了大量的人工繁育群体,但因遗传育种研究的滞后,疣吻沙蚕种质开始出现生长性能及抗逆性下降等退化现象[4]。为建立疣吻沙蚕资源管理策略并促进其可持续开发利用,特别是避免常年的产业化增养殖引起遗传结构单一化,防止人工繁育群体污染自然种群,有必要采用分子手段科学地分析及评估其种质现状,揭示种群遗传多样性水平与遗传结构,并以此指导新品种的培育与遗传改良研究。
目前,国内外针对疣吻沙蚕的研究主要集中在形态学、生活史、繁殖生物学、增养殖技术、营养与活性成分分析等方面[3,5]。Chen 等[5-6]完成了疣吻沙蚕线粒体基因组测序,并基于线粒体COI 序列分析了7 个地理群体的遗传结构,除此之外,遗传学相关的研究鲜有报道;分子遗传信息的匮乏制约了疣吻沙蚕资源保护利用研究的深入开展。微卫星标记具有多态性丰富、共显性遗传、基因组覆盖广泛等优点[7],该技术操作简便、检测周期短、稳定性高、重复性好,已广泛应用于谱系鉴定、亲缘关系检测、遗传多样性分析、种质资源评价、遗传图谱构建及基因定位等领域[8]。基因组survey 是利用高通量测序技术实施小片段、低深度测序,然后基于Lander-Waterman 模型进行k-mer 分析,并根据k-mer 频率和深度的统计结果评估物种基因组的大小与复杂程度等关键特征[9]。基因组survey 不仅能为全基因组测序及高质量组装提供科学依据,而且对无参考基因组序列的物种而言,利用基因组survey 数据大规模开发微卫星标记是当前最高效的策略之一,较传统方法具有效率高、周期短、成本低的优势[9]。基因组微卫星鉴定研究在水产经济动物中已广泛开展,筛选的标记获得了良好的遗传分析效果。例如,张永德等[9]在卵形鲳鲹 (Trachinotus ovatus) 基因组survey 数据中检测到190 121 个微卫星位点并成功开发了多态分子标记,为全基因组测序与组装、渔业资源保护利用及良种选育奠定了基础;上官清等[10]分析了斑鳢 (Channamaculata) 基因组微卫星特征,并筛选到20 个多态性位点用于群体遗传多样性和遗传结构分析,为该物种的遗传监测、亲缘关系鉴定、种质资源养护及管理提供了技术支持。
本研究对疣吻沙蚕基因组进行低深度高通量测序,通过k-mer 分析预测基因组大小、杂合度和重复比例等信息,对测序数据初步组装后搜索组装序列中的微卫星位点,分析其特征与分布规律,初步验证标记的有效性和多态性,以期指导基因组精细图谱的绘制,并为群体遗传学研究提供可靠的标记资源。
1 材料与方法
1.1 实验材料
疣吻沙蚕采集自阳江市广东阳海农业技术发展有限公司疣吻沙蚕增养殖试验基地(111°55'23"E、21°49'16"N) 保种的越南海防群体。用于基因组测序的疣吻沙蚕幼体用适量无菌水洗涤3 次,解剖后剪取体壁肌肉组织装入2 mL 冻存管,液氮速冻后置于-80 ℃保存。微卫星标记多态性验证群体的30 尾个体经无菌水洗涤后,解剖剪取体壁肌肉组织,置于体积分数为95%的乙醇中在-20 ℃下保存。
1.2 基因组测序及生物信息学分析
运用苯酚-氯仿法提取疣吻沙蚕基因组DNA,经1.0% (w) 琼脂糖凝胶电泳检测完整度后,用NanoDrop 2000 超微量分光光度计 (ThermoFisher,美国) 检测浓度和纯度。利用Covaris 超声波破碎仪将基因组DNA 片段化,筛选合适长度的DNA片段,经末端修复、加A 尾、添加测序接头、纯化及PCR 扩增等步骤建立350 bp 小片段文库。测序文库经Qubit 2.0 (ThermoFisher,美国) 和Agilent 2100 Bioanalyzer (Agilent,美国) 检测浓度和插入片段大小后,用Illumina HiseqTMX Ten 平台进行双末端测序。基因组测序工作委托北京诺禾致源科技股份有限公司完成。原始数据经质控和过滤后,用GCE 1.0.0[11]软件对有效数据进行k-mer 分析。运用SOAPdenovo 2.01[12]软件,选择k-mer=41 将有效数据组装至contig 和scaffold 级别,并统计GC 含量和覆盖深度等信息。
1.3 微卫星位点搜索与引物设计
使用MISA (Microsatellite identification tool) 软件(http://pgrc.ipk-gatersleben.de/misa/misa.html)在长度大于500 bp 的序列中搜索微卫星位点。运行参数为:重复基序长度1~6 bp,单碱基重复次数≥12 次,二碱基重复次数≥6 次,三碱基、四碱基重复次数≥5 次,五碱基、六碱基重复次数≥4 次;将间隔区域长度小于100 bp 的相邻微卫星归为1 个复合型位点。基于微卫星侧翼序列,用Primer 5[13]批量设计引物,主要参数为:引物长度18~27 bp,扩增产物100~300 bp,退火温度(Tm) 55~65 ℃,GC 含量40%~60%,正、反向引物退火温差≤5 ℃;尽量避免出现发卡结构、二聚体、错配和引物二聚体;每个微卫星位点生成3~5 对候选引物。随机选取50 对微卫星引物委托生工生物工程(上海)股份有限公司合成。
1.4 多态微卫星标记的验证与筛选
使用天根生化科技(北京)有限公司的海洋动物组织基因组DNA 提取试剂盒从疣吻沙蚕体壁肌肉组织中提取基因组DNA。PCR 反应体系为20 μL,正、反向引物 (10 μmol·L-1) 各0.5 μL,DNA 模板(15 ng·μL-1) 1.0 μL,2×PCR Mix 10 μL,用超纯水补至20 μL。扩增反应由Bio-Rad My Cycler Thermal Cycler (Bio-Rad,美国) 完成,运行程序为:95 ℃预变性5 min;95 ℃变性30 s,59~60 ℃退火30 s,72 ℃延伸30 s,25 个循环;72 ℃延伸5 min。PCR 产物先进行1.5% (w) 琼脂糖凝胶电泳分析,参照预期片段大小筛选出有效扩增引物。有效扩增引物加荧光接头后 (正向引物5'端添加FAM 荧光素),在30 尾疣吻沙蚕个体的基因组DNA 中扩增以验证其多态性。PCR 产物送至上海翼禾应用生物技术有限公司用3730XL 测序分析仪 (Applied Biosystems,美国) 进行毛细管电泳分析,使用GeneMapper 3.2 软件 (Applied Biosystems,美国) 进行基因分型。
1.5 数据分析
使用Excel 2016 软件完成微卫星位点分布特征信息的统计分析和图表绘制。微卫星发生频率=含微卫星的序列总数/序列总数×100%;微卫星出现频率=微卫星总数/序列总数×100%;微卫星丰度(个·Mb-1)=微卫星总数/序列总长度[14]。用GenAlEx 6.5 软件[15]计算等位基因数 (Na)、有效等位基因数(Ne)、观测杂合度 (Ho)、期望杂合度 (He)、多态信息含量 (PIC)。用Genepop 在线软件 (https://genepop.curtin.edu.au/) 检验位点间的连锁不平衡及群体的哈迪-温伯格平衡 (Hardy-Weinberg Equilibrium,HWE),并用Bonferroni 法对显著性阈值进行校正。
2 结果
2.1 疣吻沙蚕的基因组survey 测序
低深度高通量测序产生的原始数据经质控后共获得57.48 Gb 有效数据,碱基错误率为0.05%,Q20 和Q30 分别为95.56%和89.70%,表明基因组测序质量较高 (表1)。对有效数据进行k-mer 分析(k=17),结果显示在深度为57 时出现主峰值(图1),总k-mer 为44 257 233 158,排除错误k-mer的误差影响后得到修正的基因组大小为759.53 Mb,杂合率为1.41%,重复序列比例为45.92%。选择k-mer=41 将有效读段初步组装至contig 和scaffold 水平,最终获得的contig 总长度为821 637 022 bp,最大长度为84 713 bp,N50 为548 bp;scaffold 总长度为840 375 821 bp,最大长度为89 326 bp,N50 为662 bp。

表1 疣吻沙蚕基因组 survey 测序数据统计Table 1 Statistics of genomic survey sequencing data of T.heterochaetus
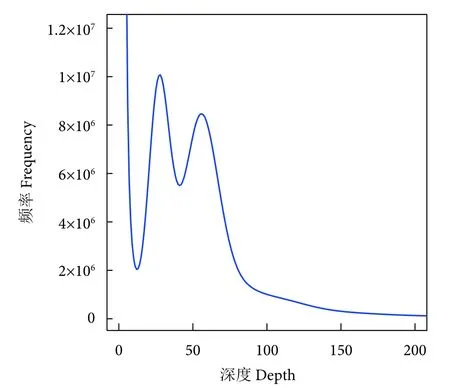
图1 疣吻沙蚕基因组 k-mer 种类频率分布Fig.1 Frequency distribution of k-mer species in genome of T.heterochaetus
2.2 疣吻沙蚕基因组微卫星位点特征
用MISA 软件在109 881 条组装序列中检测到130 216 个微卫星位点,长度共计2 341 179 bp;微卫星发生频率5.04%,出现频率5.97%,分布丰度为154.9 个·Mb-1。疣吻沙蚕微卫星位点以单碱基和二碱基重复最为丰富,分别有45 582 和42 298 条,各占35.00%和32.48%;其次是三碱基重复 (18 782条),占14.42%;六碱基数量最少,仅占2.44%(图2)。微卫星序列的重复数范围为4~56 拷贝,主要集中在4~18 拷贝,重复19 次及以上的有3 703 条,仅占3.50%;单碱基重复以12~16 次最常见 (40 706条,89.30%),二碱基重复集中在6~18 次 (42 035条,99.38%),三碱基重复以5~12 次为主 (18 526条,98.64%),四碱基重复以5~10 次为主 (12 173条,98.91%),而五碱基和六碱基重复主要为4~8 次 (共11 176 条,99.37%) (图2—图3)。
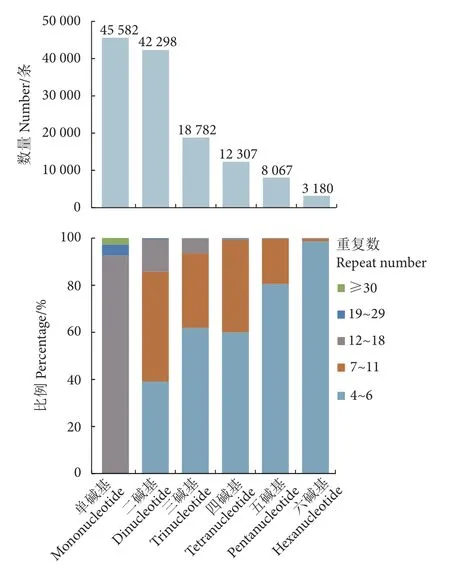
图2 疣吻沙蚕基因组 6 种类型微卫星的数量与比例Fig.2 Number and proportion of six motif types of microsatellite loci in genome of T.heterochaetus
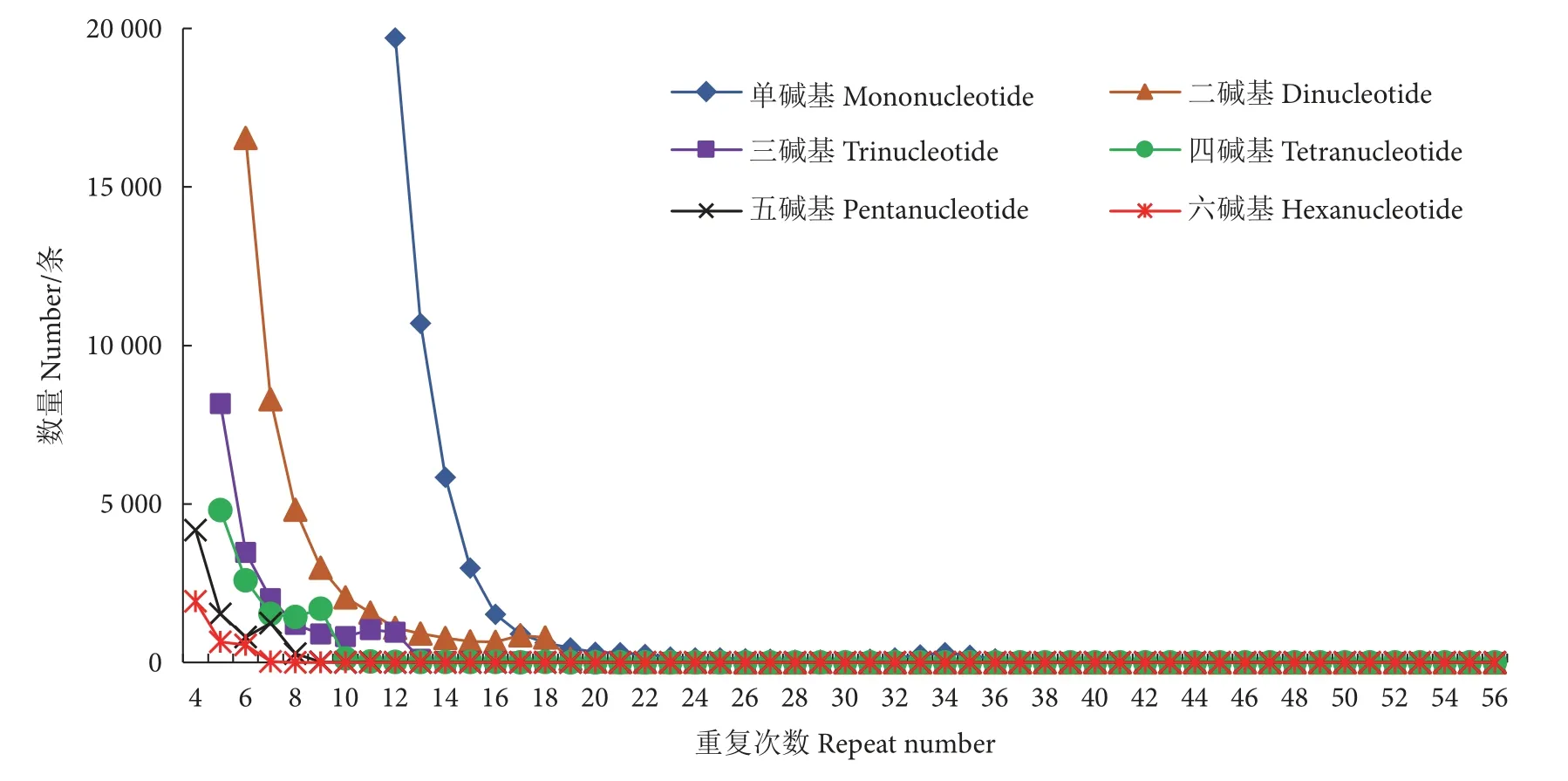
图3 疣吻沙蚕基因组微卫星重复数分布特征Fig.3 Distribution pattern of microsatellite repeat number in genome of T.heterochaetus
疣吻沙蚕基因组微卫星共包含320 种重复基序类型:单碱基2 种、二碱基4 种、三碱基10 种、四碱基31 种、五碱基91 种、六碱基182 种。单碱基重复基序拷贝数集中在12~15 次,以C/G 为主,占比58.02%;二碱基重复拷贝数多为6~10 次,以AT/AT 最为丰富,占比62.38%,其次是AC/GT(27.37%)、AG/CT (10.22%),而CG/CG 数量稀少(0.02%);在三碱基重复中,拷贝数多为5~12 次,占比最高的是AAT/ATT,有6253 条 (33.29%),其次是ATC/GAT (32.15%),而CCG/CGG 数量最少,仅占0.06%;四碱基拷贝数多为5~8 次,优势重复基序是AAAT/ATTT,有3567 条 (28.98%),其次是AATC/GATT (14.08%)、ACTC/GAGT(9.60%)、ATCC/GGAT (7.38%);五碱基、六碱基拷贝数集中在4~7 次,优势重复基序分别是AAAAT/ATTTT (17.07%) 和AACCCT/AGGGTT (11.73%)(图4)。
2.3 疣吻沙蚕基因组微卫星位点长度特征
根据重复序列长度可将微卫星位点分为两类:一类是长度达20 bp 及以上的高度多态I 型,另一类则是长度介于12~19 bp 的中度多态II 型[16]。疣吻沙蚕基因组微卫星位点的长度分布区间为12~336 bp,I 型微卫星位点有43 240 条,占33.21%,剩余的II 型位点占66.79%;绝大部分I 型微卫星位点的长度为20~39 bp,超过50 bp 的位点仅占0.51% (图5-a)。进一步分析发现,在I 型微卫星位点中,二、三、四、五碱基是最主要的重复类型,占85.92%,在后续多态标记的筛选中有较高的开发价值(图5-b)。
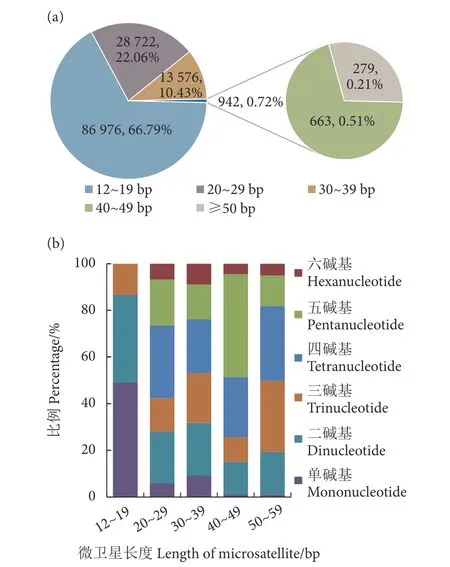
图5 疣吻沙蚕基因组微卫星长度分布特征注:a.不同长度区间微卫星数量及比例;b.不同类型微卫星长度分布特征。Fig.5 Distribution pattern of length of microsatellite loci genome of T.heterochaetusNote: a.Number and percentage of microsatellite loci at different length intervals; b.Length distribution of the six motif types of microsatellite loci.
2.4 疣吻沙蚕基因组微卫星多态标记验证筛选
使用Primer 5 成功对37 370 个微卫星位点设计了引物。随机选取50 个位点合成引物并进行PCR 验证,共获得41 对 (82%) 有效扩增引物,表明MISA 软件鉴定的微卫星位点具有较高有效性。多态性筛选结果显示,15 个 (30%) 微卫星位点的引物表现出稳定且可重复的多态性 (表2,图6)。在30 尾疣吻沙蚕中,15 对引物共检测到87 个等位基因,平均等位基因数5.800,等位基因频率为0.060~0.400,其中ThGM021 位点的等位基因数最少 (Na=2.000),ThGM004 位点的等位基因数最多(Na=12.000)。Ne为1.164~6.713,平均值为3.328;Ho为0.050~0.879,平均值为0.487;He为0.141~0.789,平均值为0.561;PIC 为0.136~0.776,平均值为0.511 (表3)。在15 个位点中,8 个属高度多态性位点 (PIC>0.5),5 个属中度多态性位点(0.25<PIC≤0.5),2 个属低度多态性位点 (PIC≤0.25),表明该群体的遗传多样性较丰富。经Bonferroni 校正后,有3 个位点 (ThGM006、ThGM011、ThGM040) 偏离HWE,其中ThGM040 属于高度多态性位点。各位点间无连锁不平衡现象。

表2 疣吻沙蚕 15 对多态微卫星引物信息Table 2 Information of 15 polymorphic microsatellite loci in genome of T.heterochaetus

表3 15 个多态微卫星位点在疣吻沙蚕群体中的遗传特征Table 3 Genetic characteristics of 15 polymorphic microsatellite loci in a T.heterochaetus population

图6 部分多态微卫星标记的毛细管电泳分型结果Fig.6 A set of polymorphic microsatellite loci visualized by high-resolution capillary electrophoresis
3 讨论
3.1 疣吻沙蚕基因组基本特征
不同物种间基因组的大小和复杂程度有明显差异,会直接影响到测序策略的选择及基因组的组装效果。因此,进行全基因组测序前须先评估物种基因组的基本特征。疣吻沙蚕基因组survey 测序及k-mer 分析估计其基因组为759.53 Mb,远大于水蛭 (Helobdellarobusta)[17]和宽体金线蛭 (WhitmaniaPigra)[18]等所有已报道的蛭纲物种,也较多毛纲的海蠕虫 (Capitellateleta)[17]、欧文虫 (Owenia fusiformis)[19]、巨型管虫 (Riftiapachyptila)[20]、Lamellibrachialuymesi[21]的大;与寡毛纲通俗腔蚓(Metaphirevulgaris)[22]及多毛纲搓稚虫 (Streblospio benedicti)[23]的大小 (约0.7 Gb) 相当;但明显小于安德爱胜蚓 (Eiseniaandrei)[24]、赤子爱胜蚓 (E.fetida)[25],以及多毛纲的旋鳃虫(Spirobranchuslamarcki)[26]和深海管虫 (Paraescarpiaechinospica)[27](1.0~1.3 Gb)。物种间基因组大小的差异性在一定程度上与重复序列比例的高低有关。疣吻沙蚕基因组的重复序列比例 (45.92%) 低于旋鳃虫[26]和深海管虫[27],但显著高于海蠕虫[17]、巨型管虫[20],以及所有已公布的蛭纲动物 (重复比例均低于34%)[15-18];其基因组杂合率 (1.41%) 与安德爱胜蚓[24]和赤子爱胜蚓[25]相当,远远高于L.luymesi(0.60%)[21]、搓稚虫 (0.29%)[23]与深海管虫 (0.63%)[27],是目前已报道的杂合率最高的多毛类。此外,组装的contig和scalffold 总长分别为821 637 022 和840 375 821 bp,N50 分别为548 和662 bp,提示过高的杂合率会造成组装序列长度大于预估基因组大小,并导致显著偏低的N50 指标[28]。综上所述,疣吻沙蚕基因组属复杂基因组类型,绘制染色体级别高质量全基因组图谱应优先考虑“PacBio+Illumina+Hi-C”策略。该研究数据为后续全基因组测序与组装提供了基础资料。
3.2 疣吻沙蚕基因组微卫星分布特征
水生动物基因组中单碱基重复微卫星占优势的现象较少,已报道的有中华绒螯蟹 (Eriocheirsinensis)[29]、鲤 (Cyprinuscarpio)[30]、脊尾白虾 (Exopalaemoncarinicauda)[31]和胡鲶 (Clariasbatrachus)[32]。在疣吻沙蚕基因组中检测到 130 216 个微卫星位点,位点数量随重复基序碱基数的增加而迅速减少,单碱基重复微卫星最丰富 (35.00%),其次为二碱基重复 (32.48%)、三碱基重复 (14.42%),这与以二碱基或三碱基为优势类型的鱼[9,33-35]、虾[36-37]、贝类[38]明显不同,特别是与亲缘关系较近的蛭类、寡毛类相比也表现出较大差异。宽体金线蛭[39]、天锡杜拉蚓 (Drawidagisti)[40]基因组中三碱基重复占绝对优势,主要基序类型分别是AAT、ATA 和ATT、AAT。王斌等[41]对4 种蛭类的转录组数据进行分析,同样发现三碱基重复微卫星占优势,在宽体金线蛭中甚至高达68.00%。作为最特殊的微卫星重复类型,三碱基重复微卫星可形成复杂的环-折叠构型来稳定DNA 结构,从而更有利于转录过程中的解旋和蛋白质识别[33]。此外,疣吻沙蚕转录组中二碱基AT/TA 重复基序频率 (32.19%) 最高,单碱基A/T 重复类型(21.17%)其次,之后是三碱基AGC/GCT (29.06%) 基序 (未发表数据),表明微卫星类型特征在转录组和基因组水平上存在差异,类似的差异性在圆鳍鱼 (Cyclopteruslumpus)[42]、凡纳滨对虾[36,43]、宽体金线蛭[39,41]中也被证实。不同物种表现出不同的微卫星优势类型分布规律,可能与其进化水平有关。
疣吻沙蚕基因组微卫星核心区集中在4~18 拷贝,随着重复次数的增加,微卫星数量呈显著降低趋势,这可能与重复单元长度的不断增加使得微卫星稳定性降低或基序高频次重复导致更高的突变率有关[44]。进一步分析发现,二碱基至六碱基微卫星重复单元中A/T 含量显著高于G/C 含量,即微卫星序列表现出明显的A/T 碱基优势,这与许多水生动物如脊尾白虾[31]、凡纳滨对虾[36]、虾夷扇贝 (Mizuhopectenyessoensis)[38]、宽体金线蛭[39]及旧金山湾卤虫 (Artemiafranciscana)[45]中的研究结果一致。早期观点认为,微卫星富含A/T 的原因可能是由于CpG 甲基化后的C 易脱氨基转变为T,导致G/C 比例不断缩小,而突变的A/T 碱基类型相应增多[33];后来却发现这也可能与微卫星位点的产生方式,即与DNA 的复制滑动存在一定关系[29,46]。微卫星富含A/T 的原因可能是A/T 含量高则Tm值降低,其序列容易发生DNA 解链,并通过复制滑动机制和重组机制产生高A/T 含量的重复类型的概率更高[38]。
3.3 疣吻沙蚕微卫星标记开发及其遗传分析效果
微卫星是重要的分子遗传学研究工具,开发多态标记是其广泛应用的关键。与传统方法相比,基于高通量测序技术开发微卫星标记更具优势,产生的基因组或转录组数据是标记开发的重要资源。然而与转录组微卫星相比,基因组微卫星的多态性往往更高且分布更广泛,可获得更优的基因组覆盖率[42]。Temnykh 等[16]认为长度≥20 bp 的微卫星位点多态性较高,长度10~20 bp 的为中等多态性,小于10 bp 的多态性低。在疣吻沙蚕基因组中共筛选到43 240 条序列长度≥20 bp 的I 型微卫星位点,占比超过33%,远高于其转录组序列中20%的I 型位点比例 (未发表数据)。挑选50 个微卫星位点进行有效性及多态性验证,有41 个 (82%)位点的引物可扩增出特异性条带,其中15 个 (30%)表现出稳定且可重复的多态性。疣吻沙蚕基因组多态微卫星标记的筛选成功率与卵形鲳鲹[9]、凡纳滨对虾[36]、宽体金线蛭[39]中的结果相当。由此可见,疣吻沙蚕微卫星标记筛选成功率较高,获得的微卫星位点具有良好的开发潜力,是丰富且可靠的标记资源。
用微卫星标记开展群体遗传分析时,一般认为有效等位基因数越接近观测等位基因数,群体等位基因分布就越均匀;然而在实际分析中,通常会将全部条带视为有效等位基因,无效等位基因过剩便会造成等位基因分布不均[47]。从疣吻沙蚕基因组筛选的15 个多态微卫星位点中,仅3 个位点(ThGM021、ThGM035、ThGM041) 的等位基因数接近有效等位基因数,提示大部分位的等位基因分布不均,这可能是因为验证群体的样本容量偏小引起了主效等位基因的缺失[9]。有研究表明,群体的最小样本容量一般与遗传分析选用的参数有关。利用He、PIC 以及香农指数等评估遗传多样性时,群体样本容量达到27 便能使分析结果接近总体水平的95%;但选择Na时,样本容量则需达到52 以上[48]。疣吻沙蚕的群体样本量为30,故而选择He、PIC 可更好地评价微卫星位点的多态性。15个位点的平均Ho(0.487)、平均He(0.561)和平均PIC (0.511)共同表明,疣吻沙蚕验证群体具有较丰富的遗传多样性,这与Chen 等[5]利用COI 标记的分析结果并不完全一致,可能是因为微卫星属于核标记,多态性更高且受选择性作用更小,其揭示遗传变异的灵敏度高于线粒体标记。通常认为,PIC 越接近1,则群体杂合个体的比例越大、多态性越高:PIC<0.25 为低度多态性,0.25≤PIC<0.5 为中度多态性,PIC≥0.5 为高度多态性[47]。在15 个多态微卫星位点中,仅2 个为低度多态性,其余13 个 (86.7%) 均为高度或中度多态性。因此,基于疣吻沙蚕基因组序列筛选的微卫星标记多态性高且遗传信息丰富,可为群体遗传分析、种质资源评价、分子育种研究提供优质工具。
4 结论
疣吻沙蚕基因组属于高杂合、高重复的复杂基因组,其微卫星位点的类型丰富且具备较好的多态性潜能,可作为有效资源用于微卫星标记的大规模开发,对种质资源评价与保护利用、种群遗传学以及分子育种研究具有实际价值。

