后基因组时代发展抗除草剂作物的机遇及挑战
吴元龙,惠凤娇,潘振远,尤春源,林海荣,李志博,金双侠,聂新辉
后基因组时代发展抗除草剂作物的机遇及挑战
吴元龙1,惠凤娇2,潘振远1,尤春源3,林海荣1,李志博1,金双侠2,聂新辉1
1石河子大学农学院/新疆生产建设兵团绿洲生态农业重点实验室,新疆石河子 832003;2华中农业大学植物科学技术学院/作物遗传改良全国重点实验室,武汉 430070;3石河子农业科学院棉花研究所,新疆石河子 832011
全球农业正在面临着严峻的挑战,育种技术是种业发展的基础和关键。基因编辑技术是指对目标基因进行定点修饰,实现对特定靶标基片段的删除、插入和替换。利用基因编辑技术可以精准修改目标基因或将某种优良基因引入到作物中产生优良农艺性状,在分子设计育种中具有巨大潜力,对保障粮食安全具有重大意义。杂草危害对作物的产量和品质影响巨大,如何高效、安全、可持续地防治草害一直是研究的热点。目前,全球市场已经出现超过200种的化学除草剂,利用化学方法来防治草害已成为现代化农业的重要组成部分,抗除草剂作物的推广也显著降低了杂草防治成本,但随着抗除草剂作物的大面积推广和长期使用单一除草剂,杂草抗/耐药性和抗性基因逃逸等环境安全问题逐渐被发现。目前,功能基因组学、生物信息学、基因工程技术的发展(特别是基因编辑技术在植物中的广泛应用),为创制抗除草剂作物和新型高效的除草剂系统创造了条件。本文首先介绍抑制植物氨基酸生物合成、植物脂类代谢、植物类胡萝卜素、质体醌和生育酚生物合成途径除草剂的主要作用靶标基因及其作用机制。其次,介绍2种挖掘新型抗除草剂基因与除草剂系统的方法,包括基于CRISPR/Cas系统对作物内源的除草剂抗性基因进行定向突变方法和基于天然产物与生物体在自然界中存在共同进化理论的抗性基因导向方法。同时,介绍3种培育抗除草剂作物方法的研究进展,包括常规育种培育法、转基因育种培育法和基于CRISPR/Cas基因组编辑技术培育法。其中,重点介绍CIRSPR/Cas系统、碱基编辑技术和Prime-editing系统在培育抗除草剂作物中的研究进展。针对抗性杂草的产生及环境安全问题是当前化学防治杂草面临的主要挑战,以及抗除草剂作物所面临的主要挑战是基因逃逸问题。目前,快速发展的基因组编辑技术为后基因组时代发展抗除草剂作物提供了新的解决策略和新的机遇。最后,对除草剂作物的未来进行了展望。
基因编辑技术;除草剂;抗除草剂基因;抗除草剂作物育种
0 引言
人类为了获取食物进行农业生产,随着世界人口的不断增长和环境问题的日趋严峻,农作物产量和品质的提高成为亟须解决的重要科学问题,任何影响作物生产力的因素都应该被高度重视[1]。其中,农田杂草是影响作物产量和品质的重要因素,杂草防治是作物生产中的关键环节。杂草具有较强的环境适应性和抗逆性[2],与农作物竞争生长资源(包括光照、水分和养分等)和空间条件[3],增加田间病害虫的发生,释放有毒物质造成粮食减产[4]。因此,杂草会直接影响作物产量,阻碍农业的快速、健康、可持续性发展。世界农田杂草种类繁多,超过上百种杂草具有较大危害性,造成作物平均减产可达15%以上,造成的直接经济损失近千亿元[5-7]。有报道显示,杂草危害水稻产量可达40%以上[8]。开发高效,可持续的草害防治措施成为现代农业发展的重要战略目标之一,也一直是研究者研究的热点问题。
杂草防治有多种方法,人工除草可以有效减少杂草危害,但是这种方法费时费力,效率低下,不符合现代农业的发展趋势。化学除草由于操作简单,经济有效而被广泛应用。除草剂不仅具有除草速度快和效率高的特点,而且适应于机械化种植、病虫害综合防治和耕作制度的改革。因此,喷施除草剂是防治杂草的主要手段。目前,全球市场已经出现超过200种的化学除草剂,除草剂的年产量已居农药之首,利用化学方法来防除草害已成为现代化农业的重要组成部分,对全球粮食生产作出了重大贡献[1, 9]。除草剂的分类方式多样,如根据作用方式可以将除草剂分为选择性除草剂(如苯磺隆、敌稗和苯达松等)和灭生性除草剂(如草甘膦和草铵膦等);根据有效成分可以将除草剂分为以无机物或有机物为原料,经过化学反应或机械加工而制成的化学除草剂(具有不同化学结构的除草剂类型已超过20余类)和利用生物活体或生物代谢产物制成的生物除草剂;根据在作物体内的移动情况可以将除草剂分为触杀型除草剂(百草枯、乙氧氟草醚等)和内吸型除草剂(2,4-D丁酯、双草醚、草甘膦)等等。
世界范围内长期、广泛使用某一类型的除草剂,造成的杂草抗药性和基因逃逸、药害残留等环境安全问题日益显现,备受全球关注,因此,有必要研发出更高效的除草剂,以及培育出抗除草剂的作物新种质[1]。基于环境友好与可持续性考虑,对于新型除草剂的要求也越来越严苛,它不仅要高效杀灭杂草,还需要考虑安全无公害,适应环境和不影响作物本身的生长发育等因素。因此,如何获得选择性强,作用机理独特,安全高效,对环境和人类的影响极小的除草剂就显得尤为重要。另一方面,这也对抗除草剂作物发展提出了新的要求。针对目前杂草防治的瓶颈问题,一方面已经从一些微生物、动物、植物中挖掘到一些抗除草剂基因。同时,基于杂交育种以及植物基因工程的发展,成功获得了一些可以耐受特定除草剂的作物,为一些灭生性除草剂的大面积推广提供了条件,极大地降低了杂草防治的成本[10]。随着功能基因组学,代谢组学和蛋白质组学以及基因工程技术(特别是基因编辑技术)的快速发展,为解决杂草危害提供了新的切入点和发展方向,为解决除草剂面临的挑战创造了条件。为了阐明抗除草剂作物的发展,本文将对除草剂的主要类型及其作用机制进行简介;进一步介绍新型抗除草剂及其基因系统的挖掘;最后介绍抗除草剂作物的培育及基于后基因组时代发展抗除草剂作物的机遇与挑战,以期为环境友好型除草剂的开发和抗除草剂作物的培育提供理论参考。
1 除草剂作用主要靶标基因及其作用机制
研究除草剂的作用靶标基因,可以为研制新型除草剂与抗除草剂作物的培育提供重要的参考价值。植物为了维持其正常的生长发育,需要不断地进行呼吸作用、光合作用、蛋白质和脂类等物质的代谢,这些代谢通路的维持对于植物生长发育具有重要作用[11-12]。通过阻碍这些代谢通路来达到杀死杂草的目的是许多除草剂的主要作用机制。目前,主要有以下几种不同作用机制的除草剂,抑制植物氨基酸生物合成途径、抑制植物光合作用、抑制植物呼吸作用、抑制植物微管与组织发育的化学除草剂和干扰植物激素平衡、利用生物活体或生物代谢产物制成的生物除草剂。
1.1 抑制植物氨基酸生物合成
氨基酸是蛋白质的基本组成成分,而蛋白质是生命的执行者,目前,抑制氨基酸和蛋白质代谢的除草剂主要有3种类型,分别是抑制支链氨基酸(branched chain amino acid,BCAA)生物合成类、抑制芳香族氨基酸合成类和抑制植物氮代谢类。
BCAA是指分子结构中的R基团具有支链碳链,包括亮氨酸、缬氨酸和异亮氨酸,均属于人体必需氨基酸[13-14]。由于植物无法像动物一样从食物中吸收支链氨基酸,必须自身合成,因此,支链氨基酸生物合成途径对于植物生长至关重要。同时,动物体内不存在该代谢途径,因此,靶向该途径的除草剂具有高度特异性与安全性是目前仅次于草甘膦的重要化学除草剂之一。支链氨基酸生物合成途径是由丙酮酸经乙酰乳酸合酶(acetolactate synthase,ALS)、乙酰羟酸异构还原酶(aceto hydroxyacid isomeroreductase,AHIR)和二羟酸脱水酶(dihydroxyacid dehydratase,DHAD)催化形成缬氨酸和亮氨酸,以及由α-酮丁酸经上述3种酶催化形成异亮氨酸2条代谢通路构成(图1-A),其中,ALS、AHIR和DHAD酶即为支链氨基酸生物合成途径的关键酶[14]。ALS也称为乙酰羟基酸合成酶(acetohydroxy acid synthase,AHAS,EC2.2.1.6)具有催化形成乙酰羟基丁酸酯和乙酰乳酸酯的功能。目前,已经开发出许多靶向ALS的除草剂,这类除草剂具有广谱性、活性高、选择性强、对动物低毒性的特点,使其在全球范围内得到了广泛应用。其作用机制均是抑制ALS活性,阻碍植物支链氨基酸的合成,进而影响蛋白质合成、细胞分裂和植物生长,最终导致植物枯萎死亡[15]。而靶向AHIR和DHAD的除草剂还处于研究阶段,其中,已有研究表明,aspterric acid(AA)可以靶向DHAD,阻碍支链氨基酸生物合成途径,是具有广谱、高效、低毒性的新型绿色除草剂。相信在不久的将来靶向DHAD的除草剂将会被推广应用[16]。
芳香族氨基酸(aromatic amino acids)是指分子结构中带有苯环结构的氨基酸,如酪氨酸(tyrosine)、苯丙氨酸(phenylalanine)和色氨酸(tryptophan)。其生物合成是通过莽草酸途径(shikimate pathway)形成[17]。其中,叶绿体酶5-烯醇式丙酮酰莽草酸-3-磷酸合成酶(5-enolpyruvylshikimate-3-phosphatesynthase,EPSPS,EC2.5.1.19),催化莽草酸-3-磷酸(shikimate- 3-phosphate,S3P)和磷酸烯醇式丙酮酸(phosphoenolpyruvate,PEP)形成5-烯醇式丙酮酰莽草酸-3-磷酸(5-enolpyruvylshikimate 3-phosphate,EPSP)(图1-B)。目前,世界上使用最广泛和最重要的除草剂草甘膦(glyphosate)的靶向位点就是上述的叶绿体酶EPSPS[18]。草甘膦自1974年问世以来,已在全球广泛使用,是一种具有稳定C-P键的磷酸盐化合物,是具有环境友好性的非选择性除草剂,可以广泛控制一年生和多年生杂草。其作用机制是当草甘膦经植物茎叶吸入体内后,由于草甘膦的化学结构与PEP相似,二者竞争结合EPSPS,但不会影响S3P和EPSPS的结合,最终会形成草甘膦:EPSPS:S3P三元复合物,阻断EPSPS与PEP的结合从而抑制EPSPS的活性,导致前体物质莽草酸大量积累,植物所需的芳香族氨基酸无法正常合成,影响植株正常的氮代谢活动并最终导致死亡[18-19]。
谷氨酰胺合成酶(glutamine synthetase,GS,EC6.3.1.2)具有将谷氨酸和氨转化为谷氨酰胺的酶活,是谷氨酰胺合成关键酶,也是植物氮代谢的关键酶(图1-C)。靶向GS的化合物有L- phosphinothricin、phosalacine、tabtoxinine-β-lactam和oxetin[20]。其中,作为除草剂应用比较广泛的就是草铵膦(phosphinothricin,PPT),其作用机制是草铵膦为谷氨酸的类似物,会与谷氨酸竞争GS结合位点,抑制氮代谢,导致细胞内氨积累,最终导致植物枯萎死亡[21]。
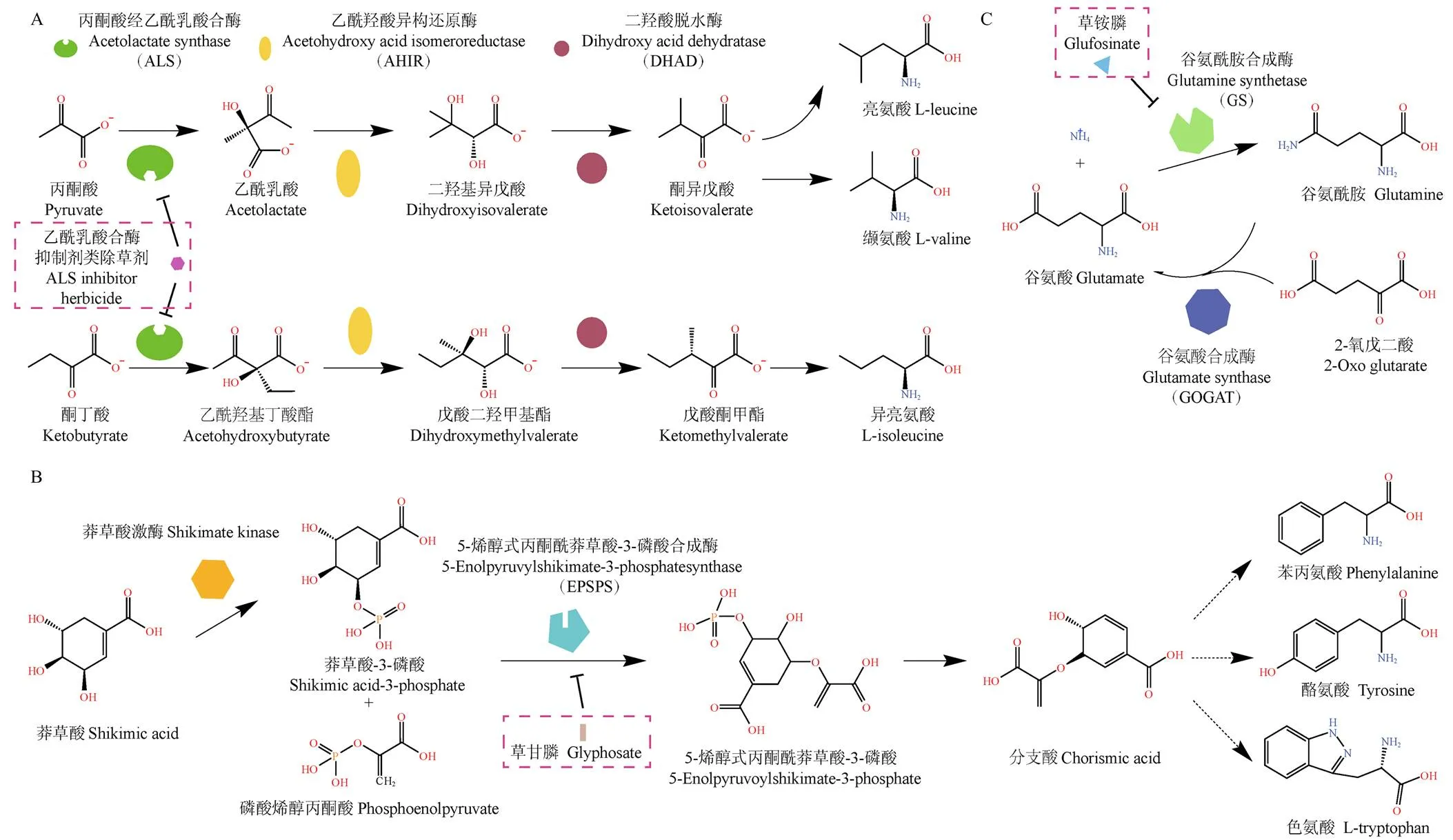
A:支链氨基酸生物合成途径及抑制乙酰羟基酸合成酶除草剂的作用位置模式图;B:抑制5-烯醇式丙酮酰莽草酸-3-磷酸合成酶除草剂的作用位置模式图;C:抑制谷氨酰胺合成酶除草剂的作用位置模式图。GS:谷氨酰胺合成酶;GOGAT:谷氨酸合成酶
1.2 抑制植物脂类代谢
脂类对于植物细胞维持正常功能具有重要作用,乙酰辅酶A羧化酶(acetyl CoA carboxylase,ACCase,EC.6.4.1.2)将乙酰CoA羧化生成丙二酰CoA(图2),该步骤是脂类和脂肪酸合成的关键限速步骤[22]。在植物中,ACCase有2种同功异构酶,其中,对除草剂敏感的异构酶是一个分子量约为220 kDa多官能团蛋白质,包含4种截然不同的蛋白质的多酶复杂结构。目前,已知有许多靶向ACCase的除草剂,包括芳氧苯氧丙酸(aryloxy phenoxy propionate,APP)、环己二酮(cyclohexanedione,CHD)和苯吡唑啉(phenyl pyrazoline,PPZ)的化学基团等[1]。这些除草剂的作用机制是通过抑制植物脂类合成途径中的关键酶ACCase酶活性,影响脂类合成而导致植物枯萎死亡。

图2 抑制乙酰辅酶A羧化酶除草剂的作用位置模式图[4]
1.3 抑制植物类胡萝卜素、质体醌和生育酚生物合成途径
类胡萝卜素、质体醌和生育酚生物合成途径之间具有相关性(图3),其中,类胡萝卜素在光合作用、光保护和抗氧化作用中的辅助采光作用中发挥着重要作用;质体醌在光合磷酸化中起重要作用;生育酚是重要的抗氧化剂。在该生物合成途径中具有一些重要的关键基因,包括对羟苯基丙酮酸双氧化酶(4-hydroxyphenylpyruvate dioxygenase,HPPD)、1-脱氧-D-木酮糖5-磷酸合成酶(1-deoxy-D-xylulose 5-phosphate synthase,DXS)、茄尼基焦磷酸合酶(solanyl diphosphate synthase,SPS)、尿黑酸茄尼酯转移酶(homogentisate solanesyl transferase,HST)和八氢番茄红素脱氢酶(phytoene desaturase,PDS),均是重要的除草剂靶标基因(图3)。其中,研究比较多的靶向基因为。HPPD是生物体内参与酪氨酸代谢的关键酶,催化羟基苯丙酮酸(4-hydroxyphenylpyruvate,HPPA)转化为尿黑酸(homogentisic acid,HGA),HGA是植物生物合成质体醌和生育酚的重要前体,质体醌是类胡萝卜素的生物合成前体,因此,在光合作用中发挥着重要作用[23]。生育酚是植物体内重要的抗氧化物质。HPPD抑制剂类除草剂的作用机制是可以与HPPD酶活性位点的Fe2+螯合,阻断HPPD与底物HPPA的结合,形成对HPPD的竞争性抑制,阻断植物中质体醌和生育酚的形成[4]。
2 挖掘新型抗除草剂基因与除草剂系统
抗除草剂作物(指在通常致死剂量的除草剂作用后仍然能够存活并再生的作物)的推广会导致杂草抗药性产生和基因逃逸等环境问题,单一种类除草剂的使用也会加速杂草抗药性的进化[3]。因此,挖掘新型作用机制的抗除草剂基因(型)和新型高效的除草剂系统就显得尤为重要。
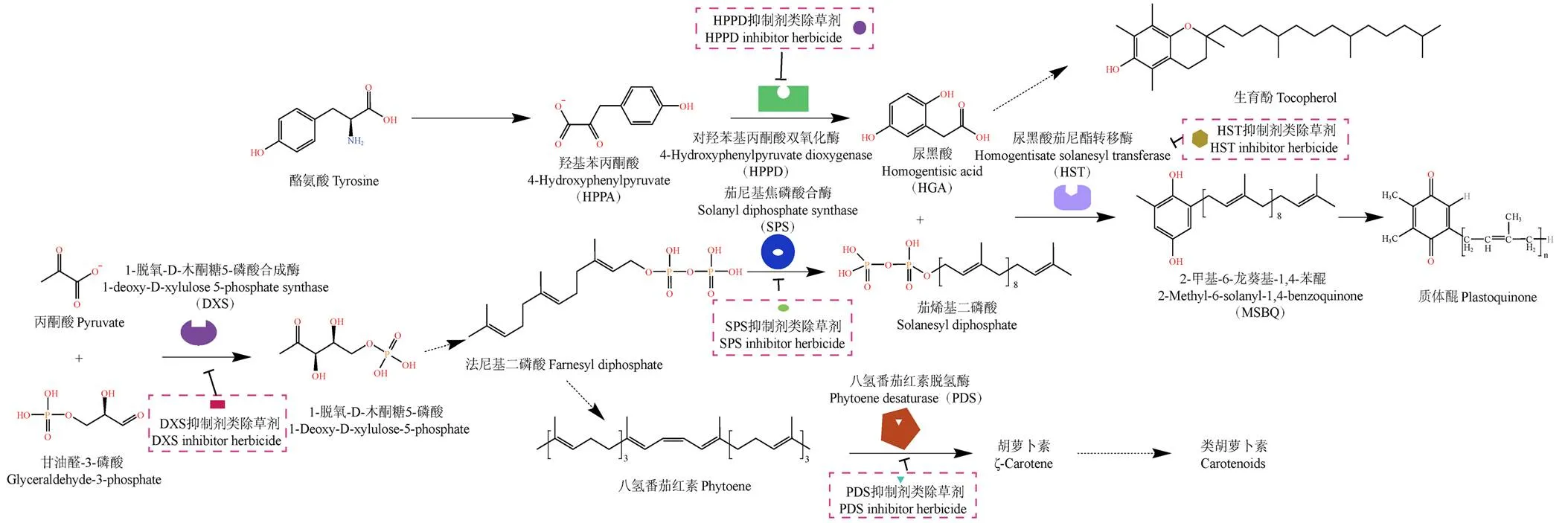
HPPD:4-羟基苯基丙酮酸双氧化酶;DXS:1-脱氧-D-木酮糖5-磷酸合成酶;SPS:茄尼基焦磷酸合酶;HST:尿黑酸茄尼酯转移酶;PDS:八氢番茄红素脱氢酶
传统抗除草剂基因的挖掘是在除草剂环境条件下进行物理或化学诱变,获得目标抗性材料后利用分子育种手段定位获得目的基因,或者是从天然微生物中挖掘抗性基因如鼠伤寒沙门氏菌中克隆到的抗草甘膦的aroA基因[24],来自吸水链霉菌的抗草铵膦的BAR(bialaphos resistance)和PAT(phosphinthricin acetyltransferase)基因[25]。但这些传统的育种方法存在效率较低,育种周期长,工作量大的弊端。目前,随着功能基因组学和生物技术的快速发展,育种技术也由传统的分子育种迅速地转型进入到后基因组学时代,为综合利用基因组数据和生物信息学数据鉴定新型抗除草剂基因和开发新作用机制的除草剂提供了新的方法和思路。
针对已知的除草剂抗性基因,一方面研究者利用clustered regularly interspaced palindromic repeats/ CRISPR-associated proteins system(CRISPR/Cas)系统对作物内源的除草剂抗性基因进行定向的饱和突变,发现了大量新的抗除草剂基因型[26-27]。如:有研究者利用基于prime editors系统的prime editing library-mediated saturation mutagenesis(PLSM)方法,对水稻ACCase基因CT结构域进行了饱和突变,筛选出16种具有不同氨基酸替换类型的新型抗除草剂基因[28]。另一方面,针对单一除草剂抗药性的问题,可以利用基因编辑技术在作物中同时聚合多个除草剂抗性,创制多抗除草剂作物,通过多种除草剂的复合交替使用,可以有效避免单一抗性的弊端。如,基于单碱基编辑技术的发展,周焕斌团队利用开发出的TadA9腺嘌呤单碱基编辑器,成功对水稻主栽品种南粳46中的4个除草剂靶标基因实现了一次性的同时改造,4个靶点共编辑效率高达56.25%[29]。除此之外,日本埼玉大学首次鉴定出的水稻广谱抗除草剂基因HIS1(HPPD inhibitor sensitive 1,HPPD抑制剂敏感1),赋予了水稻对双环磺草酮(benzobicyclon,BBC)和其他β-三酮类除草剂的抗性,为抗除草剂基因的挖掘也带来了希望与启发[30]。
据最新统计,全球已发现268种(154种双子叶,114种单子叶)杂草的521个生物型,杂草已经进化出对31种已知除草剂作用位点中的21种和165种不同除草剂的抗性(国际抗除草剂杂草数据库,https://weedscience.org/Home.aspx)。因此,研制新型抗除草剂基因与除草剂系统,就显得十分迫切。研究表明,天然产物与生物体在自然界中存在共同进化理论,该理论认为天然产物和生物之间存在共进化关系,该理论已经在医药健康及农业生产中得到了广泛的应用[17]。据此,研究者提出了基于抗性基因导向方法挖掘新型作用机制的除草剂系统的方法。如,研究者以支链氨基酸通路中的关键酶DHAD为研究对象,利用已公布的真菌基因组数据,筛选获得含有天然产物(可转化成除草剂)合成基因与DHAD同源基因(astD)的保守基因簇[17]。在保守基因簇中的DHAD基因型和产生的天然产物构成了潜在的新型作用机制除草剂系统。接着研究者试验证明天然产物aspterric acid可以作用于DHAD抑制植物生长,利用真菌自身的抗性基因可培育具有抗aspterric acid的抗性植物。最终证明天然产物aspterric acid可以作为除草剂且鉴定到其抗性基因(astD)[16]。中国农业科学院烟草研究所从海洋真菌中筛选到除草活性显著的Xanthone类化合物,对以反枝苋、绿苋等为代表的苋属杂草的最小抑草浓度达到8 μg·ml-1,活性达到传统草甘膦的4倍,是一种新型的天然产物除草剂[31]。华中农业大学从海洋细菌sp. YM12中克隆到抗草铵膦基因,编码的蛋白在体外酶活试验中发现与bar基因或pat基因编码的蛋白具有不同的酶学特征,氨基酸序列同源性仅为37%,属于一种全新的抗草铵膦基因[32],该团队同时从中克隆得到新型抗性基因Ⅰvariabilis- EPSPS,其编码的蛋白对草甘膦具有一定的抗性,且与Ⅱ类EPSPS基因同源性较低,是一种新型的草甘膦抗性基因[33]。这种基于基因组学与生物信息学的挖掘方法大大提高了发现新型除草剂系统的有效性与高效性。基于化学工程技术,利用氮化碳纳米材料可实现多种磺酰脲类除草剂的可见光催化降解,通过单原子修饰可显著提高纳米材料的性能,使目标除草剂降解速率提高4倍,有效地减轻磺酰脲类除草剂对后茬敏感作物的药害,为环境友好型除草剂的开发提供了新的思路[34]。基于结构生物学,已有研究者对乙酰羟酸合成酶AHAS突变体(W574L和P197T)与除草剂CE或BS结合的晶体结构进行解析,发现这些突变可降低除草剂与AHAS的结合亲和力,并阻碍除草剂对乙酰羟酸合成酶的时间性氧化失活,进一步揭示了杂草通过AHAS突变获得除草剂抗药性的结构机制,为新型除草剂的设计与开发奠定理论基础[35]。
3 抗除草剂作物的培育
目前,创制抗除草剂作物的主要途径包含常规育种、转基因育种和基于CRISPR/Cas基因组编辑技术育种。
3.1 常规育种培育抗除草剂作物
常规培育抗除草剂作物新品种的方法主要有杂交选育法、自然选择培育法、物理或者化学手段(辐射、花粉诱变、种子诱变、体细胞诱变和化学诱变剂等)组织培养法等。杂交选育主要是育种家在获得抗性作物材料后,通过将其与优良的栽培品系进行杂交,从中筛选聚集双亲优良性状的抗除草剂品种。诱变育种是指通过化学、物理(紫外线和X射线等)或可移动的遗传因子等因素诱发生物体的遗传物质产生遗传变异,并筛选出具有目标性状突变个体进行新品种培育的过程。利用突变育种技术已经在玉米、小麦、水稻、油菜、向日葵等作物中培育了许多抗除草剂作物,如:抗磺酰脲的小麦[36],抗咪唑啉类和磺酰脲类的向日葵[37]和抗磺酰脲类除草剂大豆[38]等。诱变育种培育的抗除草剂作物为非转基因作物,推广应用阻力较小[26]。综上,这些方法操作简单,可获得的突变范围相对较大,但获得抗性作物的周期较长,工作量较大,突变的低概率和随机性使得培育过程变得低效,且几乎不可能同时诱导产生多个特定突变体材料[39]。
3.2 转基因育种培育抗除草剂作物
转基因育种是指通过引入一个或多个优良基因,来改良目标农艺性状的育种方法。目前,大部分商业化的抗除草剂作物均是通过这种技术获得,约占全部转基因作物的25%[39]。针对靶标抗性主要是通过超量表达外源抗性靶标基因或修饰除草剂靶标蛋白(降低酶与除草剂的结合亲和力)的方式来创制抗除草剂作物。目前,超量表达外源抗性靶标基因主要有CP4菌株的、(,叶绿体转运肽)等变体、草甘膦-N-乙酰转移酶(glyphosate N-acetyltransferase,GAT)基因和草甘膦氧化还原酶(glyphosate oxidoreductase,GOX)基因等[34, 40-42],并成功培育了抗溴苯腈棉花、抗草胺膦的油菜和抗草甘膦大豆等[43-44],在大豆、油菜、玉米、苜蓿、水稻和棉花等农作物中得到了广泛应用[45-46]。如:在大豆中超表达G2-EPSPS和GAT基因,创制了既能抗草甘膦又能够以排毒机制降解体内残留草甘膦的大豆材料[47]。针对非靶标抗性的引入降解/转移除草剂的酶或酶系统方式,包括膦丝菌素乙酰转移酶(PAT)基因、双丙氨磷抗性(Bar)基因和细胞色素P450等[48]。此外,也有过表达外源抗性靶标基因和非靶标抗性基因相结合的应用[49]。然而,转基因育种培育抗除草剂作物受限于操作繁琐、劳动密集型、成本高和食品、环境与生物安全监管程序繁琐等因素。据农业农村部的数据(http://www.moa.gov.cn/ztzl/zjyqwgz/ spxx/index.htm),目前,我国获得转基因生物安全证书的抗除草剂作物只有玉米(、、和、和、和、、、maroACC基因)和大豆(、和)。
3.3 基于CRISPR/Cas基因组编辑技术培育抗除草剂作物
基因组编辑技术是一种以插入、缺失或碱基替代的方式在目标序列中产生突变以实现DNA修饰的技术。基因组编辑由多种基本技术组成,如ZFNs(锌指核酸酶)、TALENs(转录激活因子效应物核酸酶)和CRISPR/Cas9(聚类规则间隔短回文重复序列及相关蛋白9)系统。目前,基于CRISPR-Cas系统的基因组编辑技术正以前所未有的速度、深度和广度影响着生命科学基础研究、应用研究及人类健康等领域,使精确和高效地编辑目标基因序列由可能变成了现实。CRISPR-Cas系统是原核生物的一种获得性免疫系统,由单链RNA引导的内切酶可以在基因组的特定靶标位点引起变异,经过人工改造后主要包含向导RNA(gRNA)和Cas蛋白两部分[50-51]。
3.3.1 基于CIRSPR/Cas系统培育抗除草剂作物 Cas蛋白在切割DNA双链后会造成双链断裂(double- strand break,DSB),紧接着引发生物体内存在的修复机制来修复损伤DNA:一种是非同源末端连接(non-homologous end joining,NHEJ),这种修复机制不精确,在修复过程不可避免地发生碱基插入或缺失,引起基因的移码突变;另一种是同源重组修复(homology-directed repair,HDR),以供体DNA作为模板进行修复,借助这种修复机制可以根据所提供DNA模板的不同实现精准基因插入或碱基修饰等[52]。在高等动植物中,以NHEJ机制进行修复的频率显著高于以HDR的频率,因为NHEJ机制可在细胞处于任何时期时发生,而HDR机制则仅在特定的细胞周期(S及G2期)发生[53]。
目前,应用范围广泛、高效的基因编辑工具主要包括CRISPR-Cas9和CRISPR-Cas12a[54]2种,这两种系统目前已经在水稻、小麦、烟草、玉米、马铃薯、番茄、向日葵、大豆、苜蓿等多种作物中得到了广泛的应用。利用CRISPR/Cas9介导的基因编辑技术,基于NHEJ修复方式,以为靶点进行精准突变,成功在水稻中筛选获得一个新的除草剂(咪唑乙烟酸)耐受性等位基因[55]。但另一方面,基于HDR的技术挑战性更大,相对于动物细胞和人类细胞,植物中存在细胞壁的阻碍,在进行HDR过程中面临的巨大挑战就是如何把足够的供体分子输送到细胞中去,且保证供体分子在植物细胞中不被降解或被转移到胞质中[56]。但也有一些成功的案例值得借鉴。目前,已经开发了不同的方法提高HDR的效率,包括细胞周期的调控[57]、修复途径关键蛋白表达的调节、提高修复模板的供应量[58]、优化供体修复模板(donor repair template,DRT)的设计[59]。例如,在玉米中,利用单链寡核苷酸(single stranded oligonucleotide,SSON)或双链DNA载体作为DRT,实现了ALS2中P165S的氨基酸精准替换,获得了对氯磺隆抗性的玉米编辑植物。Sun等[60]利用CRISPR/Cas9系统,设计了2个sgRNA(small guide RNA)和一个供体修复模板,通过在供体两端添加靶标序列来增加游离的供体数量,以基因枪轰击的方式在水稻中实现了ALS基因中W548L和S627I 2个氨基酸残基的同时精确替换;2018年,该团队又利用CRISPR/Cas12a基因编辑系统,以DNA作为HDR的模板,实现了ALS中相同氨基酸位点的精确替换[61];2019年,夏兰琴团队利用CRISPR/Cpf1基因编辑系统,以RNA作为HDR的模板,对ALS的2个氨基酸位点(W548L和S627I)进行定点替换,成功获得无转基因成分的抗ALS抑制剂类除草剂水稻植株[56]。该研究是在植物中首次成功利用RNA作为同源重组修复模板,开辟了利用植物RNA作为同源供体模板进行同源修复的新思路。以上都是基于基因组编辑技术创制抗除草剂作物的成功案例,除此之外,在方法创新上,通过改造小麦矮缩病毒(wheat dwarf virus,WDV),利用其能侵染大多数单子叶和双子叶植物,以及病毒能够大量复制的特性,转化水稻愈伤组织发现其提供供体分子的数量是传统T-DNA的数百倍。该研究进一步把CRISPR/Cas9系统和WDV系统整合,辅以定向筛选的方式,对水稻内源的ACT1和GST位点定点插入GFP标记,对转化植株的鉴定表明外源基因定向敲入的效率最高可达到19%[58];2020年,朱健康院士团队又在基因打靶方面取得新的进展,使用化学修饰的DNA为供体模板,通过粒子轰击法将多达2 049个碱基对的序列以25%的效率插入水稻基因组。同时,还报道了一种巧妙的基因替换方法,该方法依赖于同源导向修复、化学修饰的供体DNA和靶位点串联重复序列的存在,以6.1%的效率实现了高达130 bp序列的替换[62]。
3.3.2 基于碱基编辑技术培育抗除草剂作物 基于CRISPR-Cas9发展而来的碱基编辑系统在创制抗除草剂材料方面发挥了重要作用。单碱基编辑系统元件主要包含nCas9或dCas9与相应的脱氨酶,能够在不引入DNA双链断裂也不需要重组修复模板的情况下实现高效、精准的单碱基水平修饰,主要有3种类型:胞嘧啶碱基编辑器(cytosine base editor,CBE,C-to-T)[63]、腺嘌呤碱基编辑器(adenine base editor,ABE,A-to-G)[64]和糖基化酶碱基编辑器(glycosylase base editor,GBE,C-to-G)[65]。LI等[66]利用碱基编辑技术对玉米乙酰乳酸合成酶ZmALS1和ZmALS2基因进行靶向突变,发现目标单碱基双突变体可以达到并超过转基因同类产品的抗性水平,除草剂抗性水平达到大田生产推荐上限15倍剂量或一般推荐35倍剂量水平;扬州大学王幼平团队利用适用于油菜的碱基编辑系统成功对BnALS1基因Pro197进行靶向突变,共获得BnALS1基因的2种类型(P197F和P197S)突变体,喷施苯磺隆在3倍田间推荐喷施剂量的浓度下均未表现任何药害症状[67]。李家洋团队通过碱基编辑小麦的乙酰乳酸合成酶和乙酰辅酶A羧化酶基因,生成了具有除草剂耐受性突变的无转基因小麦种质,赋予对磺酰脲类,咪唑啉酮类和芳氧基苯氧基丙酸酯类除草剂的耐受性[68]。利用腺嘌呤碱基编辑器对小麦的α类tubulin基因Met-268位点进行编辑,成功地获得了有效耐受二硝基苯胺类除草剂的小麦种质材料[69]。
现有的单碱基编辑器CBE和ABE只能催化单一类型碱基的转换,不能实现2种类型的同时转换。2020年,中国科学院遗传与发育生物学研究所高彩霞、李家洋课题组将胞嘧啶脱氨酶APOBEC3A和腺嘌呤脱氨酶ABE7.10同时融合在nCas9的N端,构建了4种形式的新型饱和靶向内源基因突变的双碱基编辑器STEME-1—STEME-4(saturated targeted endogenous mutagenesis editors),新型双碱基编辑器可以只在一个sgRNA引导下诱导靶位点C-T和A-G的同时突变,显著增加了靶基因碱基突变的饱和度及突变类型的多样性。利用STEME-1系统成功对水稻进行饱和突变,扩大了基因编辑的范围,成功地创制了抗除草剂材料,同时,也加速了抗除草剂基因的定向进化[70]。
3.3.3 基于Prime-editing系统培育抗除草剂作物 Prime-editing系统是一种基于CRISPR的新型编辑器,在不产生DNA双链断裂的情况下,无须添加外的供体模板,在活细胞DNA中就可实现几乎任何替换(substitution)、微小插入(micro insertion)和微小缺失(microdeletion)。该系统包含Cas9-逆转录酶(nCas9与逆转录酶的融合蛋白)和pegRNA(包含目标序列逆转录模板),具有编辑精准、位置灵活、类型多样等特点,有极为重要的应用潜力[71]。Prime editing系统自被开发利用以来,经历了多代的优化升级,主要包括使用增强的prime editors效应蛋白[72]、优化改进pegRNAs的设计[73-74]、减少prime editing的副产物,改进DNA修复[75]。其中,pegRNA的设计合理与否对于精准编辑的影响似乎更大。pegRNA的切点选择、元件长度乃至序列碱基构成都会对prime editing效率造成重大影响。这也暗示在没有有效指导方法的情况下,植物基因组prime editing系统可能需要构建由不同参数元件组成的pegRNA阵列并加以试验筛选才可能实现有效编辑(development of a plant prime editing system for precise editing in the rice genome)。对于pegRNAs的设计,高彩霞老师课题组已经开发了高效的pegRNA设计网站PlantPegDesigner(http://www.plantgenomeediting.net/)及脱靶评估系统,为高效PE系统的应用奠定基础[76]。另一方面,在pegRNA设计方面效果比较好的是通过在pegRNA的3′末端添加RNA motifs,一方面可以对3′进行保护,另一方面可以阻碍pegRNA的降解,这种策略将PE的编辑效率提升到原来的3—4倍[74]。目前,已有研究利用prime-editing系统对进行定向氨基酸替换,创制了具有咪唑啉酮和双草醚抗性的水稻材料[43, 45]。通过删除M-MLV-RT的RNase H结构域或在M-MLV-RT的N端融合病毒核衣壳蛋白(nucleocapsid,NC),开发出了升级版新型引导编辑器ePPE,成功创制了可抗甲咪唑烟酸和烟嘧磺隆2种除草剂的水稻新材料[77]。中国农业大学陈其军课题组在玉米中测试了3种优化策略的PE系统,结果表明,优化的PE能够高效诱导产生可遗传的纯合和杂合玉米突变株系,为培育抗除草剂玉米品种奠定了基础[78]。基于PE系统的不断优化与改造,将为基因编辑工具的开发以及抗除草剂种质的创制提供稳固的技术支持。
综上,在以上所描述的利用生物技术创制抗除草剂作物的类型中,抗ALS抑制类的除草剂作物材料最丰富,在水稻、小麦、玉米、油菜和烟草等均有相关报道(表1)。
4 机遇与挑战
目前,我国采用化学除草的面积已达到播种面积的60%,基本上形成了以化学药剂防除为主的田间杂草防除技术体系[99]。转基因作物自1996年开始商业化种植以来,全球转基因作物的种植面积总体上呈逐年攀升趋势。2019年全球转基因作物种植面积超过1.9亿hm2,是1996年种植面积170万hm2的112倍。抗除草剂是转基因大豆、油菜、玉米、苜蓿和棉花的主要性状,单一抗除草剂转基因作物的种植面积为8 150万hm2,约占全球转基因作物种植面积的43%,这就意味着抗除草剂作物有着巨大的市场发展潜力与占有率[100]。
随着除草剂的大面积使用,抗性杂草的产生是当前化学防治杂草面临的主要挑战,培育复合抗除草剂作物、优化除草剂配套使用方法是解决该问题的有效途径。同时,基因逃逸以及环境安全问题也是面临的一个重要挑战。如果一些作物的近缘种植株获得了抗除草剂基因而变成了“超级杂草”,或者是农民为了快速达到消除杂草的目的加大药剂的使用量,甚至是随意丢弃废弃的农药包装,都将会对农田杂草的防治、环境安全带来新的难题。基于此,研制开发一种新型安全的生物型除草剂及其抗除草剂作物系统将有助于问题的解决。
CRISPR-Cas系统与诱变育种相比可节省大量的人力物力,且更高效和精准。相较于转基因作物,目前,已经有国家逐渐出台了对于基因编辑作物的监管政策,相较于对转基因作物的严格管控,美国监管机构对基因编辑作物的态度相对“温和”,让人感到欣喜的是经基因编辑的西红柿和鱼已经正式在日本上市。对于抗除草剂作物的创制,结合已有的研究表明,大多数抗除草剂作物的作用机理主要是某一位点的氨基酸发生特定突变,导致与除草剂的结合活性降低,基于CRISPR-Cas9系统发展的碱基编辑技术和Prime editing技术在创制抗除草剂作物上就作出了重要贡献,创制了不同类型的单抗除草剂材料,以及具有复合性状的多抗除草剂材料。同时,CRISPR-Cas系统也存在着一些局限性,主要包括(1)PAM位点的限制:目前,常用的Cas9和Cas12a主要识别的PAM是NGG和TTTV,但是针对一些特定的除草剂突变位点却没有合适的PAM,因此,就需要开发一些无PAM限制的Cas变体,拓宽全基因组编辑的范围;(2)转化受体材料和转化方法的限制:目前,植物中常用的转化系统包括农杆菌介导法、基因枪轰击法及PEG转化法(polyethylene glycol,PEG),其中,农杆菌介导法是最成熟的转化途径,但由于宿主范围的限制,只适用于部分植物物种。对于受体材料具有较强的基因型的差异,如:在棉花中只有JIN668、YZ-1和中棉24具有较强的再生能力,其余的棉花种质例如海岛棉则难再生,限制了基因编辑的广泛应用;(3)脱靶问题:基因编辑工具除了要具备高效性之外,还应具有一定的精准性,尽可能提高DNA识别的特异性,减少在全基因组范围内的脱靶;(4)原创性以及专利问题:在我国科学研究中所使用的基因编辑技术体系,其相关核心专利大都掌握在欧美相关研究机构和公司手中,博德研究所的张峰团队已经申请了CRISPR/Cas9的专利,未来我国如果要利用其进行分子育种、医疗诊断等商业化应用的话,存在被“卡脖子”的巨大风险。但目前随着基因组学、结构生物学、宏基因组学、生物信息学的快速发展,基因编辑技术也在以前所未有的态势不断发展,新型的基因编辑工具不断被发掘,基因编辑技术将为抗除草剂作物的培育提供更多的技术手段。
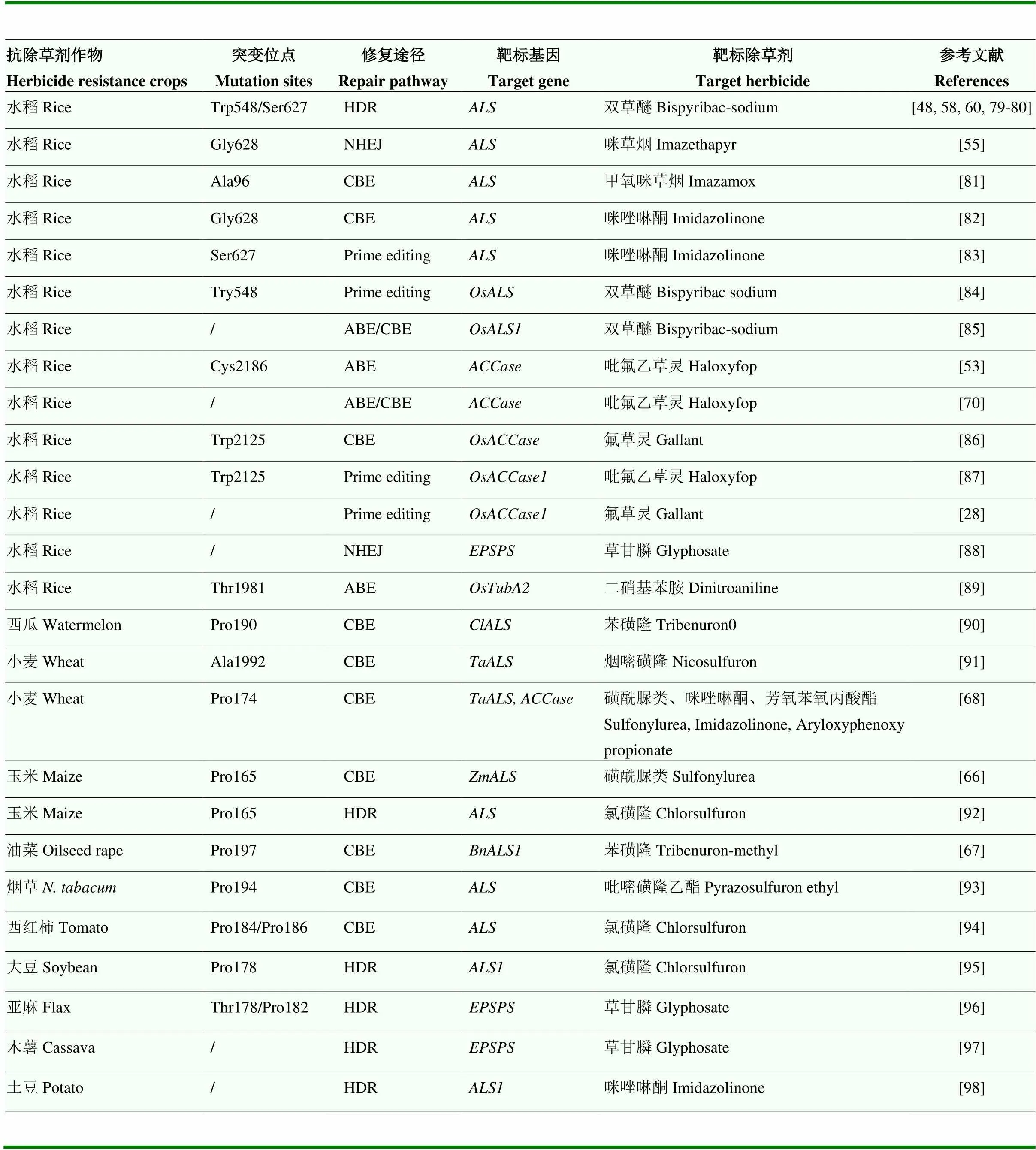
表1 基于基因编辑培育抗除草剂作物
HDR:同源重组修复;NHEJ:非同源末端连接;ABE:腺嘌呤碱基编辑;CBE:胞嘧啶碱基编辑
HDR: homology-directed repair; NHEJ: Non-homologous end joining; ABE: adenine base editing; CBE: cytidine base editing
5 展望
目前,抗除草剂作物的种植一方面减少了劳动力和财力的投入,降低了生产成本,另一方面,这也促进了规模化与机械化的发展,为大规模、集约化的现代化农场的建设创造了条件。但随之而来的是除草剂的大量施用和抗除草剂作物大面积推广,导致杂草对除草剂抗性,更有甚者部分杂草对多种除草剂产生了多重抗性,这就为杂草管理和抗除草剂作物的发展提出了新的挑战。针对这一挑战,需要进行科学的杂草管理、合理的种植制度、挖掘与现有除草剂没有交叉抗性的新型除草剂基因并研究其作用模式、借助转基因技术及基因编辑技术创制满足生产需要的抗除草剂新种质[101]。寻找新型的生物除草剂及其抗性基因与作用机制也是未来一个重要的发展方向,这样既能够最大限度地发挥现有除草剂的经济效益,也能优化现有的杂草防治体系和普及保水、保土及低投入的少耕、免耕、密植的耕种模式,同时优化抗除草剂作物的种植方式。此外,基于环境友好与可持续性的考虑(绿水青山就是金山银山),就要求新型除草剂应该具有高效,可持续,安全无公害和适应环境等特性。现在,也出现了一些解决这些问题的方法。如:基于抗性基因导向方法挖掘新型作用机制的除草剂系统的方法[16];综合运用生物信息学、分子生物学和药理学的方法为发现除草剂原始靶标提供了便利。基于这些方法,还开发出一些新型除草剂标靶基因,如:SPS、HST、DHODH、FAT和DHAD等[102]。目前,全球已经成功商业化或正式获得登记的生物除草剂产品已经有20余种[21]。其中利用生物活体的生物除草剂类型,由于需要较为严格的环境条件才能起作用,限制了其应用。这些问题的解决均有赖于功能基因组学研究、生物基因工程技术和生产工艺的创新[3]。因此,为了克服生物除草剂产品发展的成本与环境制约因素,应加大力度研制和发展生物除草剂生产工艺、新剂型及应用技术。
转基因技术与基因编辑技术是创制抗除草剂材料的2种最优方法,但两者之间存在着本质的区别。基因编辑作物育种用于编辑作物内源基因,且后代可分离出外源调控基因序列,经过连续多代分离后可以获得完全不含外源基因、且具有目的性状的目标材料。面对基因编辑技术所存在的一些限制性问题,目前也有了一些应对措施,例如,目前已经挖掘了一些Cas蛋白的变体,既可以拓宽基因组编辑的范围,又具有高效的靶向特异性,如xCas9[103]、Cas9-NG[104]、SpRY[105]、SpG[106]、ttLbCas12a[107]和enAsCas12a[108]等;转化受限于基因型的植物,一方面可以运用纳米颗粒,或病毒载体进行基因编辑元件的递送方法打破基因型限制,目前在烟草[109]、小麦[110]、甘薯[111]中均有相关报道;另一方面,通过利用一些再生因子提高转化的效率,比如BBM、WUS等关键再生因子[112];在原创性的基因编辑工具挖掘方面我国也取得了一定的进步,中国农业大学的赖锦盛教授利用生物信息学成功挖掘到一种新的CRISPR/Cas12j基因编辑系统。目前,各国对于基因编辑技术的监管问题也不尽相同。2022年1月24日,中国农业农村部制定公布了《农业用基因编辑植物安全评价指南(试行)》,主要针对没有引入外源基因的基因编辑植物,依据可能产生的风险申请安全评价。这对我国基因编辑技术从业者将是一个极大鼓舞。对于我国生物育种技术研发与产业推动意义重大。
基因编辑技术对于我国的作物改良具有重要的推动作用,体现在作物的高产、抗逆、单倍体诱导、品质改良等一系列角度,这对于抗除草剂作物的发展也将是里程碑式的。相信在后基因组时代,伴随着基因编辑技术以及多种组学的发展,我国的抗除草剂育种将迎来一个崭新的时代。
[1] Powles S B, Yu Q. Evolution in action: plants resistant to herbicides. Annual Review of Plant Biology,2010, 61: 317-347.
[2] 杨红梅, 冯莉, 陈光辉. 作物田杂草种子库研究进展. 广东农业科学, 2008, 35(10): 57-60, 67.
Yang H M, FENG L, Chen G H. Research development of crop field weed seed-bank. Guangdong Agricultural Sciences, 2008, 35(10): 57-60, 67. (in Chinese)
[3] 孙金秋, 任相亮, 胡红岩, 姜伟丽, 马亚杰, 王丹, 宋贤鹏, 马艳, 马小艳. 农田杂草群落演替的影响因素综述. 杂草学报, 2019, 37(2): 1-9.
Sun J Q, Ren X L, Hu H Y, Jiang W L, Ma Y J, Wang D, Song X P, Ma Y, Ma X Y. The factors influencing weed community succession in the crop field. Journal of Weed Science, 2019, 37(2): 1-9. (in Chinese)
[4] Jin M, Chen L, Deng X W, Tang X Y. Development of herbicide resistance genes and their application in rice. The Crop Journal, 2022, 10(1): 26-35.
[5] 李扬汉. 中国杂草志. 北京: 中国农业出版社, 1998.
Li Y H. Weeds of China. 北京: China Agriculture Press, 1998. (in Chinese)
[6] Oerke E C. Crop losses to pests. The Journal of Agricultural Science, 2006, 144(1): 31-43.
[7] 李香菊. 近年我国农田杂草防控中的突出问题与治理对策. 植物保护, 2018, 44(5): 77-84.
Li X j. Main problems and management strategies of weeds in agricultural fields in China in recent years. Plant Protection, 2018, 44(5): 77-84. (in Chinese)
[8] Burgos N R, Singh V, Tseng T M, Black H, Young N D, Huang Z, Hyma K E, Gealy D R, Caicedo A L. The impact of herbicide-resistant rice technology on phenotypic diversity and population structure of United States weedy rice. Plant Physiology, 2014, 166(3): 1208-1220.
[9] 檀琮萍, 江昌俊. 作物抗除草剂基因工程与抗除草剂转基因小麦的研究进展. 安徽农业大学学报, 2003, 30(1): 27-33.
Tan C p, Jiang C j. Research progress on herbicide resistant gene engineering of crop and transgenic herbicide resistant wheat. Journal of Anhui Agricultural University, 2003, 30(1): 27-33. (in Chinese)
[10] 强胜, 宋小玲, 戴伟民. 抗除草剂转基因作物面临的机遇与挑战及其发展策略. 农业生物技术学报, 2010, 18(1): 114-125.
Qiang S, Song X l, Dai W M.The opportunity and challenge faced by transgenic herbicide-resistant crops and their development strategy. Journal of Agricultural Biotechnology, 2010, 18(1): 114-125. (in Chinese)
[11] Zhang D B, Shi J X, Yang X J. Role of lipid metabolism in plant pollen exine development. Sub-cellular Biochemistry,2016, 86: 315-337.
[12] Pichersky E, Lewinsohn E. Convergent evolution in plant specialized metabolism. Annual Review of Plant Biology, 2011, 62: 549-566.
[13] Nakka S, Jugulam M, Peterson D, Asif M. Herbicide resistance: Development of wheat production systems and current status of resistant weeds in wheat cropping systems. The Crop Journal, 2019, 7(6): 750-760.
[14] Galili G, Amir R, Fernie A R. The regulation of essential amino acid synthesis and accumulation in plants. Annual Review of Plant Biology, 2016, 67: 153-178.
[15] Tan S Y, Evans R R, Dahmer M L, Singh B K, Shaner D L. Imidazolinone-tolerant crops: history, current status and future. Pest Management Science, 2005, 61(3): 246-257.
[16] Yan Y, Liu Q K, Zang X, Yuan S G, Bat-Erdene U, Nguyen C, Gan J H, Zhou J H, Jacobsen S E, Tang Y. Resistance-gene-directed discovery of a natural-product herbicide with a new mode of action. Nature, 2018, 559(7714): 415-418.
[17] Amrhein N, Deus B, Gehrke P, Steinrucken H C. The site of the inhibition of the shikimate pathway by glyphosate: II. Interference of glyphosate with chorismate formation in vivo and in vitro. Plant Physiology, 1980, 66(5): 830-834.
[18] Duke S O, Powles S B. Glyphosate: a once-in-a-century herbicide. Pest Management Science,2008, 64(4): 319-325.
[19] Dill G M. Glyphosate-resistant crops: history, status and future. Pest Management Science, 2005, 61(3): 219-224.
[20] 陈世国, 强胜. 生物除草剂研究与开发的现状及未来的发展趋势. 中国生物防治学报, 2015, 31(5): 770-779.
Chen S G, Qiang S. The status and future directions of bioherbicide study and development. Chinese Journal of Biological Control, 2015, 31(5): 770-779. (in Chinese)
[21] James D, Borphukan B, Fartyal D, Ram B, Singh J, Manna M, Sheri V, Panditi V, Yadav R, REDDY M K. Concurrent overexpression of OsGS1;1 and OsGS2 genes in transgenic rice (L.): impact on tolerance to abiotic stresses. Frontiers in Plant Science, 2018, 9: 786.
[22] Tang W, Zhou F Y, Chen J, Zhou X G. Resistance to ACCase-inhibiting herbicides in an Asia minor bluegrass () population in China. Pesticide Biochemistry and Physiology, 2014, 108: 16-20.
[23] Ndikuryayo F, Moosavi B, Yang W C, Yang G F. 4- hydroxyphenylpyruvate dioxygenase inhibitors: From chemical biology to agrochemicals. Journal of Agricultural and Food Chemistry, 2017, 65(39): 8523-8537.
[24] Comai L, Sen L C, Stalker D M. An altered aroa gene product confers resistance to the herbicide glyphosate. Science, 1983, 221(4608): 370-371.
[25] Miki B, McHugh S. Selectable marker genes in transgenic plants: applications, alternatives and biosafety. Journal of Biotechnology,2004, 107(3): 193-232.
[26] Dong H R, Huang Y, Wang K J. The development of herbicide resistance crop plants using CRISPR/Cas9-mediated gene editing. Genes, 2021, 12(6): 912.
[27] Hussain A, Ding X, Alariqi M, Manghwar H, Hui F J, Li Y P, Cheng J Q, Wu C L, Cao J L, Jin S X. Herbicide resistance: another hot agronomic trait for plant genome editing. Plants, 2021, 10(4): 621.
[28] Xu R F, Liu X S, Li J, Qin R Y, Wei P C. Identification of herbicide resistance OsACC1 mutations via in planta prime-editing-library screening in rice. Nature Plants, 2021, 7(7): 888-892.
[29] Yan D R, Ren B, Liu L, Yan F, Li S F, Wang G R, Sun W X, Zhou X P, Zhou H B. High-efficiency and multiplex adenine base editing in plants using new TadA variants. Molecular Plant, 2021, 14(5): 722-731.
[30] Maeda H, Murata K, Sakuma N, Takei S, Yamazaki A, AHIRm M R, Kawata M, Hirose S, Kawagishi-Kobayashi M, Taniguchi Y, SUZUKI S, SEKINO K, OHSHIMA M, KATO H, YOSHIDA H, TOZAWA Y. A rice gene that confers broad-spectrum resistance to β-triketone herbicides. Science, 2019, 365(6451): 393-396.
[31] Zhao D L, Han X B, Wang M, Zeng Y T, Li Y Q, Ma G Y, Liu J, Zheng C J, Wen M X, Zhang Z F, ZHANG P, ZHANG C S. Herbicidal and antifungal xanthone derivatives from the-derived fungusD5. Journal of Agricultural and Food Chemistry, 2020, 68(40): 11207-11214.
[32] Wu G B, Yuan M R, Wei L, Zhang Y, Lin Y J, Zhang L L, Liu Z D. Characterization of a novel cold-adapted phosphinothricin N-acetyltransferase from the marine bacteriumsp. strain YM12. Journal of Molecular Catalysis B: Enzymatic, 2014, 104: 23-28.
[33] Yi S y, Wu G b, Lin Y j, Hu N, Liu Z d. Characterization of a new type of glyphosate-tolerant 5-enolpyruvyl shikimate-3-phosphate synthase from. Journal of Molecular Catalysis B: Enzymatic, 2015, 111: 1-8.
[34] Liu X, Wang S J, Yu W S, Zhang J Q, Fang S, Zhang J G, Qiu J, Kong F Y, Duan X G. Single platinum atoms anchored on holy carbon nitride for efficient photodegradation of sulfonylurea herbicide. Chemical Engineering Journal, 2022, 446: 137426.
[35] Lonhienne T, Cheng Y, Garcia M D, Hu S H, Low Y S, Schenk G, Williams C M, Guddat L W. Structural basis of resistance to herbicides that target acetohydroxyacid synthase. Nature Communications, 2022, 13(1): 3368.
[36] NEWHOUSE K E, SMITH W A, STARRETT M A, SCHAEFER T J, SINGH B K. Tolerance to imidazolinone herbicides in wheat. Plant physiology, 1992, 100(2): 882-886.
[37] Sala C A, Bulos M, Altieri E, Ramos M l. Genetics and breeding of herbicide tolerance in sunflower. Helia, 2012, 35(57): 57-69.
[38] Sebastian S A, Fader G M, Ulrich J F, Forney D R, Chaleff R S. Semidominant soybean mutation for resistance to sulfonylurea herbicides. Crop Science, 1989, 29(6): 1403-1408.
[39] Endo M, Toki S. Creation of herbicide-tolerant crops by gene targeting. Journal of Pesticide Science, 2013, 38(2): 49-59.
[40] Castle L A, Siehl D L, Gorton R, Patten P A, Chen Y H, Bertain S, Cho H J, Duck N, Wong J, Liu D L, LASSNER M W. Discovery and directed evolution of a glyphosate tolerance gene. Science, 2004, 304(5674): 1151-1154.
[41] Thompson C J, Movva N R, Tizard R, Crameri R, Davies J E, Lauwereys M, Botterman J. Characterization of the herbicide-resistance gene bar from. The EMBO Journal, 1987, 6(9): 2519-2523.
[42] Cui Y, Liu Z D, Li Y, Zhou F, Chen H, Lin Y J. Application of a novel phosphinothricin N-acetyltransferase (RePAT) gene in developing glufosinate-resistant rice. Scientific Reports, 2016, 6(1): 21259.
[43] Beckie H J, Hall L M. Genetically-modified herbicide-resistant (GMHR) crops a two-edged sword? An Americas perspective on development and effect on weed management. Crop Protection, 2014, 66: 40-45.
[44] Dun B Q, Wang X J, Lu W, Chen M, Zhang W, Ping S Z, Wang Z X, Zhang B M, Lin M. Development of highly glyphosate-tolerant tobacco by coexpression of glyphosate acetyltransferase gat and EPSPS G2-aroA genes. The Crop Journal, 2014, 2(2/3): 164-169.
[45] Yi S Y, Cui Y, Zhao Y, Liu Z D, Lin Y J, Zhou F. A novel naturally occurring class I 5-enolpyruvylshikimate-3-phosphate synthase fromsp. confers high glyphosate tolerance to rice. Scientific Reports, 2016, 6: 19104.
[46] 崔永祯, 杨晓云, 李绍祥, 浦秋红, 杨忠慧, 李宏生, 丁明亮. 转基因技术与基因编辑技术在抗除草剂农作物上的应用. 中国种业, 2022(4): 19-22.
Cui Y Z, Yang X Y, Li S X, Pu Q H, Yang Z H, Li H S, Ding M L. Application of transgenic technology and gene editing technology in crops with herbicide resistants. China Seed Industry, 2022(4): 19-22. (in Chinese)
[47] Guo B F, Guo Y, Hong H L, Jin L G, Zhang L J, Chang R Z, Lu W, Lin M, Qiu L J. Co-expression of G2-EPSPS and glyphosate acetyltransferasegenes conferring high tolerance to glyphosate in soybean. Frontiers in Plant Science, 2015, 6: 847.
[48] 邱龙, 马崇烈, 刘博林, 章旺根. 耐除草剂转基因作物研究现状及发展前景. 中国农业科学, 2012, 45(12): 2357-2363.
Qiu L, Ma C L, Liu B L, Zhang W G. Current situation of research on transgenic crops with herbicide tolerance and development prospect. Scientia Agricultura Sinica, 2012, 45(12): 2357-2363. (in Chinese)
[49] Fartyal D, Agarwal A, James D, Borphukan B, Ram B, Sheri V, Yadav R, Manna M, Varakumar P, REDDY M K. Co-expression of P173S mutant rice EPSPS andgenes results in higher glyphosate tolerance in transgenic rice. Frontiers in Plant Science, 2018, 9: 144.
[50] Manghwar H, Lindsey K, Zhang X L, Jin S X. CRISPR/Cas system: recent advances and future prospects for genome editing. Trends in plant science, 2019, 24(12): 1102-1125.
[51] Horvath P, Barrangou R. CRISPR/Cas, the immune system of bacteria and archaea. Science,2010, 327(5962): 167-170.
[52] Wang J Y, Doudna J A. CRISPR technology: A decade of genome editing is only the beginning. Science, 2023, 379(6629): eadd8643.
[53] Puchta H, Fauser F. Synthetic nucleases for genome engineering in plants: prospects for a bright future. The Plant Journal, 2014, 78(5): 727-741.
[54] Zetsche B, Gootenberg J S, Abudayyeh O O, Slaymaker I M, Makarova K S, Essletzbichler P, Volz S E, Joung J, van der Oost J, Regev A, KOONIN E V, ZHANG F. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell, 2015, 163(3): 759-771.
[55] Wang F Q, Xu Y, Li W Q, Chen Z H, Wang J, Fan F J, Tao Y J, Jiang Y J, Zhu Q H, Yang J. Creating a novel herbicide-tolerance OsALS allele using CRISPR/Cas9-mediated gene editing. The Crop Journal, 2021, 9(2): 305-312.
[56] Li S Y, Li J Y, He Y B, Xu M L, Zhang J H, Du W M, Zhao Y D, Xia L Q. Precise gene replacement in rice by RNA transcript- templated homologous recombination. Nature Biotechnology, 2019, 37(4): 445-450.
[57] Reiss B, Klemm M, Kosak H, Schell J. RecA protein stimulates homologous recombination in plants. Proceedings of the National Academy of Sciences of the United States of America, 1996, 93(7): 3094-3098.
[58] Wang M g, Lu Y m, Botella J R, Mao Y f, Hua K, Zhu J k. Gene targeting by homology-directed repair in rice using a geminivirus-based CRISPR/Cas9 system. Molecular plant, 2017, 10(7): 1007-1010.
[59] Song F, Stieger K. Optimizing the DNA donor template for homology-directed repair of double-strand breaks. Molecular Therapy-Nucleic Acids, 2017, 7: 53-60.
[60] Sun Y w, Zhang X, Wu C y, He Y b, Ma Y z, Hou H, Guo X p, Du W m, Zhao Y d, Xia L q. Engineering herbicide-resistant rice plants through CRISPR/Cas9-mediated homologous recombination of acetolactate synthase. Molecular Plant, 2016, 9(4): 628-631.
[61] Li S y, Li J y, Zhang J h, Du W m, Fu J d, Sutar S, Zhao Y d, Xia L q. Synthesis-dependent repair of Cpf1-induced double strand DNA breaks enables targeted gene replacement in rice. Journal of Experimental Botany, 2018, 69(20): 4715-4721.
[62] Lu Y m, Tian Y f, Shen R d, Yao Q, Wang M g, Chen M, Dong J s, Zhang T e, Li F, Lei M g, zhu j k.Targeted, efficient sequence insertion and replacement in rice. Nature Biotechnology, 2020, 38(12): 1402-1407.
[63] Komor A C, Kim Y B, Packer M S, Zuris J A, Liu D R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature, 2016, 533(7603): 420-424.
[64] Gaudelli N M, Komor A C, Rees H A, Packer M S, Badran A H, Bryson D I, Liu D R. Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature, 2017, 551(7681): 464-471.
[65] Tong H w, Wang X c, Liu Y h, Liu N n, Li Y, Luo J m, Ma Q, Wu D n, Li J y, Xu C l, yang h.Programmable A-to-Y base editing by fusing an adenine base editor with an N-methylpurine DNA glycosylase. Nature Biotechnology, 2023.
[66] Li Y m, Zhu J j, Wu H, Liu C l, Huang C l, Lan J h, Zhao Y m, Xie C x. Precise base editing of non-allelic acetolactate synthase genes confers sulfonylurea herbicide resistance in maize. The Crop Journal, 2020, 8(3): 449-456.
[67] Wu J, Chen C, Xian G y, Liu D x, Lin L, Yin S l, Sun Q f, Fang Y j, Zhang H, Wang Y p. Engineering herbicide-resistant oilseed rape by CRISPR/Cas9-mediated cytosine base-editing. Plant Biotechnology Journal, 2020, 18(9): 1857-1859.
[68] Zhang R, Liu J x, Chai Z z, Chen S, Bai Y, Zong Y, Chen K l, Li J y, Jiang L j, Gao C x. Generation of herbicide tolerance traits and a new selectable marker in wheat using base editing. Nature Plants, 2019, 5(5): 480-485.
[69] Han H n, Wu Z w, Zheng L, Han J y, Zhang Y, Li J h, Zhang S j, Li G y, Ma C l, Wang P p. Generation of a high-efficiency adenine base editor with TadA8e for developing wheat dinitroaniline-resistant germplasm. The Crop Journal, 2022, 10(2): 368-374.
[70] Li C, Zhang R, Meng X b, Chen S, Zong Y, Lu C j, Qiu J L, Chen Y H, Li J y, Gao C x. Targeted, random mutagenesis of plant genes with dual cytosine and adenine base editors. Nature biotechnology, 2020, 38(7): 875-882.
[71] Anzalone A V, Randolph P B, Davis J R, Sousa A A, Koblan L W, Levy J M, Chen P J, Wilson C, Newby G A, Raguram A, liu d r. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature,2019, 576(7785): 149-157.
[72] Zhuang Y, Liu J l, Wu H, Zhu Q g, Yan Y c, Meng H w, Chen P R, Yi C q. Increasing the efficiency and precision of prime editing with guide RNA pairs. Nature Chemical Biology, 2022, 18(1): 29-37.
[73] Li X s, Zhou L n, Gao B Q, Li G y, Wang X, Wang Y, Wei J, Han W y, Wang Z x, Li J F, GAO R Z, ZHU J J, XU W C, WU J, YANG B, SUN X D, YANG L, CHEN J. Highly efficient prime editing by introducing same-sense mutations in pegRNA or stabilizing its structure. Nature Communications, 2022, 13(1): 1669.
[74] Nelson J W, Randolph P B, Shen S P, Everette K A, Chen P J, Anzalone A V, An M, Newby G A, Chen J C, Hsu A, liu d r. Engineered pegRNAs improve prime editing efficiency. Nature Biotechnology, 2022, 40(3): 402-410.
[75] CHEN P J, HUSSMANN J A, YAN J, KNIPPING F, RAVISANKAR P, CHEN P F, CHEN C D, NELSON J W, Newby G A, SAHIN M, OSBORN M J, WEISSMAN J S, ADAMSON B, LIU D R. Enhanced prime editing systems by manipulating cellular determinants of editing outcomes. Cell,2021, 184(22): 5635-5652. e29.
[76] Jin S, Lin Q p, Gao Q, Gao C x. Optimized prime editing in monocot plants using PlantPegDesigner and engineered plant prime editors (ePPEs). Nature Protocols, 2023, 18(3): 831-853.
[77] Zong Y, Liu Y j, Xue C x, Li B s, Li X y, Wang Y p, Li J, Liu G w, Huang X x, Cao X f, gao c x.An engineered prime editor with enhanced editing efficiency in plants. Nature Biotechnology, 2022, 40(9): 1394-1402.
[78] Qiao D x, Wang J y, Lu M H, Xin C p, Chai Y p, Jiang Y y, Sun W, Cao Z h, Guo S y, Wang X C, chen q j. Optimized prime editing efficiently generates heritable mutations in maize. Journal of Integrative Plant Biology, 2023, 65(4): 900-906.
[79] Endo M, Mikami M, Toki S. Biallelic gene targeting in rice. Plant Physiology, 2016, 170(2): 667-677.
[80] Shimatani Z, Kashojiya S, Takayama M, Terada R, Arazoe T, Ishii H, Teramura H, Yamamoto T, Komatsu H, Miura K, EZURA H, NISHIDA K, ARIIZUMI T, KONDO A. Targeted base editing in rice and tomato using a CRISPR-Cas9 cytidine deaminase fusion. Nature Biotechnology, 2017, 35(5): 441-443.
[81] Shimatani Z, Fujikura U, Ishii H, Matsui Y, Suzuki M, Ueke Y, Taoka K i, Terada R, Nishida K, Kondo A. Inheritance of co-edited genes by CRISPR-based targeted nucleotide substitutions in rice. Plant Physiology and Biochemistry, 2018, 131: 78-83.
[82] Zhang R, Chen S, Meng X B, Chai Z Z, Wang D L, Yuan Y G, Chen K L, Jiang L J, Li J, Gao C X. Generating broad-spectrum tolerance to ALS-inhibiting herbicides in rice by base editing. Science China Life Sciences, 2021, 64(10): 1624-1633.
[83] Hua K, Jiang Y W, Tao X P, Zhu J K. Precision genome engineering in rice using prime editing system. Plant Biotechnology Journal, 2020, 18(11): 2167-2169.
[84] Butt H, Rao G S, Sedeek K, Aman R, Kamel R, Mahfouz M. Engineering herbicide resistance via prime editing in rice. Plant Biotechnology Journal, 2020, 18(12): 2370-2372.
[85] Kuang Y J, Li S F, Ren B, Yan F, Spetz C, Li X J, Zhou X P, Zhou H B. Base-editing-mediated artificial evolution of OsALS1 in planta to develop novel herbicide-tolerant rice germplasms. Molecular Plant, 2020, 13(4): 565-572.
[86] Liu X S, Qin R Y, Li J, Liao S X, Shan T F, Xu R F, Wu D X, Wei P C. A CRISPR-Cas9-mediated domain-specific base-editing screen enables functional assessment of ACCase variants in rice. Plant Biotechnology Journal, 2020, 18(9): 1845-1847.
[87] Xu R F, Li J, Liu X S, Shan T F, Qin R Y, Wei P C. Development of plant prime-editing systems for precise genome editing. Plant Communications, 2020, 1(3): 100043.
[88] Li J, Meng X B, Zong Y, Chen K L, Zhang H W, Liu J X, Li J Y, Gao C X. Gene replacements and insertions in rice by intron targeting using CRISPR-Cas9. Nature Plants, 2016, 2(10): 16139.
[89] Liu L, Kuang Y J, Yan F, Li S F, Ren B, Gosavi G, Spetz C, Li X J, Wang X F, Zhou X P, ZHOU H B. Developing a novel artificial rice germplasm for dinitroaniline herbicide resistance by base editing of OsTubA2. Plant Biotechnology Journal, 2021, 19(1): 5-7.
[90] Tian S W, Jiang L J, Cui X X, Zhang J, Guo S G, Li M Y, Zhang H Y, Ren Y, Gong G Y, Zong M, LIU F, CHEN Q J, XU Y. Engineering herbicide-resistant watermelon variety through CRISPR/Cas9-mediated base-editing. Plant Cell Reports, 2018, 37(9): 1353-1356.
[91] Zong Y, Song Q N, Li C, Jin S, Zhang D B, Wang Y P, Qiu J L, Gao C X. Efficient C-to-T base editing in plants using a fusion of nCas9 and human APOBEC3A. Nature Biotechnology, 2018, 36(10): 950-953.
[92] Svitashev S, Young J K, Schwartz C, Gao H R, Falco S C, Cigan A M. Targeted mutagenesis, precise gene editing, and site-specific gene insertion in maize using Cas9 and guide RNA. Plant Physiology, 2015, 169(2): 931-945.
[93] Kang B C, Woo J W, Kim S T, Bae S J, Choi M, Kim J S, Kim S G. Guidelines for C to T base editing in plants: base-editing window, guide RNA length, and efficient promoter. Plant Biotechnology Reports, 2019, 13(5): 533-541.
[94] Veillet F, Perrot L, Chauvin L, Kermarrec M P, Guyon-Debast A, Chauvin J E, Nogué F, Mazier M.Transgene-free genome editing in tomato and potato plants using-mediated delivery of a CRISPR/Cas9 cytidine base editor. International Journal of Molecular Sciences, 2019, 20(2): 402.
[95] LiZ s, Liu Z B, Xing A q, Moon B P, Koellhoffer J P, Huang L x, Ward R T, Clifton E, Falco S C, Cigan A M.Cas9-guide RNA directed genome editing in soybean. Plant Physiology, 2015, 169(2): 960-970.
[96] Sauer N J, Narváez-Vásquez J, Mozoruk J, Miller R B, Warburg Z J, Woodward M J, Mihiret Y A, Lincoln T A, Segami R E, Sanders S L, WALKER K A, BEETHAM P R, SCHÖPKE C R, GOCAL G F W. Oligonucleotide-mediated genome editing provides precision and function to engineered nucleases and antibiotics in plants. Plant Physiology, 2016, 170(4): 1917-1928.
[97] Hummel A W, Chauhan R D, Cermak T, Mutka A M, Vijayaraghavan A, Boyher A, Starker C G, Bart R, Voytas D F, Taylor N J. Allele exchange at the EPSPS locus confers glyphosate tolerance in cassava. Plant Biotechnology Journal, 2018, 16(7): 1275-1282.
[98] BUTLER N M, BALTES N J, VOYTAS D F, DOUCHES D S. Geminivirus-mediated genome editing in potato (L.) using sequence-specific nucleases. Frontiers in Plant Science, 2016, 7: 1045.
[99] 强胜, 陈世国. 生物除草剂研发现状及其面临的机遇与挑战. 杂草科学, 2011, 29(1): 1-6.
Qiang S, Chen S g. Current status of bioherbicide research and development and its opportunities and challenges. Journal of Weed Science, 2011, 29(1): 1-6. (in Chinese)
[100] 蒋田田, 文君慧. 我国抗除草剂转基因作物面临的机遇和挑战. 安徽农业科学, 2021, 49(22): 239-242.
Jiang T t, Wen J h. Opportunities and challenges facing genetically modified herbicide-resistant crops in china. Journal of Anhui Agricultural Sciences, 2021, 49(22): 239-242. (in Chinese)
[101] He B, Hu Y h, Wang W, Yan W, Ye Y h. The Progress towards novel herbicide modes of action and targeted herbicide development. Agronomy, 2022, 12(11): 2792.
[102] Duke S O, Stidham M A, Dayan F E. A novel genomic approach to herbicide and herbicide mode of action discovery. Pest Management Science, 2019, 75(2): 314-317.
[103] Hu J H, Miller S M, Geurts M H, Tang W, Chen L, Sun N, Zeina C M, Gao X, Rees H A, Lin Z, LIU D R. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature, 2018, 556(7699): 57-63.
[104] ZENG D C, LI X T, HUANG J F, LI Y Y, CAI S Q, YU W Z, LI Y L, HUANG Y P, XIE X R, GONG Q,TAN J T, ZHENG Z Y, GUO M H, LIU Y G, ZHU Q L. Engineered Cas9 variant tools expand targeting scope of genome and base editing in rice. Plant Biotechnology Journal, 2020, 18(6): 1348-1350.
[105] Christie K A, Guo J A, Silverstein R A, Doll R M, Mabuchi M, Stutzman H E, Lin J, Ma L, Walton R T, Pinello L, ROBB G B, KLEINSTIVER B P. Precise DNA cleavage using CRISPR-SpRYgests. Nature Biotechnology, 2023, 41(3): 409-416.
[106] WALTON R T, CHRISTIE K A, WHITTAKER M N, KLEINSTIVER B P. Unconstrained genome targeting with near-PAMless engineered CRISPR-Cas9 variants. Science, 2020, 368(6488): 290-296.
[107] Schindele P, Merker L, Schreiber T, Prange A, Tissier A, Puchta H. Enhancing gene editing and gene targeting efficiencies inby using an intron-containing version of ttLbCas12a. Plant Biotechnology Journal, 2023, 21(3): 457-459.
[108] KLEINSTIVER B P, SOUSA A A, WALTON R T, TAK Y E, HSU J Y, CLEMENT K, WELCH M M, Horng J E, MALAGON-LOPEZ J, SCARFÒ I, MAUS M V, PINELLO L, ARYEE M J, JOUNG J K. Engineered CRISPR-Cas12a variants with increased activities and improved targeting ranges for gene, epigenetic and base editing. Nature Biotechnology, 2019, 37(3): 276-282.
[109] Liu Q, Zhao C L, Sun K, Deng Y L, Li Z H. Engineered biocontainable RNA virus vectors for non-transgenic genome editing across crop species and genotypes. Molecular Plant, 2023, 16(3): 616-631.
[110] Li T D, Hu J C, Sun Y, Li B S, Zhang D L, Li W L, Liu J X, Li D W, Gao C X, Zhang Y L, WANG Y P. Highly efficient heritable genome editing in wheat using an RNA virus and bypassing tissue culture. Molecular Plant, 2021, 14(11): 1787-1798.
[111] Cao X S, Xie H T, Song M L, Lu J H, Ma P, Huang B Y, Wang M G, Tian Y F, Chen F, Peng J, LANG Z B, LI G F, ZHU J K. Cut-dip-budding delivery system enables genetic modifications in plants without tissue culture. The Innovation, 2023, 4(1): 100345.
[112] Maher M F, Nasti R A, Vollbrecht M, Starker C G, Clark M D, Voytas D F. Plant gene editing through de novo induction of meristems. Nature Biotechnology, 2020, 38(1): 84-89.
Opportunities and challenges for developing herbicide-resistance crops in the post-genomic era
WU YuanLong1, HUI FengJiao2, PAN ZhenYuan1, YOU ChunYuan3, LIN HaiRong1, LI ZhiBo1, JIN ShuangXia2, NIE XinHui1
1Agricultural College, Shihezi University/Key Laboratory of Oasis Ecology Agriculture, Xinjiang Production and Construction Corps, Shihezi 832003, Xinjiang;2College of Plant Sciences & Technology, Huazhong Agricultural University/National Key Laboratory of Crop Genetic Improvement, Wuhan 430070;3Cotton Research Institute of Shihezi Academy of Agricultural Sciences, Shihezi 832011, Xinjiang
Global agriculture is facing severe challenges, and breeding technology is the foundation and key to the development of the seed industry. Gene editing technology refers to the precise modification of target genes to achieve deletion, insertion, and replacement of specific target gene fragments. It can precisely modify target genes or introduce certain excellent genes into crops to produce crops with excellent agronomic traits, which has great potential in molecular design breeding and is of great significance to ensuring food security. Weed damage has a huge impact on the yield and quality of crops. To control weed damage efficiently, safely and sustainably has always been a hot research topic. Currently, more than 200 types of chemical herbicides have emerged in the global market. Using chemical methods to control weeds has become an important part of modern agriculture, and the cost of weed control has been significantly reduced by promoting herbicide-resistant crops. However, with the large-scale promotion of herbicide-resistant crops and the long-term use of single herbicides, environmental safety problems such as weed resistance and escape of resistant genes have gradually been discovered. Currently, the development of functional genomics, bioinformatics and genetic engineering technology (especially the widespread application of gene editing technology in plants) has created conditions for the creation of herbicide-resistant crops and new efficient weed control systems. In this article, the main target genes of herbicides that inhibit amino acid biosynthesis, lipid metabolism, carotenoid, plastoquinone and tocopherol biosynthesis pathways and their action mechanisms are introduced at first. Secondly, two methods for mining new herbicide resistance genes and herbicide systems are introduced, including the directed mutation method of herbicide resistance genes within crops based on CRISPR/Cas system and the resistance gene guidance method based on the co-evolution theory of natural product and organisms in nature. Moreover, the research progress of three breeding methods for herbicide resistant crops was reviewed, including conventional breeding, transgenic breeding and CRISPR/Cas genome editing based breeding. Among them, the research progress of CIRSPR/Cas system, base editing technology, and prime editing system in cultivating herbicide resistant crops were highlighted. The main challenge faced by chemical control of weeds and herbicide resistant crops is resistant weeds and environmental safety issues, and gene escape, respectively. At present, the rapid development of genome editing technology provides new solutions and new opportunities for the development of herbicide resistant crops in the post genome era. Finally, the prospects for the future of herbicide-resistant crops were provided.
gene editing technology; herbicides; herbicide-resistant genes; breeding of herbicide-resistant crops
2023-05-03;
2023-06-25
兵团科技创新人才计划-科技特派员(S2019CB1877)、兵团科技创新人才计划-强青(2021CB028)、石河子大学青年创新拔尖人才计划-拔尖人才(CXBJ202208)
吴元龙:wyl19880322@163.com。惠凤娇:17806278731@163.com。吴元龙和惠凤娇为同等贡献作者。通信作者金双侠,E-mail:jsx@mail.hzau.edu.cn。通信作者聂新辉,E-mail:xjnxh2004130@126.com
10.3864/j.issn.0578-1752.2023.17.005
(责任编辑 李莉)

