柚果肉颜色遗传变异分析及候选基因挖掘
程莉,杨胜男,朱延松,王旭,赵婉彤,李仁静,李沛,苑忠杰,江东
柚果肉颜色遗传变异分析及候选基因挖掘
程莉,杨胜男,朱延松,王旭,赵婉彤,李仁静,李沛,苑忠杰,江东
西南大学柑桔研究所,重庆 400712
【目的】果肉颜色是柚类品种重要的外观性状以及品质性状,挖掘与柚果肉颜色显著相关的变异位点及相关基因,为进一步理解柚类品种果肉呈色机理以及分子标记辅助育种奠定基础。【方法】通过表型调查及色差仪测量,对100份柚类种质的果肉颜色进行评价和分类,利用GBS(genotyping-by-sequencing)测序技术对所有材料进行简化基因组测序。将测序得到的基因型数据通过GCTA软件计算特征值及特征向量分析群体结构,利用plink 2.0软件计算不同果肉颜色群体的遗传分化系数(Fst),选用GEMMA软件中GLM模型进行全基因组关联分析,选取与颜色表型显著关联的变异位点进行等位变异分析,根据柑橘LD大小对变异位点邻近位置进行基因注释,筛选出与果肉颜色形成相关的候选基因,随机选择4份白肉柚和4份红肉柚材料在不同发育时期对候选基因表达进行初步验证。【结果】根据果肉颜色将100份柚类种质分为白肉柚和红肉柚两大类,其中包含58份白肉柚和42份红肉柚。通过Fst遗传分化指数分析和GWAS全基因组关联分析共筛选到Fst系数大于0.4且-log10(P)>9的位点6个,对6个变异位点进行基因分型,根据基因型能够预测柚品种的果肉颜色。对变异位点侧翼25 kb序列进行基因注释共筛选出14个基因,其中包含类异戊二烯合成、叶绿体氧化还原反应、质体发育、脱落酸信号调节、乙烯响应等相关基因和转录因子。基因表达分析显示,(促分裂原活化蛋白激酶激酶激酶MAPKKK17)、(叶绿体卟啉原脱氨酶)、(转录因子TCP7)、(叶绿体超氧化物歧化酶SOD3)、(FAD合成酶2)与果肉颜色形成存在一定关联。【结论】经过GWAS和Fst两种分析方式共鉴定到6个与果肉颜色显著关联的SNP位点,对该位点上下游25 kb的距离进行基因注释共筛选到了14个基因,通过基因表达模式分析初步判定其中5个基因参与了柚果肉颜色的形成。
柚;果肉颜色;Fst;关联分析;候选基因
0 引言
【研究意义】柚是芸香科柑橘属植物的一个基本种,经过长期的生物进化和栽培育种形成了丰富多样的种质资源[1-2],对现有资源进行鉴定、评价、分析和利用对于柚类育种改良具有重要意义。随着我国柚类产业的发展和消费需求的多样化,对于柚类品种的果实品质要求不断提高。果肉颜色是柚类品种重要的品质性状之一,在长期的栽培育种和人为选择过程中,产生了果肉颜色丰富的变异材料[3]。近年来,研究者们在解析果肉呈色机理上已经做了大量工作,包括柚在内的柑橘属植物果实呈色的关键结构基因已十分清晰[4]。但由于柑橘果树童期长、杂交群体占地面积大、遗传背景复杂、可利用的分子标记较少等多因素影响[5-7],对于果实颜色的代谢调控网络和相关基因还知之甚少,且通过构建遗传杂交群体来对果肉颜色的相关调控因子进行精细挖掘及定位耗时长且难度大[8-9]。因此,本研究利用国家柑橘种质资源圃(重庆)中保存的丰富柚类资源,通过高通量测序技术结合关联分析和群体遗传分化分析两种方式,以挖掘与果肉呈色显著关联的变异位点和相关基因,为理解果肉颜色这个复杂的表型性状提供思路,进一步为分子标记辅助育种和柚果肉颜色改良提供参考。【前人研究进展】果实颜色的形成是一个复杂的生物学过程[10],包含柚在内的柑橘属植物,果实着色主要涉及到叶绿素、类胡萝卜素和花青素3大类,3类色素化合物的成分和比例是决定果实呈色的关键指标[11-13]。在柚类品种中,除紫皮柚[14-15]外,鲜有果实在成熟过程中存在花青素沉着现象,绝大多数果实颜色形成受叶绿素和类胡萝卜素的影响。类胡萝卜素的合成、降解以及在质体中的储藏水平均会影响类胡萝卜素的积累,进而导致不同果肉颜色的呈现[16]。随着测序技术的不断发展、测序成本的逐渐降低,对于果实着色的研究已从对合成通路上结构基因的功能解析发展到挖掘更多调控因子。Minamikawa等[17]通过二代测序分析了787份柑橘材料,发现在chr 2、chr 4、chr 6上均存在与果皮和果肉颜色显著关联的SNP位点,且与仅有17 kb的距离。王霞[18]利用191份柚类资源对果肉颜色进行了GWAS关联分析,确定在7号染色体上存在显著关联位点。【本研究切入点】果实颜色代谢相关的调控网络和关键基因有待研究。【拟解决的关键问题】通过Fst分析及GWAS关联分析方法鉴定与柚果肉颜色形成显著关联的SNP位点,确定果肉颜色形成的关键基因,为柚的品质改良奠定基础。
1 材料与方法
试验于2021年5月至2023年2月在西南大学/中国农业科学院柑桔研究所进行,所用100份柚类种质材料均来自于国家柑橘种质资源圃(重庆),材料名称见表1。

表1 100份柚果肉颜色统计
续表1 Continued table 1
1.1 果肉颜色鉴定
在果实成熟期,每份材料采集3个大小均匀的柚果进行颜色统计并拍照记录,白色果肉表型记为A,红色果肉表型记为B。使用colormeter pro手持色差仪测定果实横切面L(亮度)、a(从红色至绿色的范围)、b(从红色至绿色的范围)数值,每份材料重复测定3次,每个横切面重复测定4次。运用饱和度C=(a2+b2)1/2,色差值CI=1000×a/(L×C)对果肉颜色进行评价。
1.2 全基因组测序
将采集的100份柚春梢叶片用75%酒精擦拭干净,采用改良CTAB法提取基因组DNA,用1.0%的琼脂糖凝胶电泳和Nanodrop ND_1000分光光度计进行DNA浓度和质量检测。将所有通过质检的样品委托北京诺禾致源生物信息科技有限公司进行GBS简化基因组测序。测序得到的原始图像数据经base calling转化为序列数据raw data,raw data经fastp软件质控后比对到柚参考基因组(https:// www.citrusgenomedb.org/analysis/187),采用GATK- HaplotypeCaller和Samtools软件提取测序结果中的多态性SNP位点,设置dp 5、Miss 0.2、maf 0.01的条件参数进行过滤,最后得到包含100份材料高质量SNP数据的vcf文件用作后续分析。
1.3 主成分分析
用GCTA软件(http://cnsgenomics.com/software/ gcta/)计算特征值及特征向量进行主成分分析,R软件中的ggplot2程序包将结果可视化。
1.4 优异变异位点筛选
遗传分化指数Fst可用于评价群体间的分化程度[19]。将测序得到的vcf文件通过plink软件转换为二进制bed文件,根据红肉柚和白肉柚的表型分组文件逐位点计算群体间的遗传分化系数Fst,利用R软件中的ggplot2程序包进行可视化,合理设置阈值,筛选Fst指数较大的位点用于后续分析。
全基因组关联分析GWAS能够将表型数据和高通量测序结果关联分析,从而鉴定与目标性状相关的变异位点。使用GEMMA软件(https://gitee. com/khjia/GEMMA)中的GLM模型进行柚果肉颜色关联分析,根据分析结果合理设置阈值,统计-log10(P)较大的位点并利用qqman程序包绘制manhattan-plot。
1.5 候选基因预测及初步验证
通过Fst计算结果以及GWAS关联分析结果两种方式筛选优异变异位点,将变异位点侧翼25 kb序列用柑橘基因组数据库(https://www.citrusgenomedb. org/)和蛋白质数据库(https://www.uniprot.org/)进行基因注释,结合前人研究以及基因功能注释结果筛选出候选基因。为验证候选基因与果肉颜色形成的关系,将候选基因的CDS序列上传到NCBI(https:// www.ncbi.nlm.nih.gov/tools/primer-blast/index.cgi?LINK_LOC=BlastHome)网站进行荧光定量PCR(qPCR)引物设计,引物序列见表2。随机选择4份白肉柚(卫寺蜜柚、通贤柚、左氏柚、垫江白柚)和4份红肉柚(垫江红心杂种3号、平阳柚、菊花芯柚、江津红心柚)用作验证材料。采集果实膨大期(S1)、转色期(S2)、成熟期(S3)3个时期的柚果,对其颜色参数a、b及色差值CI进行统计,取适量果肉于液氮中充分研磨,用Biospin多糖多酚植物总RNA提取试剂盒提取果肉RNA,用Hifair® III 1st Strand cDNA Synthesis SuperMix for qPCR逆转录试剂将RNA逆转录成cDNA,用Hieff UNICON® Universal Blue qPCR SYBR Green Master Mix进行荧光定量。选择为内参基因,用2-△△CT计算候选基因相对表达水平,GraphPad Prism软件作图分析。
表2 qPCR相关引物核苷酸序列
2 结果
2.1 果肉颜色鉴定
经过调查统计,100份柚类种质资源中有58份材料为白肉柚,42份材料为红肉柚。色差测定结果见表3,白肉柚色差指数CI介于-3.27—65.00,颜色变异系数为73.21%;红肉色差指数CI介于18.37—65.00,颜色变异系数为27.38%。以饱和度C为横坐标,色差值CI为纵坐标绘制散点图(图1),可见白肉群体被聚为一类,红肉群体聚集为一类。
2.2 主成分分析
经过GBS测序后获得大量SNP位点,过滤筛选后共得到包含100份材料的121 726个高质量SNP数据。基于过滤后的SNP位点主成分分析结果显示(图2),100份材料中红肉和白肉群体没有明显的群体分层现象,表明红肉和白肉柚类品种群之间并不存在明显的遗传差异。若群体之间产生分层,则说明群体间存在太多的遗传差异,则不利于性状的关联分析和基因定位。因而果肉颜色这个数量性状可以利用100份柚类自然资源群体进行表型和基因型的关联分析。
2.3 优异变异位点筛选
为挖掘白肉柚与红肉柚群体果肉颜色的变异情况,根据红肉柚(58)和白肉柚(42)表型分组用plink软件进行全基因扫描得到Fst值,阈值设置为0.4。Fst结果显示(图3),利用白肉和红肉两种表型鉴定到的遗传分化指数最大位点均分布在chr 7上,且数值达到0.4以上,而已有研究表明Fst指数大于0.25即说明群体间存在明显分化[19]。这表明白肉柚群体和红肉柚群体在该位点存在明显的分化现象,且与果肉颜色性状高度相关。高于阈值线的6个SNP位点可用于揭示表型性状的变异关系,详细信息见表4。
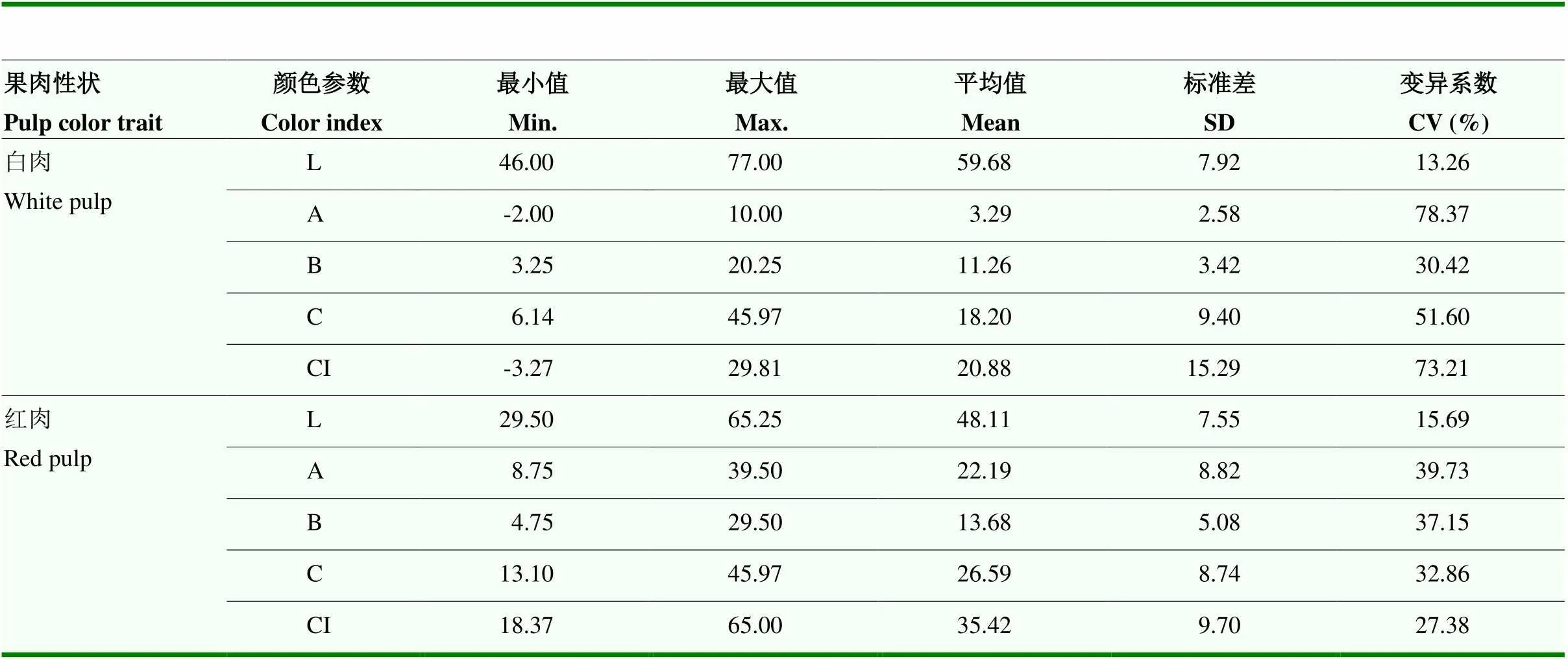
表3 100份柚类种质果肉色差CI变化分布及分布特征
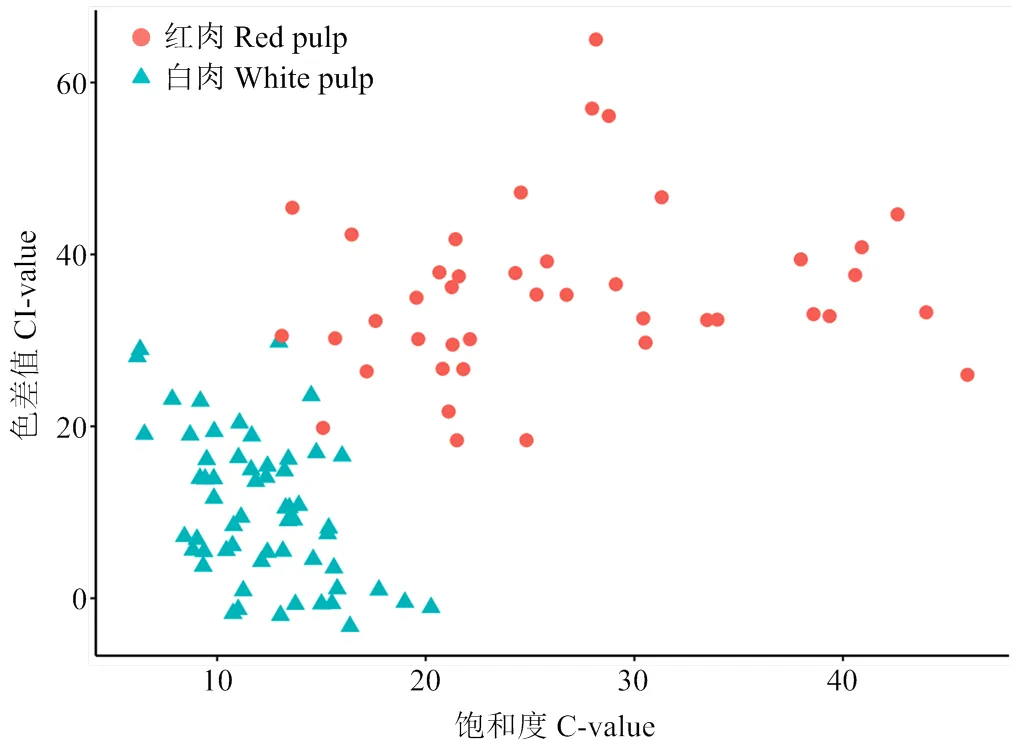
图1 100份柚类种质的果肉色差值散点图
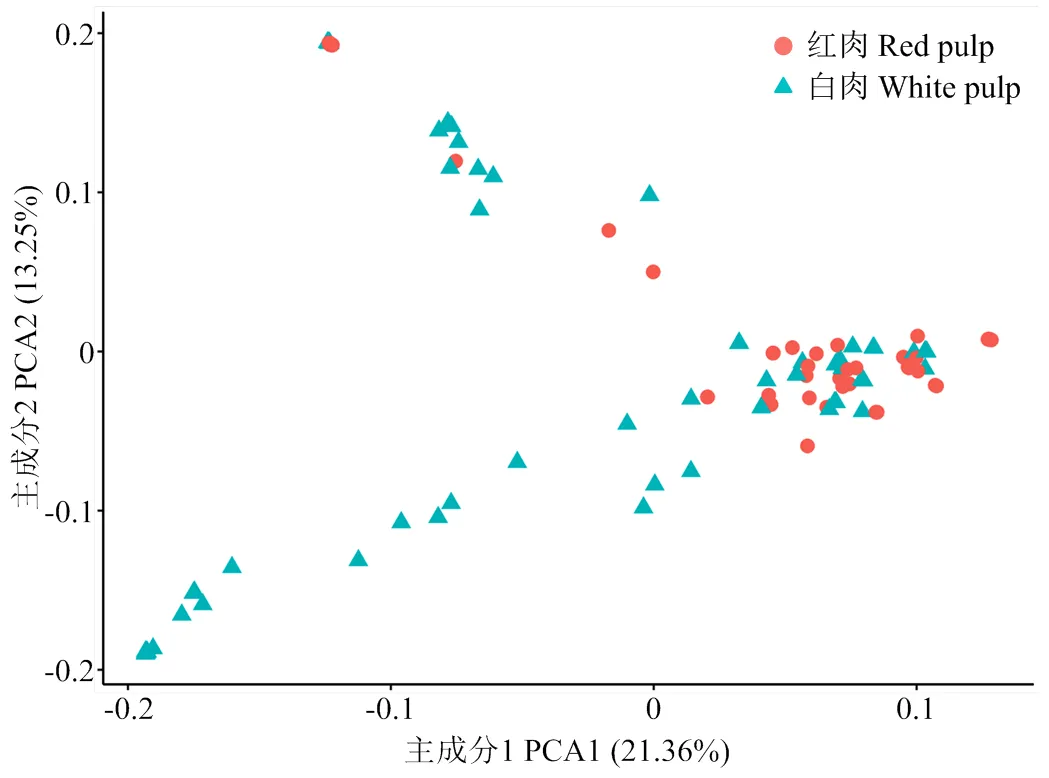
图2 100份柚主成分分析图
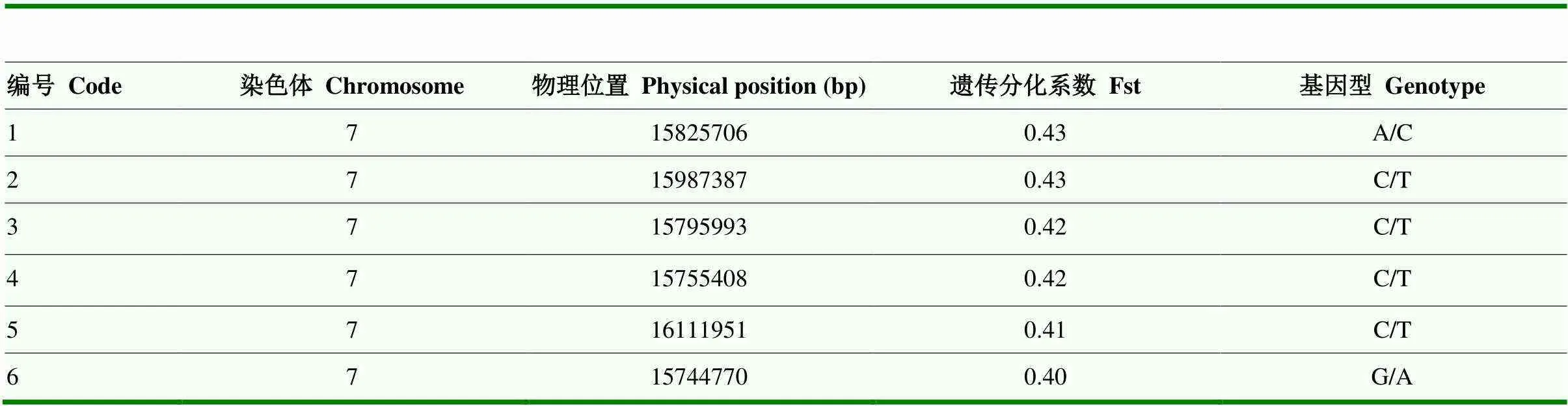
表4 Fst计算获得的6个SNP位点信息
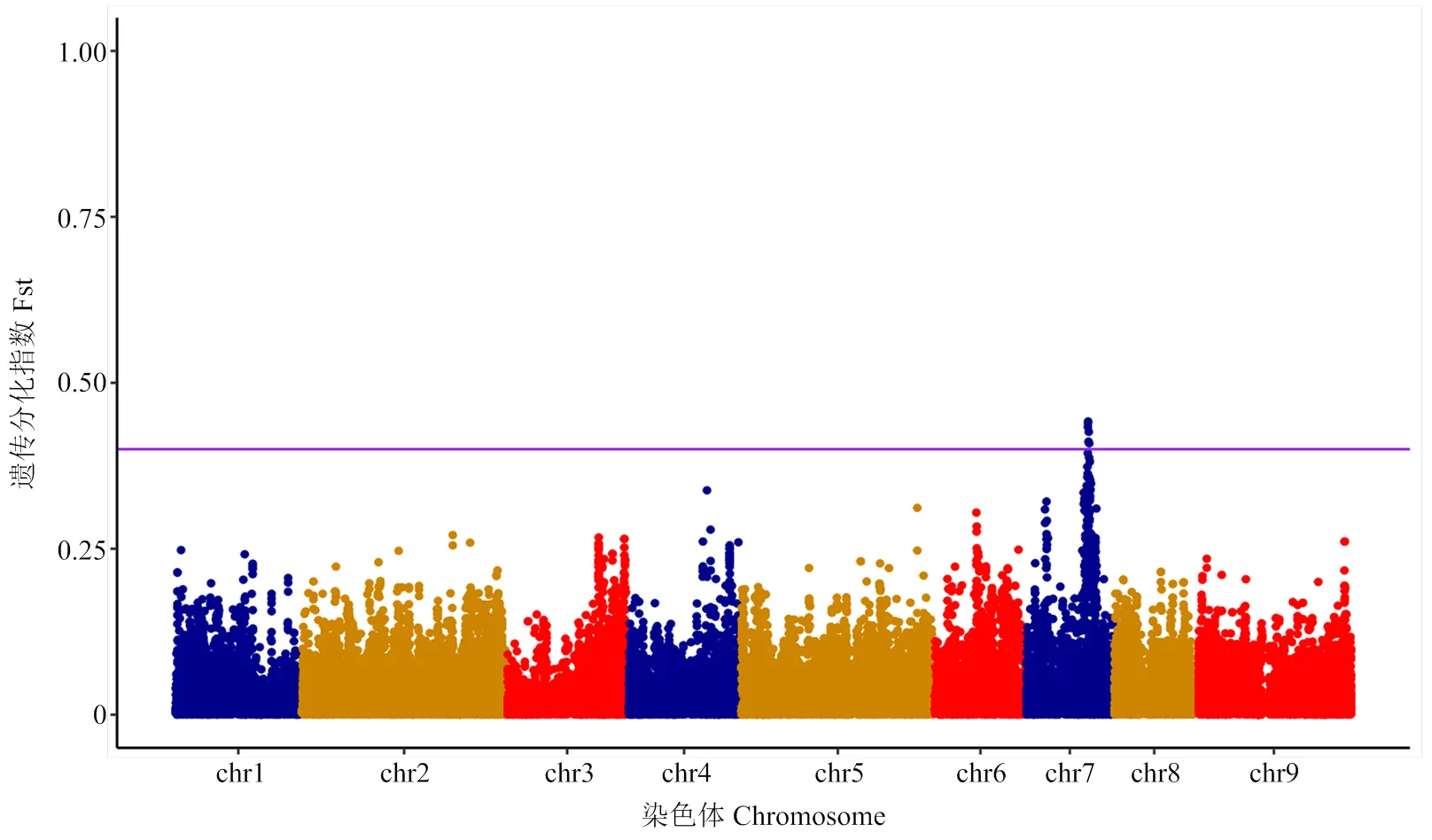
图3 白肉柚和红肉柚群体Fst分析
选用一般线性模型GLM进行GWAS关联分析,阈值设置为-log10(P)=9。曼哈顿图(图4-A)显示,与果肉颜色关联的位点共12个(表5),均位于7号染色体上,而Q-Q plot(图4-B)结果显示前半部分与观测值一致,后半部分微微上翘,说明高出期望值的位点可能与果肉颜色相关。
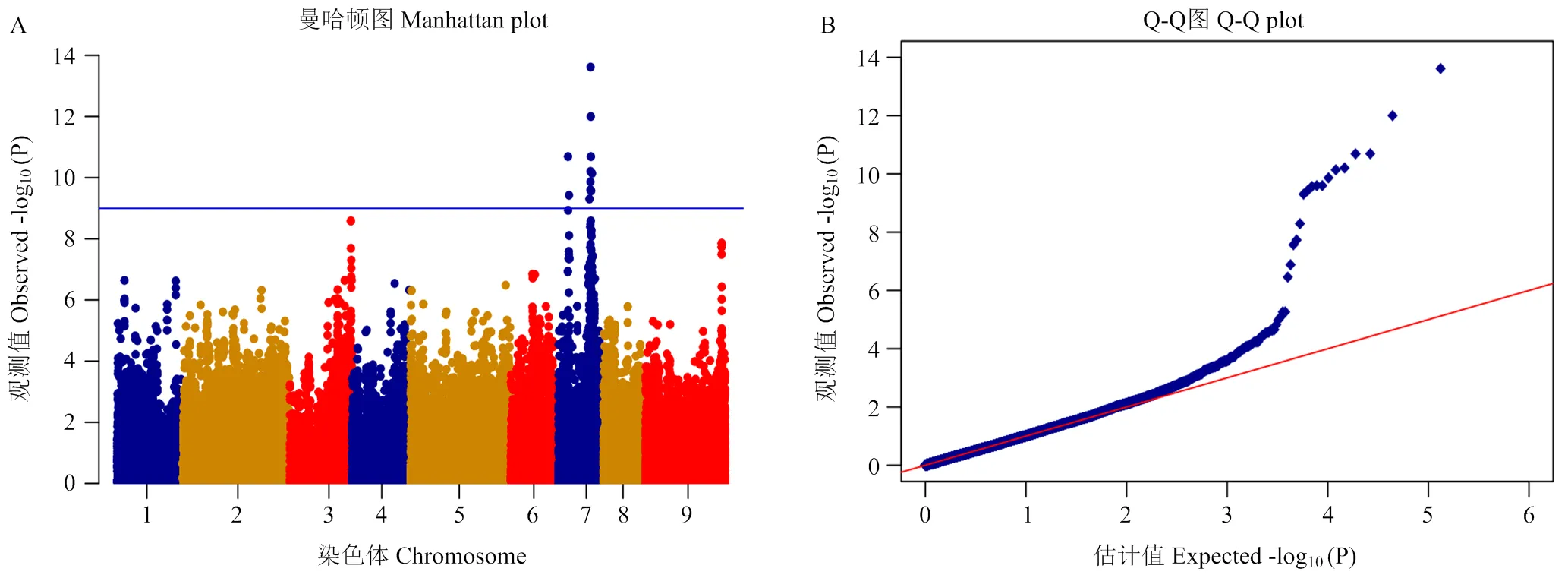
图4 利用100份柚果肉颜色进行GWAS关联分析
经过Fst遗传分化分析以及GWAS全基因组关联分析两种方式发现在chr 7上存在Fst>0.4且-log10(P)>9的相同变异位点6个,分别是SNP01: 15825706、SNP02: 15987387、SNP03: 15795993、SNP04: 15755408、SNP05: 15744770、SNP06: 16111951。6个SNP在的基因型分别是:A/C、C/T、C/T、C/T、G/A、C/T,其等位分析显示(图5),SNP01中基因型为AA的28份材料全部为白肉,基因型为CC的材料中果肉颜色呈红色的占87%,而基因型为AC的材料有65%果肉为白色;SNP02中基因型为CC和CT的大部分材料果肉颜色为白色,而基因型为TT的红肉材料有38份,占所有红肉材料的90%;SNP03基因型中CC基因型只有2份材料为白肉,CT和TT基因型则只有少数材料为红肉;SNP04中基因型为CT和TT的材料绝大部分果肉为白色,而基因型为CC的材料中,有82%为红色;SNP05中基因型为GG的材料果肉全部为白色,而GA基因型材料中,95%果肉为白色,AA基因型中有41份材料为红肉,仅有1份红肉材料非AA基因型;SNP06中CC基因型材料全部为白色果肉,CT基因型材料中有84%为白肉,TT基因型材料中有39份材料果肉呈红色,占所有红肉材料比例的93%。以上结果表明,6个SNP与果肉颜色存在不同程度的关联,与其关联的候选基因可能参与果肉颜色形成。
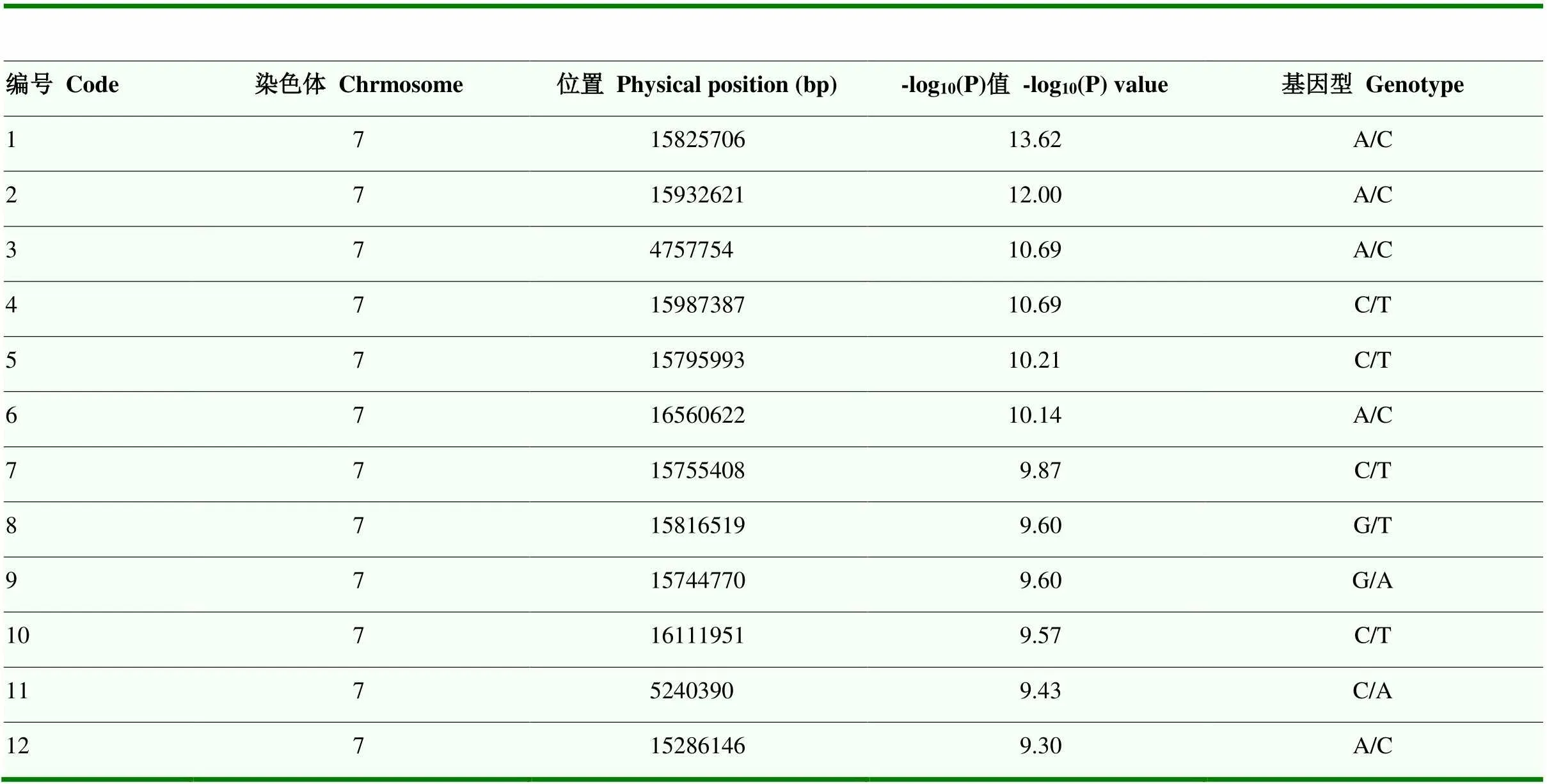
表5 GWAS分析获得的12个SNP位点信息
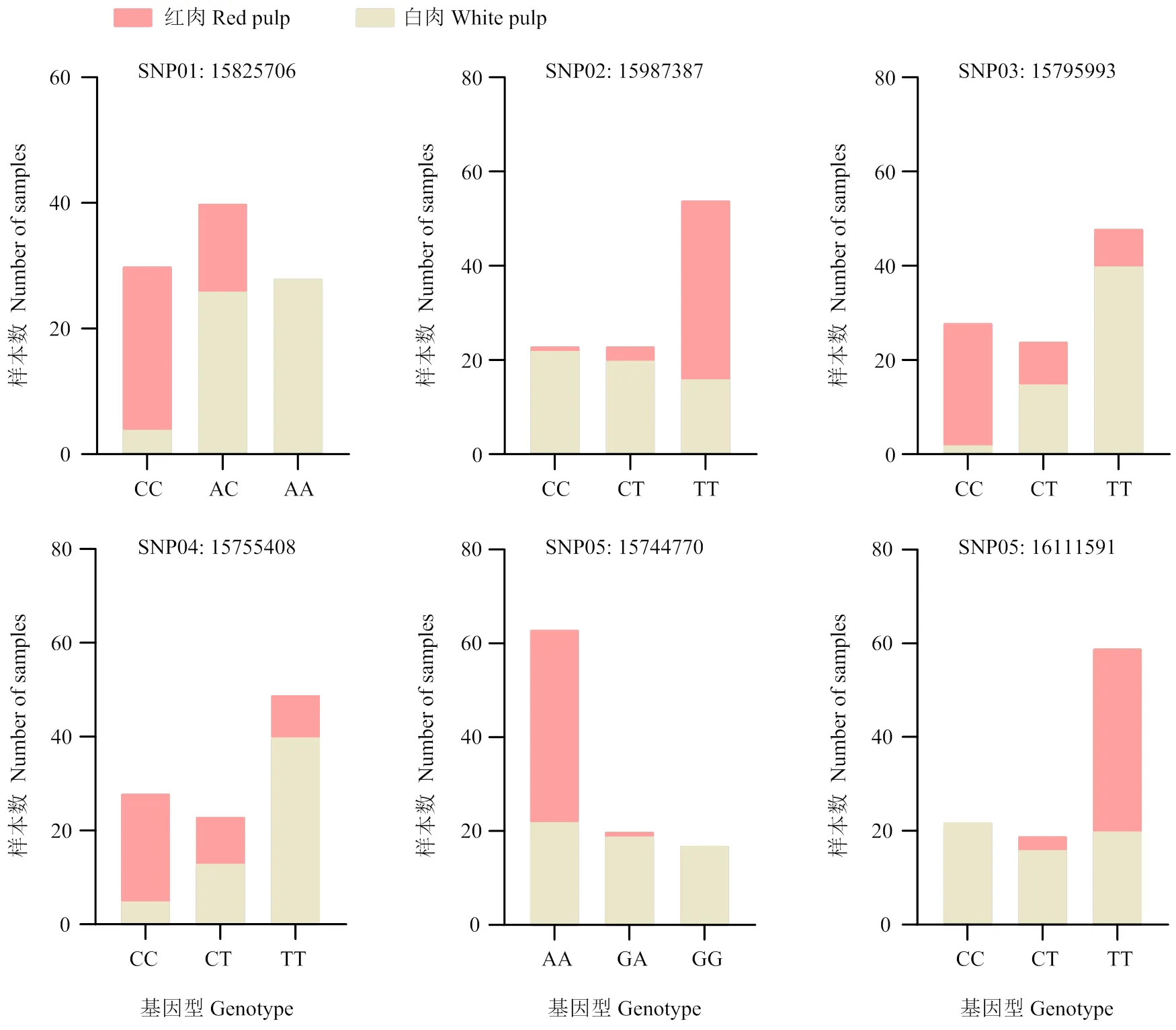
图5 100份柚类种质6个SNP基因型的分析
2.4 候选基因预测及初步验证
根据类胡萝卜素合成代谢途径相关基因的调控研究及变异位点临近基因的功能注释信息,筛选到14个基因作为果肉颜色形成的候选基因(表6)。为进一步验证候选基因是否参与果肉颜色形成,分别选取4个白肉柚材料和4个红肉柚材料3个发育时期的果实进行荧光定量分析,果肉呈色情况及颜色指数变化分别见图6和图7,随着果实发育成熟,白肉和红肉果肉颜色差异逐渐明显,白肉柚在整个发育过程其a值和色差值变化不大,而红肉柚的a值和色差值随果肉颜色加深而显著升高。
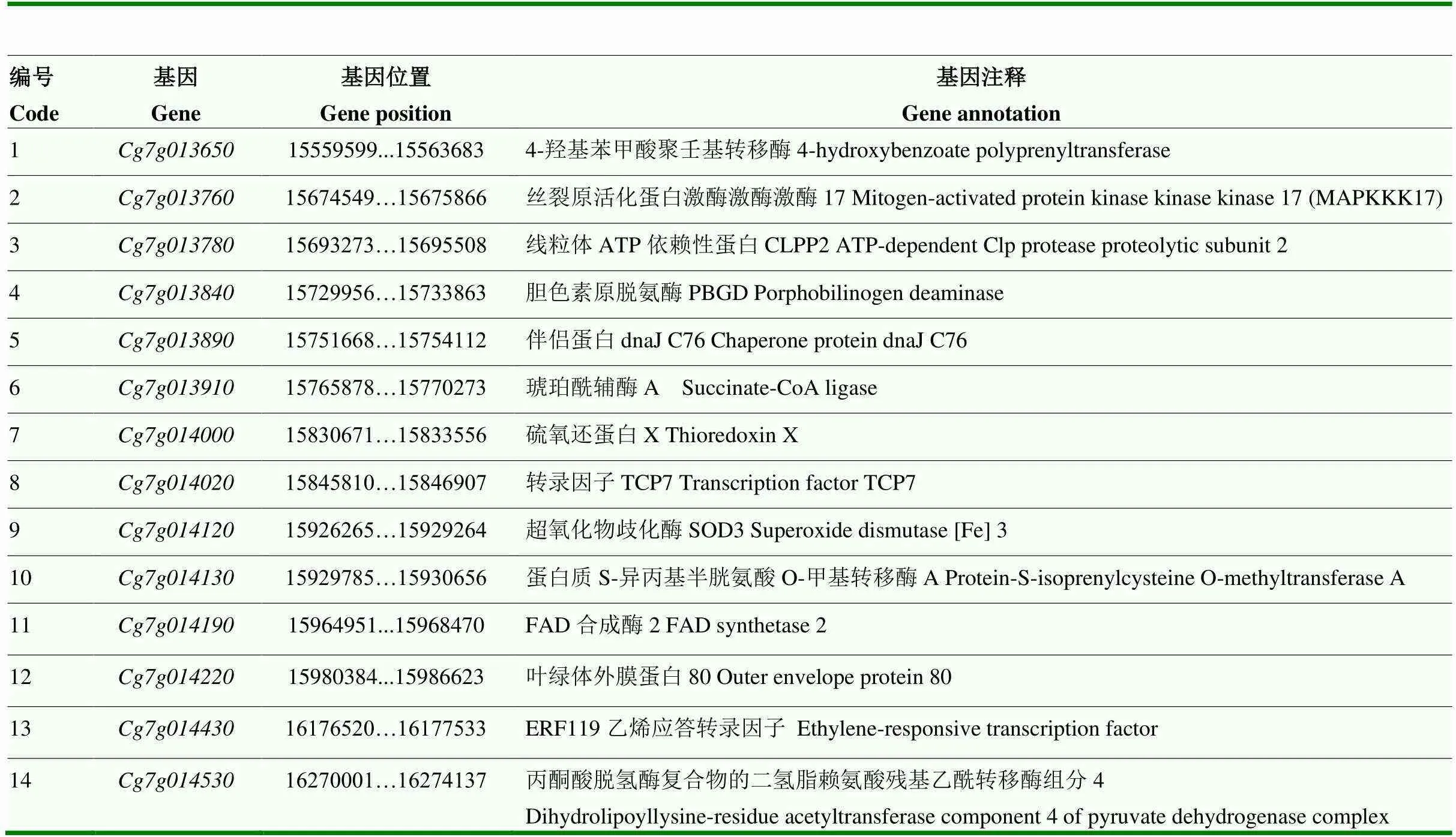
表6 14个候选基因注释信息
将14个基因在柚类果实的不同发育时期进行荧光定量分析(图8)。在白肉品种总体呈现出先增后减的趋势,尤其在S2时期表达量较高,而在4个红肉材料中无明显表达差异。和随着果实转色成熟,大体呈现出先增高后降低的趋势,在S2及S3时期红肉材料相对白肉材料平均相对表达更高,在S2时期白肉的‘卫寺蜜柚’和‘左氏柚’的表达量显著提高。在4个白肉柚中呈现先增高后下降的趋势,在4个红肉柚中呈现逐渐降低的趋势,总体上看,白肉柚材料的表达量相对较高。在S2、S3时期表达量较高,但在两种果肉表型材料中无明显表达差异。在S1时期的白肉材料中表达量显著低于红肉材料,总体来看,该基因在两种果肉颜色柚果中无明显差异。在成熟过程中总体呈现先上升后下降的趋势,但在两种颜色果肉材料中表达趋势无明显规律。在红肉品种中的表达量均很低。在白肉材料中表达高于红肉材料,在S1时期及S3时期差异更显著。大体呈现先增后减的规律,但在两种果肉表型材料中的表达趋势无明显规律。在试验材料中均呈现先增后减的表达规律,且在S1、S2、S3三个时期白肉品种的表达量显著高于红肉品种。在两种颜色果肉材料中表达趋势无明显规律。在S1时期白肉中的表达量较高于红肉,但总体来看,在整个发育过程,该基因在两种材料中无明显的表达差异。在两种颜色果肉材料中表达趋势无明显规律。综上,推测、、、、参与果肉颜色形成。
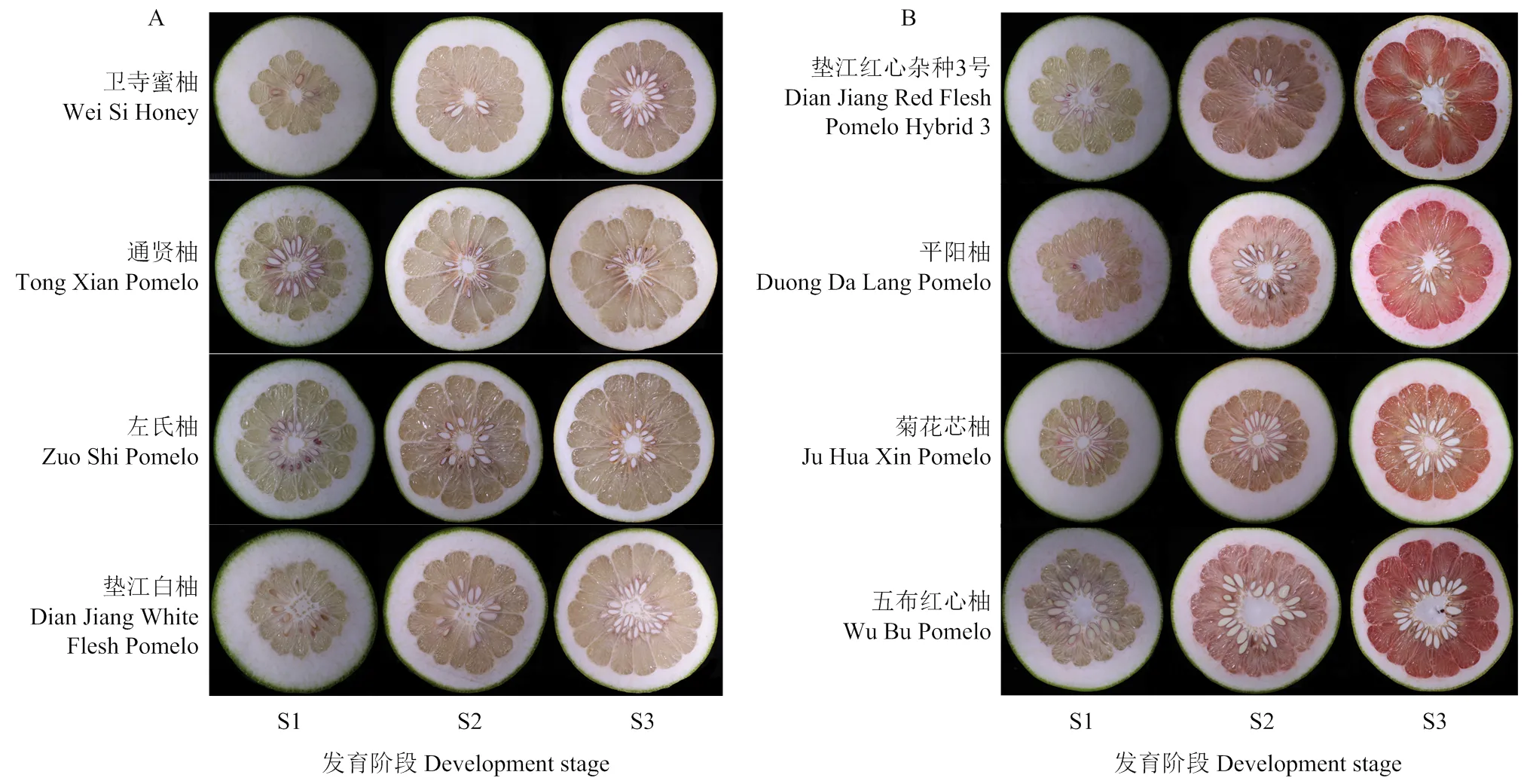
S1:膨大期;S2:转色期;S3:成熟期。下同 S1: The expansion period; S2: The turning period; S3: The mature period. The same as below
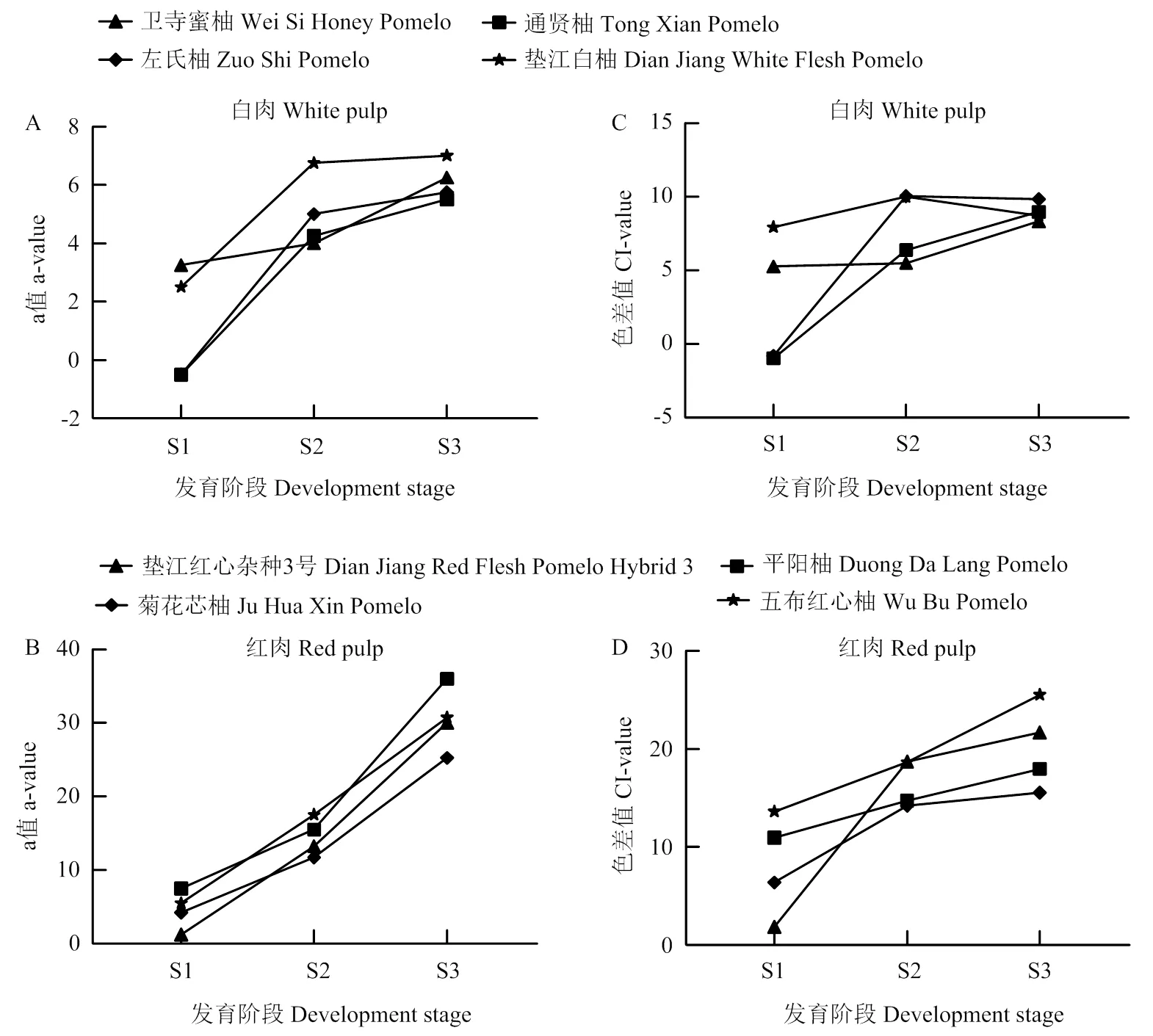
图7 候选基因验证所用材料果肉颜色参数变化
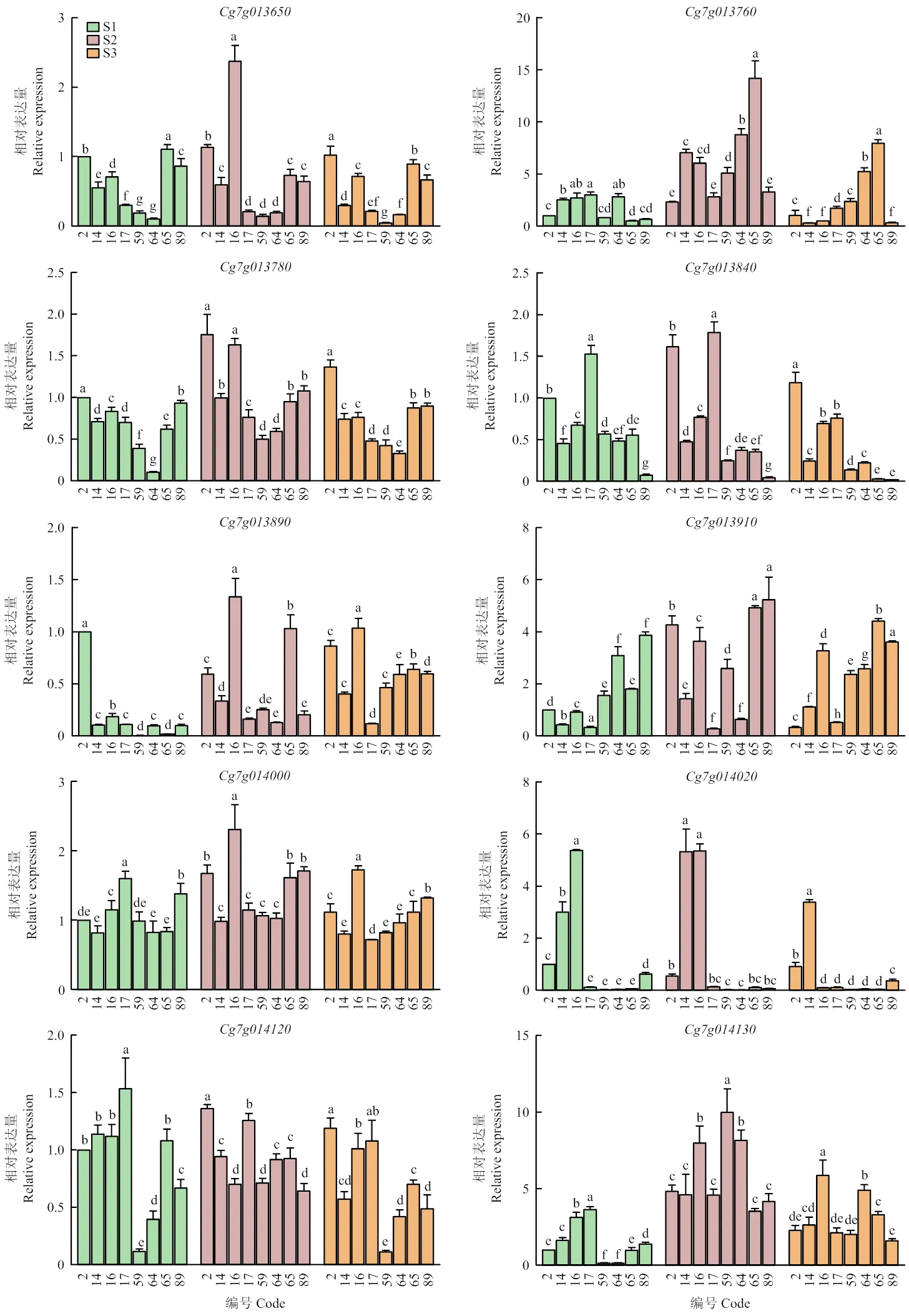
每个基因均以‘卫寺蜜柚’S1时期的相对表达量为1。编号所指的品种同表1
The expression level of each gene in Wei Si Honey pomelo S1 period was set as 1. The cultivar indicated by the code is the same as table 1
图8 14个基因在4个红肉和4个白肉柚种质果实不同发育时期的相对表达量
Fig. 8 The relative expression of 14 genes in 4 red and 4 white pulp of pomelo accessions at different fruit development stages
3 讨论
我国是柚生产和消费的大国,果实品质是柚在市场上竞争的重要因素[20]。果肉颜色不仅是柚重要的外观品质,也是人们评判其营养价值的重要参考指标[21]。在现有的柚类群体中,产生了众多红色果肉的表型变异品种,因其亮丽的颜色和丰富的营养价值受到消费者青睐和育种工作者的关注,因此,丰富果肉颜色对于柚的品种改良具有非常重要的意义。
3.1 群体结构分析
本研究从果肉色差和GBS测序结果分析了100份柚类种质的红肉和白肉两种呈色现象,并对其进行了群体结构划分。根据果肉色差测定结果可见白肉柚和红肉柚存在明显的聚类现象;而利用测序得到的SNP标记数据进行主成分分析发现,白肉柚和红肉柚群体结构较为混杂,没有明显的分层现象,可能是本研究所用SNP数据较多,无法将两个群体显著地区分。
3.2 变异位点分析
果肉颜色作为一个复杂的品质性状,受外界条件和遗传因子综合作用[22]。尽管果肉呈色的主要色素类胡萝卜素的代谢通路已非常清晰,但作为一个由多个基因共同决定的性状,类胡萝卜素的积累不仅受其结构基因影响,其他激素代谢以及转录调控等均有可能影响类胡萝卜素相关基因的表达进而影响色素的积累[23]。在前人的研究中,利用191份柚共定位了4个SNP位点与果肉颜色相关,其中2号染色体上1个,7号染色体上2个,9号染色体上1个[18]。邬庆江[24]通过白肉柚和红肉柚杂交父母本及子代群体对关联位点进行验证,但发现经过BSA重测序的结果与王霞的结果无重叠关联区间。基于此,利用自然群体对于果肉颜色表型进行关联位点验证和候选基因挖掘仍显得十分重要。本研究通过红肉和白肉两种材料共定位到位于7号染色体上的6个SNP位点,与王霞[18]结果基本相符,且在变异位点上游发现直接参与类胡萝卜素代谢的基因。通过对6个变异位点的等位变异分析发现,这些位点的不同基因型可以用于果肉呈色的判断。
3.3 候选基因预测
对显著关联位点邻近区域进行注释后筛选了14个基因作为候选基因,经过基因表达量分析发现,(促分裂原活化蛋白激酶激酶激酶MAPKKK17)、(胆色素原脱氨酶PBGD)、(转录因子TCP7)、(超氧化物歧化酶SOD3)、(FAD合成酶2)5个基因的表达模式与果肉着色存在显著关联。MAPKKK17是蛋白质磷酸化信号通路中的重要激酶,参与植物生物和非生物胁迫[25],研究证明MAPKKK17级联途径参与ABA的信号转导[26],而类胡萝卜素的降解产物可作为ABA合成的前体物质[27],结合定量分析结果,发现MAPKKK17在红肉材料中较白肉材料高表达,推测其参与了果肉颜色的形成。果肉的呈色由叶绿素和类胡萝卜素等的含量和成分决定,叶绿素和类胡萝卜素之间存在相互协调影响的关系[28],与叶绿素合成、降解相关基因的表达对类胡萝卜素的积累能产生一定影响[29]。胆色素原脱氨酶PBGD位于叶绿素合成通路上游,是叶绿素合成途径中的关键酶之一[30]。在本研究中,PBGD所编码的基因在果实成熟过程中的表达量先增后减,且白肉组织中表达量相对红肉较高,推测其通过控制叶绿素的合成影响了叶绿素与类胡萝卜素的占比关系进而影响到果实着色。TCP转录因子是一类参与植物众多生长发育过程的转录因子,前人发现参与柑橘果实成熟过程,超表达番茄植株较野生型植株转色提前[31-32]。本研究中,在白肉材料中较红肉材料高表达,初步判断其参与了果实着色过程。超氧化物歧化酶具有抗氧化作用,研究表明,拟南芥中SOD2和SOD3可充当叶绿体活性氧消除剂,介导叶绿体的早期发育[33]。在白肉中3个时期较红肉材料均存在高表达现象,推测其参与了果实颜色的形成。FAD还原酶涉及到植物体内多种氧化还原反应,而研究证明FAD作为CRISTO、LCYB、LCYE、ZEP等多种酶的辅助因子,在色素的积累过程中发挥作用,低含量的FAD更有利于果实的着色[33-34]。本研究中,在4个白肉材料中表达量较高,而在红肉材料中表达量较低,推测其通过影响类胡萝卜素的积累进而影响果肉颜色。
4 结论
通过GWAS关联分析以及Fst方式鉴定到6个与果肉颜色显著关联的SNP位点,通过基因表达量分析筛选到5个与呈色有关的基因,分别是(促分裂原活化蛋白激酶激酶激酶MAPKKK17)、(胆色素原脱氨酶PBGD)、(转录因子TCP7)、(超氧化物歧化酶SOD3)、(FAD合成酶2),初步推测这些基因参与了柚果肉颜色的形成。
[1] YAN J, LIU J, XIONG Y B, QIN W, TANG C. Identification of the geographical origins of pomelos using multielement fingerprinting. Journal of Food Science, 2015, 80(2): C228-C233.
[2] 张太平, 彭少麟. 柚的起源、演化及分布初探. 生态学杂志, 2000, 19(5): 58-61, 66.
ZHANG T P, PENG S L. Introduction to the origin and evolution of pomelo and ItsDistribution in China. Chinese Journal of Ecology, 2000, 19(5): 58-61, 66. (in Chinese)
[3] JIANG Q H, YE J L, ZHU K J, WU F F, CHAI L J, XU Q, DENG X X. Transcriptome and co-expression network analyses provide insights into fruit shading that enhances carotenoid accumulation in pomelo (). Horticultural Plant Journal, 2022, 8(4): 423-434.
[4] KATO M, IKOMA Y, MATSUMOTO H, SUGIURA M, HYODO H, YANO M. Accumulation of carotenoids and expression of carotenoid biosynthetic genes during maturation in citrus fruit. Plant Physiology, 2004, 134(2): 824-837.
[5] 张太平, 彭少麟, 王峥峰, 陈碧琛. 柚类种质资源研究与保护概况. 生态科学, 2001, 20(3): 8-13.
ZHANG T P, PENG S L, WANG Z F, CHEN B C. Introduction to the study and protection of pomelo germplasm. Eological Science, 2001, 20(3): 8-13. (in Chinese)
[6] GILL K, KUMAR P, KUMAR A, KAPOOR B, SHARMA R, JOSHI A K. Comprehensive mechanistic insights into the citrus genetics, breeding challenges, biotechnological implications, and omics-based interventions. Tree Genetics & Genomes, 2022, 18(2): 9.
[7] WU G A, PROCHNIK S, JENKINS J, SALSE J, HELLSTEN U, MURAT F, PERRIER X, RUIZ M, SCALABRIN S, TEROL J,. Sequencing of diverse mandarin, pummelo and orange genomes reveals complex history of admixture during citrus domestication. Nature Biotechnology, 2014, 32(7): 656-662.
[8] YU Y, CHEN C X, GMITTER F G. QTL mapping of mandarin () fruit characters using high-throughput SNP markers. Tree Genetics & Genomes, 2016, 12(4): 77.
[9] BERNET G P, FERNANDEZ-RIBACOBA J, CARBONELL E A, ASINS M J. Comparative genome-wide segregation analysis and map construction using a reciprocal cross design to facilitate citrus germplasm utilization. Molecular Breeding, 2010, 25(4): 659-673.
[10] MA G, ZHANG L C, KATO M. Molecular regulation of carotenoid biosynthesis in citrus juice sacs: New advances. Scientia Horticulturae, 2023, 309: 111629.
[11] FUJII H, NONAKA K, MINAMIKAWA M F, ENDO T, SUGIYAMA A, HAMAZAKI K, IWATA H, OMURA M, SHIMADA T. Allelic composition of carotenoid metabolic genes in 13 founders influences carotenoid composition in juice sac tissues of fruits among Japanese citrus breeding population. PLoS ONE, 2021, 16(2): e0246468.
[12] IKOMA Y, MATSUMOTO H, KATO M. Diversity in the carotenoid profiles and the expression of genes related to carotenoid accumulation among citrus genotypes. Breeding Science, 2016, 66(1): 139-147.
[13] 徐娟. 几个柑桔产区果实色泽评价及红肉脐橙(L.cv.Cara cara)果肉呈色机理初探[D]. 武汉: 华中农业大学, 2002.
XU J. Evaluation of fruit color in severalproducing areas and preliminary study on the mechanism of pulp color ofL. cv. Cara cara [D]. Wuhan: Huazhong Agricultural University, 2002. (in Chinese)
[14] 袁悦. 血橙和紫皮柚果实花青苷积累响应光和低温的机制研究[D]. 武汉: 华中农业大学, 2019.
YUAN Y. Study on the mechanism of anthocyanin accumulation in blood orange and purple pomelo fruits in response to light and low temperature [D]. Wuhan: Huazhong Agricultural University, 2019. (in Chinese)
[15] HUANG D, WANG X, TANG Z Z, YUAN Y, XU Y T, HE J X, JIANG X L, PENG S, LI L, BUTELLI E, DENG X X, XU Q. Subfunctionalization of the Ruby2-Ruby1 gene cluster during the domestication of citrus. Nature Plants, 2018, 4(11): 930-941.
[16] SUN T H, TADMOR Y, LI L. Pathways for carotenoid biosynthesis, degradation, and storage. Methods in Molecular Biology, 2020, 2083: 3-23.
[17] MINAMIKAWA M F, NONAKA K, KAMINUMA E, KAJIYA- KANEGAE H, ONOGI A, GOTO S, YOSHIOKA T, IMAI A, HAMADA H, HAYASHI T, MATSUMOTO S, KATAYOSE Y, TOYODA A, FUJIYAMA A, NAKAMURA Y, SHIMIZU T, IWATA H. Genome-wide association study and genomic prediction in citrus: potential of genomics-assisted breeding for fruit quality traits. Scientific Reports, 2017, 7: 4721.
[18] 王霞. 柑橘精细定位平台构建及其在多胚和色泽芽变研究中的应用[D]. 武汉: 华中农业大学, 2018.
WANG X. Construction of citrus fine positioning platform and its application in the study of multi-embryo and color bud change [D]. Wuhan: Huazhong Agricultural University, 2018. (in Chinese)
[19] WRIGHT S. The genetical structure of populations. Annals of Eugenics, 1951, 15(4): 323-354.
[20] 周蕊. 我国柚类市场与产业调查分析报告. 农产品市场, 2021(24): 47-49.
ZHOU R. Investigation and analysis report on China’s pomelo market and industry. Agricultural Products Market, 2021(24): 47-49. (in Chinese)
[21] 张亚飞, 彭洁, 朱延松, 杨胜男, 王旭, 赵婉彤, 江东. 柑橘CCD基因家族鉴定及Cc4a对果肉颜色的影响. 中国农业科学, 2020, 53(9): 1874-1889. doi: 10.3864/j.issn.0578-1752.2020.09.014.
ZHANG Y F, PENG J, ZHU Y S, YANG S N, WANG X, ZHAO W T, JIANG D. Genome wide identification of CCD gene family inand effect ofon the color offlesh. Scientia Agricultura Sinica, 2020, 53(9): 1874-1889. doi: 10.3864/j.issn.0578- 1752.2020.09.014. (in Chinese)
[22] LADO J, ALÓS E, MANZI M, CRONJE P J R, GÓMEZ-CADENAS A, RODRIGO M J, ZACARÍAS L. Light regulation of carotenoid biosynthesis in the peel of mandarin and sweet orange fruits. Frontiers in Plant Science, 2019, 10: 1288.
[23] LU P J, WANG S S, GRIERSON D, XU C J. Transcriptomic changes triggered by carotenoid biosynthesis inhibitors and role ofphosphate transporter 4;2 (CsPHT4;2) in enhancing carotenoid accumulation. Planta, 2019, 249(1): 257-270.
[24] 邬庆江. 柚果肉色泽和果形调控基因的克隆与鉴定[D]. 武汉: 华中农业大学, 2021.
WU Q J. Cloning and identification of genes regulating flesh color and fruit shape in pummelo [D]. Wuhan: Huazhong Agricultural University, 2021. (in Chinese)
[25] ROMERO-HERNANDEZ G, MARTINEZ M. Opposite roles of MAPKKK17 and MAPKKK21 againstin. Frontiers in Plant Science, 2022, 13: 1038866.
[26] DANQUAH A, DE ZÉLICOURT A, BOUDSOCQ M, NEUBAUER J, FREI DIT FREY N, LEONHARDT N, PATEYRON S, GWINNER F, TAMBY J P, ORTIZ-MASIA D, MARCOTE M J, HIRT H, COLCOMBET J. Identification and characterization of an ABA- activated MAP kinase cascade in. The Plant Journal, 2015, 82(2): 232-244.
[27] TAN B C, JOSEPH L M, DENG W T, LIU L J, LI Q B, CLINE K, MCCARTY D R. Molecular characterization of the9-cis epoxycarotenoid dioxygenase gene family. The Plant Journal, 2003, 35(1): 44-56.
[28] XIE J, YAO S X, MING J, DENG L L, ZENG K F. Variations in chlorophyll and carotenoid contents and expression of genes involved in pigment metabolism response to oleocellosis in citrus fruits. Food Chemistry, 2019, 272: 49-57.
[29] YUAN H, ZHANG J X, NAGESWARAN D, LI L. Carotenoid metabolism and regulation in horticultural crops. Horticulture Research, 2015, 2: 15036.
[30] LIM S H, WITTY M, WALLACE-COOK A D M, ILAG L I, SMITH A G. Porphobilinogen deaminase is encoded by a single gene inand is targeted to the chloroplasts. Plant Molecular Biology, 1994, 26(3): 863-872.
[31] LIN Y F, CHEN Y Y, HSIAO Y Y, SHEN C Y, HSU J L, YEH C M, MITSUDA N, OHME-TAKAGI M, LIU Z J, TSAI W C. Genome-wide identification and characterization ofgenes involved in ovule development of. Journal of Experimental Botany, 2016, 67(17): 5051-5066.
[32] 周延培. 柑橘TCP家族生物信息学分析及CsTCP1转录因子参与果实成熟的功能验证 [D]. 武汉: 华中农业大学, 2016.
ZHOU Y P. Bioinformatics analysis ofTCP family and functional verification of CsTCP1 transcription factor involved in fruit ripening [D]. Wuhan: Huazhong Agricultural University, 2016. (in Chinese)
[33] BYCHKOV I A, ANDREEVA A A, KUDRYAKOVA N, POJIDAEVA E S, KUSNETSOV V V. The role of PAP4/FSD3 and PAP9/FSD2 in heat stress responses of chloroplast genes. Plant Science, 2022, 322: 111359.
[34] YU Q J, SCHAUB P, GHISLA S, AL-BABILI S, KRIEGER- LISZKAY A, BEYER P. The lycopene cyclase CrtY from(formerly) catalyzes and FADred- dependent non-redox reaction. Journal of Biological Chemistry, 2010, 285(16): 12109-12120.
Genetic Variation Analysis and Candidate Genes Mining of Regulating Flesh Color in Pomelo
CHENG Li, YANG ShengNan, ZHU YanSong, WANG Xu, ZHAO WanTong, LI RenJing, LI Pei, YUAN ZhongJie, JIANG Dong
Citrus Research Institute of Southwest University, Chongqing 400712
【Objective】The flesh color is an important appearance and quality trait of pomelo varieties. The aim of this study was to excavate variation loci and genes significantly related to the flesh color of pomelo varieties, so as to lay a foundation for further understanding the flesh color development mechanism and marker-assisted breeding of pomelo varieties. 【Method】100 pomelo accessions were used to identify their flesh color with a color difference meter, and classified based on their phenotype results. The simplified genome sequencing of 100 pomelo accessions was carried out using GBS (genotyping-by-sequencing) technology. The eigenvalues and eigenvectors of the genotype data obtained from sequencing were calculated by GCTA software to analyze the population structure, and the genetic differentiation index (Fst) of two different flesh color populations were calculated using Plink 2.0 software. The GLM model in GEMMA software was adopted to perform genome-wide association analysis, and the variation loci significantly associated with color phenotype were identified for further allelic variation analysis. According to the citrus linkage disequilibrium (LD) distance, the candidate genes located in the 25 kb adjacent region around the variation loci that might be related to the forming of flesh color were screened out, four white and four red pomelo accessions were randomly selected to perform qPCR to obtain the candidate genes expression at different fruit development stages. 【Result】According to the phenotype of flesh color, 100 accessions were divided into two categories corresponding to white and red pomelo accessions, including 58 white flesh pomelos and 42 red flesh pomelos. Six SNP sites with Fstindex greater than 0.4 and -log10(P)>9 were screened out after Fst genetic differentiation analysis and GWAS genome-wide association analysis. The genotypes of six variation SNPs sites were extracted from 100 pomelos accessions and the genotypes of these SNPs could be used to predict the flesh color of pomelos. A total of 14 genes that function annotated as isoprene synthesis, plastid development, abscisic acid signal regulation, ethylene response related genes and transcription factors were screened and selected. Genes expression analysis showed that(mitogen-activated protein kinase kinase MAPKKK17),(chloroplast porphyrinogen deaminase),(transcription factor TCP7),(chloroplast superoxide dismutase SOD3),(FAD synthetase 2) were related to the forming of flesh color in pomelos. 【Conclusion】 A total of 6 SNP sites significantly associated with flesh color in pomelos accessions were identified by GWAS and Fst analysis. A total of 14 genes within the 25 kb regions around the mutation SNPs site were screened and annotated. The gene expression analysis showed that five genes might be involved in the forming of flesh color in pomelo accessions.
pomelo; flesh color; Fst; GWAS; candidate genes
2023-02-13;
2023-05-17
国家科技重点研发项目(2019YFD1001400)、柑橘种质资源精准鉴定项目(19211142)、西南大学实验技术研究项目(SYJ2023021)
程莉,E-mail:chengliak@163.com。通信作者江东,E-mail:jiangdong@cric.cn
10.3864/j.issn.0578-1752.2023.17.015
(责任编辑 赵伶俐)

