酱香大曲中高温放线菌的筛选及基因组解析
田浩杰,李豆南,2,*,邱树毅,周剑丽,龙则河,王珂佳,4,刘茂强,陈 杰,程 度,潘凤爽
(1.贵州大学酿酒与食品工程学院,贵州省发酵工程与生物制药重点实验室,贵州 贵阳 550025;2.酿酒生物技术及应用四川省重点实验室,四川 宜宾 644000;3.贵州省仁怀市天邦酿酒有限公司,贵州 仁怀 564500;4.贵州轻工职业技术学院轻工化工系,贵州 贵阳 550025)
“曲乃酒之骨”,酱香型高温大曲在发酵条件下富集了丰富的功能微生物,在生产过程中为整个酱酒酿造体系提供了微生物、酶系、风味及其前体物[1-3]。其独特的微生物群落结构对酱香型白酒的酒体风味形成有重要影响。研究表明,细菌类群在酱香大曲中具有举足轻重的作用[4],诸如厚壁菌门、变形菌门、拟杆菌门和放线菌门为酱香大曲中的优势细菌类群[5-6],其中,芽孢杆菌已经被证实与酱香风味产生密切相关,是乙酸乙酯、异戊醇、糠醇及吡嗪类物质等的重要来源[7-9]。
高温放线菌是一类革兰氏阳性、化能异养的微生物类群,能够在极端环境下生存和繁殖[10],能代谢产生酯化酶、淀粉酶、磷酸酶等多种酶和风味及其前体物[8]。该菌在酱香型白酒酿造体系中属于一类具有重要应用潜力的微生物资源,其在白酒酿造体系中的功能尚不清楚,有待进一步研究确定。近年来,随着微生物多样性分析技术的发展,越来越多的研究表明高温放线菌为高温大曲中的优势细菌类群[6,11-12],这也说明高温放线菌在酱香型白酒酿造体系中具有重要的潜在功能。
随着可培养技术的进步,针对酿造体系内高温放线菌未培养菌种资源的开发也逐步开展。王芙蓉[13]和白成松[14]等分别从酱香大曲和酒糟中分离鉴定了高温放线菌,并对其功能特性进行了初步研究,证实其与淀粉、纤维素、蛋白质的降解存在关联。本课题组在前期研究中也成功建立了一种从酱香大曲中分离、筛选高温放线菌的方法,从酱香型大曲中分离鉴定了多株高温放线菌,证实这些菌株具有良好的产吡嗪风味组分能力,并对其代谢产吡嗪的途径及机理进行解析[15-16]。这些工作的开展有助于发掘酱香大曲中高温放线菌的功能和应用价值,为后续高温放线菌功能特性的确定与挖掘奠定了基础。
基于课题组前期的工作,本研究对酱香型高温大曲中的高温放线菌进行分离筛选,结合多相分类学技术进行菌株鉴定,并利用第二代Illumina和第三代PacBio结合的测序技术对所筛选菌株进行全基因组测序,从基因组层面解析其潜在的代谢功能特性,分析该菌株代谢典型风味组分的潜在机理,旨在为今后继续深入解析高温放线菌类群微生物产风味物质,特别是四甲基吡嗪的生物合成代谢途径及机理提供参考。
1 材料与方法
1.1 材料与试剂
酱香大曲来源于贵州省茅台镇某酿酒企业。为保证样品的代表性,所有样品均在白酒酿造车间取粉碎后待投料的陈曲粉,并于曲堆中随机取20 个样品混匀包装,保存于4 ℃冰箱备用。
细菌基因组DNA提取试剂盒 美国Biomiga公司;制霉菌素、新生霉素、琼脂糖、Tris、EDTA-2Na北京索莱宝科技有限公司;聚合酶链式反应(polymerase chain reaction,PCR)引物 上海派森诺生物科技有限公司;D4030A dNTP、DRR20AM LATaqTM聚合酶 宝生物工程(大连)有限公司;061180琼脂糖 生工生物工程(上海)股份有限公司;其他化学试剂均为国产分析纯。
分离筛选培养基[17]:葡萄糖1 g、蛋白胨0.5 g、胰蛋白胨0.3 g、氯化钠0.5 g、复合维生素2.75 mg(包括VB1、VB2、VB3、VB5、VB6各0.5 mg,VB70.25 mg)、琼脂 20.0 g、蒸馏水1 000 mL,pH 7.2~7.4,121 ℃灭菌20 min。
ISP2培养基(用于菌株纯化、保藏):酵母膏4 g、麦芽汁10.0 g、葡萄糖4.0 g、琼脂20.0 g、蒸馏水1 000 mL,pH 7.0~7.2,121 ℃灭菌20 min。
液体培养基:葡萄糖10 g、小麦浸出物20 mL、氯化钠5 g、磷酸氢二钾4 g、磷酸二氢钾2 g、七水硫酸镁0.5 g、蛋白胨4 g、蒸馏水1 000 mL,pH 7.0~7.2,115 ℃灭菌30 min。
1.2 仪器与设备
高速冷冻离心机 美国贝克曼库尔特有限公司;核酸凝胶电泳仪、凝胶成像仪 德国耶拿分析仪器股份公司;S-3400N型扫描电子显微镜 日本日立公司;旋涡振荡器 江苏省海门市麒麟医用仪器厂;超净工作台 江苏净化设备有限公司;Illumina NovaSeq 5000测序平台、Pacbio Sequel第三代单分子实时测序系统美国Thermo公司。
1.3 方法
1.3.1 高温放线菌的分离筛选
参考李豆南等[15]的方法对酱香型高温大曲中的高温放线菌进行分离筛选。
1.3.2 菌种鉴定
1.3.2.1 菌株形态学观察鉴定
将分离到的菌株接种于ISP2固体培养基上,于45 ℃恒温培养箱中倒置培养3 d后分别从菌落形态、基内菌丝、气生菌丝颜色及可溶性色素产生情况等方面观察并记录实验结果,同时经菌株插片培养后,制片于扫描电子显微镜下观察其菌丝体及孢子结构特征,并根据《伯杰氏细菌鉴定手册》完成菌株形态学鉴定。
1.3.2.2 菌株温敏性分析
将菌株分别置于30、37、45、50、55、60 ℃培养箱中培养48 h,观察菌株生长状况。
1.3.2.3 菌株生理生化鉴定
分别设计明胶液化、碳源利用、牛奶凝固与胨化、淀粉水解、硝酸盐还原和纤维素降解等生理生化实验,观察菌株的生理特性。
1.3.2.4 菌株分子生物学鉴定
参考李豆南等[15]方法对分离筛选出的菌株进行分子生物学鉴定。
1.3.3 菌株全基因组测序及序列分析
1.3.3.1 菌株基因组DNA提取及测序文库构建
菌株FBKL4.014接种于100 mL液体培养基中,45 ℃培养24 h后,使用细菌基因组DNA提取试剂盒提取基因组DNA,操作步骤参照试剂盒说明书,并对抽提的样品进行检测。使用紫外分光光度计,分别在260 nm和280 nm波长处测定DNA的吸光度,并通过0.8%琼脂糖凝胶电泳检测提取DNA的质量。
1.3.3.2 全基因组测序完成图、组装及数据质控分析
菌株FBKL4.014的全基因组测序委托上海派森诺生物科技有限公司完成,提取的基因组经质检合格达到测序要求后,利用第二代Illumina NovaSeq和第三代PacBio Sequel结合的测序技术进行完成图测序。制备好的二代测序文库在Illumina HiSeq测序平台上进行双端测序(2×150 bp),测序深度433×,三代测序文库在PacBio Sequel测序平台上进行单分子测序。Illumina NovaSeq测序原始数据以双端(Paired-end)FASTQ格式保存,并对下机数据过滤,采用Adapter Removal去除3’端接头污染,采用SOAPec基于Kmer频率对所有原始reads进行质量校正。利用HGAP和Canu软件对PacBio下机数据进行拼装,并用二代的高质量数据通过pilon软件对三代contig结果进行校正,最终拼接得到完整序列。
1.3.3.3 基因预测及功能注释分析
利用Glimmer 3.02对基因组中的基因进行预测[18]。分别使用TRNAscan-SE[19]、Barrnap[20]、Rfam[21]等软件进行tRNA、rRNA和ncRNA预测。成簇的规律间隔短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPRs)通过CRISPR识别工具进行鉴定[22]。基因功能注释通过BLAST将菌株预测的基因序列结果分别与非冗余(Non-Redundant,NR)蛋白质数据库[23]、基因本体论[24](Gene Ontology,GO)、京都基因与基因组百科全书[25](Kyoto Encycolpedia of Gene and Genomes,KEGG)、直系同源聚类群[26](Cluster of Orthologous Groups,COG)、欧洲蛋白质数据库Swiss-Prot[27]、碳水化合物活性酶[28](Carbohydrate-Active Enzymes,CAZy)数据库进行比对,得到相应的基因功能注释结果,采用CGView(http://stothard.afns.ualberta.ca/cgview_server/)绘制基因组圈图。通过KEGG数据库对碳水化合物代谢和氨基酸代谢途径进行挖掘。
2 结果与分析
2.1 菌株分离筛选及鉴定
2.1.1 菌株的形态学特征
从酱香高温大曲中分离得到1 株纯种菌株FBKL4.014,根据菌落形态特征初步判断其可能归属于高温放线菌,并保藏于贵州省发酵工程与生物制药重点实验室。
菌株宏观形态学观察结果显示(图1A),该菌株长势良好,在培养基正反两面表现出的生长趋势不同,菌落总体呈缺刻圆状、突起、有较多皱褶、质地干燥致密,基内菌丝呈浅黄色,气生菌丝呈乳白色且较为发达,无可溶性色素产生,该菌株的菌落形态具有高温放线菌培养的典型特征。高温放线菌FBKL4.014的微观形态特征如图1B所示,结合《伯杰氏细菌鉴定手册》[29]分析,发现其菌丝体较短,形状不一且黏在一起呈簇状,呈圆球形的分生孢子直接着生于菌丝体上,分布于菌丝体两侧,其中有分支的孢子梗长度较短。
2.1.2 菌株的最适生长温度
表1结果表明,菌株FBKL4.014在45~50 ℃条件下生长最旺盛,在55~60 ℃高温范围内仍能够缓慢生长,37 ℃时该菌株能正常生长,但在30 ℃时不生长。Wang Xueshan等[30]和颜林春[31]的研究表明高温放线菌属大多可以生长在最高温度可达60~65 ℃的高温发酵环境中,因此,菌株FBKL4.014在耐热性方面与高温放线菌具有很高的相似度,而这也与其分离环境——酱香大曲的高温发酵环境密切相关。
2.1.3 菌株的生理生化特征
生理生化分析(表2)表明,高温放线菌FBKL4.014为革兰氏阳性菌,该菌株可将硝酸盐还原为亚硝酸盐,并能使牛奶凝固但不胨化,使明胶产生液化现象,同时该菌株能水解淀粉和纤维素,由此推测该菌株能够代谢产生淀粉酶和纤维素酶,具备将大分子原料降解为可发酵性糖的潜能,有助于推动整个酿造过程的顺利进行。碳源利用实验结果显示,该菌株能够有效利用葡萄糖、海藻糖、D-棉子糖、D-甘露醇和D-山梨醇等碳源进行生长代谢,对麦芽糖表现出弱利用特征,不能利用乳糖和肌醇两种碳源。此外,该菌株能够利用硫酸铵和磷酸氢二铵两种无机氮源和酪氨酸,对尿素表现出弱利用性,不能利用脯氨酸、苏氨酸、谷氨酸和甘氨酸。
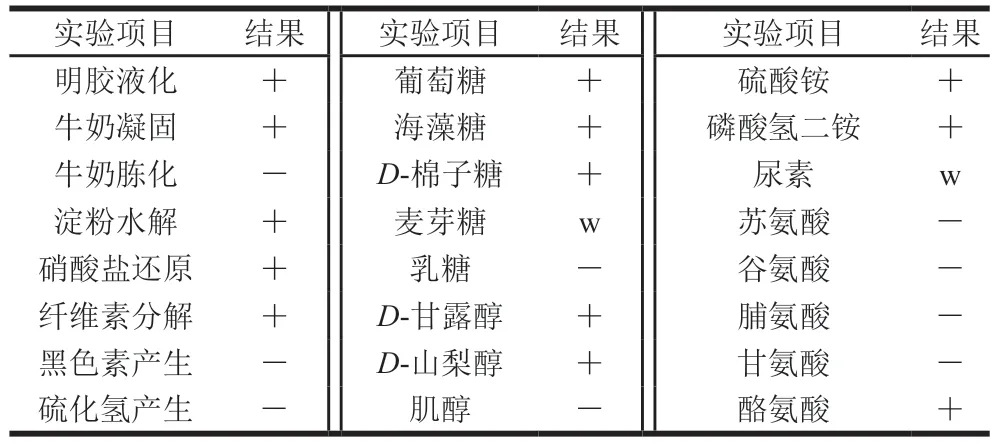
表2 菌株FBKL4.014的生理生化特征Table 2 Physiological and biochemical characteristics of strain FBKL4.014
2.1.4 菌株16S rRNA基因系统发育分析
高温放线菌FBKL4.014基因组DNA片段长度约23 kb,经PCR扩增、拼接后的16S rDNA序列有效片段长度约为900 bp,与GenBank、EzTaxon等数据库中相似菌株的16S rDNA序列进行比对分析,并在种分类学水平搜索序列相似性,结果显示菌株FBKL4.014与莱斯氏属下糖莱斯氏菌(Laceyella sacchari)序列相似性最高;通过构建系统发育树(图2),发现高温放线菌FBKL4.014与L.sacchariJF819833聚类在同一个分支上,其序列同源性为99.78%,因此判断该菌株在种水平上极有可能为L.sacchari。
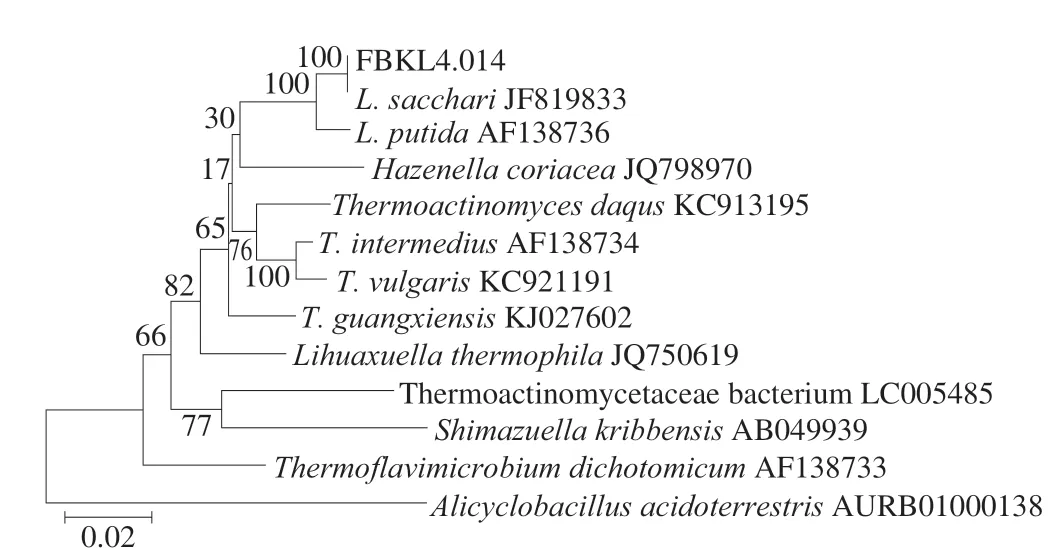
图2 菌株FBKL4.014基于16S rRNA基因序列的系统发育树Fig.2 Neighbor-Joining phylogenetic tree based on 16S rRNA gene sequence of strain FBKL4.014
2.2 L. sacchari FBKL4.014全基因组结果与分析
2.2.1 菌株FBKL4.014基因组的一般特征
L.sacchariFBKL4.014基因组信息已上传NCBI GenBank数据库中,登录号为CP103866.1。对Illumina NovaSeq和PacBio Sequel下机测序数据进行联合拼装,得到完整的基因组序列。L.sacchariFBKL4.014基因组序列全长3.35 Mb,GC含量为48.67%,共预测到3 328 个蛋白编码基因,包括97 个tRNA、31 个rRNA(10 个5S rRNA、11 个16S rRNA、10 个23S rRNA)和36 个ncRNA编码基因,预测到11 条CRISPRs结构,测序深度为433×(表3)。利用CGView软件绘制了FBKL4.014基因组圈图(图3)。此外,通过NCBI平台查阅到其他糖莱西氏菌的GC含量大多在47%~49%之间,与本研究菌株所测得的GC含量(48.67%)接近,进一步证实该菌符合高温放线菌菌属GC含量低的特征。
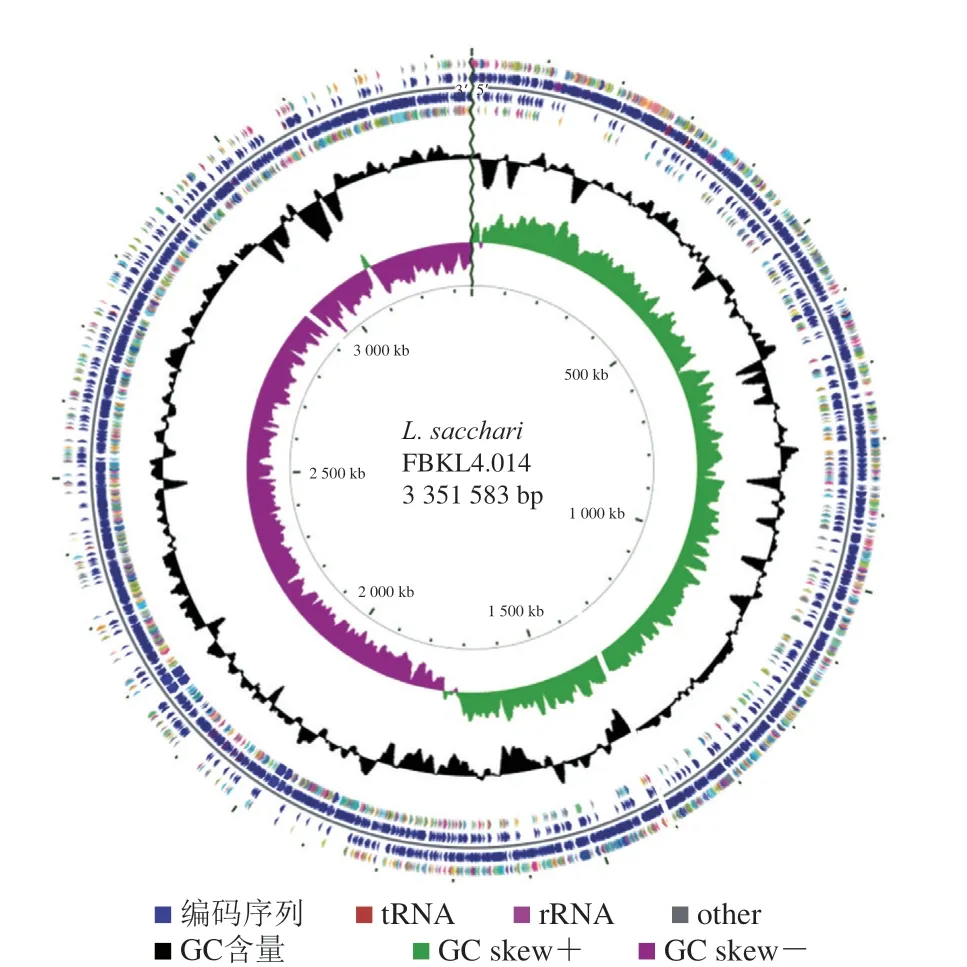
图3 菌株FBKL4.014基因组圈图Fig.3 Circos plot of the genome of strain FBKL4.014
2.2.2 菌株FBKL4.014基因组功能注释结果分析
利用BLAST在线比对分析工具将FBKL4.014的测序结果分别与NR、直系同源蛋白分组比对(evolutionary genealogy of genes: Non-supervised Orthologous Groups,eggNOG)、KEGG、Swiss-Prot、GO和Pfam数据库进行比对分析,获取注释到的蛋白编码基因数量分别为3 325、2 580、1 649、2 301、2 150 个和2 497 个;其中,在TCDB和PHI数据库中注释到的基因数较少,分别为474 个和553 个。此外,在CAZy和CARD数据库中分别注释到72 条和56 条基因信息。FBKL4.014基因组功能注释总体情况如图4所示。
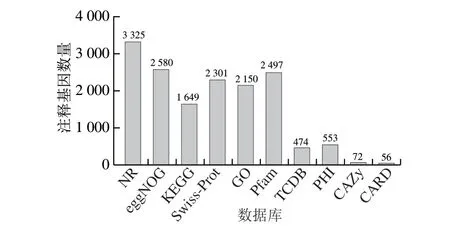
图4 菌株FBKL4.014基因组功能注释总体结果统计Fig.4 Overall statistics of gene function annotation of strain FBKL4.014
2.2.2.1 NR数据库基因注释分析
FBKL4.014共有3 225 个蛋白编码基因在NR数据库中得到注释,占总预测编码蛋白基因总数的96.91%,其中注释到与L.sacchari同源的基因数量最多,共2 614 个,占全部注释结果的81.5%,这表明FBKL4.014在基因组的结构、同源性上与L.sacchari高度相似,也与前述16S rRNA基因分类鉴定结果一致。
2.2.2.2 eggNOG(COG)数据库基因注释分析
eggNOG(COG)数据库的比对分析结果显示( 图5 ) , 在 数 据 库 的 所 有2 5 个 功 能 分 类中,FBKL4.014共有19 类2 580 个基因得到了注释,占注释基因总数的77.52%。其中具有潜在生物学功能的基因注释结果最为丰富,共721 个,占注释基因总数的27.95%,这也为今后针对高温放线菌未知基因组信息的进一步挖掘提供可能。其次,氨基酸转运与代谢存在215 个注释结果,占注释基因总数8.33%;有研究表明酱香大曲中氨基酸代谢的活跃程度总体较高[2],酿酒原料中含有丰富的蛋白质,微生物通过氨基酸代谢、转运途径分解原料中的蛋白质,不仅可以为微生物生长提供必须的能量物质,也可以为酒体提供风味及其前体物。结合课题组前期的工作,这一研究结果的发现也说明在发酵体系中菌株FBKL4.014可能与酱香型白酒中含氮化合物的形成密切相关。
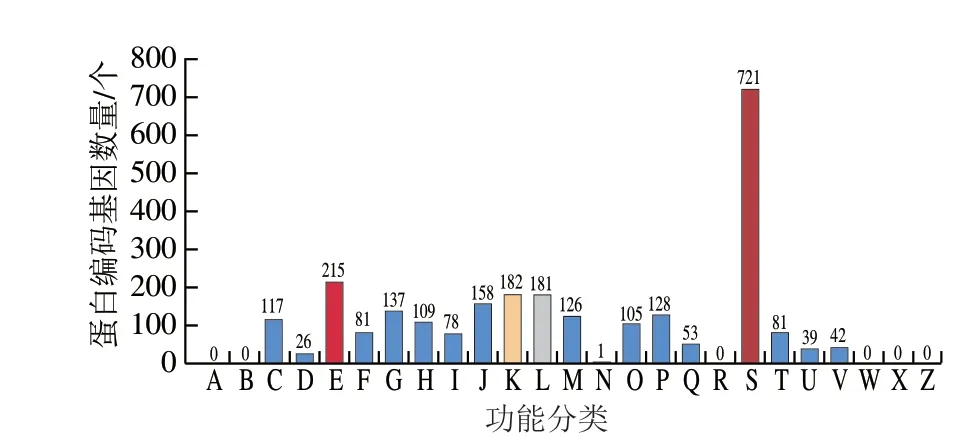
图5 FBKL4.014基因组eggNOG数据库比对分析结果Fig.5 Results of alignment of FBKL4.014 genome with eggNOG database
2.2.2.3 GO数据库基因注释分析
GO数据库的比对结果表明(图6),菌株基因组共有2 150 个蛋白编码基因得到注释,占注释基因总数的64.60%。在GO数据库生物过程、细胞组成、分子功能三大功能分类数据库中,生物过程、细胞氮化合物代谢过程、生物合成过程和小分子代谢过程这4 个途径富集的基因数量最多,分别富集到2 030、789、753 个和579 个基因。有研究表明,在酱香型白酒高温酿造体系下,随着发酵温度升高,大曲中微生物丰度随之降低[5],因此以菌株FBKL4.014为代表的一类嗜热菌的正常生物合成和代谢活动对推动酱香型白酒整体酿造进程意义重大,尤其是细胞氮复合代谢过程富集到较多的基因,也与前期该菌株能将硝酸盐还原为亚硝酸盐以及利用硫酸铵和磷酸氢二铵等无机氮源的研究结果高度相关,初步揭示该菌株具有将无机氮转化为如游离氨等某些含氮风味物质前体物的潜力。碳水化合物代谢过程和分解代谢过程也分别富集了113 个和272 个基因,表明该菌株可能参与到酿酒原料中淀粉、纤维素及其他有机大分子物质的分解代谢过程,是酒体中风味及其前体物的潜在来源。此外,细胞、细胞组成、细胞内和细胞质4 个途径分别富集到686、523、510 个和396 个基因;分子功能、离子结合和DNA结合3 个途径也分别富集到1968、690 个和336 个基因。
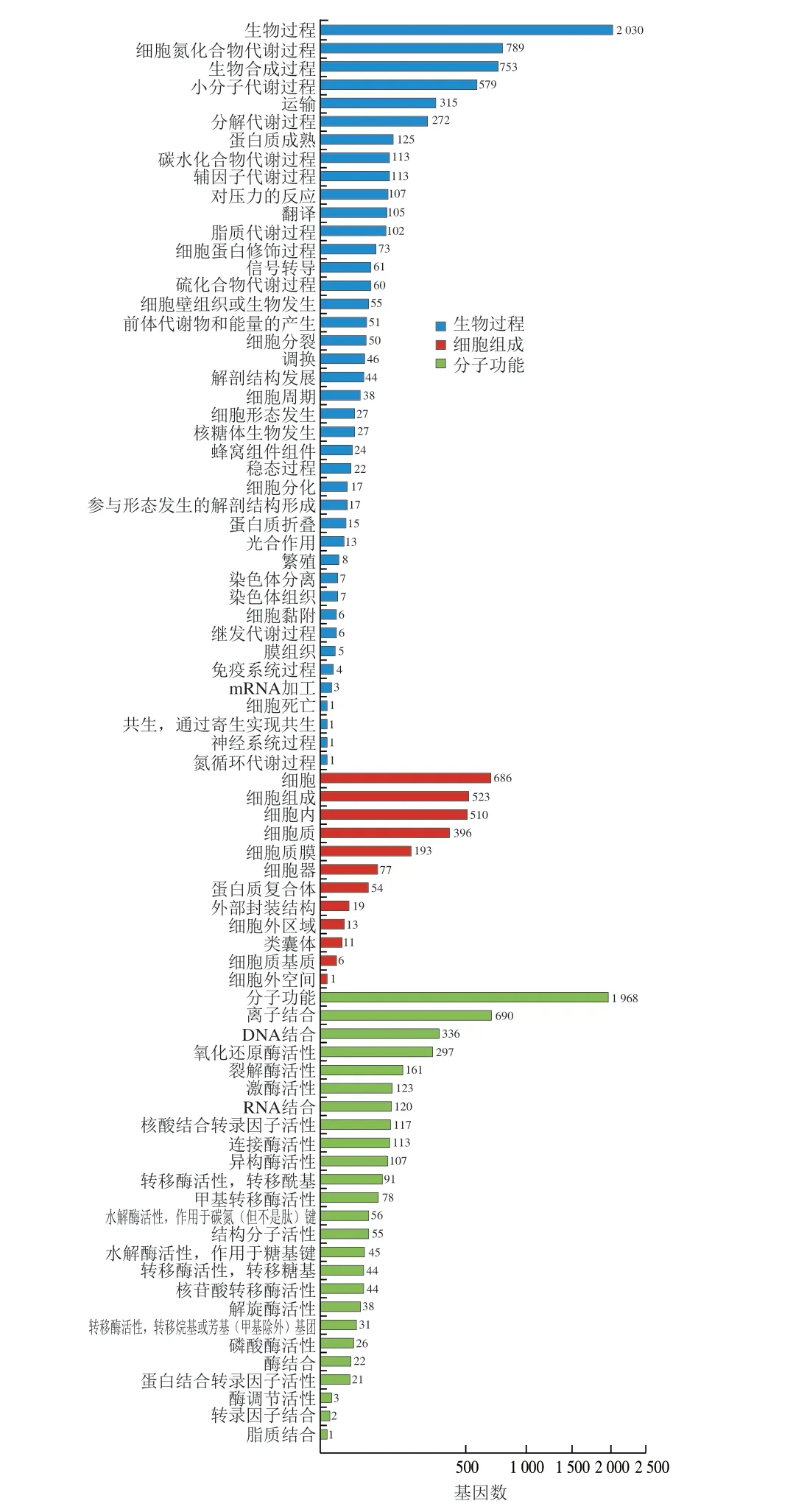
图6 FBKL4.014基因组的GO功能分类图Fig.6 GO function classification map of FBKL4.014 genome
2.2.2.4 KEGG数据库注释分析
KEGG数据库中FBKL4.014共有1 649 个蛋白编码基因注释到8 个层级的205 条通路中,占基因总数的49.55%,结果如图7所示。分析结果表明代谢途径富集到的基因数量最多,共1 085 个,其中有19 个较为完整的代谢途径(图8),总体上表现出氨基酸代谢活动较为活跃的结果。
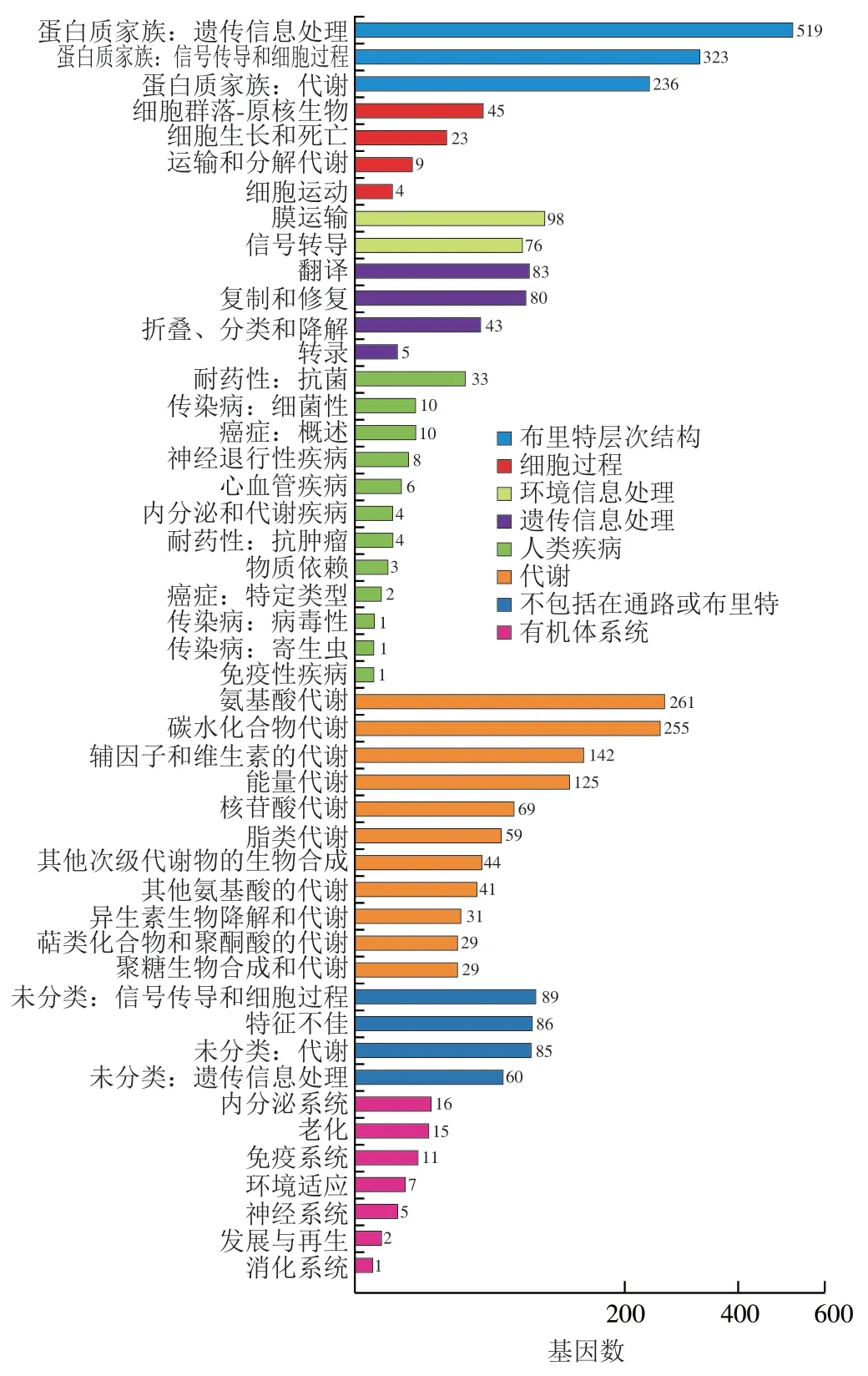
图7 菌株FBKL4.014的KEGG功能分类图Fig.7 KEGG function classification map of strain FBKL4.014

图8 菌株FBKL4.014较完整的代谢途径Fig.8 Complete metabolism pathways in strain FBKL4.014
进一步通过KEGG数据库氨基酸代谢通路分析,发现菌株具备丙氨酸、谷氨酸、天冬氨酸、丝氨酸、甘氨酸、色氨酸和苏氨酸等19 种氨基酸代谢途径。对氨基酸代谢通路的进一步分析发现,天冬氨酸、丝氨酸、脯氨酸、苏氨酸、谷氨酸和天冬酰胺可由糖类物质生物转化而成,丙氨酸、甘氨酸、缬氨酸、异亮氨酸、酪氨酸和苯丙氨酸既可以由糖类物质生物合成,也可以由前述的6 种氨基酸通过生物转化合成。从FBKL4.014注释结果发现、亮氨酸、精氨酸、赖氨酸和酪氨酸只能通过相应氨基酸生物转化而不能由糖类物质合成,这与张巧玲等[32]的研究结果一致。氨基酸代谢在白酒酿造体系中极为重要,主要表现在3 个方面:一方面氨基酸是酱香型白酒中风味前体物的重要来源,可代谢产生相应的醇类物质作为酒体中酸类和酯类物质形成的重要前体,如亮氨酸代谢生成异戊醇、异亮氨酸代谢产生活性戊醇、酪氨酸代谢产生酪醇、缬氨酸代谢产生异丁醇、苯丙氨酸代谢产生2-苯乙醇等。另一方面氨基酸本身就是酱香型白酒中风味物质的直接来源,通过代谢产生内酯、大马士酮和香叶基内酮等风味物质。第三氨基酸与还原糖在高温条件下发生美拉德反应生成呋喃类衍生物、吡咯类衍生物和醛酮及吡嗪类衍生物[33]。因此,菌株FBKL4.014 KEGG注释分析结果也初步表明其在酱酒酿造体系中可能与一些含氮风味组分的产生密切关联。
2.2.2.5 CAZy分析
酱香型白酒酿造体系中,酿酒原料主要以高粱为主,原料中含有丰富的淀粉、纤维素等大分子物质,在微生物代谢产生的碳水化合物活性酶的作用下被分解为许多小分子化合物,为微生物生长提供能量和营养物质,同时也是酒体中风味物质的重要来源[33-34]。测序结果分析表明,FBKL4.014基因组中共有72 个基因注释到CAZy数据库中,其中包含碳水化合物结合域酶(carbohydrate-binding modules,CBMs)9 个,CBM家族包括CBM16、CBM32、CBM39、CBM50和CBM61;碳水化合物酯酶(carbohydrate esterases,CEs)17 个,CE家族包括CE1、CE3、CE4、CE8、CE9、CE10和CE14;糖基转移酶(glycosyl transferases,GTs)13 个,GT家族包括GT1、GT2、GT4、GT28和GT51;辅助活性酶(auxiliary activities,AAs)7 个,AA家族包括AA1_3、AA3、AA4、AA6和AA10;其中,糖苷水解酶(glycoside hydrolases,GHs)最多,有25 个,GH家族则包括GH3、GH4、GH13_1、GH13_29、GH13_31、GH15、GH18、GH23、GH25、GH33、GH46、GH64、G H 1 0 9、G H 1 2 3、G H 1 2 9;最少的是多糖裂解酶(polysaccharide lyases,PLs),仅注释到PL22_1中的1 个基因。CAZy 注释分析结果见表4。

表4 FBKL4.014基因的CAZy分析结果统计Table 4 Statistics of CAZy analysis of FBKL4.014 genes
FBKL4.014基因组中GH15家族的葡萄糖淀粉酶(EC 3.2.1.3)是水解淀粉的主要酶,在白酒酿造中可将原料中的淀粉水解为葡萄糖,一方面葡萄糖为酵母菌、细菌等微生物生长代谢提供营养物质和能量,推动发酵过程的进行;另一方面葡萄糖经糖酵解、三羧酸循环、丙酮酸代谢等途径产生氨基酸、有机酸等酒体风味及风味前体物[35]。此外,GH3家族的β-葡萄糖苷酶(EC 3.2.1.21)是纤维素降解的关键酶,通过水解纤维素非还原末端β-D-糖苷键形成葡萄糖[36]。这也进一步证实了前期菌株生理生化特征有关菌株能产淀粉酶和纤维素酶的推测。总之,从CAZy分析角度对菌株FBKL4.014降解淀粉、纤维素等碳水化合物能力的明确,再次表明高温放线菌类群在推动酱香型白酒酿造顺利进行的过程中具有一定的潜在作用。
2.2.2.6 FBKL4.014四甲基吡嗪合成相关基因分析
白酒酿造体系中的四甲基吡嗪可通过微生物代谢产生,乙偶姻和游离氨是其关键前体物[44]。因此四甲基吡嗪的代谢可分为两部分:1)吡嗪半环碳骨架化合物的合成;2)吡嗪含氮基团结构的形成。如图9所示,一方面,酿酒原料中的糖类物质在微生物作用下,经糖酵解(Embden-Meyerhof-Parnas,EMP)途径产生丙酮酸,丙酮酸通过乙酰乳酸合酶(EC 2.2.1.6)转化为α-乙酰乳酸,随后通过乙酰乳酸脱羧酶(acetolactate decarboxylase,ALDC)脱羧转化为乙偶姻(半环碳骨架化合物);另一方面,蛋白质被蛋白酶水解成游离氨基酸,经氨基酸代谢进一步转化为游离态氨(含氮基团);半环碳骨架化合物和含氮基团通过非酶促反应生成四甲基吡嗪[45-46]。
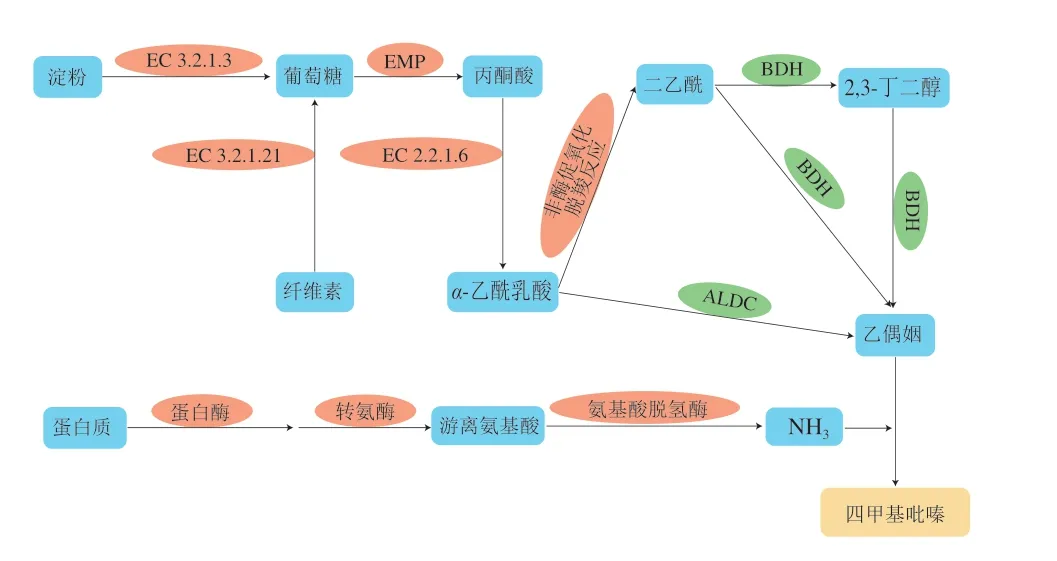
图9 四甲基吡嗪生物合成途径Fig.9 Biosynthesis pathways of tetramethyl pyrazine
乙偶姻的生物合成途径包含一系列复杂的生物过程[47],FBKL4.014基因组中所注释到的葡萄糖淀粉酶(EC 3.2.1.3)、β-葡萄糖苷酶(EC 3.2.1.21)可分别将淀粉和纤维素降解为单糖,经糖酵解途径产生丙酮酸,在乙酰乳酸合成酶(EC 2.2.1.6)作用下合成乙酰乳酸,再经ALDC或2,3-丁二醇脱氢酶(2,3-butanediol dehydrogenase,BDH)脱羧/脱氢合成乙偶姻[48]。不过基因组中没有发现ALDC和BDH蛋白编码基因,因此,推测该菌株这些蛋白编码基因发生了变异,亦或该菌株可能存在尚未被发现的四甲基吡嗪生物合成的完整途径。
四甲基吡嗪生物合成的另一个关键前体物质为游离氨,KEGG注释分析表明,FBKL4.014氨基酸代谢活动比较活跃,基因组中含有蛋白酶、肽酶及羧肽酶编码基因,具备有效将原料中的蛋白质和多肽水解成游离氨基酸的能力,同时,丙氨酸脱氢酶(EC 1.4.1.1)、谷氨酸脱氢酶(EC 1.4.1.2)、亮氨酸脱氢酶(EC 1.4.1.9)和脯氨酸脱氢酶(EC 1.5.5.2)等氨基酸脱氢酶注释结果也说明菌株可能具备将游离氨基酸进一步转化为游离态氨的能力。吴建峰[49]研究表明,固态发酵体系中氨浓度增加可提高四甲基吡嗪的含量,微生物产生的蛋白酶可将蛋白质水解为氨基酸,再经谷氨酸脱氢酶等氨基酸脱氢酶进行联合脱氨作用产生氨。综上所述,结合本课题组前期的工作,认为FBKL4.014具备生物合成四甲基吡嗪的潜力。
3 结 论
从酱香型高温大曲中筛选到1 株高温放线菌FBKL4.014,经形态学、生理生化特征、16S rRNA基因系统发育树鉴定,确定其可能为高温放线菌科、莱斯氏菌属下的L.sacchari。同时,利用第2代和第3代结合的测序技术完成L.sacchariFBKL4.014基因组测序工作,测序结果表明,L.sacchariFBKL4.014染色体基因组序列全长3.35 Mb,GC含量48.67%,测序深度433×,共预测到3 328 个蛋白编码基因,为后续进一步对其进行功能性研究提供数据支持,同时希望为酱香型白酒酿造体系中其他未培养微生物筛选方法提供参考。
通过全基因组测序及基因组解析,发现了L.sacchariFBKL4.014基因组上葡萄糖淀粉酶(EC 3.2.1.3)、β-葡萄糖苷酶(EC 3.2.1.21)等碳水化合物活性酶编码基因的存在,证实其可能为该菌株参与酿酒原料中碳水化合物分解代谢的关键证据。KEGG数据库注释分析结果进一步显示该菌株基因组中有较完整的氨基酸代谢途径,说明该菌株氨基酸代谢活动也可能非常活跃;酱香型白酒中含氮化合物高是其典型的风格特征,其中,细胞氮复合代谢过程可能是该菌株将环境中的无机氮转化为某些风味前体物的关键途径,无疑表明菌株FBKL4.014很可能也是酱香型白酒中含氮物质的重要微生物来源。此外,该菌株基因组中还存在四甲基吡嗪合成的关键酶编码基因,同时针对ALDC和2,3-BDH尚未在该菌株基因组中被发现的事实,推测该菌株可能存在尚未被发现的特殊的四甲基吡嗪生物合成完整途径,也为今后酱香型白酒中风味组分在高温放线菌中的生成机理研究提供了基因层面的数据支持。本研究有助于发掘酱香大曲中高温放线菌的功能应用价值,为后续高温放线菌功能特性的确定与资源挖掘奠定了基础。

