融合多分支特征的肝脏和肝脏肿瘤的体积分割
杨本臣,贾宇航,金海波
(辽宁工程技术大学 软件学院,辽宁 葫芦岛 125105)
0 概述
原发性肝癌是2020 年全球第六大最常诊断的癌症和第三大癌症死亡原因[1]。计算机断层扫描(CT)影像的准确测量,包括肝脏和肿瘤的位置、体积、形状,可以帮助医生作出精确的肝脏肿瘤细胞评估和治疗计划。传统的肝脏病变评估是由放射科医生通过分析计算机断层扫描影像逐层完成描绘的,这种方式成本高、耗时长、可重复性差,分割结果很大程度上依赖于操作者的技术水平。因此,临床对肝脏及肿瘤的自动分割方法提出了较高的要求。
早期的医学图像自动分割方法主要基于区域的阈值法[2]、区域生长法[3]、边缘检测滤波器[4-5]等。这些传统的图像分割方法能够取得较好的分割结果,但分割过程往往取决于操作者的先验知识,如阈值法中选取阈值、区域生长法中选取种子点等,导致分割结果受到主观因素的影响较大。
自深度学习出现以来,全卷积网络[6],特别是U 型编码器-解码器架构[7-9]在各种医学语义分割任务中取得了较好的效果。在典型的U-Net[10-11]体系结构中,编码器负责对提取的特征进行下采样来捕捉图片的上下文信息,解码器对提取的特征进行上采样来对分割部分进行精确定位,不同分辨率的编码器和解码器的输出通过跳跃连接合并以恢复在下采样期间丢失的空间信息。VALANARASU等[12]提出一种过完备卷积结构(Kite-Net),通过交叉残差特征块与U-Net 结合在一起实现了对边界精细细节甚至在更深层小结构的提取。然而,卷积层中感受野的局部性仍然将它们的学习能力限制在相对较小的区域。为了提高非局部建模能力,已有研究提出将自注意力模块与卷积层相结合的方法[13-14]。最近,完全基于自注意力的Transformer[15]设计被用于对序列到序列任务中长距离依赖关系进行建模,它能捕获序列中任意位置之间的关系并且在各种计算机视觉任务上达到先进的性能。然而,虽然Transformer具有强大的学习全局信息的能力,但它不能正确捕捉局部信息。
本文设计KTU-Net 多分支特征融合网络用于肝脏和肝脏肿瘤的精确分割。KTU-Net 通过融合3 个网络的特征实现全局和局部信息的融合,从而提高模型的分割效果。最后,使用公共数据集Liver Tumor Segmentation benchmark(LiTS)验证提出的模型在肝脏肿瘤分割任务上的有效性。
1 相关工作
1.1 基于U-Net 的分割网络
自从U-Net[11]引入医学图像分割任务取得了巨大成功,许多基于U-Net 的变体也不断出现。ZHOU等[16]提出了一种深度监督的编码器-解码器网络,其中编码器和解码器子网络通过一系列嵌套、密集的跳跃路径连接在一起,缩小了编解码子网络特征映射之间的语义鸿沟。张相芬等[9]提出了一种U 型结构UNet3+,通过深度监督从多尺度聚集的特征图中学习层次化的表征,而完全跳跃连接则将多尺度特征图中的高层次语义特征与低层次语义特征相结合。VALANARASU等[12]提出了一种双分支结构的KiU-Net,将过完备卷积结构Kite-Net 和U-Net网络通过交叉残差特征块结合在一起,然后使各自的特征信息相互传递来学习互补特征,实现了对边界精细细节以及更深层中小目标特征的提取。BENČEVIĆ等[17]提出了一种名为Polar U-Net 的架构,通过训练一个神经网络来实现对原始数据集的极坐标变换,这样变换后的极坐标原点就是对象的中心点,实现了网络的维度降低以及分割和定位任务的分离,使网络更容易收敛。尽管这些网络取得了成功,但它们在学习全局上下文信息和长距离空间依赖方面存在局限性,这可能会严重影响挑战性任务的分割性能。
1.2 基于Transformer 的分割网络
HATAMIZADEH等[18]提出了UNETR,通过利用Transformer 作为编码器来学习输入体积的序列表示,并且编码器和解码器遵循了成功的U 型网络设计,能够有效地捕捉全局多尺度信息。VALANARASU等[19]提出了一个门控的轴向注意力模型MedT,该模型通过在自我注意力中引入一个总结性的控制机制,扩展了现有的卷积神经网络架构。CAO等[20]提出了一种纯粹基 于Transformer 的U 型架构Swin-Unet,使用带有移位窗口的分层Swin Transformer 作为编码器来提取上下文特征,并且设计了一个基于Swin Transformer 带有补丁扩展层的对称解码器,用于执行上采样操作以恢复特征图的空间分辨率。LIN等[21]提出了DS-TransUNet,它进一步扩展了Swin-UNet,增加了一个编码器来处理多尺度的输入,并引入了一个融合模块,通过自注意力机制有效地建立了不同尺度特征之间的全局依赖关系。
与现有的方法不同,本文所提出的网络架构在捕获局部边缘的精细细节的同时,还能捕获全局多尺度信息,从而确保空间一致性。
2 本文方法
2.1 体积分割的KTU-Net 架构
体积分割的KTU-Net 架构如图1 所示(彩色效果见《计算机工程》官网HTML版,下同)。它是一个三分支网络,从上往下3 个分支分别为Kite-Net、U-Net、Transformer。对于U-Net 与Kite-Net 分支,输入形状为X∈RC×H×W×D的计算机断层扫描图像,其中,H、W为扫描的高度和宽度,D为扫描的二维图像切片数,C为通道数。在编码阶段,Kite-Net 通过3 个线性上采样层将输入投影到更高的维度,这样可以限制感受野在网络深层的增加来学习更精确的局部边缘特征细节。U-Net 通过3D 最大池化层不断进行下采样来学习输入图像的高级特征。在解码阶段,KiU-Net 和KTU-Net 每一层都采用跳跃连接将底层特征与高层特征相结合,从而提高特征的重用性。此外,在编解码过程中,KTU-Net 与U-Net 的每个层级都使用残差特征融合块结合起来。

图1 KTU-Net 整体架构Fig.1 Overall architecture of KTU-Net
同样地,对于Transformer 分支,将输入体X∈RC×H×W×D重塑为均匀不重叠的块X∈RN×(P3·C),其中,(P,P,P) 表示每个块的分辨率,N=为输入序列的长度。使用线性层将块投影到K维嵌入空间,然后为了对空间信息进行编码,增加了一维可学习的位置嵌入并将其添加到块嵌入中以保留位置信息。用公式可以表示为:
其中:E∈R(P3·C)×K为块嵌入投影;Epos∈RN×K为位置嵌入。
Transformer 编码器由L层多头自注意力(MSA)机制和多层感知器(MLP)模块组成。因此,第i层的输出表示为:
其中:LN()表示层归一化;MLP 由具有GeLU 激活函数的2 个线性层组成;i为中间块标识符;L为Transformer 层数。
最后,将Transformer 分支作为一个单一的网络训练特征来学习分类器,与KTU-Net 其他2 个分支学习的分类器的预测结果相结合,实现肝脏和肝脏肿瘤的分割。
2.2 交叉残差特征块
使用交叉残差特征块(Cross Residual Feature Block,CRFB)来结合Kite-Net 和U-Net 在多个尺度上的特征。在编码和解码阶段,U-Net 和Kite-Net的每一层都使用CRFB 来融合高层语义特征和局部边缘特征。CRFB 通过插值、GN 和ReLU 激活函数提取Kite-Net 的交叉残差特征,之后与U-Net的原始输入进行加性操作。CRFB 对U-Net 的操作同理。由于这两个网络学习不同的特征,因此它们可以通过CRFB 学习互补的特征,提高网络学习的特征的质量。CRFB 结构如图2 所示,输出可以表示为:
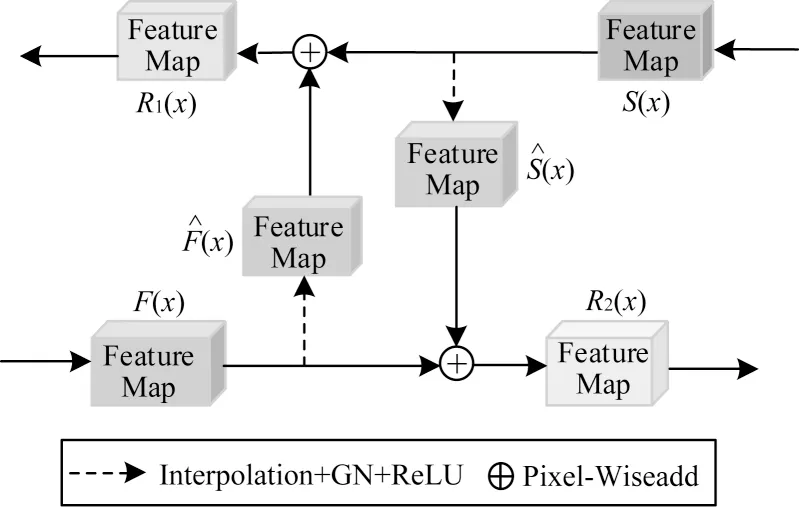
图2 交叉残差特征块结构Fig.2 Structure of CRFB
其中:F(x)和S(x)是交叉残差特征块的输入;(x)和(x)是特征图进行插值操作、ReLU 激活和组归一化后的特征映射。
2.3 融合细节和时机
在获得了全局上下文信息后,有两种通用的标准范例将其与局部特征一起使用[22]。上文描述的网络架构的融合方式为晚期融合,即全局特征和局部特征都用来学习它们自己的分类器,然后将两个预测合并成单个分类分数。另一种融合方式为早期融合,即在空间上将全局特征分解到与局部特征图相同的大小后连接起来,使用组合的特征来学习分类器。这两种方法各有利弊。对于后期融合,存在这样一种情况:只有在全局特征辅助下的局部特征才能够判断出正确的分类结果,如果两个特征分别训练再融合,可能会产生分割损失。对于早期融合,融合前不进行归一化,高层特征就会被低层的大尺度特征覆盖,所以,在合并特征前必须对每个单独的特征进行标准化,以使融合后的特征能够正常工作。
为了比较采用两种融合方式的分割性能,设计采用早期融合方式的KTU-Net,网络架构如图3 所示。在这种情况下,Transformer 在编码阶段生成输出特征图后,使用一个反卷积层来改变特征图的大小,然后与U-Net 编码阶段得到的特征图级联,提供全局多尺度信息。

图3 采用早期融合方式的KTU-Net 架构Fig.3 KTU-Net architecture with early fusion approach
解码阶段的每一层级都通过CRFB 模块将Transformer 和U-Net 级联的特征图信息与Kite-Net的局部精细边缘信息结合,以实现全局信息与局部信息的融合。
在解码阶段之后,从KTU-Net 3 个分支得到的输出被合并输入到1×1×1 3D 卷积层来生成预测体素。
2.4 损失函数
本文采用BCEWithLogits Loss 和Dice Loss 相结合的损失函数。BCEWithLogits Loss 将Sigmoid函数和二元交叉熵损失函数结合为一类,从而使计算结果更加稳定,并且有利于梯度的稳定反向传播,避免了梯度消失的问题。BCEWithLogits Loss定义为:
其中:C表示类 数;N表示体素数 量;Pi,j和Gi,j分别表示体素i第j类的预测值输出和真实标签;σ(Pi,j)为Sigmoid 函数。
Dice Loss 可以解决语义分割中正负样本之间的极端不平衡问题,避免发生过拟合现象。它通过侧重对前景区域的挖掘减少局部最小值对损失函数的影响,并且对网络的监督惩罚不会随着特征的大小而改变。计算公式为:
其中:Smooth 的值设置为1e-5。
BCEWithLogits Loss 利于对每个类别的平等评估,Dice Loss 利于优化分割网络,因此,上述损失的混合损失函数最终表示为:
其中:λ的值设置为0.5。
3 实验及结果分析
3.1 数据和评估指标
使用LiTS 数据集来训练和评估所提出的方法。LiTS 数据集来自111 个(不包括3DIRCAD-B 数据集)腹部对比计算机断层扫描图像,输入尺寸为512×512 像素,平面内分辨率范围为0.56~1.00 mm,切片厚度范围为0.45~6.00 mm。在实验过程中,随机将CT 图像划分为3 个不重叠的子集:70%的CT 图像用于训练,20%用于验证,10%用于测试。对于图像预处理,参考文献[23]中的方法,处理后的图像分辨率为48×128×128 像素。对于图像后处理,只保留分割预测的最大连接区域。
为了验证所提出的网络的基础事实和测试结果之间的性能,使用以下客观和通用的度量指标来评估分割模型:Dice 得分(DC)、Dice 全局(DG)、体积重叠误差(VOE)、平均对称表面距离(ASSD)和精度。将所提出的网络与KiU-Net、TransBTS、UNETR这三个先进的网络进行比较。
3.2 实验过程
本文基于Python 3.7、PyTorch 1.8.1 和Windows 10运行所有实验。所有训练程序都是在一个24 GB 内存的NVIDIA 2080 GPU 上执行的。在训练过程中,为了能够在模型收敛的情况下得到最优的模型,又不至于在数据量较少的情况下过拟合,在实验过程中引入Early Stop 机制,因此,最终的迭代次数不是人为设定的而是由验证集上的损失决定的,设定的最大迭代次数为200次。另外,训练过程中的batch size 大小设定为2,选择的是自适应参数的优化算法Ranger[24],学习率初始化为0.001,Early Stop 中的耐心值为40[25]。基于的Transformer包含8层,嵌入大小为512,并且使用16×16×16 像素的补丁分辨率。
3.3 对比实验
本文在肝脏肿瘤分割挑战数据集(LiTS)上进行主要的实验,将KTU-Net与以下3 个先进的3D 医学图像分割技术进行比较:1)KiU-Net[11];2)TransBTS[26];3)UNETR[18]。
肝脏分割定量指标的比较见表1,肿瘤分割定量指标的比较见表2。本文的KTU-Net 使用后期融合策略,在肝脏分割方面取得了0.967DC 和0.969DG,在肿瘤分割方面取得了0.666DC 和0.713DG。可以看到,与KiU-Net相比,添 加Transformer 作为第三分支后,模型在肝脏和肿瘤分割的准确性上都取得了更好的表现,这一结果证明了将全局信息与局部信息融合策略的有效性。由图3 可以看出,KTU-Net 在肝脏边界精细细节以及肿瘤小目标分割上表现得比TransBTS 和UNETR 更好,说明过完备卷积结构有利于边界精细细节和更深层小结构特征的提取,从而提升了模型对特征的学习能力。

表1 肝脏分割定量指标比较Table 1 Comparison of quantitative indicators of liver segmentation

表2 肿瘤分割定量指标比较Table 2 Comparison of quantitative indicators of tumor segmentation
表1 和表2 中倒数第2 行为采用早期融合策略的KTU-Net 的实验结果,可见融合全局信息后网络的性能得到了提升。而与采用后期融合策略的KTU-Net 相比,网络性能反而下降,这表明使用后期融合方法的KTU-Net 能够更有效地结合多分支网络的特征。另外,为了进行公平比较,在所有这些实验中使用了相同的管道,同时进行了相同的预处理和后处理操作。
3.4 结果可视化
图4 显示了CT 肝脏肿瘤分割结果的直观比较,每一行对应的是来自不同断层扫描的单个切片的分割预测结果,红色区域表示预测的肝脏,绿色区域表示预测的病灶,灰色区域表示真实肝脏,白色区域表示真实病灶,下同。三维分割结果用ITKSnap[26]进行了可视化。偶数行的方框内展示了肿瘤部分的放大图,Ground Truth 绿色区域表示肝脏的真实病灶位置。定性分析表明,本文方法具有更平滑的肝脏肿瘤边缘分割性能,肝脏肿瘤分割取得了比其他先进方法更好或相似的结果。融合了KiU-Net(可以提取边缘的精细细节)和Transformer(捕捉全局背景信息)的KTU-Net 网络对小规模的肿瘤和相对大规模的肝脏都取得了更好的分割效果。
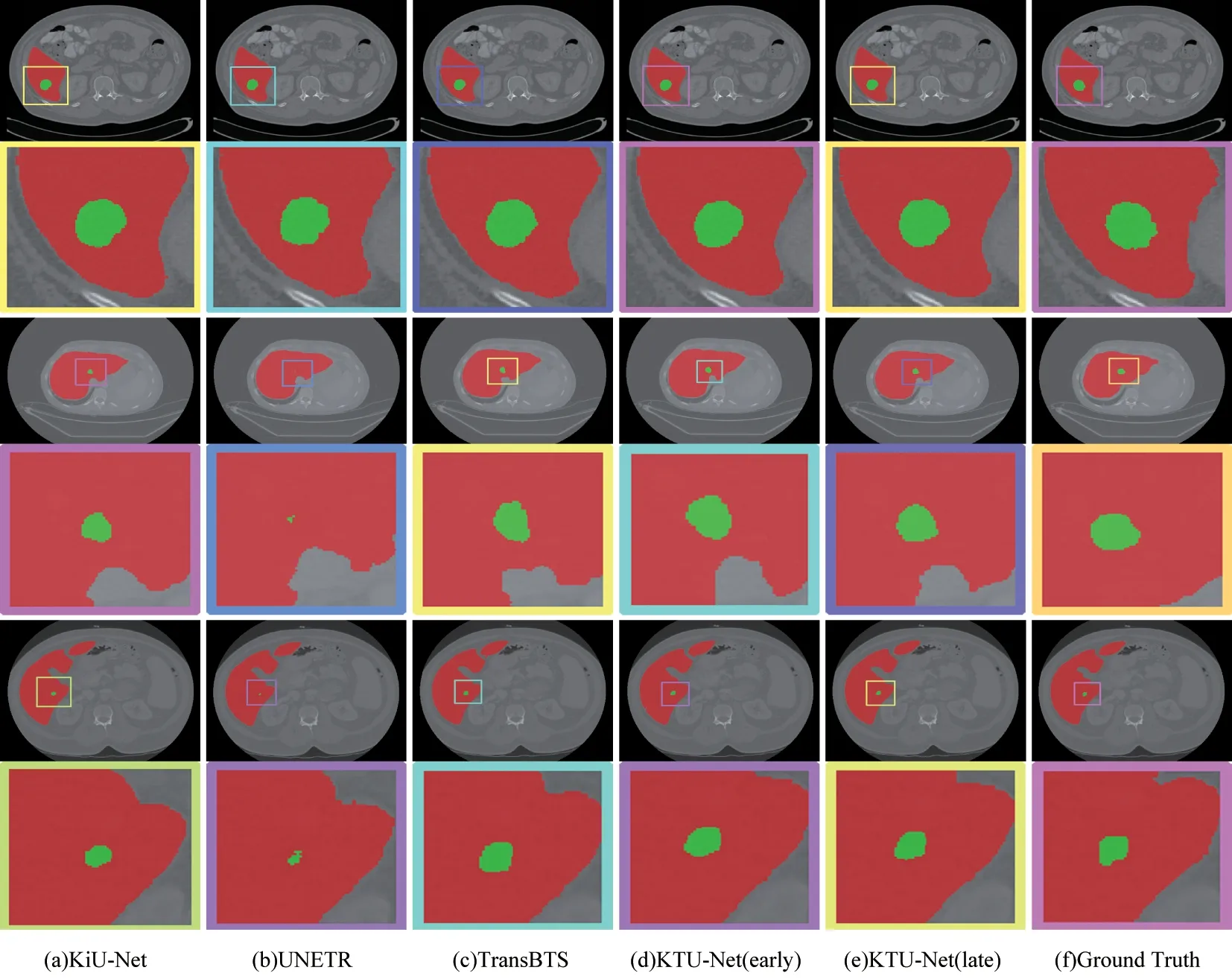
图4 CT 肝脏肿瘤分割结果的视觉对比Fig.4 Visual comparison of CT liver tumor segmentation results
图5 显示了CT 肝脏肿瘤分割结果与Ground Truth 的视觉对比。可以明显看出,采用后期融合策略的KTU-Net 充分覆盖了肝脏的病灶区域,它与采用早期融合策略的KTU-Net 相比损失值更低,说明后期融合方法能有效地结合多分支网络的特征。同时采用后融合策略的KTU-Net 与其他方法相比在肝脏肿瘤分割表现更好,例如第2 组分割结果中,本文方法在肝脏边缘分割更平滑,肿瘤分割区域与真实病灶的IOU 更高。

图5 CT 肝脏肿瘤分割结果与Ground Truth 的视觉对比Fig.5 Visual comparison of CT liver tumor segmentation results with Ground Truth
3.5 消融实验
为简单起见,表3 显示了对KTU-Net 中不同模块的消融研究结果,在3DIRCADB 数据集上进行了实验。表中第1 行最基本的基线由三分支网络(TBN)组成,其不包含任何的跳跃连接(SK)以及交叉残差块(CRFB),并且最后通过添加3 个分支的特征映射输出来融合三分支网络。与基线相比,加入跳跃连接后,平均Dice 分数分别提高了0.3% 和0.8%,说明跳跃连接使解码器在进行上采样时获得了更多高分辨率信息,更完善地恢复了原始图像中的细节信息。同样,加入CRFB 块后,肝脏肿瘤的平均Dice 分数分别提高了0.7%和2.1%,说明交叉残差块通过结合各个分支网络特征,提高了特征融合的质量。从表3 和上述分析可以得出,随着网络中每个模块的添加,性能都会得到一定提升。
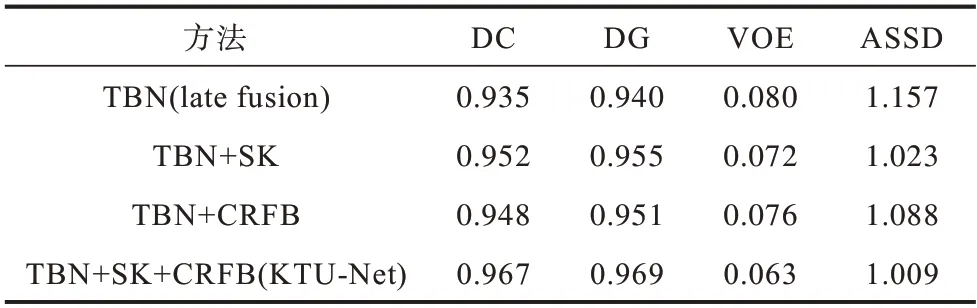
表3 KTU-Net 中使用不同区块的效果Table 3 Effect of using different blocks in KTU-Net
4 结束语
本文提出了多分支特征融合的网络结构KTU-Net,同时关注输入图像的全局和局部特征。与使用早期融合方法相比,使用后期融合方法可以更有效地将全局信息与局部信息进行融合。与先进的三维医学图像分割方法的比较结果表明,本文方法在肝脏肿瘤分割中取得了较高的精度,证明了KTU-Net 的有效性。与现有的肝脏肿瘤分割网络不同,本文通过结合多分支网络实现全局和局部信息的融合。实验结果表明,使用Transformer 作为第三分支来捕获全局背景信息,可以显著提高肝脏肿瘤的分割精度。对于临床实践,本文提出的网络架构在肝脏肿瘤分割任务上表现得更好,可以帮助医生从CT图像分割结果中作出更快的评估和诊断。然而,由于模型的复杂性和大量的参数,多分支网络在训练时产生了较大的计算成本。在未来的工作中,将设计一个轻型的网络模型,以减少模型的计算量,同时保持模型分割的准确性和所提取特征的有效性。

