大豆SWEET 基因在荚粒发育过程中与逆境胁迫下的表达
柯博洋, 李文龙, 张彩英*
(1.河北农业大学生命科学学院,河北 保定 071001; 2.河北农业大学农学院,河北 保定 071001)
光合作用产生的糖类物质不仅是植物重要的碳源和能量贮存形式,而且是重要的信号传递分子,在植物生长发育及抵御各种生物与非生物胁迫中发挥着重要作用[1-2]。然而,糖类物质在叶片中合成以后必需经过韧皮部的运输才能到达其他组织部位或器官进而发挥功能。由于糖类物质不能独立地实现跨膜运输,因而需要糖转运蛋白[3-4]。
SWEET(sugars will eventually be exported transporters)是一类重要的糖转运蛋白,一般含有7个跨膜结构域[5]。辛红佳等[6]利用拟南芥SWEET1 /2 /3突变体证实,SWEET基因具有调控角果生长发育的功能。Li 等[7]发现,水稻基因组有21 个SWEET基因,其中7个参与种子发育,SWEET基因突变后可造成植株花粉活力下降、颖果和种子发育不良。Hu等[8]发现,黄瓜基因组有17个SWEET基因,其在不同组织、器官中的表达存在较大差别,如CsSWEET3与CsSWEET15在茎中优势表达,CsSWEET1、CsSWEET9和CsSWEET10等在雄花中优势表达,CsSWEET7a和CsSWEET7b等在果实中优势表达。Qin 等[9]发现,小麦基因组有105 个SWEET基因,其中59%的基因在干旱、高温以及盐胁迫处理后表达量发生改变。Li 等[10]发现,甘薯IbSWEET10在尖孢镰刀菌侵染后上调表达,过表达IbSWEET10可提高甘薯抗病性。由此可见,糖转运蛋白对于植物正常生长发育以及抗病和抗逆具有重要意义。
与其他植物相比,目前关于大豆SWEET基因的研究较少,且主要集中在功能基因挖掘方面。Wang等[11]发现,GmSWEET10a(Glyma.15G049200)和GmSWEET10b(Glyma.08G183500)通过转运蔗糖和己糖来影响种子大小及油分含量。Zhang等[12]在15 号染色体定位到油分含量数量性状基因座(quantitative trait locus,QTL),该QTL 区间含有1个SWEET基因(GmSWEET39),进一步分析发现,GmSWEET39在大豆种皮优势表达,且通过控制糖类物质从种皮向种胚的运输影响油分含量。Miao 等[13]测定了382 份大豆品种资源的油脂及其组分含量,经群体进化与关联分析获得油脂含量显著关联位点,并在其连锁不平衡范围内寻找到1 个SWEET基因(GmSWEET39),进一步分析发现,GmSWEET39在大豆籽粒高表达,且与油脂含量显著正相关,转化GmSWEET39优势单倍型可显著提高转基因拟南芥和大豆毛状根的油脂含量。Patil 等[14]在2015 年利用已测序大豆品种‘Williams82’基因组序列(Wm82.a1.v1 版本)预测出52 个SWEET基因,但随着测序技术和基因组组装质量的提高,目前‘Williams82’基因组已更新到Wm82.a4.v1 版本[15],且野生大豆基因组也已公布[16],因此,利用最新公布基因组分析栽培大豆和野生大豆SWEET基因,并研究其在大豆生长发育尤其是豆荚和籽粒发育以及抵御生物和非生物胁迫中的作用意义重大。
鉴于此,本研究利用栽培大豆‘Williams82’和野生大豆‘W05’最新基因组数据分析SWEET基因,并利用栽培大豆豆荚和籽粒不同发育时期转录组数据,以及接种花叶病毒不同时间的叶片转录组和低磷胁迫处理不同时间的根系转录组数据分析SWEET基因表达量,筛选参与豆荚和籽粒发育以及抗病和耐低磷的候选基因,为利用SWEET基因提高豆荚和籽粒产量以及抗病、抗逆能力奠定基础。
1 材料与方法
1.1 试验材料
不同栽培大豆品种(小荚品种‘KN7’和大荚品种‘C813’)豆荚5 个不同发育时期(PT1~PT5,分别为开花后5、7、9、12、17 d)转录组数据[17];不同栽培大豆品种(‘Z92116’和‘QH30’)籽粒5个不同发育时期(ST1~ST5,分别为开花后 24、31、38、45、52 d)转录组数据[18];不同大豆品种(抗病品种‘QH30’和感病品种‘Z92116’)接种花叶病毒(soybean mosaic virus,SMV)SC7 株系7 个不同时间(0、3、10、24、120、240、336 h)叶片转录组数据[19];不同大豆品种(耐低磷品种‘ZH15’和不耐低磷品种‘NMH’)低磷胁迫处理5个不同时间(0、7、28、49、70 d)根系转录组数据[20]。
1.2 试验方法
1.2.1栽培大豆与野生大豆SWEET基因鉴定首先从NCBI 网站(https://www.ncbi.nlm.nih.gov/)下载最新版的blast-2.13.0+,然后从大豆公共数据库Soybase网站(https://www.soybase.org/)下载栽培大豆‘Williams82’最新版本基因组(Wm82.a4.v1)[15]和野生大豆‘W05’基因组(W05.a1.v1)[16]CDS(coding sequence)序列,并以上述CDS 序列为基础分别建立栽培大豆和野生大豆核酸数据库;随后以拟南芥AtSWEET11 氨基酸序列为查询序列,利用Tblastn 分别在上述2 个核酸数据库中搜寻匹配阈值小于1e-5 的核酸序列,进而获得栽培大豆和野生大豆基因组中的SWEET基因。
1.2.2栽培大豆与野生大豆SWEET基因系统进化树的构建 首先在栽培大豆‘Williams82’和野生大豆‘W05’基因组CDS 序列中查找上述SWEET基因序列,并单独提取上述序列,进而获得包含SWEET基因序列的fasta 文件;然后将该fasta 格式文件进行mafft 比对,并使用MEGA 剪切掉匹配性较差的区段,将结果保存为fas 文件;随后使用fasttree 软件构建栽培大豆与野生大豆SWEET基因的系统进化树,并利用iqtree 软件进行可视化调整,将最终结果保存为pdf 格式输出。
1.2.3栽培大豆SWEET基因在豆荚不同发育时期表达分析 利用2 个鲜荚大小不同品种(小荚品种‘KN7’和大荚品种‘C813’)开花后5个不同豆荚发育时期(PT1~PT5)的转录组数据[17],分析SWEET基因的表达情况,并依据基因表达模式进行分组,筛选影响豆荚发育的SWEET基因,采用HemI热图软件对基因表达量进行作图。
1.2.4栽培大豆SWEET基因在种子不同发育时期表达分析 利用2 个不同品种(‘Z92116’和‘QH30’)籽粒5个不同发育时期(ST1~ST5)的转录组数据[18]分析SWEET基因表达情况,并依据基因表达模式对基因进行分组,筛选影响籽粒发育的SWEET基因,采用HemI热图软件作图。
1.2.5SWEET基因在大豆叶片接种SMV 不同时间表达分析 利用抗感SMV特性不同的2个栽培大豆品种(抗病品种‘QH30’和感病品种‘Z92116’)接种大豆花叶病毒SC7 株系(黄淮海大豆生态区SMV 主要流行株系)7 个不同时间的叶片转录组数据[19]分析SWEET基因表达情况,并依据基因表达模式对其进行分组,筛选可能参与大豆抗花叶病毒反应的SWEET基因,采用HemI 热图软件作图。
1.2.6SWEET基因在大豆根系低磷处理不同时间表达分析 利用耐低磷大豆品种‘ZH15’和不耐低磷品种‘NMH’低磷处理5个不同时间的根系转录组数据[20]分析SWEET基因表达情况,并依据基因表达模式对其进行分组,筛选可能参与大豆耐低磷反应的SWEET基因,采用HemI 热图软件作图。
1.2.7SWEET基因在不同大豆品种资源间的SNP 分析 利用Soybase 网站公布的1 556 份大豆品种资源重测序数据[21],分析SWEET基因内部的非同义突变单核苷酸多态性(single nucleotide polymorphism,SNP),并根据SNP 突变位置以及基因编码蛋白的序列,在NCBI 网站(https://www.ncbi.nlm.nih.gov/)分析突变位点是否位于基因编码蛋白的保守结构域。
2 结果与分析
2.1 栽培大豆与野生大豆基因组SWEET 基因分析
2.1.1栽培大豆基因组SWEET基因分析 为提供栽培大豆SWEET基因最新信息,利用‘Williams82’最新版本基因组(Wm82.a4.v1)分析SWEET基因,获得其基因ID、起始和终止物理位置以及编码区长度等信息,共获得48个SWEET基因(表1),其编码蛋白长度在174~354 个氨基酸,位于15 条染色体(2、3、4、5、6、8、9、12、13、14、15、17、18、19和20号染色体),以8号和6号染色体分布的SWEET基因数量最多。并且发现,上述48个SWEET基因中有43 个可与已报道[14]预测的SWEET基因进行对应,但基因的物理位置以及部分基因的长度因‘Williams82’基因组版本更新而发生改变,其中一些基因的编码蛋白长度也发生变化,故本研究重新预测的SWEET基因结果将为今后克隆这些基因、分析基因表达以及研究其生物学功能提供参考。

表1 栽培大豆基因组48个SWEET基因信息Table 1 Information of 48 SWEET genes in cultivated soybean genome
2.1.2野生大豆基因组SWEET基因分析 为挖掘利用野生大豆SWEET基因,利用野生大豆‘W05’基因组分析SWEET基因,获得其基因ID、起始和终止物理位置以及编码区长度等信息,共获得51 个SWEET基因(表2),其编码蛋白长度在84~392 个氨基酸,位于16 条染色体(2、3、4、5、6、8、9、11、12、13、14、15、17、18、19和20号染色体),并以8号、6号和4号染色体分布的SWEET基因数量较多。

表2 野生大豆基因组51个SWEET基因信息Table 2 Information of 51 SWEET genes in wild soybean genome
2.1.3栽培与野生大豆SWEET基因的系统进化树分析 为进一步分析上述48 个栽培大豆SWEET基因和51 个野生大豆SWEET基因之间的关系,对这99 个SWEET基因构建系统进化树,结果(图1)发现,99 个基因可分为3 个亚组,其中第Ⅰ亚组包含16 个基因,第Ⅱ亚组包含26 个基因,第Ⅲ亚组包含57 个基因;同时发现,除野生大豆W05.11g030581和W05.u055442等个别基因外,其余基因均可在野生大豆与栽培大豆之间形成“两两配对”关系,如野生大豆W05.03g007786和栽培大豆Glyma.03G209600、野生大豆W05.19g050127和栽培大豆Glyma.19G009800等,这些“配对”基因间相似性更高,为利用野生大豆SWEET基因提供了依据,也为今后研究SWEET基因从野生大豆到栽培大豆的进化奠定了基础。
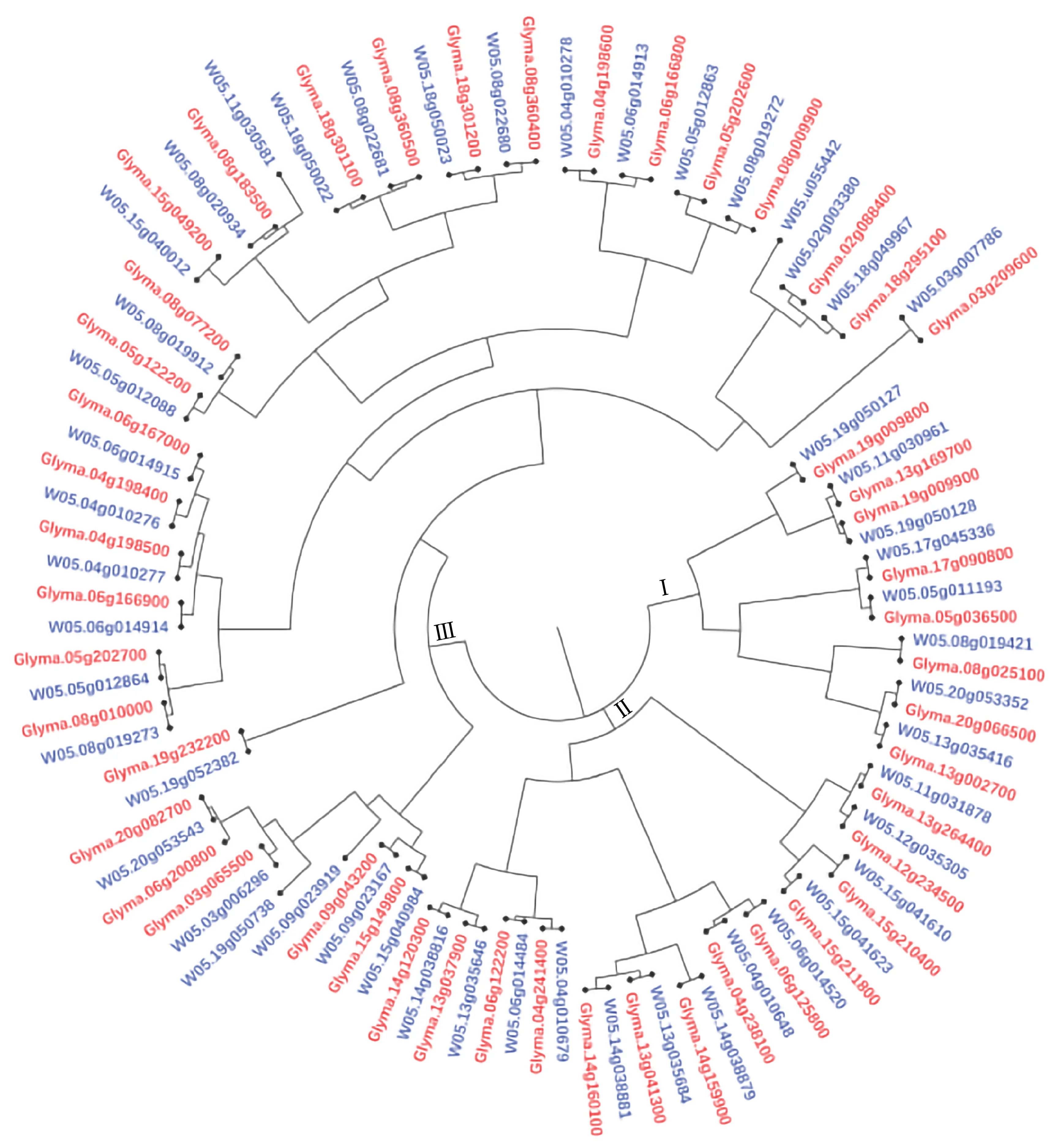
图1 栽培大豆与野生大豆SWEET基因的系统进化树Fig. 1 Phylogenetic tree of SWEET genes in cultivated and wild soybeans
2.2 栽培大豆SWEET基因表达分析
2.2.1豆荚不同发育时期SWEET基因表达分析 通过分析SWEET基因在2个大豆品种5个豆荚发育时期转录组数据,结果发现(图2),有16个基因在豆荚表达,根据其表达情况可将这16 个基因分为3 类。第1 类基因的表达量(以PT1时期为对照)随豆荚发育在2个品种中均至少有1个时期呈现增加,如Glyma.06G122200、Glyma.14G159900和Glyma.14G160100等,其中Glyma.06G122200在2 个品种的PT2、PT3、PT4和PT5时期的表达量均高于PT1时期,Glyma.06G166800在‘KN7’的PT4和PT5时期以及‘C813’的PT3、PT4和PT5时期的表达量均高于PT1时期,说明这些基因的高表达对不同类型品种豆荚发育具有重要意义。

图2 豆荚不同发育时期SWEET基因表达分析Fig. 2 Expressions of SWEET genes in different soybean pod developmental stages
第2 类基因的表达量只随其中1 个品种豆荚发育出现上升,如Glyma.05G122200、Glyma.08G077200和Glyma.19G009900等,其中Glyma.05G122200在‘KN7’的PT2和PT3时期表达量高于PT1时期,Glyma.08G077200在‘KN7’的PT2、PT3和PT4时期表达量高于PT1时期,说明这些基因的高表达只与特定品种的豆荚发育有关。第3 类基因的表达随豆荚发育没有发生明显改变,如Glyma.05G202700、Glyma.08G010000、Glyma.13G169700和Glyma.15G211800。
2.2.2籽粒不同发育时期SWEET基因表达分析 通过分析SWEET基因在2个大豆品种5个籽粒发育时期转录组数据,结果发现(图3),有12个基因在籽粒表达,并且根据其表达情况可将其分为3 类。第1 类基因的表达量(以ST1时期为对照)随籽粒发育在2个品种中均至少有1个时期呈现增加,如Glyma.08G025100、Glyma.13G041300和Glyma.14G120300,其中Glyma.13G041300在2个品种的ST2、ST3、ST4和ST5时期表达量均高于ST1时期,Glyma.08G025100和Glyma.14G120300的表达量在‘Z92116’的ST4和ST5时期以及‘QH30’的ST5时期高于ST1时期,说明这类基因的高表达对不同类型品种籽粒发育具有重要意义。
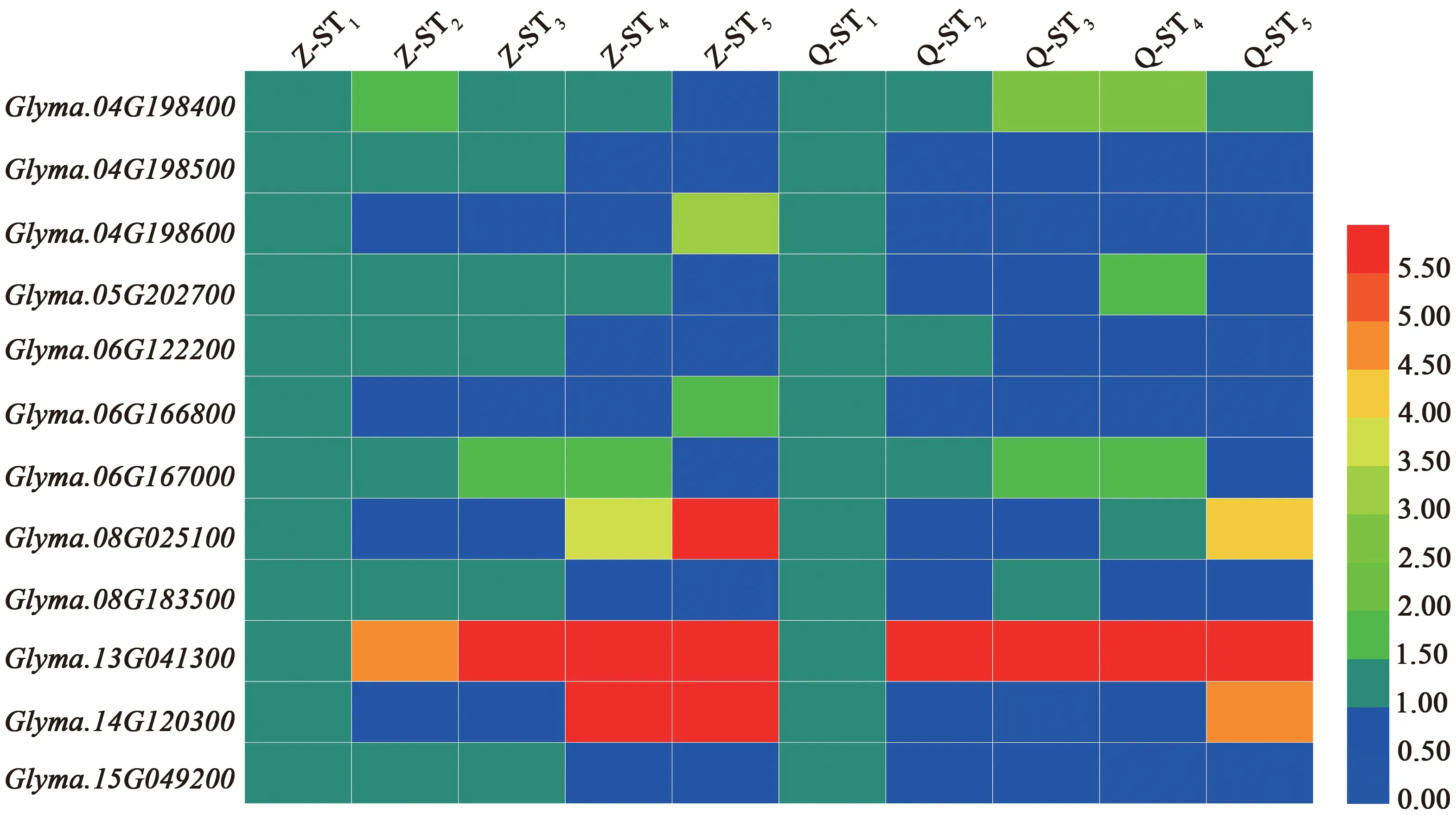
图3 籽粒不同发育时期SWEET基因表达分析Fig. 3 Expressions of SWEET genes in different soybean seed developmental stages
第2类基因的表达只随其中1个品种的籽粒发育出现上升,其中Glyma.04G198400在‘QH30’的ST3和ST4时期表达量高于ST1时期,Glyma.04G198600在‘Z92116’的ST5时期表达量高于ST1时期,说明这类基因的高表达只与特定品种籽粒发育有关。第3 类基因的表达随籽粒发育没有明显变化,如Glyma.04G198500、Glyma.06G122200、Glyma.08G183500和Glyma.15G049200等。
2.2.3接种SMV不同时间SWEET基因表达分析通过分析SWEET基因在不同抗感SMV 大豆品种叶片中的表达量,结果(图4)发现,不同基因对接种SMV 的响应存在较大差别,表明有些SWEET基因参与大豆对SMV 的抗性反应,但也有些基因可能不参与大豆抵抗SMV 侵染过程;进一步分析发现,即使是响应SMV 接种的SWEET基因,其响应时间和响应模式也存在较大差别,说明不同SWEET基因在参与大豆抗SMV 反应中发挥的功能可能不同。

图4 大豆SWEET基因在叶片接种SMV不同时间的表达Fig. 4 Expressions of SWEET genes in soybean leaves at different time-points after SMV inoculation
根据SWEET基因对大豆叶片接种SMV后的表达模式将其划分为3 类,其中,第1 类属于在抗病品种叶片高水平表达的SWEET基因,如Glyma.08G009900和Glyma.13G264400,Glyma.08G009900在抗病品种‘QH30’接种SMV后3、10、24、120、240 和336 h 的表达量均高于接种前,而感病品种中的表达量在SMV 接种前后没有发生明显改变,表明这类基因的高表达可能与大豆抗花叶病毒病相关。第2 类基因在抗感品种‘Z92116’叶片均较高水平表达,如Glyma.04G198600和Glyma.12G234500,其中Glyma.04G198600在‘QH30’接种SMV 后的120和336 h表达量高于接种前,而在‘Z92116’接种SMV 后的3、120和336 h表达量高于接种前,表明这些基因的高表达对于不同大豆品种抵御SMV 侵染具有一定作用。第3类基因在大豆接种SMV 前后表达量没有发生明显改变,如Glyma.15G211800,说明其可能不参与大豆抗花叶病毒。
2.2.4低磷胁迫不同时间根系SWEET基因表达分析 对SWEET基因在不同大豆品种低磷处理根系中的表达量进行分析(图5)发现,按照基因表达情况可将其分为3类。与胁迫前相比,第1类基因的表达量在耐低磷和不耐低磷品种根系均呈现上升趋势,如Glyma.04G198400、Glyma.14G160100和Glyma.15G211800,其中Glyma.04G198400在耐低磷品种‘ZH15’低磷处理28 和49 d 时表达量较高,在不耐低磷品种‘NMH’低磷处理7 d时表达量较高;第2 类基因的表达量仅在其中1 个品种的根系中存在上升趋势,如Glyma.04G238100和Glyma.12G 234500;第3 类基因的表达量在低磷处理前后大豆根系中没有发生明显改变,表明其可能不参与大豆抵抗低磷逆境胁迫。
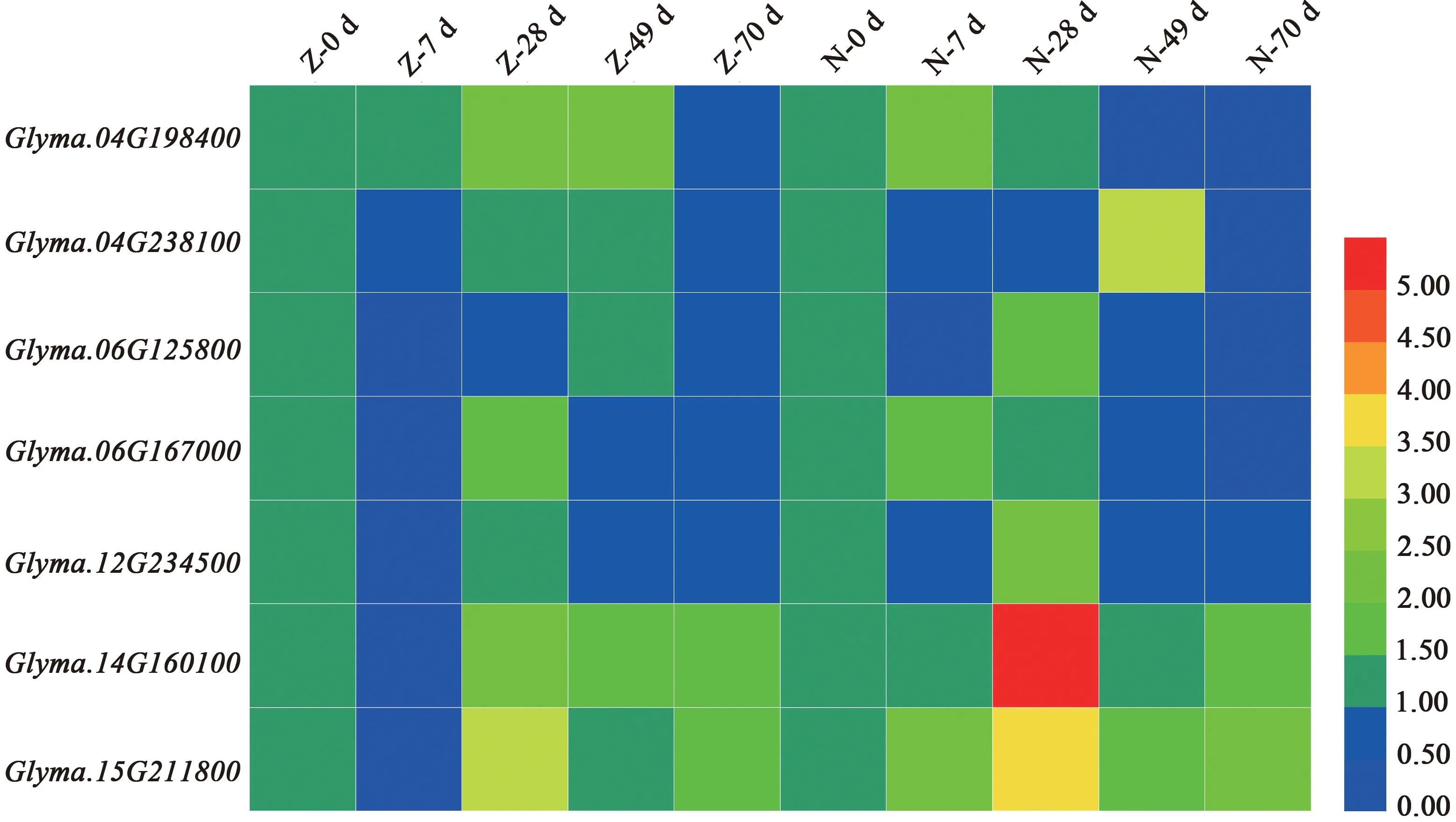
图5 大豆SWEET基因在低磷胁迫处理不同时间根系的表达Fig. 5 Expressions of SWEET genes in soybean roots at different time-points after low phosphorus stress
2.3 栽培大豆SWEET基因SNP等位变异分析
利用Soybase网站公布的1 556份大豆品种资源重测序数据[21],分析栽培大豆48个SWEET基因内部的SNP,结果(表3)发现,43 个基因总共存在220 个非同义突变SNPs(除Glyma.06g122200、Glyma.08g010000、Glyma.08g025100、Glyma.17g090800和Glyma.18g301100外),其中以Glyma.06G200800中的非同义突变SNPs 数量最多(29 个),其次为Glyma.20G082700(18 个);进一步分析发现,有103 个SNPs 位于33 个SWEET基因的保守结构域(除Glyma.05G202700、Glyma.06G167000和Glyma.08G360400等10 个基因外),可能影响基因的生物学功能,这些SNPs 对揭示SWEET基因生物学功能具有重要意义。

表3 栽培大豆SWEET基因非同义突变SNP分析Table 3 Nonsynonymous SNP of cultivated soybean SWEET genes
3 讨论
目前,关于SWEET基因在提高植物果实和籽粒产量以及抗病、抗逆等方面的研究已有报道[22-26]。大豆是世界重要粮油作物,既可采摘鲜荚食用又可收获成熟籽粒,在各国农业生产中占有不可替代的地位[27]。本研究利用大豆公共数据库公布的最新版本栽培大豆与野生大豆基因组数据分析SWEET基因,同时结合栽培大豆转录组数据,分析SWEET基因在豆荚和籽粒不同发育时期以及叶片花叶病毒侵染和根系低磷胁迫处理下的表达,明确SWEET基因在上述几种情况下的表达模式,筛选可能参与大豆荚果与种子发育以及抗病抗逆候选基因,为进一步发掘利用SWEET基因,提高菜用和粒用大豆产量以及抗病抗逆能力提供依据。
有关SWEET基因在大豆豆荚和种子发育中的功能目前报道较少。Hooker 等[28]研究SWEET基因在大豆叶片的表达以筛选在籽粒蛋白质合成和积累过程中的候选基因。Wang 等[29]研究发现,GmSWEET15a和GmSWEET15b在大豆子叶期的胚乳中优势表达,与胚乳向胚胎运输糖类物质有关,基因的双突变引起胚胎发育迟缓及种子败育。Patil 等[14]利用‘Williams82’最早版本基因组预测出52 个大豆SWEET基因(GmSWEET1~GmSWEET52),位于15 条染色体。本研究中,利用‘Williams82’最新版本基因组预测出48 个SWEET基因,也位于15 条染色体。通过比较发现,本研究中有43 个SWEET基因可对应到Patil 等[14]预测的GmSWEETs,有5 个SWEET基因属于新预测的基因,分别 为Glyma.03G065500、Glyma.05G202600、Glyma.06G200800、Glyma.08G360500和Glyma.20G082700;而Patil 等[14]预测的GmSWEETs中有9 个在‘Williams82’最新版本基因组中没有找到。Wang 等[29]曾利用37 个大豆SWEET基因与拟南芥和水稻SWEET基因构建系统进化树,其中Glyma.08G360500也不能对应到Patil 等[14]预测的GmSWEETs中,与本研究结果一致。Hooker等[28]在研究大豆SWEET基因时发现,Patil 等[14]预测的GmSWEET3、GmSWEET18和GmSWEET27在‘Williams82’第二版本基因组(Wm82.a2.v1)中找不到对应的基因ID,本研究中这3 个基因在‘Williams82’最新版本基因组(Wm82.a4.v1)中也找不到对应的ID,可见不同版本预测结果存在一定差别。分析原因,一方面可能是由于不同版本的‘Williams82’基因组测序和组装技术不同;另一方面也可能与不同版本‘Williams82’基因组测序所用材料差别有关[15,30]。
关于大豆SWEET基因在不同组织器官的表达,Patil 等[14]利用‘Williams82’转录组数据分析其预测的52 个SWEET基因发现,有23 个基因在各组织器官的表达量非常低,而GmSWEET21和GmSWEET24在各组织器官的表达量较高,并且发现很多SWEET基因在花和种子形成及发育过程中呈现高表达,暗示其在大豆生殖生长阶段具有重要功能。在本研究中,对4 个大豆品种豆荚和籽粒发育的5 个时期转录组数据分析发现,与籽粒发育ST1时期(开花后24 d)相比,Glyma.08G025100、Glyma.13G041300和Glyma.14G120300等基因的表达量随籽粒发育进程而增加;同时发现,与豆荚发育PT1时期(开花后5 d)相比,Glyma.06G122200和Glyma.14G159900等基因的表达量随豆荚发育进程而增加。进一步比较本研究和Patil等[14]研究结果发现,二者存在一定程度的一致性,如有些SWEET基因在本研究的4 个品种以及Patil 等[14]研究的Williams82 豆荚和籽粒不同发育时期的表达量都非常低,如Glyma.02G088400(GmSWEET1)、Glyma.05G036500(GmSWEET9)、Glyma.13G002700(GmSWEET32)、Glyma.19G009800(GmSWEET47)和Glyma.19G232200(GmSWEET49)等,说明这些基因可能参与其他组织器官发育过程或参与某些特定条件下的糖类运输;还有一些SWEET基因在豆荚或籽粒不同发育时期始终保持较高水平的表达量,如Glyma.08G183500(GmSWEET24)等。更重要的是,有些SWEET基因随豆荚或籽粒发育进程呈现增加,可作为进一步研究的重要候选基因。另外,本研究比较在豆荚和籽粒中随发育进程而表达量增加的基因发现,二者不尽相同,说明在大豆豆荚发育和籽粒发育过程中可能存在不同的糖类物质运输蛋白。
目前,SWEET基因是否参与大豆抗花叶病毒尚不明确。孙玥[31]利用花叶病毒JLCC 株系接种‘吉大豆1 号’14 d 后发现,叶片出现花叶,并伴随叶缘下卷、顶部叶片皱缩以及植株矮化;经检测在接种14 d 后,有33 个SWEET基因上调表达,19个下调表达,说明SWEET基因对SMV侵染的响应不同。本研究利用黄淮海生态区SMV流行株系SC7接种抗病品种‘QH30’和感病品种‘Z92116’,分析SWEET基因在不同接种时间表达量发现,不同基因对接种SC7的响应存在差别。孙玥[31]研究发现,‘吉大豆1号’在接种JLCC株系14 d出现上调表达的基因有GmSWEET6、GmSWEET9和GmSWEET20,下调表达的基因有GmSWEET4、GmSWEET12和GmSWEET17等。本研究分析SC7 株系接种336 h的SWEET基因表达发现,Glyma.04G198600(GmSWEET6)在抗病和感病品种中均上调表达,与孙玥[31]研究结果一致;重要的是,本研究发现该基因在抗病品种中的表达量明显高于感病品种,表明其可能在大豆抵抗SMV 侵染过程中发挥重要功能。同时,本研究还发现,Glyma.08G009900(GmSWEET20)在抗病品种‘QH30’接种SC7 株系3 h 出现第1 次表达高峰,说明该基因能够快速响应SC7 病毒侵染,随后在接种10、24、120 和240 h呈现下降,但在接种336 h 表达量再次出现高峰,而在感病品种‘Z92116’中的表达量在接种前后没有明显变化,说明Glyma.08G009900可能在大豆抗SC7 侵染早期及中后期发挥重要作用。孙玥[31]研究表明,Glyma.08G009900基因在感JLCC株系的‘吉大豆1 号’接种336 h 的表达量也出现上调,表明其可能同时响应JLCC株系。本研究发现了一些新的可能参与大豆抗病的基因,如Glyma.13G264400和Glyma.06G166800,为进一步研究SWEET基因在大豆抗SMV 过程中的功能奠定了基础。
有关大豆SWEET基因如何响应外界低磷逆境目前尚无报道。陈阿[32]分析大豆SWEET基因在低磷处理条件下对菌根接种的响应发现,在低磷胁迫条件下,GmSWEET6和GmSWEET15在丛枝菌根接种后诱导表达,表明其可能与大豆通过丛枝菌根途径提高耐低磷能力有关,但在不接种丛枝菌根的情况下,上述2 个基因在低磷处理的大豆根系基本不表达。本研究通过分析SWEET基因在低磷处理大豆根系的表达情况发现,G1yma.04G198600(GmSWEET6)和G1yma.06G166800(GmSWEET15)在耐低磷和不耐低磷品种根系的表达量都非常低,这与陈阿[32]研究中有关这2 个基因在不接种菌根情况下的结果一致,说明这2个基因不能直接通过大豆根系响应低磷胁迫,而是通过菌根的形式来响应低磷胁迫。本研究获得了在大豆根系中响应外界低磷胁迫处理的SWEET基因,如Glyma.04G198400、Glyma.14G160100和Glyma.15G211800等,为研究该类基因在大豆抵御外界环境低磷逆境中的作用奠定了基础。

