绿豆C3H和NBS转录因子家族成员鉴定及盐胁迫响应分析
刘金洋 林云 陈景斌 闫强 薛晨晨 吴然然 陈新 袁星星

摘要:NBS和C3H是植物體内2个重要的转录因子家族,在调控植物抗病与耐盐方面不可或缺。本研究通过转录组数据分析、qRT-PCR分析,分别鉴定出30个和289个绿豆C3H和NBS家族成员,2个基因家族各有13个基因受到纯化选择,并且C3H和NBS基因家族种内共线性关系均为片段重复。耐盐材料的转录组数据分析结果表明,VrC3H5、VrC3H7、VrC3H10和VrC3H13等4个基因的表达量在盐胁迫后发生显著改变。VrC3H5,VrC3H7和VrC3H13 3个基因对脱落酸(ABA)处理、氯化钠(NaCl)处理、干旱胁迫都有不同程度的响应,VrC3H5在ABA处理后基因表达量上调超过了10倍。在NBS基因中,有85个基因在盐胁迫10 d和15 d后出现显著差异表达,其中9个NBS基因表达变化值|log2FC|(FC为表达倍数变化)大于2。VrNBS20转录因子通过调控EVM0022385参与绿豆的耐盐功能,VrNBS20可能是绿豆抗病与耐盐调控网络中的交叉点。本研究结果为绿豆耐盐与抗病研究提供了丰富的基因资源。
关键词:绿豆;NBS基因家族;C3H基因家族;盐胁迫;VrNBS20转录因子
中图分类号:Q786文献标识码:A文章编号:1000-4440(2023)05-1097-13
Identification and salt stress response analysis of mungbean C3H and NBS transcription factor family membersLIU Jin-yang,LIN Yun,CHEN Jing-bin,YAN Qiang,XUE Chen-chen,WU Ran-ran,CHEN Xin,YUAN Xing-xing
(Institute of Industrial Crops, Jiangsu Academy of Agricultural Sciences/Jiangsu Key Laboratory of Efficient Horticultural Crop Genetic Improvement, Nanjing 210014, China)
Abstract:NBS and C3H are two important transcription factor families in plants, which are indispensable in regulating plant disease resistance and salt tolerance. In this study, through transcriptome data analysis and qRT-PCR analysis, 30 and 289 mungbean C3H and NBS family members were identified, respectively. Thirteen C3H and 13 NBS genes were purified and selected, and the intraspecific collinearity analysis of C3H and NBS gene families was fragment duplication. The transcriptome data analysis of salt-tolerant materials showed that the expression levels of VrC3H5, VrC3H7, VrC3H10 and VrC3H13 were significantly changed after salt stress. VrC3H5, VrC3H7 and VrC3H13 had different degrees of response to abscisic acid (ABA) treatment, sodium chloride (NaCl) treatment and drought stress. The expression of VrC3H5 was up-regulated by more than 10 times after ABA treatment. A total of 85 NBS genes were significantly differentially expressed after 10 days and 15 days of salt stress, of which nine NBS genes had a change in expression value |log2FC| (FC was the expression fold change) greater than two. VrNBS20 was involved in the salt tolerance function of mung bean by regulating EVM0022385, and VrNBS20 may be the intersection point in the disease resistance and salt tolerance regulatory network of mungbean. The results of this study provide abundant genetic resources for the study of salt tolerance and disease resistance of mungbean.
Key words:mungbean;NBS gene family;C3H gene family;salt stress;VrNBS20 transcription factor
绿豆 (Vigna radiata L.),属于豆科豇豆属,含有22条染色体[1]。作为功能性食品原料,绿豆籽粒富含蛋白质、多种人体必需氨基酸、碳水化合物、膳食纤维及生物活性物质。随着人们生活水平的提高和对健康饮食的需求,绿豆的高营养价值开始被人们重视,绿豆的需求量逐渐增大[2]。然而,盐胁迫与病害严重制约着温带、亚热带及热带地区绿豆的生产,尤其以印度、中国、泰国、缅甸和菲律宾等国家为代表。挖掘抗性基因并应用于抗性育种已成为主要农作物抗性育种的主要方法之一。目前,在挖掘绿豆抗性基因方面的研究还不是很深入,有关绿豆耐盐相关的全基因组关联分析和遗传定位的研究还很少。转录因子作为植物的内在调控因子,在植物应对非生物与生物胁迫中具有重要的调控作用。因此,利用比较基因组学挖掘、鉴定绿豆抗病与盐胁迫响应的转录因子对绿豆的遗传改良至关重要。
NBS(Nucleotide binding site)和C3H(CCCH)是植物体内2个重要的转录因子家族,具有独特的功能和结构,在调控植物的抗病与耐盐信号转导方面起重要作用[3-5]。NBS转录因子家族是植物中大转录因子家族之一,含有TIR、NBS和 LRR等结构域。NBS结构域主要介导下游信号转导[6],LRR结构域主要介导蛋白质之间的互作。根据转录因子N端是否含有TIR结构域将其分为两个亚类[7]。一类为含有TIR-NBS-LRR结构域的TNL型;另一类R基因编码的蛋白质通常被CC(Coiled-Coil)替代,又称CC-NBS-LRR(CNL)型[8]。植物体内NBS基因的数量从几十个到上千个不等[8-10]。同时,NBS基因组成的不同会导致植物对病原体抗性形成差异[11]。NBS基因通常包含各种类型的重复,这些重复对NBS基因的功能有着重要影响[12]。NBS转录因子基因已被证明在花生青枯病、大豆花叶病、番茄菟丝子、葡萄霜霉病抗性调控中起着重要作用[13-17]。NBS转录因子基因在植物非生物胁迫中的研究还较少,挖掘具有抗逆和抗病作用的多功能转录因子基因对绿豆的抗病和耐逆育种至关重要。
C3H家族转录因子属于锌指蛋白,包含1~6个CCCH结构,主要含有3个半胱氨酸和1个组氨酸残基[18-19]。含有C-X7-8-C-X5-C-X3-H基序的C3H蛋白是最大的一类C3H转录因子[20-21]。C3H基因家族参与植物发育和逆境适应等过程,在激素调节下对植物的生长起重要作用。在水稻中,OsDOS(C3H基因)参与茉莉酸(JA)代谢,过表达该基因能显著延缓叶片衰老[22]。在拟南芥中,C3H14和C3H15基因能够调节花药发育和雄性育性[23]。此外,一些C3H锌指蛋白还参与植物的非生物胁迫反应。ZFP1通过维持细胞中离子平衡来调节渗透胁迫,提高植物的耐盐性。C3H型锌指蛋白基因AtSZF1和AtSZF2能够正向调控拟南芥的耐盐性[24]。过表达C3H18后,甘薯对干旱和高盐环境的抗性显著增强[25]。在水稻中,zfp5突变体在盐胁迫和渗透胁迫下,萌发率、主根长、脯氨酸含量、叶绿素含量及活性氧清除酶的活性显著小于野生型,而ZFP5基因过表达株系在盐胁迫和渗透胁迫下,萌发率、主根长、脯氨酸含量、叶绿素含量及活性氧清除酶的活性显著大于野生型[26]。
转录因子在非生物胁迫调控中具有重要作用, WRKY、MYB、C3H和HDzip型转录因子含有相应的顺式作用元件可以介导ABA响应基因的表达,他们在多种植物盐胁迫逆境下被诱导[27]。此外,水稻ERF1转录因子能够与ABA信号通路下游OsABI5基因启动子结合,抑制其表达,增强水稻种子在盐胁迫下的萌发率[28]。上述研究结果表明包括转录因子在内的ABA合成表达调控基因在植物非生物逆境的响应与适应方面发挥了重要的作用。
目前,有關绿豆NBS和C3H基因家族成员组成、序列及进化特征,与盐胁迫响应相关的NBS和C3H候选基因还缺乏研究。本研究以江苏省农业科学院经济作物研究所保存的苏绿1号为材料,鉴定绿豆NBS和C3H基因家族成员,分析NBS和C3H基因家族成员碱基序列及进化特征,并挖掘盐胁迫响应相关的NBS和C3H候选基因,为绿豆的抗病与耐盐育种提供基因资源。
1材料与方法
1.1植物材料和处理
本研究使用的绿豆品种为江苏省农业科学院经济作物研究所保存的苏绿1号,将其种植于28 ℃恒温温室,16 h光期、8 h暗期培养7 d后分别用20%的PEG6000、100 μmol/L的脱落酸(ABA)和100 mmol/L的盐(NaCl)溶液对幼苗进行胁迫处理,以无胁迫为对照,在处理0 h、4 h、12 h、24 h、48 h后随机选取不同处理幼苗5株,取根部组织约0.1 g,进行qRT-PCR分析。
选取2个耐盐材料A1和A3及2个不耐盐材料C1和C2[29],幼苗出芽后第10 d,分别用100 mmol/L盐溶液处理,盐处理后10 d与15 d取上述材料的叶片用以转录组分析,分析NBS和C3H家族基因在盐胁迫下表达变化。
1.2绿豆NBS和C3H基因家族成员筛选、保守基序和进化树分析首先,从网站https://www.arabidopsis.org 和https://www.researchgate.net/publication/347076195_high-quality_genome_assembly_annotation_and_evolutionary_ analysis_of_the_mungbean_Vigna_radiata_genome下载拟南芥NBS和C3H蛋白氨基酸序列和绿豆基因组编码的蛋白质序列,利用NCBI blast软件进行序列比对,设置E-value为1×10-100,筛选得到绿豆NBS和C3H候选基因。其次,利用pfam网站(http://pfam-legacy.xfam.org)分析上述候选基因编码蛋白质的保守结构域,去除编码蛋白质中不含C-X7-8-C-X5-C-X3-H和TIR、NBS、LRR结构域的候选基因。随后利用MEME软件(https://meme-suite.org/meme/tools/meme)和MEGA7软件,采用Neighbor-Joining法,迭代1 000次,对NBS和C3H家族基因进行进化树分析,再利用TBtools软件对NBS和C3H家族基因进行保守基序的聚类分析[30]。
1.3NBS和C3H家族基因染色体定位、启动子和共线性分析利用TBtools软件的GFF3/GTF Gene Position(Info.) Parse模块获取上述候选基因在染色体上的位置。利用PlantCARE网站分析C3H基因家族成员上游启动元件(5′上游选2 000 bp),筛选与植物逆境响应相关的元件,并进行可视化作图。根据拟南芥与绿豆NBS和C3H基因家族成员的序列,分析NBS和C3H家族物种间共线性特点。同时,采用TBtools软件对绿豆NBS和C3H家族物种内共线性特点进行分析,并估算共线性基因的非同义替换率(Ka)和同义替换率(Ks),以Ka/Ks值估测基因家族在进化过程中受到的选择压力。
1.4绿豆NBS和C3H基因家族成员在盐胁迫下表达变化分析使用FlaPure Plant Total RNA Extraction Kit试剂盒(北京金沙生物科技有限公司产品)提取绿豆根部总RNA。使用UnionScript First-strand cDNA Synthesis Mix for qPCR with dsDNase (北京金沙生物科技有限公司产品) 反转录合成绿豆cDNA。使用ChamQ SYBR qPCR Master Mix 试剂(南京诺唯赞生物股份有限公司产品)及ABI prism 7500 real-time PCR System (Thermo Fisher公司产品,美国)进行qRT-PCR分析。PCR扩增程序为:95 ℃ 预变性30 s;95 ℃变性10 s,60 ℃退火10 s,40个循环。基因特异性引物设计采用 Primer 5 软件,使用的内参基因为VrACTIN3 (Vradi03g00210)[29],具体引物信息见表1。每个基因表达量检测设置3个重复。
绿豆NBS和C3H基因家族候选基因相对表达量分析采用 2-△△Ct计算方法,采用FPKM(Fragments Per Kilobase per Million)计算方法分析转录组数据[31]。使用SPSS软件中的t检验法对基因表达水平进行差异显著性分析。
1.5差异转录因子调控基因的预测
将差异表达显著的C3H和NBS转录因子氨基酸序列上传至网站https://www.string-db.org进行转录因子调控基因预测,设置显著作用值为0.40,分析转录因子显著调控的基因,利用cytoscape软件对结果进行可视化作图。
2结果与分析
2.1绿豆C3H和NBS基因家族成员分布
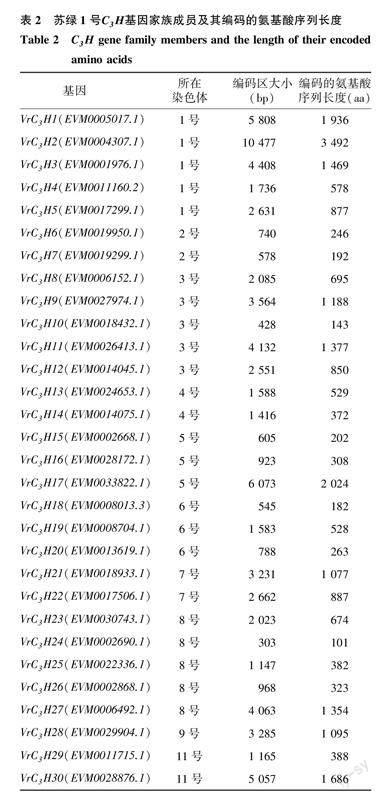
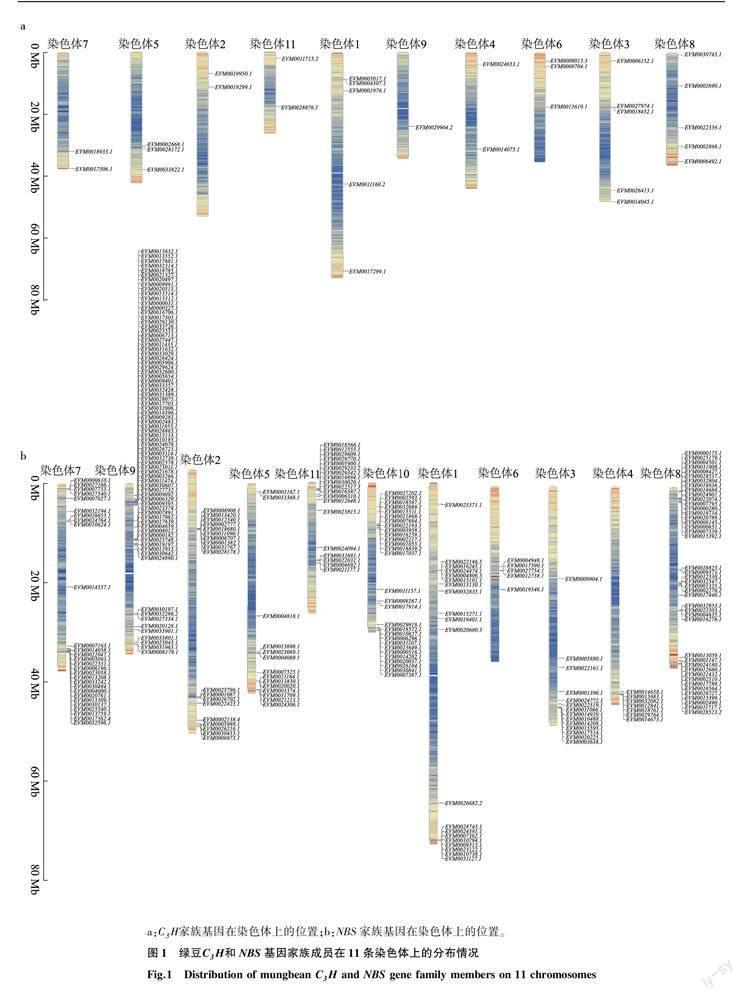
通过NCBI blast比对及MEME、pfam网站保守基序分析,在绿豆全基因组共筛选得到30个C3H基因(表2),289个NBS基因。30个C3H基因主要分布在1号、3号、5号、6号、8号染色体上,10号染色体没有C3H基因,9号染色体仅有1个C3H基因EVM0029904.1,1号、3号和8号染色体上分别有5个C3H基因(图1a)。289个NBS基因主要分布在1号、7号、8号、9号和10号染色体上,4号和6号染色体分别有7个和5个NBS基因(图1b),另有14个NBS基因分布在重叠群上。
2.2C3H和NBS基因家族进化树及保守结构分析
C3H和NBS基因家族进化树分析结果显示,C3H家族分为3个分支(图2a),位于同一分支的基因具有较高的同源性和保守型。C3H基因家族不同分支保守基序差异性较大,分支Ⅰ9个基因,含有2种保守基序(基序1和基序2);分支Ⅱ7个基因,含有保守基序4;分支Ⅲ 14个基因,含有保守基序3(图2b、图2c)。NBS基因家族分为两个分支,分支Ⅰ含有成员数为155;分支Ⅱ含有成员数为134(图3)。两分支保守基序差异性较小。虽然NBS基因家族所含的保守结构相似,但是这些基因的碱基序列长度差异性较大。在位于11条染色体上的275个基因中,分支Ⅰ基因的平均长度为3 381.5 bp,分支Ⅱ基因的平均长度为4 070.9 bp,分支Ⅰ和分支Ⅱ成员在基因碱基序列长度方面差异较大,说明NBS基因家族的功能多样性。
C3H基因家族成员上游2 000 bp碱基序列除了具有光响应、逆境和无氧响应元件外,还具有多种植物激素(茉莉酸甲酯、赤霉素、水杨酸及ABA等)响应元件(图2d)。其中有12个C3H基因的启动子区域存在ABA响应元件,14个C3H基因启动子区域存在茉莉酸甲酯响应元件,12个C3H基因的启动子区域存在赤霉素响应元件,14个C3H基因启动子区域存在干旱胁迫响应元件[MBS(CAACTG)]。这些结果说明C3H基因家族与植物的生长发育以及响应逆境有关,这种特征在其他植物中亦有体现[24]。
2.3绿豆和拟南芥C3H与NBS基因家族之间共线性分析绿豆和拟南芥C3H、NBS基因家族共线性分析结果显示,绿豆C3H基因家族和NBS基因家族分别有18个基因和7个基因与拟南芥基因具有共线性(图4a、图4b)。这说明C3H基因家族在基因序列上具有较高的保守性,而NBS基因家族在基因序列上差异性较大,表明其功能可能具有多样性。
基因组内的共线性分析结果表明,分别有13个C3H基因和13个NBS基因存在共线性(圖4c、图4d)。C3H共线性基因占所有成员的43.3%;NBS共线性基因仅占所有成员的4.5%,即NBS基因组内部共线性较差。上述的共线性关系均为片段重复(表3),并且这些基因共线性关系的Ka/Ks值(非同义替换率/同义替换率)均小于1,说明他们均受到纯化选择。
2.4C3H和NBS家族基因盐胁迫下表达特性分析
有85个NBS基因在盐处理10 d后存在显著差异表达,占总家族成员的29.41%;141个NBS基因在盐处理15 d后存在显著差异表达,占总家族成员的48.79%;有85个基因在盐处理10 d和15 d后均出现显著差异表达。在这85个差异表达基因中,有59个基因 的|log2FC|值(FC为差异倍数)大于0.5(图5A),其中46个基因的表达上调,13个基因的表达下调。此外,VrNBS20(EVM0031127.1)、VrNBS134(EVM0032953.1)、VrNBS135(EVM0022303.1)、VrNBS196(EVM0032738.1)、VrNBS167(EVM0033726.1)、VrNBS173(EVM0033029.1)、VrNBS213(EVM0000182.1),VrNBS208(EVM0007891.1)和VrNBS223(EVM0033901.1)等9个基因表达变化幅度较大(|log2FC|>2)。C3H基因家族中有4个C3H基因在盐处理10 d后存在显著差异表达,占总家族成员的13.33%。9个C3H基因在盐处理15 d后存在显著差异表达 (图5B),占总家族成员的40.00%。其中,VrC3H5(EVM0017299.1)、VrC3H7(EVM0019299.1)、VrC3H10(EVM0018432.1)和VrC3H13(EVM0024653.1)4个基因表达水平变化幅度较大。
4个表达水平差异较大的C3H基因VrC3H5、VrC3H7、VrC3H10和VrC3H13在ABA、NaCl、PEG6000处理后的相对表达量变化如图5C所示。VrC3H5、VrC3H7和VrC3H13 3个基因对ABA、NaCl、PEG6000处理有不同程度的响应。VrC3H5对ABA响应较为明显,处理4 h后达到最高值,其表达量变化超过10倍,之后出现下降。VrC3H7在ABA处理后4 h表达量达到最高值,之后出现下降;在NaCl、PEG6000处理后VrC3H7表达量下降显著。VrC3H13在ABA处理后的4 h表达量达到最高值,之后大幅下降,48 h后表达量恢复正常。
2.5差异转录因子调控基因及其互作
在显著作用值>0.4时,有2个C3H差异转录因子调控基因。其中,VrC3H5调控EVM0001847、EVM0020938和EVM0032843基因,且EVM0001847、EVM0020938和EVM0032843这3个基因之间存在显著互作关系(图6A);VrC3H7调控EVM0019967、EVM0006240、EVM0021757和EVM0031747基因,其中EVM0031747与EVM0021757、EVM0019967与EVM0006240存在显著互作关系(图6B);NBS家族中仅有VrNBS20调控EVM0022385基因,而EVM0022385与另外9个基因存在显著互作关系(图6C)。
在C3H转录因子调控的7个基因中,EVM0001847、EVM0032843和EVM0021757在盐处理后出现显著差异表达(图6D)。其中,EVM0001847基因表达量极显著上调。NBS转录因子调控的基因仅EVM0022385,但有5个基因在盐处理后与EVM0022385一样出现显著差异表达。其中EVM0009188基因表达量极显著上调(图6D)。
3讨论与结论
3.1VrC3H5、VrC3H7和VrC3H10基因参与干旱胁迫的响应从低等植物莱茵衣藻到高等植物拟南芥、水稻、玉米等都普遍存在C3H基因[32-34]。C3H型锌指蛋白在调节植物发育和胁迫响应中起着重要作用。过表达水稻OsC3H38基因能显著提高转基因水稻的耐盐性,改善水稻的生理指标[32];盐胁迫会引起拟南芥AtZFP1的表达量上调,过表达AtZFP1基因能显著提高NaCl处理下拟南芥的发芽率和出苗率[24]。在植物应对盐胁迫的代谢过程中,C3H等转录因子发挥着重要的调节功能[35],挖掘绿豆耐盐相关的C3H基因对绿豆的耐盐遗传改良至关重要。植物主要通过ABA信号途径应对外界盐胁迫[36-38],例如,在拟南芥中发现盐胁迫诱导的类胡萝卜素合成,能够提供丰富的ABA前体以确保ABA合成,增强植株的耐盐性[35]。水稻细胞壁纤维素合酶类蛋白OsCSLD4可以增强水稻ABA合成基因的表达,提高水稻耐盐性[39]。
本研究通过序列比对和系统发育树分析,发现30个C3H家族成员可划分成3个亚家族,同一亚家族的基序分布模式基本一致,如I亚家族都具有相同的3个基序,不同亚家族之间基序数量差异较大。这3个亚家族基因在进化过程中均存在片段复制现象,并且均受到纯化选择。C3H家族成员启动子区域均存在干旱胁迫响应元件。
在耐盐转录组数据分析中,发现VrC3H5、VrC3H7、VrC3H10和VrC3H13 4個基因变化幅度较大,同时在盐处理15 d后仍存在显著差异表达。这4个基因对盐处理较敏感,可能是与盐胁迫响应相关的候选基因。
在候选基因的qRT-PCR分析中,VrC3H5在ABA处理后的4 h后表达量达到最高值,表达量变化超过10倍。VrC3H7在ABA处理后的4 h后表达量达到最高值,是对照组的3倍。VrC3H10在ABA处理后的24 h后表达量达到最高值,是对照组的11倍,之后出现下降。在PEG6000和NaCl处理后VrC3H5、VrC3H7和VrC3H13 3个基因的表达量均出现下降,而VrC3H10基因的相对表达量没有显著变化。
转录因子调控基因分析结果表明VrC3H5和VrC3H7参与了耐盐调控网络。VrC3H5、VrC3H7和VrC3H10基因对盐胁迫的响应情况和转录组分析的结果较为一致,表明盐胁迫影响了这些基因的表达。
3.2绿豆VrNBS20可能同时具有抗病和耐盐功能
NBS基因家族在大豆花叶病、葡萄霜霉病、小麦叶锈病等植物病害的抗性调控中具有重要作用[40]。NBS基因是否还参与耐盐调控值得探索。
为了挖掘盐胁迫响应的NBS基因,研究发现有85个NBS基因在盐处理10 d和15 d后均出现显著差异表达。在这85个差异表达基因中,有59个基因盐处理后15 d表达变化值|log2FC|>0.5;9个NBS基因盐处理后15 d表达变化值|log2FC|>2.0。对这些基因的抗病性能还需要进一步分析。
在候选基因的互作网络分析中仅发现1个NBS转录因子(VrNBS20)调控EVM0022385基因。同時,VrNBS20的功能注释为抗烟草花叶病毒蛋白N,而与其同源的拟南芥AT5G17680基因编码的蛋白质对flg22病原菌具有免疫应答作用[41]。与EVM0022385互作的10个基因中有5个基因在盐处理后出现显著差异表达。这5个基因中EVM0009188的表达量在盐处理后出现显著上调,其他4个基因表达量显著下调。说明,VrNBS20可能不仅具有抗病的活性,同时还参与绿豆的耐盐调控。
参考文献:
[1]KANG Y J, KIM S K, KIM M Y, et al. Genome sequence of mungbean and insights into evolution within Vigna species [J]. Nat Commun, 2014, 11(5): 5443-5452.
[2]李瑞国,郭少英,王怀远. 绿豆萌发期蛋白质和维生素C含量及营养价值[J]. 食品研究与开发, 2012, 33(4):4-12.
[3]王天一,王应祥,尤辰江. 植物PHD结构域蛋白的结构与功能特性[J]. 遗传,2021,43(4):323-339.
[4]TAMELING W I, ELZINGA S D, DARMIN P S, et al. The tomato R gene products I-2 and MI-1 are functional ATP binding proteins with ATPase activity [J]. Plant Cell, 2002, 14(11): 2929-2939.
[5]REBOLEDO G, AGORIO A, PONCE DE LEON I. Moss transcription factors regulating development and defense responses to stress [J]. J Exp Bot, 2022, 73(13): 4546-4561.
[6]ZHNAG Y M, CHEN M, SUN L, et al. Genome-wide identification and evolutionary analysis of NBS-LRR genes from dioscorea rotundata[J]. Frontiers in Genetics, 2020, 11:484-495.
[7]EITAS T K, DANGL J L. NB-LRR proteins: pairs, pieces, perception, partners, and pathways [J]. Curr Opin Plant Biol, 2010, 13(4): 472-477.
[8]汪结明,江海洋,赵阳,等. 玉米自交系B73全基因组NBS类型抗病基因分析[J]. 作物学报, 2009, 35(3): 5-10.
[9]刘云飞,万红建,李志邈,等. 植物NBS-LRR抗病基因的结构、功能、进化起源及其应用[J]. 分子植物育种, 2014, 12(2):377-389.
[10]ANDERSEN E J, LINDSEY L E, NEPAL M P. Genome-wide identification of disease resistance genes (R Genes) in wheat[J/OR]. Cold Spring Harbor Laboratory, 2020. Doi:10.401/2020.07.18.210286.
[11]JUPE F. The potato NB LRR gene family. Determination, characterisation and utilisation for rapid identification of novel disease resistance genes[D]. Norwich, England: University of East Anglia, 2012.
[12]LEISTER D. Tandem and segmental gene duplication and recombination in the evolution of plant disease resistance gene[J]. Trends in Genetics Tig, 2004, 20(3):116-122.
[13]ZHANG C, CHEN H, CAI T, et al. Overexpression of a novel peanut NBS-LRR gene AhRRS5 enhances disease resistance to Ralstonia solanacearum in tobacco [J]. Plant Biotechnol J, 2017, 15(1): 39-55.
[14]DIAO P, SUN H, BAO Z, et al. Expression of an antiviral gene GmRUN1 from soybean is regulated via Intron-mediated enhancement (IME) [J]. Viruses, 2021, 13(10):2032-2046
[15]JHU M Y, FARHI M, WANG L, et al. Heinz-resistant tomato cultivars exhibit a lignin-based resistance to field dodder (Cuscuta campestris) parasitism [J]. Plant Physiology, 2022, 189(1): 129-151.
[16]QU J, DRY I, LIU L, et al. Transcriptional profiling reveals multiple defense responses in downy mildew-resistant transgenic grapevine expressing a TIR-NBS-LRR gene located at the MrRUN1/MrRPV1 locus [J]. Horticulture Research, 2021, 8(1): 161-173.
[17]TENTE E, EREFUL N, RODRIGUEZ A C, et al. Reprogramming of the wheat transcriptome in response to infection with claviceps purpurea, the causal agent of ergot [J]. BMC Plant Biology, 2021, 21(1): 316-336.
[18]WANG D, GUO Y, WU C, et al. Genome-wide analysis of CCCH zinc finger family in Arabidopsis and rice [J]. BMC Genomics, 2008, 9(1): 44-64.
[19]DANIELS B R, PERKINS E M, DOBROWSKY T M, et al. Asymmetric enrichment of PIE-1 in the Caenorhabditis elegans zygote mediated by binary counterdiffusion [J]. Journal of Cell Biology, 2009, 184(4): 473-479.
[20]皮博藝,阮颖,黄勇. 植物串联CCCH锌指蛋白RR-TZF家族研究进展及生物信息学分析[J]. 分子植物育种, 2019, 17(7):2171-2177.
[21]蒋明,刘青娥,章燕如,等. 青花菜C3H型锌指蛋白基因BoCCCH2的克隆与表达[J]. 浙江大学学报(农业与生命科学版), 2016,42(2):143-149.
[22]KONG Z, LI M, YANG W, et al. A novel nuclear-localized CCCH-type zinc finger protein, OsDOS, is involved in delaying leaf senescence in rice[J]. Plant Physiology, 2006, 141(4):1376-1388.
[23]CHAI G H, KONG Y Z, ZHU M, et al. Arabidopsis C3H14 and C3H15 have overlapping roles in the regulation of secondary wall thickening and anther development.[J]. Journal of Experimental Botany, 2015, 66(9):2595-2609.
[24]SUN J, JIANG H, XU Y, et al. The CCCH-type zinc finger proteins AtSZF1 and AtSZF2 regulate salt stress responses in Arabidopsis[J]. Plant and Cell Physiology, 2007, 48(8): 1148-1158.
[25]ZHANG H, GAO X, ZHI Y, et al. A non-tandem CCCH-type zinc-finger protein, IbC3H18, functions as a nuclear transcriptional activator and enhances abiotic stress tolerance in sweet potato[J]. New Phytologist, 2019, 223(4):1918-1936.
[26]刘冬冬. 锌指蛋白GIS和ZFP5响应非生物胁迫的分子机理研究[D].杭州:浙江大学, 2019.
[27]HOANG X L T, NHI D N H, THU N B A, et al. Transcription factors and their roles in signal transduction in plants under abiotic stresses [J]. Current Genomics, 2017, 18(6): 483-497.
[28]LI Y, ZHOU J, LI Z, et al. Salt and ABA response ERF1 improves seed germination and salt tolerance by repressing ABA signaling in rice [J]. Plant Physiology, 2022, 189(2): 1110-1127.
[29]LIU J, XUE C, LIN Y, et al. Genetic analysis and identification of VrFRO8, a salt tolerance-related gene in mungbean [J]. Gene, 2022, 836:146658-146670.
[30]MORTAZAVI A, WILLIAMS B A, MCCUE K, et al. Mapping and quantifying mammalian transcriptomes by RNA-Seq[J]. Nat Methods,2008,5(7): 621-628.
[31]LI S, LIU J, XUE C, et al. Identification and functional characterization of WRKY, PHD and MYB three salt stress responsive gene families in mungbean (Vigna radiata L.) [J]. Genes (Basel),2023, 14(2): 463-481.
[32]潘曉雪,蒋晓英,胡明瑜,等. 水稻OsCCCH基因家族的组织表达谱及胁迫诱导表达特征研究[J]. 分子植物育种, 2016,14(9):2239-2249.
[33]吴学闯,曹新有,陈明,等. 大豆C3HC4型RING锌指蛋白基因GmRZFP1克隆与表达分析[J]. 植物遗传资源学报, 2010, 11(3):343-348
[34]郭栋,宋雅菲,张佳阔,等. 玉米CCCH基因家族鉴定及分析[J]. 中国农业科技导报, 2019, 21(8): 19-27.
[35]RUIZ-SOLA M A, ARBONA V, GOMEZ-CADENAS A, et al. A root specific induction of carotenoid biosynthesis contributes to ABA production upon salt stress in arabidopsis [J]. PLoS One, 2014, 9(3): e90765.
[36]BARRERO J M, RODRIGUEZ P L, QUESADA V, et al. Both abscisic acid (ABA)-dependent and ABA-independent pathways govern the induction of NCED3, AAO3 and ABA1 in response to salt stress [J]. Plant, Cell and Environment, 2006, 29(10): 2000-2008.
[37]NAKASHIMA K, SHINWARI Z K, SAKUMA Y, et al. Organization and expression of two Arabidopsis DREB2 genes encoding DRE-binding proteins involved in dehydration- and high-salinity-responsive gene expression [J]. Plant Molecular Biology, 2000, 42(4): 657-665.
[38]GONG Z, XIONG L, SHI H, et al. Plant abiotic stress response and nutrient use efficiency [J]. Science China(Life Sciences),2020, 63(5): 635-674.
[39]ZHAO H, LI Z, WANG Y, et al. Cellulose synthase-like protein OsCSLD4 plays an important role in the response of rice to salt stress by mediating abscisic acid biosynthesis to regulate osmotic stress tolerance [J]. Plant Biotechnol J, 2022, 20(3): 468-484.
[40]兰冬雪,汤丽影,李佳,等. 禾本科植物NBS-LRR类抗病基因结构,功能和进化研究进展[J]. 中国农学通报, 2019,35(15):124-127.
[41]NAVARRO L, ZIPFEL C, ROWLAND O, et al. The transcriptional innate immune response to flg22. interplay and overlap with Avr gene-dependent defense responses and bacterial pathogenesis[J]. Plant Physiology, 2004, 135(2): 1113-1128.
(责任编辑:石春林)
——以多重共线性内容为例

