甘蓝型油菜BnKNOX基因家族的鉴定与分析
陈吴钧 刘江栋 蒋凯旋 王幼平 蒋金金
甘蓝型油菜基因家族的鉴定与分析
陈吴钧 刘江栋 蒋凯旋 王幼平 蒋金金*
扬州大学生物科学与技术学院, 江苏扬州 225009
植物特异的KNOTTED-LIKE HOMEOBOX (KNOX)蛋白属于转录调控因子家族, 该家族在植物生长发育过程和各种胁迫应答中发挥着重要的作用。KNOX蛋白中存在4个保守的结构域: TALE (three amino acid loop extension)类型的HD (homeodomain)、ELK结构域, 以及2个亚结构域KNOX1和KNOX2。目前, 甘蓝型油菜的基因家族还未有系统的研究报道。本研究通过生物信息学分析鉴定获得甘蓝型油菜的36个家族成员。通过序列比对和系统发育树分析, 将其分为3个亚家族(I、II和M类)。进化分析表明, 全基因组复制(whole genome duplication, WGD)和片段复制(segmental duplication)是基因家族扩张的主要动力。基于甘蓝型油菜不同发育时期组织/器官的RNA-seq分析发现, 该基因家族的在胚乳和种子发育过程中特异表达, 而在成熟种子中高表达。通过家族的顺式作用元件分析和非生物胁迫条件下的表达模式分析, 本文鉴定获得17个响应干旱和渗透胁迫的成员。
甘蓝型油菜; KNOX; 基因家族; 表达模式; 非生物胁迫
KNOX (KNOTTED1-LIKE HOMEOBOX)是一类编码植物特异转录因子的基因家族, 其特征是在HD (homeodomain)的螺旋1和螺旋2之间存在一个TALE (three amino acid loop extension)基序[1-3]。KNOX家族的命名源自玉米中首次鉴定的编码homeobox蛋白的()基因, 该基因的功能获得性突变可引起玉米叶片的侧脉、叶舌分化异常[4]。现有研究表明基因家族广泛存在于陆生植物和绿藻中[5]。KNOX蛋白均包含4个保守结构域: 位于C末端的TALE类型HD结构域, 负责识别下游靶基因的启动子序列; 位于HD上游的ELK结构域, 可能作为核定位信号参与转录抑制调控和蛋白质互作; MEINOX结构域位于N端, 包含2个亚结构域KNOX1和KNOX2, 可参与蛋白互作和同源二聚化、以及调控靶基因表达[6-8]。陆生植物的KNOX家族成员可分为2类(I和II), 其中I类成员主要在茎尖分生组织(shoot apical meristem, SAM)中表达, 在胚和其他组织中低表达[9]。与I类成员相比, II类基因具有更广泛的表达模式, 其在开花植物的分化组织和成熟器官中均有表达, 而在分生组织中不表达[10]。此外, 在一些双子叶植物(如拟南芥)中还存在缺乏ELK-HD结构域的KNOX亚家族, 即KNAT (knotted-like from) M类[11]。
拟南芥中的基因家族包含()、、和4个I类成员,、、和4个II类成员, 以及1个M类成员()[11]。目前对I类基因的研究较多, 该类基因主要在植物生长发育中起重要调控作用[12-16]。细胞分裂素可诱导SAM中和的表达水平;可通过促进()和()基因表达, 参与调控SAM中细胞分裂素的合成[17-19]。此外,在诱导心皮形成和发育过程中也具有重要的作用[20]。STM通过正调控和表达, 维持干细胞稳态和心皮发育, 且三者存在功能冗余[21-22]。与在维持SAM和器官分化过程中存在功能冗余, 且KNAT6可通过(-)通路参与调控胚的边界建立[23]。对被子植物叶片发育调控的研究表明, 过表达I类基因可导致叶片数量增多; 而该类基因的功能缺失会使叶片过早分化, 导致叶片发育和功能异常[24-29]。拟南芥的II类基因主要促进组织/器官分化、抑制SAM分生能力, 其功能与I类基因相反。II类基因的双突变体和多突变体的子叶叶柄增长、叶片变窄、叶齿增多[29]。家族基因在调控植物的非生物胁迫应答方面也具有重要作用[30-32]。大豆()的()可参与调控植株生长发育, 且在大豆对高温和高湿胁迫的响应中发挥重要作用[30], GmSBH1可能通过与GmBLH4 (bel1-like homeodomain 4)形成异源二聚体调控植物发育和对水分胁迫的响应[31]。杨树(×.)中,家族成员可通过抑制赤霉素(gibberellin, GA)合成, 降低杨树的株高、茎节数、节间长度和叶面积, 提高植株的耐旱性[32]。
甘蓝型油菜(, AACC, 2=38)是芸薹属的重要油料作物, 在世界各地广泛种植, 是植物油、生物柴油、生物燃料和医药产品的重要来源之一[33-34]。该物种是约7500年前由白菜型油菜(, AA, 2=20)和甘蓝(, CC, 2=18)通过自然杂交和染色体加倍产生的异源四倍体[34]。甘蓝型油菜在多倍化过程中经历了复杂的基因复制、染色体倒位和异位等事件, 促进了基因家族的多样化, 提高了物种的适应性[35-36]。目前, 关于甘蓝型油菜基因家族的研究尚未有报道。本研究全面分析了甘蓝型油菜中36个成员的序列结构、保守基序、染色体定位、表达模式及其在基因组复制过程中的共线性关系, 旨在了解响应非生物胁迫的基因家族成员, 为甘蓝型油菜耐逆基因挖掘和耐逆性状改良奠定基础。
1 材料与方法
1.1 甘蓝型油菜BnKNOX基因家族成员的鉴定
从基因组数据库(http://www.genoscope.cns.fr/ brassicanapus)[34]中提取甘蓝型油菜的基因组和蛋白序列, 以植物转录因子数据库(http://planttfdb.gao- lab.org/)中9个拟南芥KNOX蛋白为参考序列, 利用BLASTP同源检索获得甘蓝型油菜的KNOX蛋白序列。根据Pfam (http://pfam-legacy.xfam.org/)数据库中KNOX蛋白的4个保守结构域KNOX1 (PF03790)、KNOX2 (PF03791)、ELK (PF03789)和HD (PF00046)[37], 利用HMMER (https://www.ebi.ac. uk/Tools/hmmer/)鉴定油菜基因组中含有上述结构域的蛋白[38], 并利用NCBI-CDD (https://www.ncbi.nlm. nih.gov/cdd/)[39]筛选至少包含一个及以上结构域的蛋白作为甘蓝型油菜BnKNOX家族成员。
1.2 BnKNOX家族的理化性质和亚细胞定位分析
通过在线工具IPC (http://isoelectric.org/)计算BnKNOX蛋白的分子量(molecular weight, MW)和理论等电点(isoelectric point, pI), 利用在线工具WoLF PSORT (https://wolfpsort.hgc.jp/)预测BnKNOX蛋白的亚细胞定位。
1.3 KNOX家族的系统进化和基因结构分析
从芸薹属基因组数据库(https://www.brassica. info/)和拟南芥数据库(https://www.arabidopsis.org/)获得白菜型油菜、甘蓝和拟南芥家族成员的序列, 利用ClustalX (http://www.clustal.org/clustal2/)对KNOX蛋白序列进行比对, 并利用Jalview (http://www.jalview.org/)软件对比对结果进行编辑。利用MEGA7.0 (https://www.megasoftware.net/)的邻接法(Neighbor-Joining, NJ)构建KNOX的系统发育树,选择-distance和成对删除选项, 并将Bootstrap参数设置为1000。基于甘蓝型油菜基因组数据库(http://www.genoscope.cns.fr/brassicanapus)中提取的BnKNOX序列信息, 利用GSDS 2.0程序(http://gsds. gao-lab.org/)分析的外显子和内含子, 利用MEME (http://meme-suite.org/tools/meme)分析BnKNOX蛋白的保守基序, 并通过TBtools软件对基因结构和蛋白保守基序进行可视化分析[40]。
1.4 BnKNOX基因的启动子顺式作用元件分析
从甘蓝型油菜基因组数据库中获得基因编码序列(coding sequence, CDS)上游2000 bp的基因组DNA序列, 然后利用PlantCare (http://bio- informatics.psb.ugent.be/webtools/plantcare/html/)对基因启动子区域的顺式作用元件进行分析, 使用TBtools软件对元件的数量与分布进行可视化分析[40]。
1.5 BnKNOX基因的染色体定位和共线性分析
从甘蓝型油菜基因组数据库获得每条染色体的长度和基因的位置信息, 利用MG2C (http://mg2c.iask.in/mg2c_v2.0/)绘制的染色体定位图。利用MCScanX分析甘蓝型油菜、拟南芥、白菜型油菜和甘蓝中基因的复制事件和共线性关系。利用TBtools对基因家族组间和组内的共线性关系进行可视化分析[40]。
1.6 BnKNOX基因的组织表达分析
本研究收集了甘蓝型油菜株系“J9712”不同发育期的组织/器官, 包括根、子叶、下胚轴、真叶、茎、茎尖分生组织(shoot apical meristem, SAM)、花苞(3 mm)、花、胚乳、授粉后(days after pollination, DAP)14 d的角果、以及不同发育时期(21、28、35、42和50 DAP)的种子, 基于不同样本的RNA-seq分析(3个生物学重复), 根据每个基因的FPKM (fragments per kilobase of transcript per million fragments mapped)的log10转化值分析的组织表达模式。
1.7 植物材料生长、干旱和渗透胁迫处理
为了研究基因在干旱和渗透胁迫下的表达模式, 本研究将“J9712”种子经70%乙醇和10%次氯酸钠消毒后, 分别接种于含15% PEG-6000或150 mmol L–1甘露醇的1/2 MS (Murashige and Skoog)培养基、以及1/2 MS培养基(对照组), 于16 h光照/ 8 h黑暗、22℃培养14 d, 并分别于接种后第0、3、5、7、9和14天收集叶片, 每个时间点包括3个生物学重复, 所收样本用于基因的表达水平分析。
1.8 RNA提取、cDNA合成和qPCR分析
利用RNA isolater Total RNA Extraction Reagent (Vazyme, 中国)试剂盒提取叶片总RNA, 利用HiScript III RT SuperMix (Vazyme, 中国)试剂盒反转录获得cDNA。利用Primer Premier 5设计基因的qPCR引物(表1), 用AceQ Universal SYBR qPCR Master Mix (Vazyme, 中国)进行基因的qPCR分析。以为内参基因, 使用2–ΔΔCt法计算基因的相对表达量, 每个样本包含3个生物学重复。

表1 本研究中用到的qPCR引物
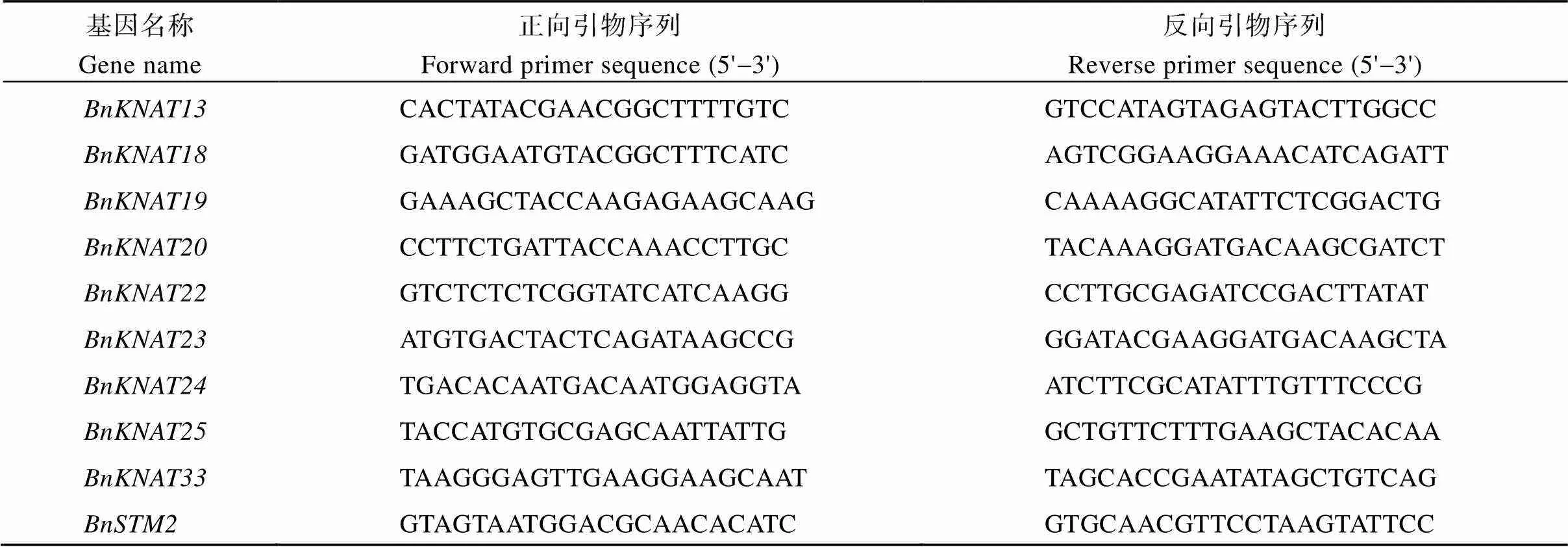
(续表1)
2 结果与分析
2.1 BnKNOX家族的全基因组鉴定与蛋白理化性质分析
甘蓝型油菜的KNOX蛋白包含4个保守结构域, 从N端到C端依次为KNOX1、KNOX2、ELK和HD结构域(图1)。油菜中包含KNOX1、KNOX2、ELK或HD结构域的成员数分别为31、31、22、328个, 但含HD结构域的基因中仅26个成员含其他结构域, 最终获得36个BnKNOX成员(图1)。本研究从植物转录因子数据库中获得9个拟南芥的AtKNOX蛋白, 通过序列同源比对也从甘蓝型油菜基因组获得36个BnKNOX成员。结合上述2种分析方法, 本研究最终鉴定到36个甘蓝型油菜KNOX蛋白(表2), 其中11、6、17个成员分别包含2、3和4个结构域, 而BnKNAT27/29仅具有KNOX1结构域。BnKNOX蛋白长度为102~444个氨基酸不等, 平均蛋白长度为286个氨基酸, 其中BnKNAT9最长,而BnKNAT29最短; 分子质量为11.09~48.91kD, 平均分子质量32.24 kD。BnKNOX蛋白的理论等电点为4.35~8.25。通过WoLF PSORT预测表明36个BnKNOX蛋白都定位于细胞核内(表2)。
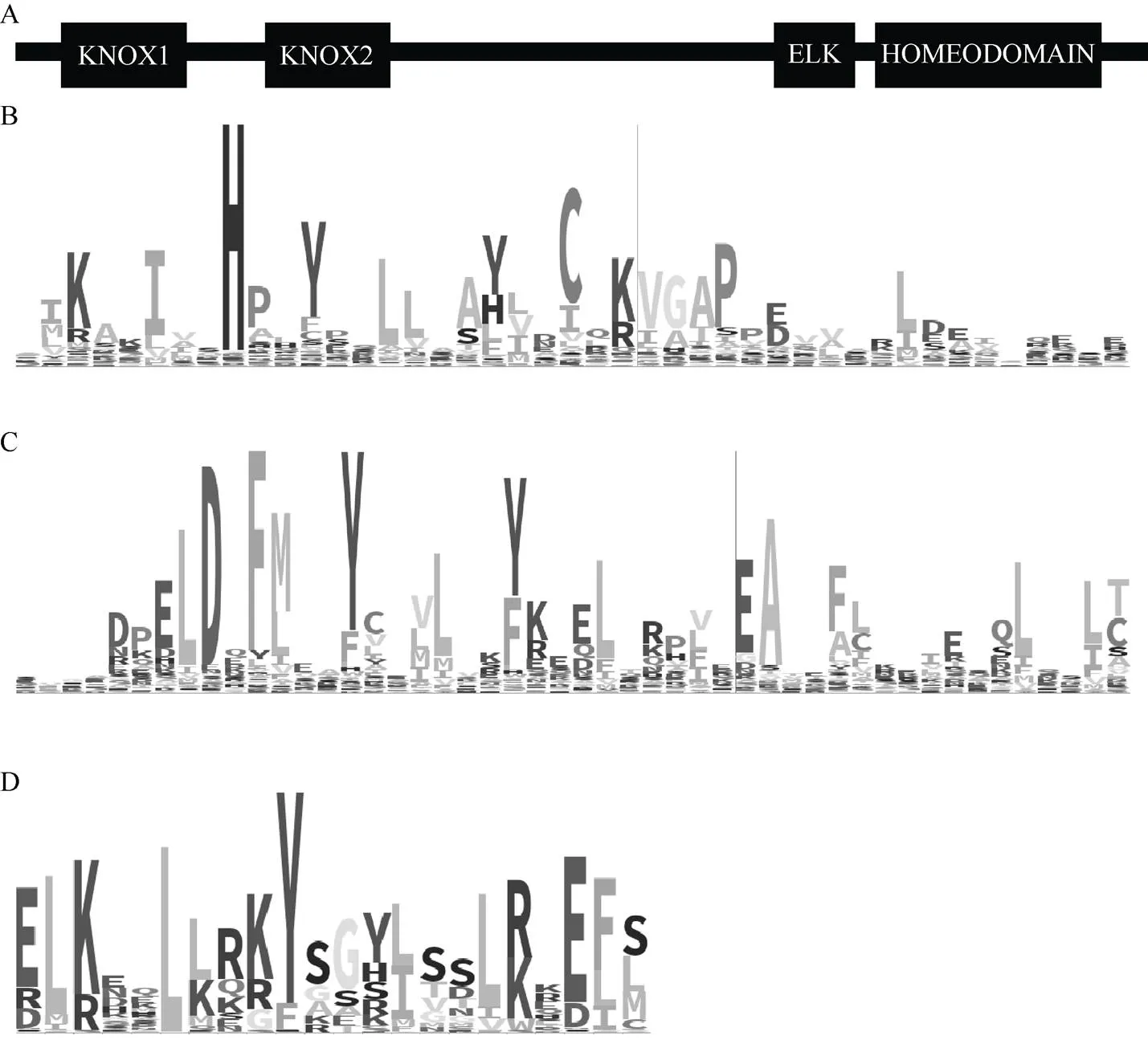
(图1)
A: BnKNOX蛋白结构示意图; B: KNOX1结构域; C: KNOX2结构域; D: ELK结构域; E: HD结构域。
A: the structure diagram of motifs in BnKNOX proteins; B: KNOX1 domain; C: KNOX2 domain; D: ELK domain; E: HD domain.
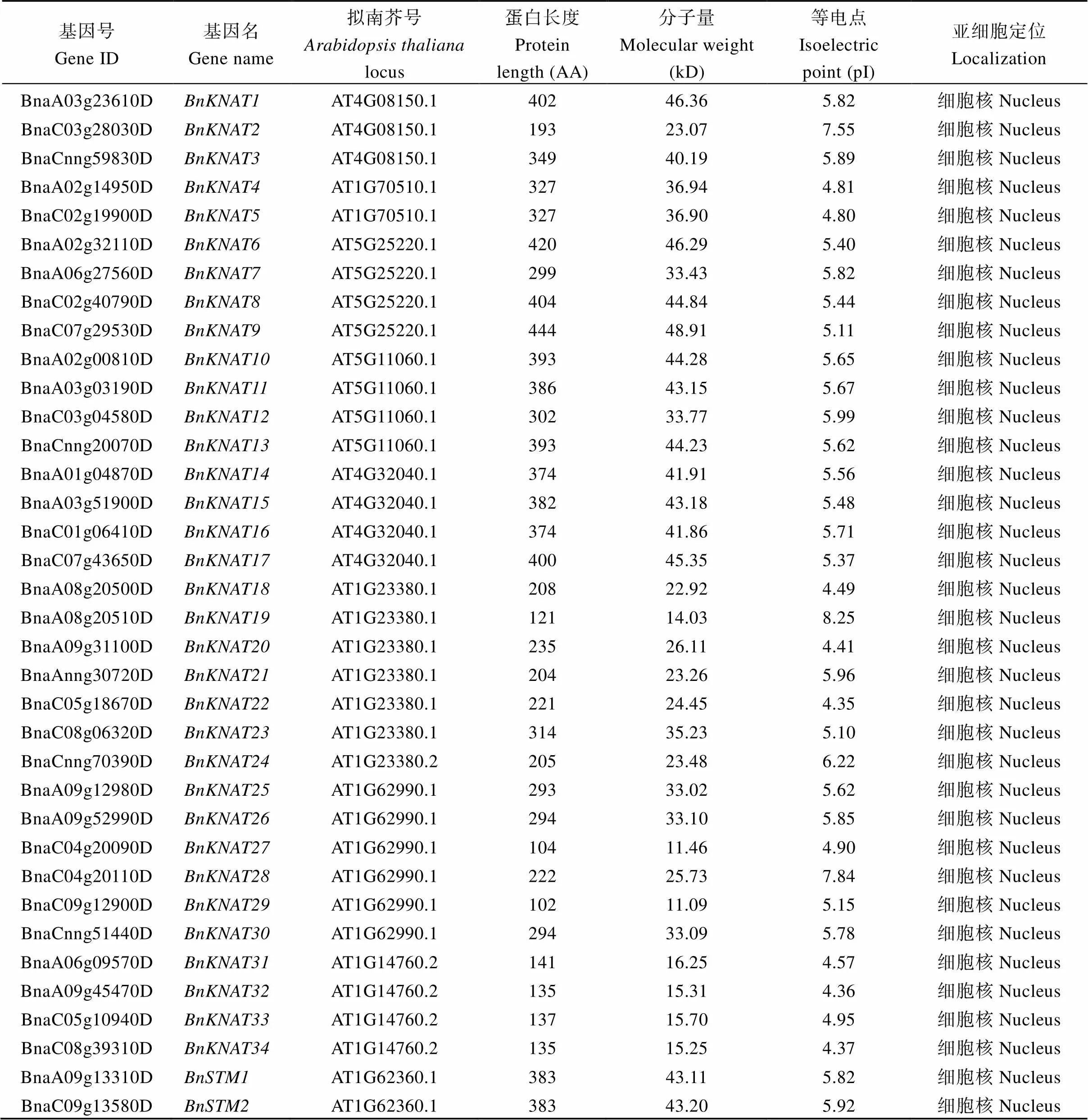
表2 甘蓝型油菜BnKNOX蛋白家族的基本信息
2.2 BnKNOX家族的系统进化分析
本研究构建了BnKNOX成员与9个拟南芥AtKNOX、17个甘蓝BoKNOX和16个白菜型油菜BrKNOX的系统发育树, 表明KNOX蛋白可分为3类, 即I类、II类和M类(图2)。其中, II类的KNOX成员最多, 包括18个BnKNOX、4个AtKNOX、8个BoKNOX和7个BrKNOX, 且BnKNAT9和BnKNAT7、BnKNAT8和BnKNAT6、BnKNAT17和BoKNAT15、BnKNAT15和BrKNAT7有较高的相似性。II类亚族成员中BnKNAT22与BoKNAT9、BnKNAT4与BrKNAT2、BnKNAT5与BoKNAT2具较高的相似性。在M亚族中, BnKNAT33和BoKNAT14、BnKNAT32和BrKNAT15、BnKNAT34和BoKNAT15有较高的相似性。
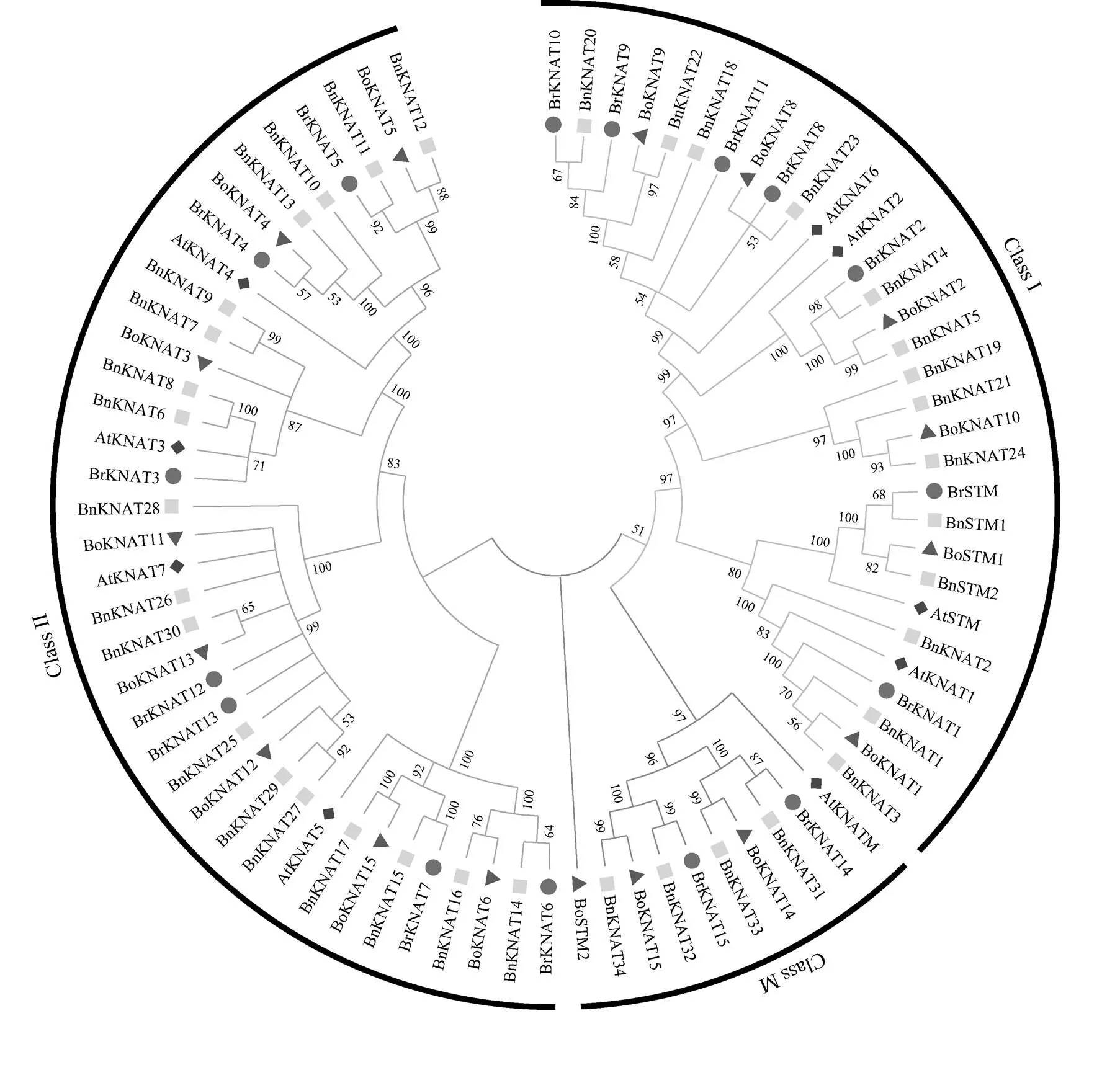
图2 甘蓝型油菜、拟南芥、甘蓝和白菜型油菜中KNOX蛋白的系统进化分析
2.3 BnKNOX基因结构和保守基序分析
外显子-内含子结构变化在基因功能分化中起着重要作用[41]。基因结构分析表明,的外显子数目为1~7个。其中仅含有1个外显子, 而含有7个外显子(图3)。BnKNOX家族成员包含15个保守的蛋白质基序, 每个Motif由10~50个氨基酸构成。在同一亚族中, BnKNOX的保守基序的结构相似, 其中Motif2和Motif3在BnKNOX成员中普遍存在(BnKNAT19/21/24/27/29除外) (图3)。表明甘蓝型油菜基因在进化上较为保守, 且可能存在个别成员的功能分化。
2.4 BnKNOX基因的染色体分布和共线性分析
本研究根据甘蓝型油菜19条染色体的注释信息, 对所鉴定的36个基因进行染色体定位分析(图4), 发现分别有15个基因定位于A和C亚基因组上, 另有6个基因并未定位于特定的染色体位置, 包括1个位于A09_random染色体、1个位于Ann_random染色体、4个位于Cnn_random染色体上的基因。A09染色体包含的数目最多, 包括//和; 而A04、A05、A07、A10和C06染色体上均没有基因。
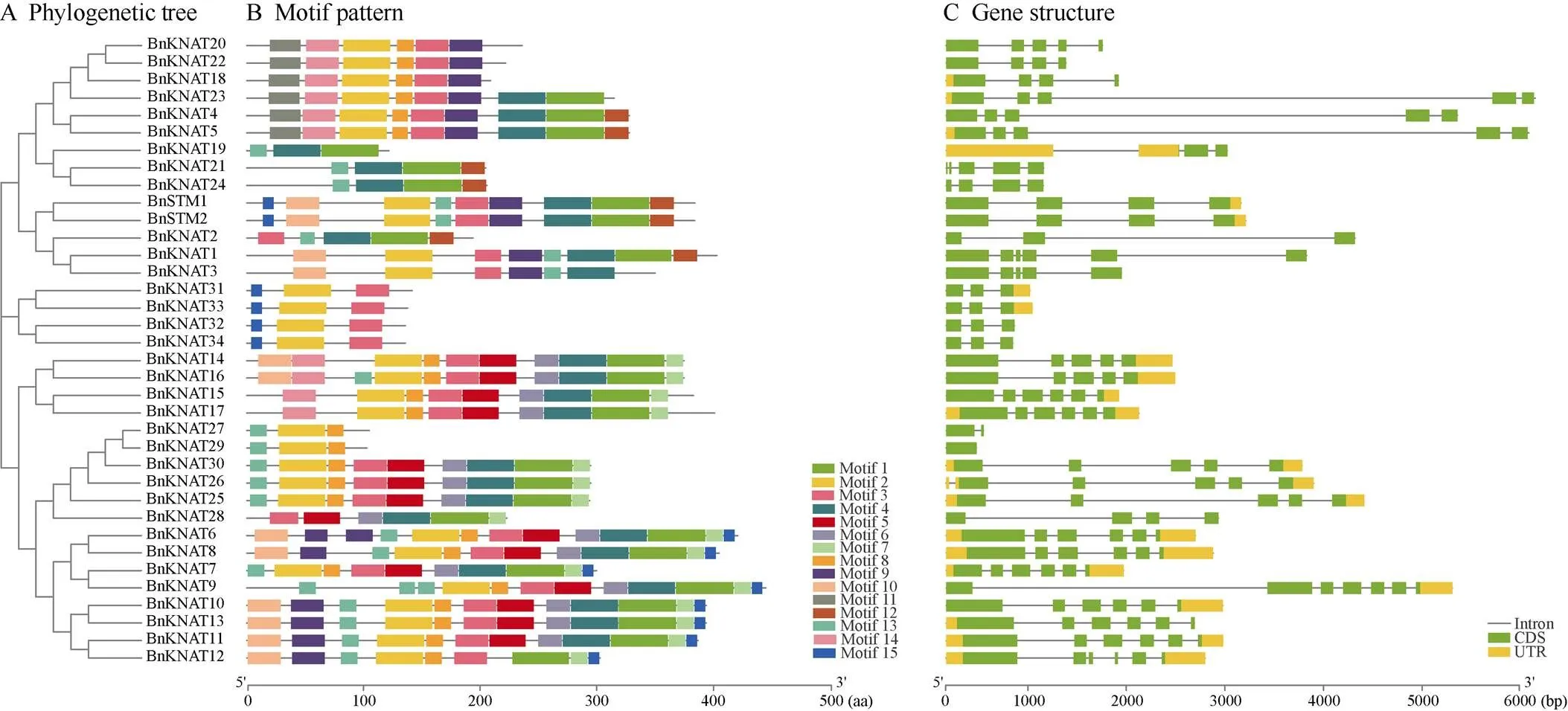
图3 甘蓝型油菜BnKNOX基因家族的基因结构和保守基序分析
A: BnKNOX蛋白的系统进化分析; B: BnKNOX蛋白的保守基序分析, Motif 1~15为不同颜色代表的基序组成; C:基因结构。UTR: 非翻译区; CDS: 编码区。
A: the phylogenetic tree of BnKNOX proteins; B: the conserved motifs of KNOX proteins, Motif 1–15 in different colored blocks represent the motif composition; C: gene structure ofgenes. UTR: untranslated region; CDS: coding sequence.
基因复制现象在植物进化中普遍存在, 并起着不可或缺的作用[42]。本研究对36个基因的复制类型进行分析发现, 其中28个基因源自全基因组复制或片段复制, 其余8个基因来自串联复制事件(附表1)。此外, 对36个基因进行物种内共线性分析, 发现了33个共线性基因对(图4)。为了进一步了解基因家族在十字花科中的进化关系, 本研究分析了其与白菜型油菜、甘蓝和拟南芥中基因的同源关系。共线性分析表明22、27和18个基因分别与、和存在共线性关系(图5)。表明基因复制事件在基因的扩张中起着重要的作用, 其中全基因组复制或片段复制起主要作用。
Ka/Ks可用于计算基因在进化过程中是否受自然选择作用, 即Ka/Ks>1表示存在正选择效应, Ka/Ks=1为中性选择, Ka/Ks<1表示有纯化选择作用。本研究利用TBtools的Ka/Ks Calculator对甘蓝型油菜进行分析发现, 所有基因对的Ka/Ks均小于1。表明在进化过程中可能经历了严格的纯化选择作用, 以确保该基因家族的进化保守性、结构稳定性和功能一致性。
2.5 BnKNOX基因的组织表达分析
基于甘蓝型油菜中不同发育阶段各组织/器官的RNA-seq数据, 本研究分析了基因家族成员的组织表达模式(图6和附表2)。的表达模式大致可分为3种类型: 第1类是在所有组织中均低表达, 如//; 第2类几乎在所有组织中都高表达, 如; 第3类是在大多数组织中表达, 但在特定组织中高表达, 如在下胚轴和茎中高表达,在胚乳和种子发育过程中高表达,在成熟种子中高表达。此外, II类基因的表达水平普遍高于I类基因, 表明II类中的基因可能对油菜的生长发育更为重要。
2.6 BnKNOX基因的启动子顺式作用元件分析
顺式作用元件在调节基因表达中起着重要作用,具有相似功能的基因可能在启动子中包含相同的调节元件[43-44]。基因的启动子区域存在4种类型的顺式作用元件, 包括与植物生长发育、非生物胁迫反应、激素反应相关的启动子元件, 以及真核生物中的基本启动子元件(图7)。本研究发现36个基因的启动子均含有光响应元件, 与激素(如脱落酸、赤霉素、茉莉酸甲酯)响应相关的元件也普遍存在于基因的启动子区域。其中, 27个成员的启动子区含有脱落酸(ABA)相关响应元件, 25个成员含有茉莉酸甲酯(MeJA)响应元件, 14个成员含有水杨酸(SA)响应元件, 5个成员含有赤霉素(GA)响应元件。此外, 部分基因(如)的启动子区域也包含与非生物逆境胁迫(如干旱、低温、厌氧诱导、损伤等)相关的响应元件。综上,基因可能在调节植物生长发育和非生物胁迫应答过程中发挥重要作用。
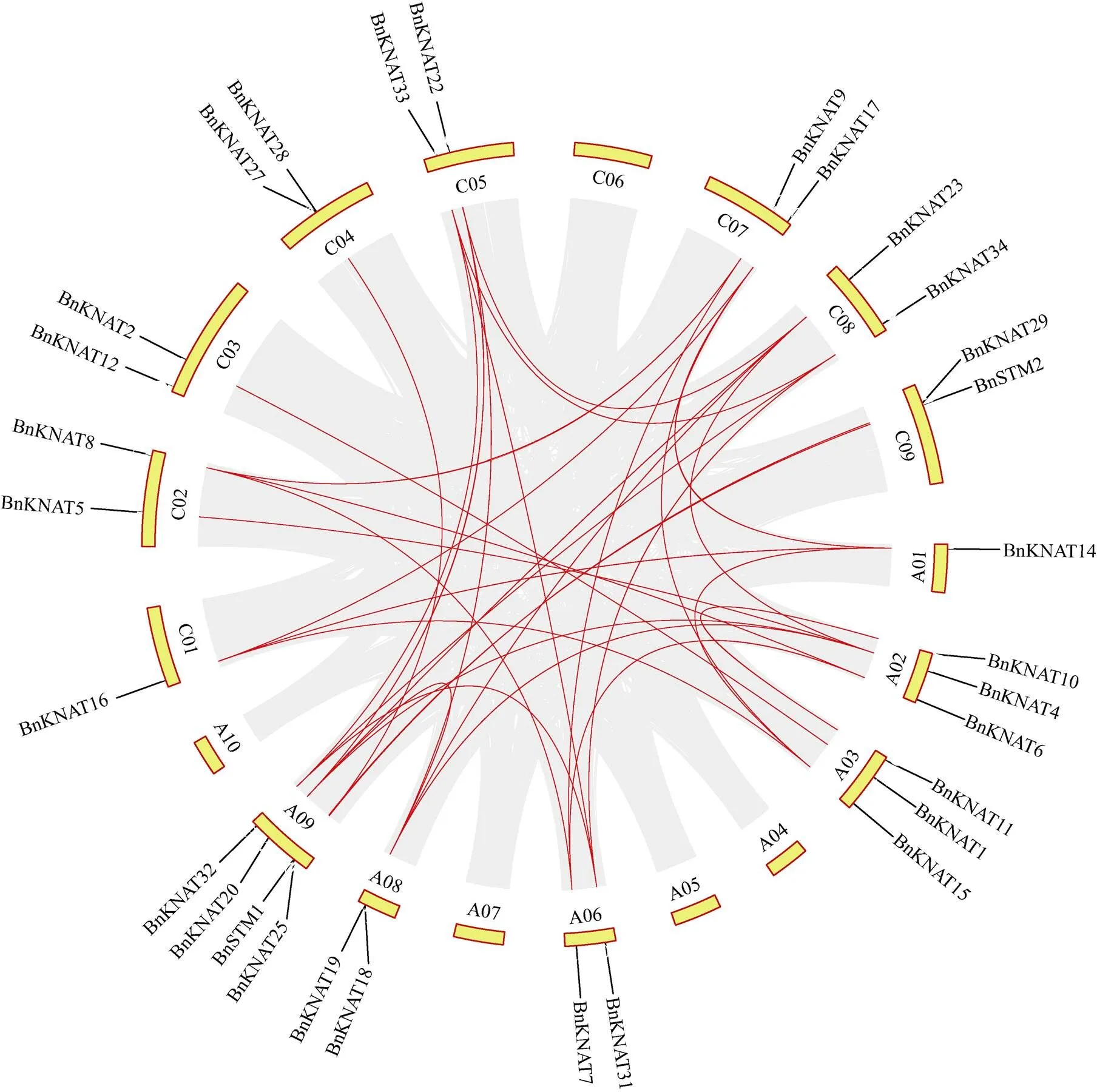
图4 BnKNOX基因的染色体分布及组内共线性分析
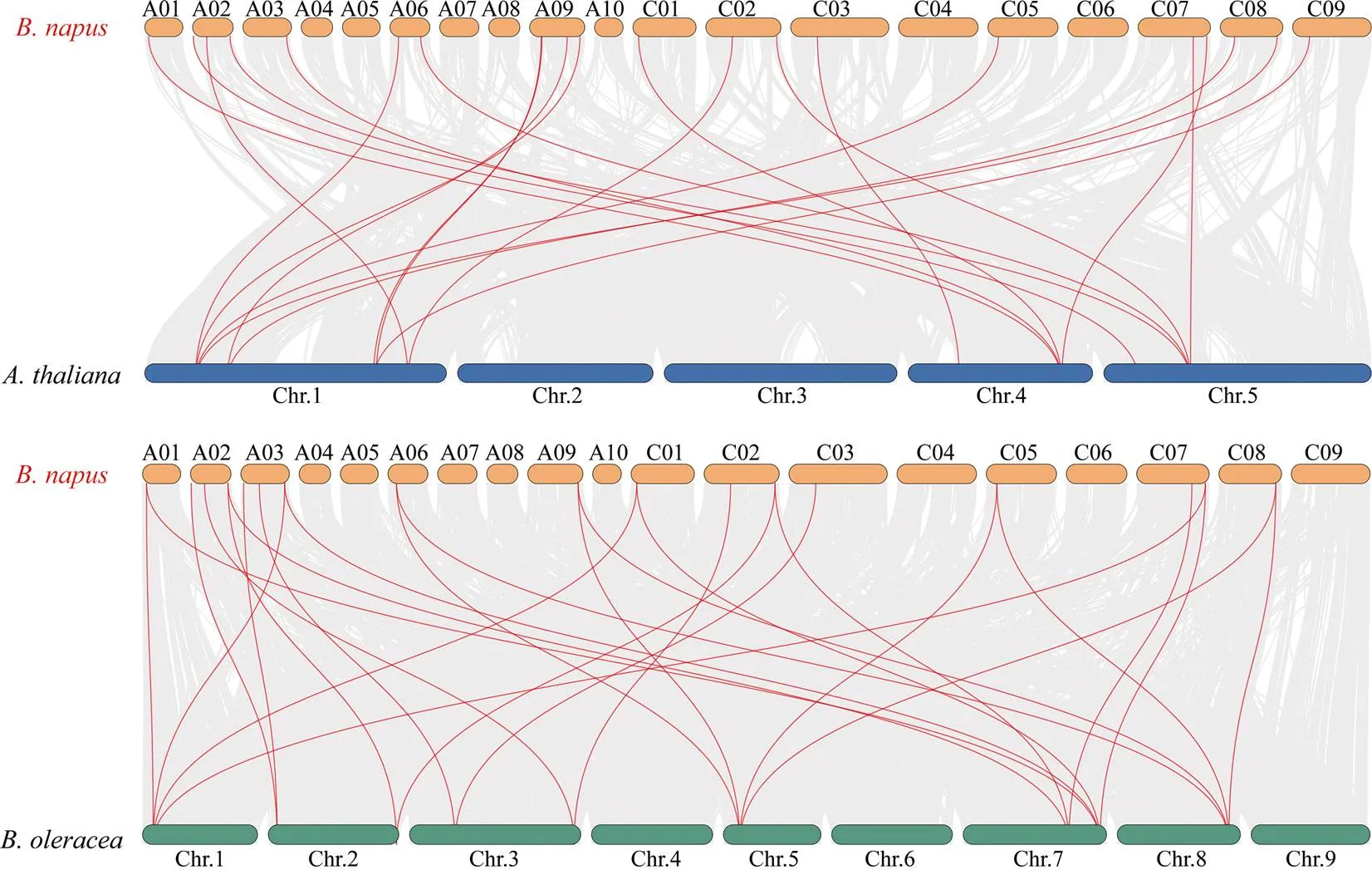
(图5)

图6 BnKNOX基因在甘蓝型油菜不同组织和发育阶段的表达模式
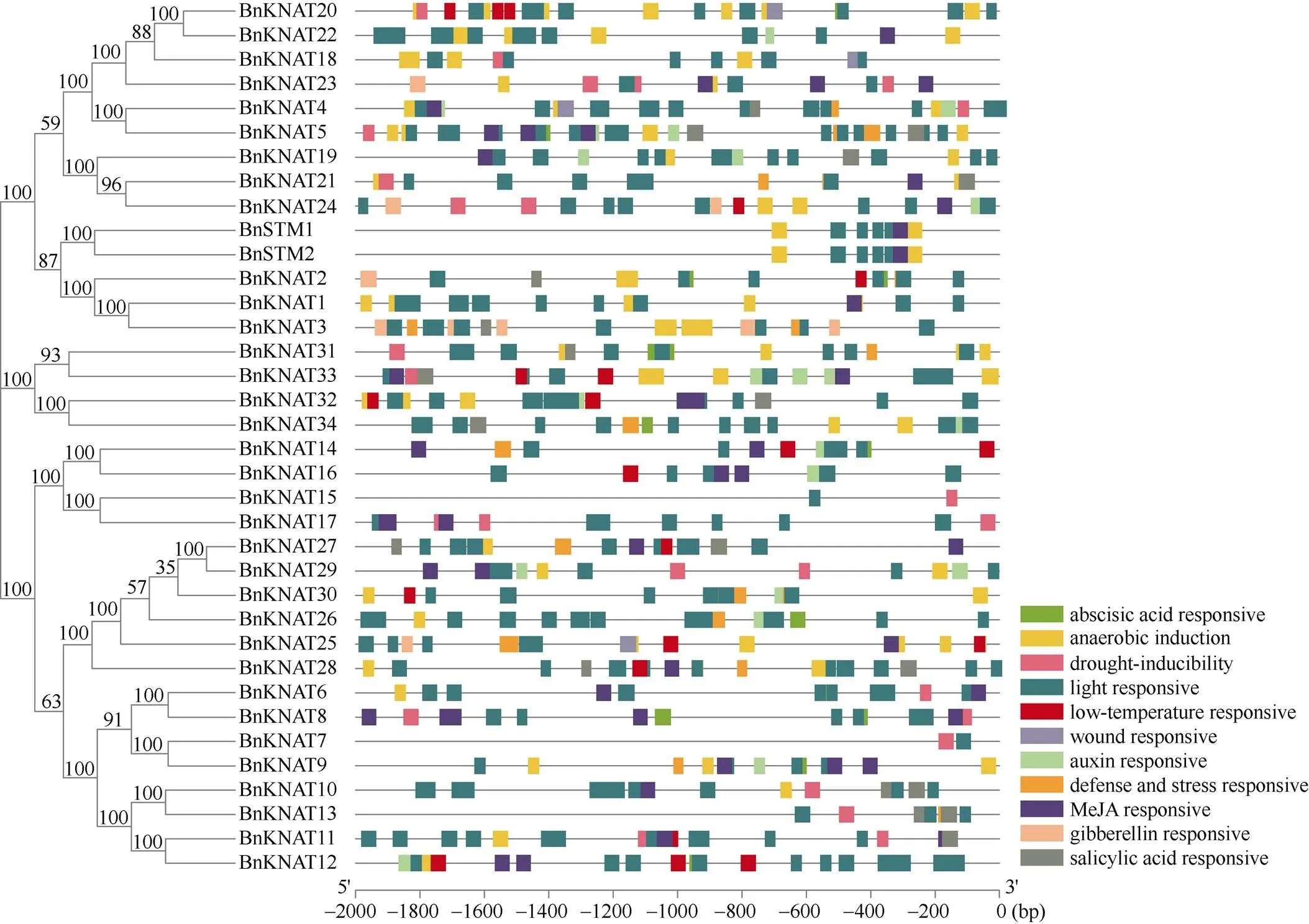
图7 BnKNOX基因的启动子顺式作用元件分析
2.7 干旱和渗透胁迫下BnKNOX基因的表达模式分析
鉴于的启动子顺式作用元件分析表明该基因家族可能在非生物胁迫响应中发挥重要作用。本研究对干旱和渗透胁迫处理后油菜叶片中的表达模式进行了分析(图8和图9)。其中, 17个基因在胁迫处理后3~14 d上调表达, 且部分基因的表达随胁迫时间而上升。其中/和的表达量随着干旱处理时间的增加而上升, 且在胁迫第14天表达量到达最大值;///的表达量也在干旱处理后第14天达到最大值;在干旱处理后第5、第9天表达量最高;在胁迫后第7天表达量最高;在胁迫后第3、第9天表达量最高;/在干旱处理后第9天表达量最高。在渗透胁迫条件下,和的表达量随着胁迫处理时间的增加而上升, 且在胁迫第14天达到最大值;///的表达量也在渗透胁迫处理后第14天达到最大值;在胁迫后第5、第9天表达量最高;在胁迫后第5、第14天表达量最高;在胁迫后第9天表达量最高。表明上述可能参与甘蓝型油菜对干旱和渗透胁迫的响应过程, 这与顺式作用元件分析的结果相一致。
3 讨论
基因在植物基因组中广泛存在, 可参与调控植物激素信号转导、非生物胁迫响应、植物生长发育等多个方面[45]。目前, 关于甘蓝型油菜基因家族尚未有研究报道。本研究通过KNOX的结构域分析和序列比对共鉴定获得36个甘蓝型油菜基因家族成员。BnKNOX成员包含4个保守的结构域KNOX1、KNOX2、ELK和HD, 且大多数成员均含有2个及以上高度保守的结构域, 而BnKNAT27/29只包含KNOX1结构域。此外, 不同亚组所包含的保守结构域的数量也不同, 这可能是导致基因功能分化的主要原因。
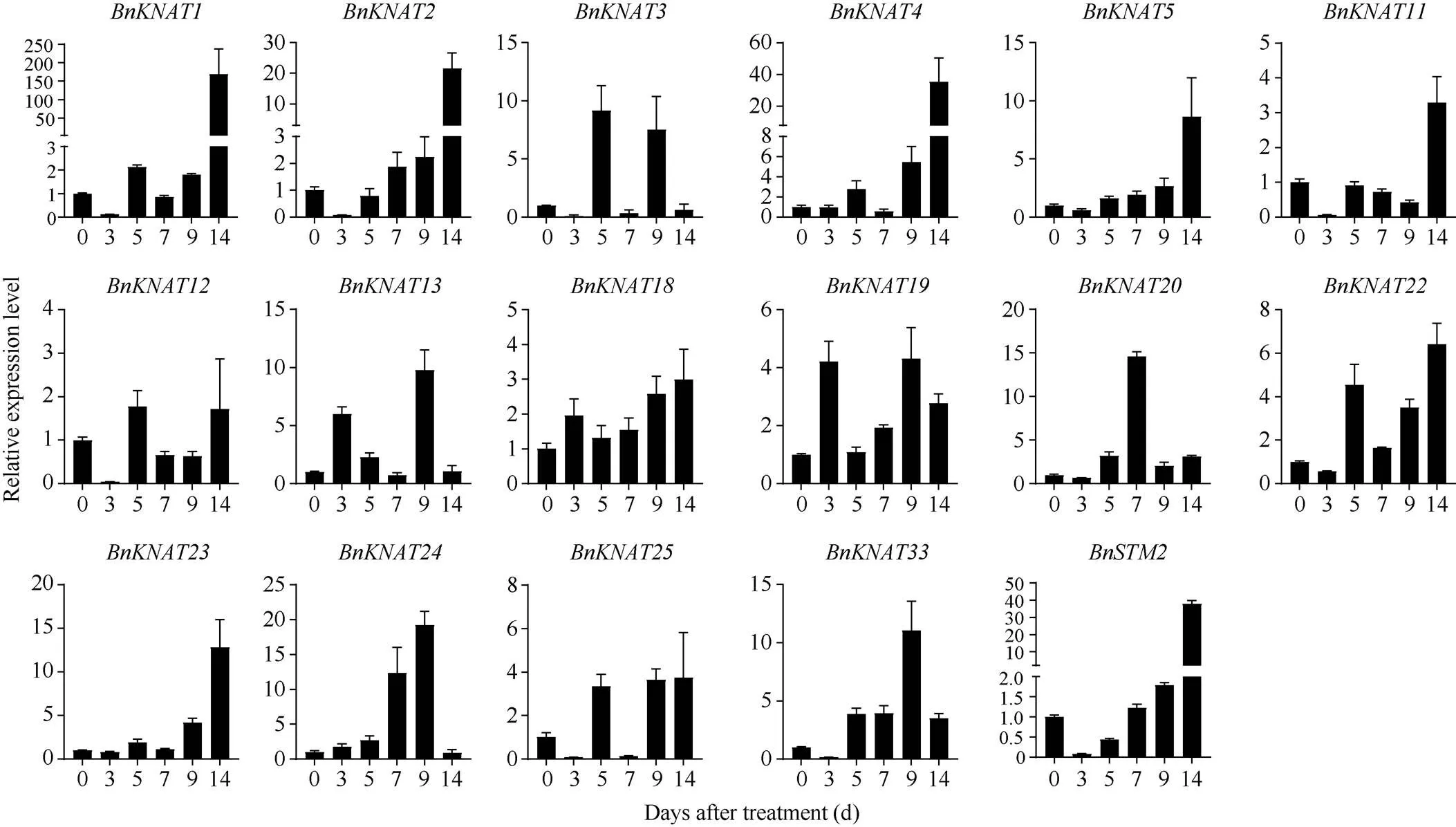
图8 干旱胁迫下甘蓝型油菜叶片中BnKNOX的表达模式
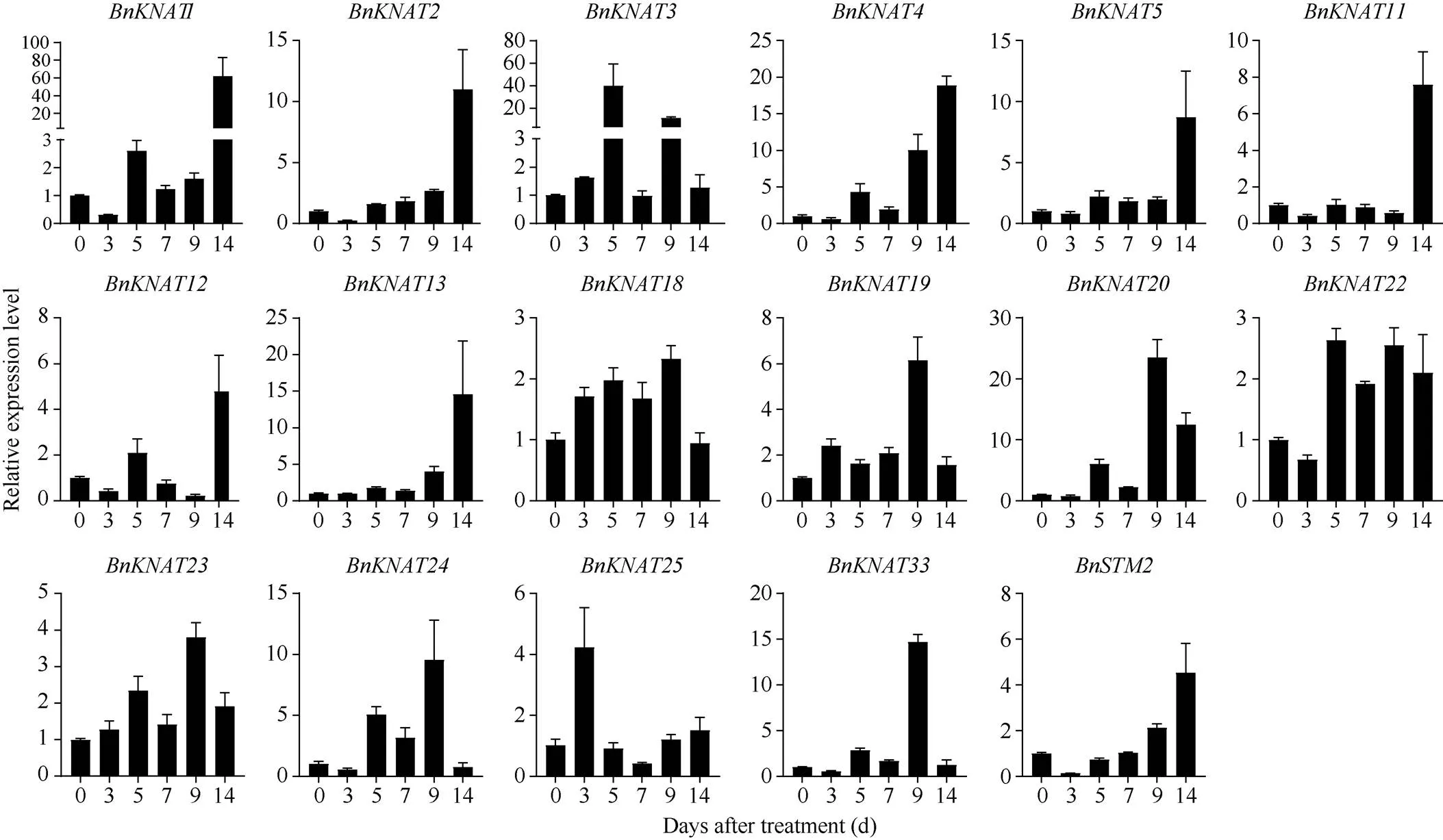
图9 渗透胁迫下甘蓝型油菜叶片中BnKNOX的表达模式
基因复制在物种进化中具有重要的推动作用, 主要包括全基因组复制/片段复制和串联复制2种机制[46]。甘蓝型油菜源于2个二倍体种(白菜型油菜和甘蓝)的自然杂交, 且其二倍体亲本也经历了基于拟南芥的多倍体化事件[34]。理论上, 每个拟南芥基因经过全基因组多倍化后在甘蓝型油菜中应存在6个同源基因, 但本研究仅鉴定到36个基因, 部分基因在进化过程中已丢失。这表明基因在油菜进化过程中可能经历了强烈的纯化选择, 而保留的基因成员在油菜中应具有重要的功能。尽管基因在甘蓝型油菜中均有一个以上的直系同源基因, 但仅/存在6个同源基因, 表明基因在甘蓝型油菜进化过程中发生了基因的扩张和丢失等复杂事件。共线性结果分析也表明基因家族成员都是基因复制事件产生的, 其中28个成员来自全基因组复制或片段复制, 其余8个成员来自串联复制。此外, 本研究发现所有基因都经过了严格的纯化选择, 这对于维持该家族的成员数量尤为重要。
本研究发现基因家族的启动子区域存在ABA、MeJA、SA和GA 4种顺式作用元件, 其中分别有27、25、14和5个成员含有这些响应元件。此外, 部分基因(如)的启动子区域具有与非生物逆境胁迫(如干旱、低温、厌氧诱导、损伤等)相关的响应元件, 我们推测基因可能在调节植物生长发育、对非生物胁迫的反应和激素反应中发挥作用。这与前人研究发现大豆成员参与植株对高温和高湿胁迫应答的报道是一致的[30-31]。此外, 在棉花中的研究表明和对植物应答非生物胁迫(如盐、干旱胁迫)的过程具有调控作用, 沉默该基因均会导致叶片细胞死亡, 气孔开放程度增加; 推测和可能通过调控气孔开闭影响棉花对干旱和盐胁迫的响应[47]。在拟南芥中异源表达小麦-可提高植株丙二醛和脯氨酸含量, 增强其耐旱性和耐盐性[48]。本研究基于家族成员的胁迫表达模式分析, 鉴定获得17个响应干旱和渗透胁迫的成员, 表明该基因家族成员可能参与甘蓝型油菜对非生物胁迫(如干旱)的应答过程。通过对这些胁迫应答基因的功能研究, 有望为甘蓝型油菜的抗逆育种奠定基础。
4 结论
本研究系统分析了甘蓝型油菜的36个基因, 并通过共线性分析和进化分析表明油菜的基因家族来源于物种的多倍化, 该基因家族在多倍化扩张的同时也发生了丢失, 且全基因组复制和片段复制是家族扩张的主要动力。通过该基因家族的组织表达模式、胁迫应答模式和启动子元件分析, 表明该基因家族成员在进化过程中可能发生了功能分化。此外, 本研究鉴定获得17个参与油菜对干旱和渗透胁迫响应的成员,这对于油菜的抗逆基因挖掘和性状改良具有一定的意义。
[1] Bürglin T R. Analysis of TALE superclass homeobox genes (MEIS, PBC, KNOX, Iroquois, TGIF) reveals a novel domain conserved between plants and animals., 1997, 25: 4173–4180.
[2] Mukherjee K, Bürglin T R. Comprehensive analysis of animal TALE homeobox genes: new conserved motifs and cases of accelerated evolution., 2007, 65: 137–153.
[3] Mukherjee K, Brocchieri L, Bürglin T R. A comprehensive classification and evolutionary analysis of plant homeobox genes., 2009, 26: 2775–2794.
[4] Vollbrecht E, Veit B, Sinha N, Hake S. The developmental geneis a member of a maize homeobox gene family., 1991, 350: 241–243.
[5] Gao J, Yang X, Zhao W, Lang T, Samuelsson T. Evolution, diversification, and expression of KNOX proteins in plants., 2015, 6: 882.
[6] Sakamoto T, Nishimura A, Tamaoki M, Kuba M, Tanaka H, Iwahori S, Matsuoka M. The conserved KNOX domain mediates specificity of tobacco KNOTTED1-type homeodomain proteins., 1999, 11: 1419–1432.
[7] Nagasaki H, Sakamoto T, Sato Y, Matsuoka M. Functional analysis of the conserved domains of a rice KNOX homeodomain protein, OSH15., 2001, 13: 2085–2098.
[8] Scofield S, Murray J A.gene function in plant stem cell niches., 2006, 60: 929–946.
[9] Kerstetter R, Vollbrecht E, Lowe B, Veit B, Yamaguchi J, Hake S. Sequence analysis and expression patterns divide the maize-like homeobox genes into two classes., 1994, 6: 1877–1887.
[10] Furumizu C, Alvarez J P, Sakakibara K, Bowman J L. Antagonistic roles forandgenes in patterning the land plant body plan following an ancient gene duplication., 2015, 11: e1004980.
[11] Magnani E, Hake S.lost the: thegene defines a novel class of KNOX transcriptional regulators missing the homeodomain., 2008, 20: 875–887.
[12] Bueno N, Alvarez J M, Ordás R J. Characterization of the() gene family inAit., 2020, 301: 110691.
[13] Clark S E, Jacobsen S E, Levin J Z, Meyerowitz E M. Theandloci competitively regulate meristem activity in., 1996, 122: 1567–1575.
[14] Chuck G, Lincoln C, Hake S.induces lobed leaves with ectopic meristems when overexpressed in., 1996, 8: 1277–1289.
[15] Endrizzi K, Moussian B, Haecker A, Levin J Z, Laux T. Thegene is required for maintenance of undifferentiated cells inshoot and floral meristems and acts at a different regulatory level than the meristem genesand., 1996, 10: 967–979.
[16] Long J A, Moan E I, Medford J I, Barton M K. A member of the KNOTTED class of homeodomain proteins encoded by thegene of., 1996, 379: 66–69.
[17] Rupp H M, Frank M, Werner T, Strnad M, Schmülling T. Increased steady state mRNA levels of theandhomeobox genes in cytokinin overproducingindicate a role for cytokinins in the shoot apical meristem., 1999, 18: 557–563.
[18] Jasinski S, Piazza P, Craft J, Hay A, Woolley L, Rieu I, Phillips A, Hedden P, Tsiantis M. KNOX action inis mediated by coordinate regulation of cytokinin and gibberellin activities., 2005, 15: 1560–1565.
[19] Yanai O, Shani E, Dolezal K, Tarkowski P, Sablowski R, Sandberg G, Samach A, Ori N.KNOXI proteins activate cytokinin biosynthesis., 2005, 15: 1566–1571.
[20] Scofield S, Dewitte W, Murray J A. Thegeneis required for the development of reproductive meristematic tissues in., 2007, 50: 767–781.
[21] Scofield S, Dewitte W, Murray J A. A model forclass-1gene function., 2008, 3: 257–259.
[22] Scofield S, Dewitte W, Murray J A. STM sustains stem cell function in theshoot apical meristem and controlsgene expression independently of the transcriptional repressor AS1., 2014, 9: e28934.
[23] Belles-Boix E, Hamant O, Witiak S M, Morin H, Traas J, Pautot V.: anhomeobox gene involved in meristem activity and organ separation., 2006, 18: 1900–1907.
[24] Hareven D, Gutfinger T, Parnis A, Eshed Y, Lifschitz E. The making of a compound leaf: genetic manipulation of leaf architecture in tomato., 1996, 84: 735–744.
[25] Bharathan G, Goliber T E, Moore C, Kessler S, Pham T, Sinha N R. Homologies in leaf form inferred fromgene expression during development., 2002, 296: 1858–1860.
[26] Hay A, Tsiantis M. The genetic basis for differences in leaf form betweenand its wild relative., 2006, 38: 942–947.
[27] Nikolov L A, Tsiantis M. Interspecies gene transfer as a method for understanding the genetic basis for evolutionary change: progress, pitfalls, and prospects., 2015, 6: 1135.
[28] Rast-Somssich M I, Broholm S, Jenkins H, Canales C, Vlad D, Kwantes M, Bilsborough G, Dello Ioio R, Ewing R M, Laufs P, Huijser P, Ohno C, Heisler M G, Hay A, Tsiantis M. Alternate wiring of agenetic network underlies differences in leaf development of.and.., 2015, 29: 2391–2404.
[29] Das Gupta M, Tsiantis M. Gene networks and the evolution of plant morphology., 2018, 45: 82–87.
[30] Shu Y, Tao Y, Wang S, Huang L, Yu X, Wang Z, Chen M, Gu W, Ma H., a homeobox transcription factor gene, relates to growth and development and involves in response to high temperature and humidity stress in soybean., 2015, 34: 1927–1937.
[31] Tao Y, Chen M, Shu Y, Zhu Y, Wang S, Huang L, Yu X, Wang Z, Qian P, Gu W, Ma H. Identification and functional characterization of a novel BEL1-LIKE homeobox transcription factor GmBLH4 in soybean., 2018, 134: 331–344.
[32] Song X, Zhao Y, Wang J, Lu M Z. The transcription factor KNAT2/6b mediates changes in plant architecture in response to drought via down-regulatingin×., 2021, 72: 5625–5637.
[33] 王汉中. 以新需求为导向的油菜产业发展战略. 中国油料作物学报, 2018, 40: 613–617. Wang H Z. New-demand oriented oilseed rape industry developing strategy., 2018, 40: 613–617 (in Chinese with English abstract).
[34] Chalhoub B, Denoeud F, Liu S, Parkin I A, Tang H, Wang X, Chiquet J, Belcram H, Tong C, Samans B, Corréa M, Da Silva C, Just J, Falentin C, Koh C S, Le Clainche I, Bernard M, Bento P, Noel B, Labadie K, Alberti A, Charles M, Arnaud D, Guo H, Daviaud C, Alamery S, Jabbari K, Zhao M, Edger P P, Chelaifa H, Tack D, Lassalle G, Mestiri I, Schnel N, Le Paslier M C, Fan G, Renault V, Bayer P E, Golicz A A, Manoli S, Lee T H, Thi V H, Chalabi S, Hu Q, Fan C, Tollenaere R, Lu Y, Battail C, Shen J, Sidebottom C H, Wang X, Canaguier A, Chauveau A, Bérard A, Deniot G, Guan M, Liu Z, Sun F, Lim Y P, Lyons E, Town C D, Bancroft I, Wang X, Meng J, Ma J, Pires J C, King G J, Brunel D, Delourme R, Renard M, Aury J M, Adams K L, Batley J, Snowdon R J, Tost J, Edwards D, Zhou Y, Hua W, Sharpe A G, Paterson A H, Guan C, Wincker P. Plant genetics. Early allopolyploid evolution in the post-Neolithicoilseed genome., 2014, 345: 950–953.
[35] Cheng F, Wu J, Wang X. Genome triplication drove the diversification ofplants., 2014, 1: 14024.
[36] Akter A, Itabashi E, Kakizaki T, Okazaki K, Dennis E S, Fujimoto R. Genome triplication leads to transcriptional divergence ofgenes during vernalization in the genus., 2021, 11: 619417.
[37] El-Gebali S, Mistry J, Bateman A, Eddy S R, Luciani A, Potter S C, Qureshi M, Richardson L J, Salazar G A, Smart A, Sonnhammer E L L, Hirsh L, Paladin L, Piovesan D, Tosatto S C E, Finn R D. The Pfam protein families database in 2019., 2019, 47: D427–D432.
[38] Potter S C, Luciani A, Eddy S R, Park Y, Lopez R, Finn R D. HMMER web server: 2018 update., 2018, 46: W200–W204.
[39] Marchler-Bauer A, Bryant S H. CD-search: protein domain annotations on the fly., 2004, 32: W327–W331.
[40] Chen C, Chen H, Zhang Y, Thomas H R, Frank M H, He Y, Xia R. TBtools: an integrative toolkit developed for interactive analyses of big biological data., 2020, 13: 1194–1202.
[41] Xu G, Guo C, Shan H, Kong H. Divergence of duplicate genes in exon-intron structure., 2012, 109: 1187–1192.
[42] Lynch M, Conery J S. The evolutionary fate and consequences of duplicate genes., 2000, 290: 1151–1155.
[43] Kong W, Ding L, Cheng J, Wang B. Identification and expression analysis of genes with pathogen-inducible-regulatory elements in the promoter regions in., 2018, 11: 52.
[44] Ho C L, Geisler M. Genome-wide computational identification of biologically significant-regulatory elements and associated transcription factors from rice.(Basel), 2019, 8: 441.
[45] Meng L, Liu X, He C, Xu B, Li Y, Hu Y. Functional divergence and adaptive selection ofgene family in plants., 2020, 15: 346–363.
[46] Zhu Y, Wu N, Song W, Yin G, Qin Y, Yan Y, Hu Y. Soybean () expansin gene superfamily origins: segmental and tandem duplication events followed by divergent selection among subfamilies., 2014, 14: 93.
[47] Sun R, Qin T, Wall S B, Wang Y, Guo X, Sun J, Liu Y, Wang Q, Zhang B. Genome-wide identification of KNOX transcription factors in cotton and the role of-and-in response to salt and drought stress., 2023, 226: 1248–1260.
[48] Han Y, Zhang L, Yan L, Xiong X, Wang W, Zhang X H, Min D H. Genome-wide analysis of TALE superfamily inreveals-is involved in abiotic stress response., 2022, 23: 89.
附表1 甘蓝型油菜基因的复制类型
Table S1 Duplication type ofgenes in
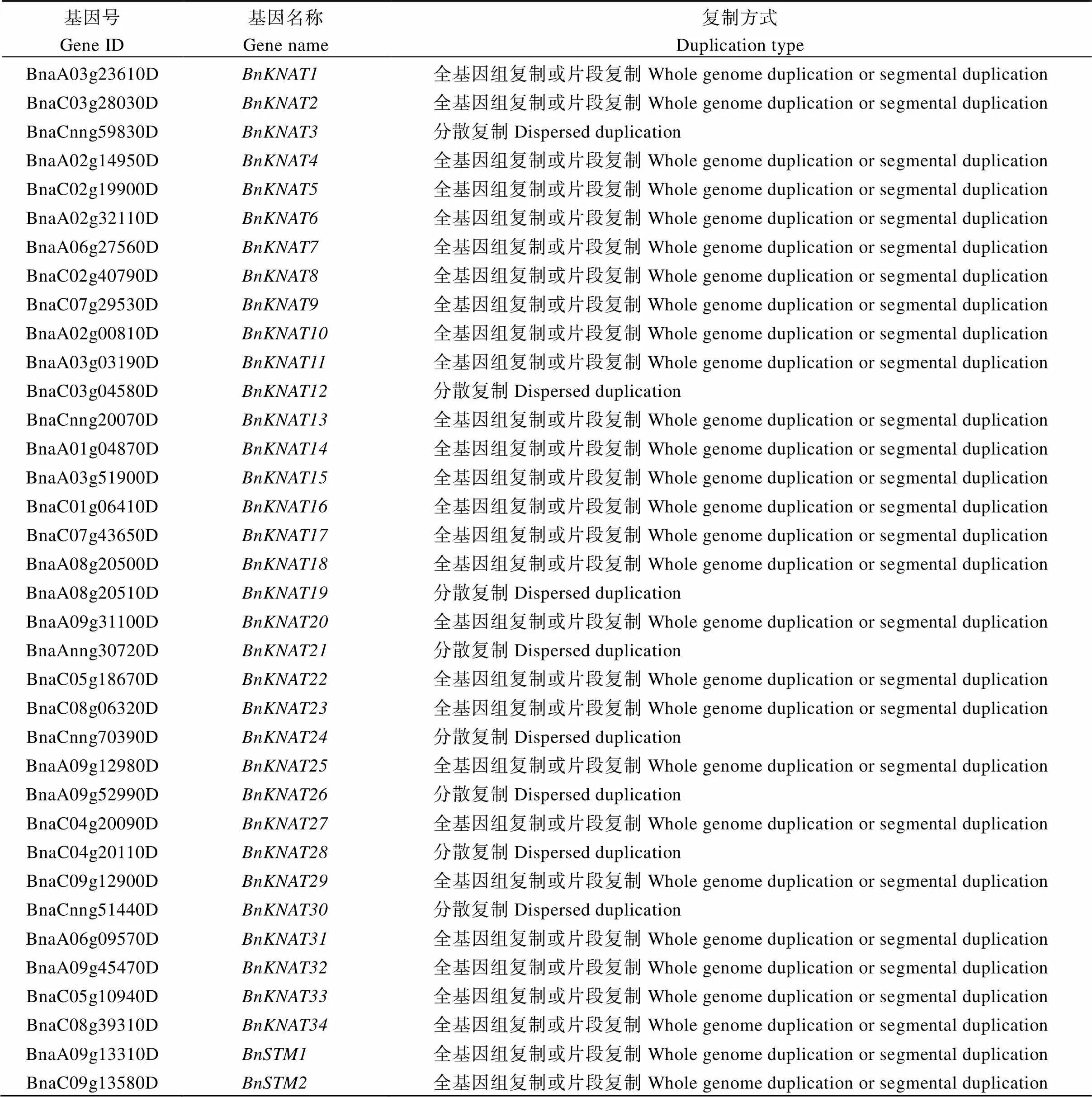
基因号Gene ID基因名称Gene name复制方式Duplication type BnaA03g23610DBnKNAT1全基因组复制或片段复制Whole genome duplication or segmental duplication BnaC03g28030DBnKNAT2全基因组复制或片段复制Whole genome duplication or segmental duplication BnaCnng59830DBnKNAT3分散复制Dispersed duplication BnaA02g14950DBnKNAT4全基因组复制或片段复制Whole genome duplication or segmental duplication BnaC02g19900DBnKNAT5全基因组复制或片段复制Whole genome duplication or segmental duplication BnaA02g32110DBnKNAT6全基因组复制或片段复制Whole genome duplication or segmental duplication BnaA06g27560DBnKNAT7全基因组复制或片段复制Whole genome duplication or segmental duplication BnaC02g40790DBnKNAT8全基因组复制或片段复制Whole genome duplication or segmental duplication BnaC07g29530DBnKNAT9全基因组复制或片段复制Whole genome duplication or segmental duplication BnaA02g00810DBnKNAT10全基因组复制或片段复制Whole genome duplication or segmental duplication BnaA03g03190DBnKNAT11全基因组复制或片段复制Whole genome duplication or segmental duplication BnaC03g04580DBnKNAT12分散复制Dispersed duplication BnaCnng20070DBnKNAT13全基因组复制或片段复制Whole genome duplication or segmental duplication BnaA01g04870DBnKNAT14全基因组复制或片段复制Whole genome duplication or segmental duplication BnaA03g51900DBnKNAT15全基因组复制或片段复制Whole genome duplication or segmental duplication BnaC01g06410DBnKNAT16全基因组复制或片段复制Whole genome duplication or segmental duplication BnaC07g43650DBnKNAT17全基因组复制或片段复制Whole genome duplication or segmental duplication BnaA08g20500DBnKNAT18全基因组复制或片段复制Whole genome duplication or segmental duplication BnaA08g20510DBnKNAT19分散复制Dispersed duplication BnaA09g31100DBnKNAT20全基因组复制或片段复制Whole genome duplication or segmental duplication BnaAnng30720DBnKNAT21分散复制Dispersed duplication BnaC05g18670DBnKNAT22全基因组复制或片段复制Whole genome duplication or segmental duplication BnaC08g06320DBnKNAT23全基因组复制或片段复制Whole genome duplication or segmental duplication BnaCnng70390DBnKNAT24分散复制Dispersed duplication BnaA09g12980DBnKNAT25全基因组复制或片段复制Whole genome duplication or segmental duplication BnaA09g52990DBnKNAT26分散复制Dispersed duplication BnaC04g20090DBnKNAT27全基因组复制或片段复制Whole genome duplication or segmental duplication BnaC04g20110DBnKNAT28分散复制Dispersed duplication BnaC09g12900DBnKNAT29全基因组复制或片段复制Whole genome duplication or segmental duplication BnaCnng51440DBnKNAT30分散复制Dispersed duplication BnaA06g09570DBnKNAT31全基因组复制或片段复制Whole genome duplication or segmental duplication BnaA09g45470DBnKNAT32全基因组复制或片段复制Whole genome duplication or segmental duplication BnaC05g10940DBnKNAT33全基因组复制或片段复制Whole genome duplication or segmental duplication BnaC08g39310DBnKNAT34全基因组复制或片段复制Whole genome duplication or segmental duplication BnaA09g13310DBnSTM1全基因组复制或片段复制Whole genome duplication or segmental duplication BnaC09g13580DBnSTM2全基因组复制或片段复制Whole genome duplication or segmental duplication
Identification and analysis ofgene family in
CHEN Wu-Jun, LIU Jiang-Dong, JIANG Kai-Xuan, WANG You-Ping, and JIANG Jin-Jin*
College of Bioscience and Biotechnology, Yangzhou University, Yangzhou 225009, Jiangsu, China
The plant specific KNOTTED-LIKE HOMEOBOX (KNOX) protein is a transcription factor family that plays an important role in plant growth and development, as well as responses to various stresses. KNOX proteins contain four conserved domains [a three amino acid loop extension (TALE) type homeodomain (HD), ELK domain, and two subdomains (KNOX1 and KNOX2)]. At present, the comprehensive study ofgenes inhas not been reported yet. In this study, 36family members were identified ingenomebioinformatic analyses, which were classified into three sub-families (I, II, and M-type) based on sequence alignment and phylogenetic analyses. Evolutionary analysis showed that the whole genome duplication (WGD) and segmental duplication were the main driving forces for the expansion ofgene family. Based on the RNA-seq data of developing tissues and organs of,were specifically expressed in endosperm and developing seeds, whilewere highly expressed in mature seeds. We analyzed the-acting elements and expression pattern ofunder various abiotic stresses, and identified 17members that might be involved in drought and osmotic stress responses.
; KNOX; gene family; expression pattern; abiotic stress
10.3724/SP.J.1006.2023.34027
本研究由国家自然科学基金项目(32272055, 31972963), 江苏省研究生实践创新计划项目(SJCX21_1602)和江苏省“青蓝工程”项目资助。
This study was supported by the National Natural Science Foundation of China (32272055, 31972963), the Graduate Training Program for Innovation and Entrepreneurship (SJCX21_1602), and the Qinglan Project of Jiangsu Province.
蒋金金, E-mail: jjjiang@yzu.edu.cn
E-mail: chenwujun0921@163.com
2023-02-10;
2023-04-17;
2023-04-24.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20230423.1902.004.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).

