原生质体再生技术复壮金针菇退化菌株的研究
张 丹,朱秋瑾,谭 琦,宋春艳,于海龙,王瑞娟,尚晓冬,刘建雨
(上海市农业科学院食用菌研究所,农业农村部南方食用菌资源利用重点实验室,国家食用菌工程技术研究中心,上海市农业遗传育种重点开放实验室,上海 201403)
金针菇(Flammulina filiformis)学名毛柄金钱菌,又称毛柄小火菇、构菌、朴菇、冬菇、朴菰等,其口感脆嫩,营养丰富,深受消费者喜爱[ 1]。 金针菇是我国主要的栽培食用菌之一,近5 年的年产量维持在260 万t左右[ 2]。 菌种是金针菇生产的核心要素,菌种退化是产业长期关注的焦点。 菌种退化是食用菌的生物学特征之一,菌种具有的典型优良性状发生明显弱化或丧失的现象称为退化,这是一个从量变到质变的逐步演化过程。 菌种退化常表现为菌丝生长势弱、基质降解能力下降、抗性弱、子实体产量明显下降等[ 3-4]。金针菇菌种具有生长速率快、对培养环境敏感、易产生粉孢子等特性,生产用菌种较易发生菌种退化[ 5]。为了保持菌种的优良性状,对常用菌种进行提纯复壮是保证金针菇高产稳产的重要措施之一。
常用的提纯复壮方法有尖端菌丝分离、组织分离以及原生质体再生等,其中原生质体再生被认为是提纯复壮的较好方法,在双孢蘑菇、白灵菇、平菇等食用菌上已有研究报道[ 6-8]。 原生质体再生技术在金针菇的诱变育种、细胞融合育种、菌丝脱毒以及遗传学分析等方面也有应用[ 1,9-11]。 陈鹏等[ 12]对金针菇原生质体制备及再生技术进行了正交优化,完善了金针菇原生质体的制备方法。 在金针菇菌种复壮方面,鲜见有关原生质体再生技术的应用及遗传机理的研究报道。
随着分子生物学的发展,广泛分布于基因组的单核苷酸多态性(Single nucleotide polymorphism,SNP)标记被大量应用于遗传研究。 SNP 主要是指在基因组水平上由单个核苷酸的变异所引起的DNA 序列多态性,包括单碱基的缺失、插入、转换及颠换等[ 13]。 根据对生物遗传性状的影响,可将SNP 分为蛋白编码SNP 和非蛋白编码SNP[ 14]。 转录组是开发标记的理想资源,基于转录组建立的标记是根据基因的差异而建立的标记,也称为基因内部标记,除具有一般分子标记的特点外,还有信息量大、通用性好等优势[ 15]。由于转录本保守性较高,如果发现一个基因内部标记与某一性状连锁,那么该转录本就可能与控制该性状的基因相关[ 16]。
本研究采用原生质体再生技术对工厂化白色金针菇退化菌株进行复壮,同时基于转录组序列开发SNP 标记对复壮前后的菌株进行遗传背景分析,并对复壮前后菌株转录组的特异表达基因进行筛选和功能分析,以期探索原生质体再生技术复壮金针菇退化菌株的分子机理,为金针菇菌种退化与提纯复壮的深入研究提供参考。
1 材料与方法
1.1 供试菌株与试剂
试验于2019 年5 月在上海市农业科学院食用菌研究所进行,供试金针菇菌株为H25 及退化的H25-d 菌株,由上海市农业科学院食用菌研究所菌种保藏中心提供。 退化菌株H25-d 为H25 菌株在PDA 培养基继代培养20 代时产生的退化菌株,其外观表征为菌落不规则、菌丝稀疏且生长速率慢,无法正常栽培出菇。
试剂:溶壁酶购于广东省科学院微生物研究所;甘露醇购于生工生物工程(上海)股份有限公司;蔗糖、碳酸钙、硫酸镁、磷酸二氢钾均为分析纯,购于国药集团化学试剂有限公司。
1.2 培养基
母种培养采用PDA 培养基,摇瓶采用PDB 培养基,培养基均为美国BD 公司生产。 木屑培养基:78%木屑,20%麸皮,2%碳酸钙,含水量为60%。 原生质体再生培养基:3.9%PDA 培养基,2%蔗糖。 工厂化液体培养基:2%白砂糖、0.4%豆粕粉、0.05%硫酸镁、0.05%磷酸二氢钾。 工厂化瓶栽培养基:59%木屑、25%玉米芯、15%米糠、1%碳酸钙,含水量为60%。
1.3 原生质体再生菌株的制备
金针菇退化菌株H25-d 的菌丝原生质体提取参照前人研究方法进行[ 12,17-18]。
在原生质体再生培养基上有星芒状菌落出现时,按出现时间先后依次挑取单菌落进行单独培养。 对再生菌株进行镜检时,筛选不具有锁状联合的原生质体单核体菌株,将单核体菌株进行交配型鉴定并两两杂交获取杂交双核菌株。
1.4 菌丝生长测试
PDA 培养基菌丝生长测定:使用5 mm 直径打孔器取接种块,接种于直径为9 cm 的培养皿中央,25 ℃暗培养,当菌丝从接种块上生长到PDA 平板培养基时,在菌丝前端划线,在菌丝即将长满平板时再次在菌丝前端划线,两次划线间的直线距离与培养天数的比值为菌丝生长速率,每个平板测定4 个方向的生长速率,取平均值,每个处理设3 个生物学重复。
木屑培养基菌丝生长测定:将直径为5 mm 的PDA 菌种块接种至装有木屑培养基的200 mm×30 mm玻璃大试管中(每管培养基干质量35 g),25 ℃暗培养,待菌丝长到料面2 cm 以下时沿菌丝前端划线,培养14 d 后再次在菌丝前端划线。 测量两次划线之间的长度,计算菌丝的平均生长速率,每个处理设3 个生物学重复。
液体培养基生长测定:利用5 mm 打孔器分别从PDA 培养基上挑取5 个菌种块,接种到装有600 mL液体培养基的1 L 三角瓶中,在黑暗条件下22 ℃、120 r∕min 振荡培养。 7 d 后检测培养液的pH 和菌丝生长量(体积测量法:取10 mL 培养液放入有刻度的离心管中,5 000 r∕min 离心5 min,倒出上清液并测定体积,培养液与上清液的体积差值占培养液体积的比值为菌丝的体积含量,每个样品测定6 次,取平均值),每个处理设3 个生物学重复。
1.5 工厂化栽培测试
栽培测试在上海福茂食用菌有限公司进行,将培养好的液体菌种接种于1 400 mL 栽培瓶中(每瓶培养料湿质量为1 000 g,在121 ℃高压条件下灭菌2 h,冷却后接种)。 测定的农艺性状指标包括生育期、原基形成时间、菇盖直径、菌柄长度、菌柄数量以及单瓶产量,所有指标测定均设3 次生物学重复。
1.6 样品总RNA 提取及转录组测序
供试退化菌株H25-d 及其复壮菌株样品由上海美吉生物医药科技有限公司进行总RNA 提取、转录组文库构建和测序。 具体参考Trapnell 等[ 19-20]的方法。 采用Illumina TruseqTM RNA sample prep Kit 方法构建文库,使用Illumina Hiseq 2000 平台测序。 测序得到原始序列,使用软件SeqPrep(https:∕∕github.com∕jstjohn∕SeqPrep)和sickle(https:∕∕github.com∕najoshi∕sickle)对其进行质量控制,得到高质量的清洁序列。
1.7 表型数据分析及转录组SNP 分析
利用SPSS 19.0 软件对金针菇表型生长数据进行差异性分析。 使用Trinity 软件(https:∕∕github.com∕trinityrnaseq∕trinityrnaseq∕wiki∕)对所有测序的清洁序列进行从头组装,得到转录组完整序列,并统计转录本及单基因簇的各项基础数据。 以组装好的转录组为模板序列,分别将供试菌株H25-d 及其复壮菌株的原始序列与其进行比对,利用SAMtools(https:∕∕samtools.sourceforge.net∕)和VarScan v2.2.7(https:∕∕varscan.sourceforge.net∕)软件挖掘SNP 位点。
1.8 特异表达基因的筛选及功能分析
筛选的特异表达基因为仅在退化或复壮菌株中表达且转录本数量大于600 的基因。 基因功能分析采用NCBI 网站的blastx 搜索数据库中相似功能基因,选取匹配度最高项。
2 结果与分析
2.1 原生质体再生菌株筛选
当原生质体再生培养基上有星芒状菌落出现时,按时间先后依次挑取单菌落进行培养,在新挑选的菌落中镜检出30 个无锁状联合的、生长性状较为优良的单核体菌株。 对原生质体单核体菌株进行交配型鉴定后,采用单单杂交的方式获取5 个具有锁状联合的杂交双核菌株。 获得的再生双核体菌株为原生质体再生菌株,称为H25-dD。 在PDA 培养基上的H25-dD 菌落外观与正常菌株H25 相似,长势明显强于退化菌株H25-d(图1)。
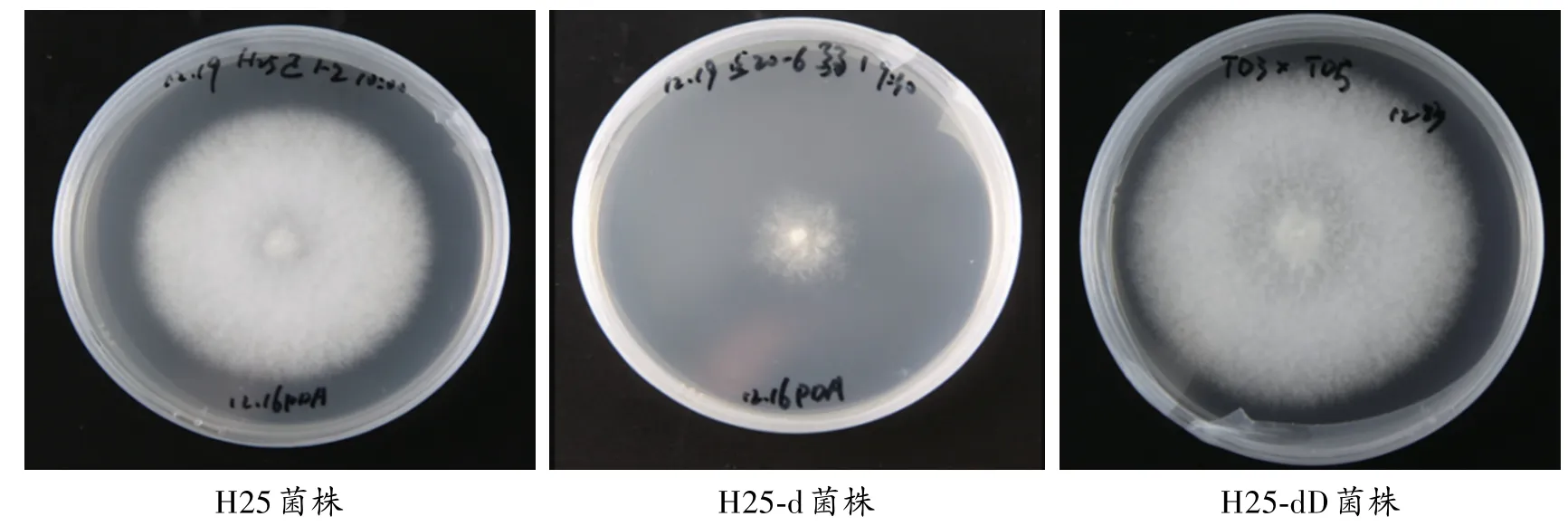
图1 供试菌株在PDA 培养基上的菌落形态Fig.1 Colony morphology of the tested strains on PDA medium
2.2 菌丝生长性状的观察
为进一步检测原生质体再生技术对H25-d 菌株的复壮效果,观察相同培养条件下H25 和H25-dD 菌株在PDA 培养基、木屑培养基、液体菌种培养基中的菌丝生长情况。 H25 与H25-dD 菌株在PDA 培养基上的生长速率分别为(4.63 ±0.15)mm∕d 和(4.68 ±0.14)mm∕d;在木屑培养基中的生长速率分别为(3.51 ±0.07)mm∕d 和(3.53 ±0.08) mm∕d;摇瓶培养7 d 后的培养液pH 分别为(6.28 ±0.01)和(6.30 ±0.07),菌丝量分别为(21.13 ±0.21)%和(21.76 ±0.92)%。 以上4 个指标之间的差异均未达显著水平,表明H25-dD 的菌丝生长性状恢复到原有正常菌株水平。
2.3 菌株栽培农艺性状的观察
菌株成熟后的子实体农艺性状是非常重要的指标,特别是产量。 试验进一步观察了H25 与H25-dD菌株在工厂化条件下的出菇情况。 从表1 可以看出,H25 和H25-dD 菌株的菌丝生育期、原基形成时间均完全一致,分别为23 d 和5 d;菌盖直径平均值分别为5.32 mm 和5.29 mm;菌柄长度平均值分别为134.27 mm和132.54 mm;单瓶子实体平均数量分别为1 228.65 个和1 315.84 个;单瓶平均产量分别为478.67 g 和476.30 g。 以上6 个指标之间均没有显著差异,表明H25-dD 菌株可以恢复到与H25 菌株相当的农艺性状。 如图2 所示,H25-dD 菌株的子实体形态与H25 菌株无明显差异,复壮效果理想。

表1 供试菌株在工厂化培养条件下的农艺性状Table 1 Agronomic traits of the tested strains under factory culture conditions

图2 供试菌株的子实体形态Fig.2 Fruiting body morphology of the tested strains
2.4 转录组序列的组装及SNP 分析
为了进一步探索原生质体再生技术复壮金针菇退化菌株的内在机理,本研究基于转录组挖掘的SNP位点对比了H25-d 及H25-dD 菌株的遗传背景差异。 转录组数据经组装共得到27 736 个转录本(Transcript)以及18 064 个单基因簇(Unigene)。 转录本的平均长度为1 444.65 bp,单基因簇的平均长度为1 227.17 bp。
以组装好的转录组序列为模板,将H25-d 及H25-dD 菌株的测序数据进行比对,挖掘SNP 位点。 结果显示,H25-d 菌株与模板比对共产生12 423 个SNP 位点,平均每kb 出现的频率为0.31 个。 SNP 在转录组单基因簇中的位置分布表明,有4 041 个位于基因的第一密码子区域,有3 950 个位于第二密码子区域,有4 115 个位于第三密码子区域,在3’和5’端非编码区均未发现SNP 位点,有317 个SNP 没有明确的分布区域。 H25-dD 菌株与模板比对共产生13 093 个SNP 位点,平均每kb 出现的频率为0.32 个。 SNP 在单基因簇中的位置分布表明,有4 240 个位于第一密码子区域,有4 045 个位于第二密码子区域,有4 324个位于第三密码子区域,在3’和5’端非编码区均未发现SNP 位点,有484 个SNP 没有明确的分布区域。
分析SNP 的碱基序列发现,其包含置换和颠换两种类型的多态性位点。 H25-d 的SNP 中发生置换的碱基类型为C∕T 和A∕G,数量分别为4 558 个和4 768 个,转录组每kb 出现的频率均为0.11 个。 发生颠换的碱基有4 种类型,分别为A∕T、A∕C、T∕G 和C∕G,对应的数量分别为979 个、689 个、856 个和573 个,在转录组上每kb 出现的频率分别为0.02 个、0.01 个、0.02 个和0.01 个。 H25-dD 菌株的SNP 中发生置换的碱基类型为C∕T 和A∕G,数量分别为4 765 个和4 975 个,转录组每kb 出现的频率分别为0.11 个和0.12 个。 发生颠换的碱基类型分别为A∕T、A∕C、T∕G 和C∕G,对应的数量分别为1 029 个、768 个、850 个和706 个,在转录组上每kb 出现的频率分别为0.02 个、0.01 个、0.02 个和0.01 个。
H25-d 与H25-dD 菌株的SNP 分布情况比较表明,两个菌株在SNP 数量及位置分布上均存在差异。在SNP 数量方面,H25-dD 菌株比H25-d 菌株多670 个,其中有199 个位于基因的第一密码子区域,有95个位于第二密码子区域,有209 个位于第三密码子区域,有167 个没有明确的分布位置。 在SNP 碱基类型方面两类菌株也有较大差异,H25-dD 菌株比H25-d 菌株发生C∕T 型碱基转换的数量多207 个、A∕G 型多207 个。 H25-dD 菌株比H25-d 菌株发生颠换的A∕T 类型多50 个、A∕C 类型多79 个、T∕G 类型少5 个、C∕G 类型多133 个。 上述差异SNP 位点表明,原生质体再生后菌株的遗传背景产生了变化,且这些变化多位于基因的密码子区域。
2.5 特异表达基因分析
基于SNP 位点的检测结果,进一步对H25-d 及H25-dD 菌株中特异表达基因进行挖掘,筛选出仅在各自转录组特异表达的基因。 结果表明,复壮菌株的特异表达基因数量远高于退化菌株。 如表2 所示,H25-d 菌株转录本数量大于600 的特异表达基因仅有c1661_g1,且未匹配到已知功能的相似基因。H25-dD 菌株中转录本数量大于600 的特异表达基因数量有15 个。 功能分析表明,H25-dD 菌株的基因表达在细胞能量代谢、抗氧化、细胞骨架构建以及细胞DNA 甲基化修饰等方面发生了较大变化。 其中基因c8593_g1、c12033_g1 与细胞内ATP 的合成有关[ 21];c10839_g1、c4223_g1、c10584_g3、c11072_g1、c11025_g3 分别为细胞色素C 氧化酶、NADH 脱氢酶、细胞色素b-c1 复合体的结构单元,参与细胞能量代谢中重要的氧化磷酸化代谢途径[ 22-25]。 c12025_g1 为磷酸烯醇式丙酮酸羧化酶相关基因,参与细胞内三羧酸循环代谢途径[ 26]。 c11989_g2 为烯醇化酶相关基因,烯醇化酶是催化2-磷酸甘油酸形成高能化合物磷酸烯醇式丙酮酸的酶,是糖酵解的关键酶之一[ 27]。 c12016_g1 为热激蛋白家族相关基因,编码70 ku 热激蛋白,该蛋白是细胞内抵抗氧化胁迫和参与细胞信号传导的重要物质[ 28]。 c11822_g3 为谷氨酰胺合成酶相关基因,与细胞内重要的抗氧化物谷胱甘肽的合成有关[ 29]。 c12031_g1 为微管蛋白相关基因,主要参与细胞骨架构建及细胞分裂等生物学过程[ 30]。 c11997_g2 为半胱氨酸蛋白酶相关基因,半胱氨酸蛋白酶一般存在于细胞的溶酶体中,主要参与细胞吞噬和细胞内多余物质的清除和消化[ 31]。 c11938_g1 为S-腺苷甲硫氨酸合成酶相关基因,是催化腺苷三磷酸(ATP)和甲硫氨酸形成S-腺苷甲硫氨酸的酶,而S-腺苷甲硫氨酸是细胞DNA 甲基化的主要供体[ 32]。 c12006_g1 为半胱氨酸甲基转移酶相关基因,同样参与细胞的DNA 甲基化修饰作用[ 33]。
3 结论与讨论
工厂化栽培的白色金针菇优良品种在使用一段时间后会出现大量粉孢子产生、菌丝生长不一致、出菇不齐、产量下降等退化现象。 菌种退化会给金针菇生产企业带来较大损失,如何有效缓解菌种退化是金针菇产业需要研究的重要问题。
金针菇菌种的提纯复壮是缓解菌种退化的有效方法之一,常用方法主要有增加菌丝营养、尖端菌丝分离、组织分离及原生质体制备等。 陈志松[ 34]研究认为,挑取菌褶处的组织块进行分离可有效复壮退化的金针菇菌种。 李雪飞等[ 35]采用组织分离和菌丝尖端分离的方法,结合构树基质培养和蓝光处理对退化金针菇菌丝进行了复壮。 刘昆昂等[ 36]利用结晶紫PDA 培养基结合原生质体制备技术对黄色金针菇退化菌株进行了复壮。 本研究制备的原生质体单核体杂交菌株可有效恢复白色金针菇退化菌株的农艺性状,与前人的研究结果较为一致。 本研究获得的原生质体再生菌株的产量等指标并没有显著超过对照菌株,其原因是对照菌株为液氮保藏的原始母种,尚未经历长期的继代培养,其菌株性状优良,复壮后的菌株在同样培养条件下较难超越原始母种。 若想利用原生质体再生技术获得产量等指标上超过原菌株的菌株,可在原生质体再生过程中进行诱变育种,筛选出产量等指标超越原始母种的优良变异菌株。 另外,也可尝试采用加富培养基对获得的原生质体再生菌株进行培养。
基于转录组序列开发的SNP 标记对复壮前后菌株的遗传背景分析显示,原生质体再生菌株的遗传背景发生了变化,复壮后菌株的SNP 位点比退化菌株的SNP 位点多670 个,这些位点大部分位于基因转录表达的密码子区域,说明原生质体再生对菌株的遗传背景产生了影响,与表型相关基因的转录表达发生了变化。 本研究进一步筛选了退化和复壮菌株中转录本数量超过600 的特异表达基因,通过基因功能分析明确了细胞能量代谢、抗氧化、细胞骨架构建以及细胞DNA 甲基化修饰等方面相关的基因在复壮菌株中特异表达,这些基因的表达对细胞的正常代谢和清除有害物质具有重要作用,这与Wang 等[ 28]在金龟子绿僵菌(Metarhizium anisopliae)中的研究结果较为一致。
基于上述分析,初步认为原生质体再生技术能够增强细胞的抗氧化能力及对有毒有害物质的清除能力,打破有害物质导致的细胞糖酵解、三羧酸循环、氧化磷酸化、ATP 合成以及DNA 甲基化修饰、骨架构建等重要代谢途径功能紊乱的恶性循环,使细胞的生长发育重回正轨,从而达到菌株复壮的效果。

