中国荷斯坦奶牛乳脂中miR-2285f生物信息学分析及靶基因验证
张 迪 刘佳敏 禹保军 李德生 顾亚玲 温 万 张 娟*
(1.宁夏大学 农学院,银川 750021;2.宁夏回族自治区反刍动物分子细胞育种重点实验室,银川 750021;3.宁夏畜牧工作站,银川 750002)
乳脂肪是乳中的主要能量物质和重要的营养成分,由编码转录调节因子和非编码因子共同调控表达,并涉及到一个庞大、复杂的分子调控网络,受到多个激素及信号分子等调控,包括脂肪酸、甘油三酯的摄入、转运、活化、合成、脂滴的形成和分泌等过程[1]。随着候选基因筛选测序和比较基因组学研究方法的发展,更多的乳脂代谢相关基因及调控因子被发现并验证其功能[2],miRNA便是其中一种重要的调控因子。
MicroRNA(miRNA)是长度为18~24个核苷酸的非编码RNA分子,通过碱基互补配对的方式调控生物个体的某些生理生化指标[3],并在调节乳腺发育[4]、泌乳过程[5]、脂肪代谢[6]、天然免疫[7]等生物学功能方面发挥一定作用。Bu等[8]在牛乳腺组织中获得99条miRNA,其中miR-23a、miR-24、miR-143和miR-103的表达水平较高。miR-33a、miR-103过表达可促进乳腺上皮细胞(Bovine mammary epithelial cells,BMECs)中甘油三酯的合成[9-10],miR-25、miR-454都可抑制靶基因PPARγ(Peroxisome proliferator activated receptor gamma)的表达从而降低细胞内甘油三酯的合成[11-12]。还有研究发现,羊乳腺组织中miR-103、miR-200a、miR-27a及miR-23a协同调控了乳腺脂肪酸合成过程[13]。通过对山羊泌乳期差异表达的miRNA并定位于与乳脂含量相关的QTL中发现miR-2285家族在其中显著表达[14]。同时,miR-2285家族也是一类高表达的牛特异性miRNA,miR-2258o-3p通过负调控靶基因同源性磷酸酶PTEN(Phosphatase and tensin homolog)和极低密度脂蛋白受体VLDLR(Very low density lipoprotein receptor)缓解低氧引起的细胞凋亡[15]。同时,通过课题组前期工作,对乳脂率极端差异的8头宁夏中国荷斯坦奶牛乳腺上皮细胞转录组测序,miR-2285f在高乳脂组表达量较高且显著差异,并且转录组测序通过荧光定量的方法进行了验证,数据可靠性高[16]。但是miR-2285f是否影响中国荷斯坦奶牛乳脂代谢尚不清楚。因此本研究通过对中国荷斯坦奶牛miR-2285f生物信息学分析以及靶向关系验证,进一步了解miR-2285f在乳脂代谢中的作用,为后续miR-2285f对牛乳腺上皮细胞乳脂代谢功能验证提供重要依据。
1 材料与方法
1.1 中国荷斯坦奶牛miR-2285f基因组位置分析
通过NCBI数据库(https:∥www.ncbi.nlm.nih.gov/)和UCSC数据库(UCSC Genome Browser Home)查找miR-2285f在牛基因组的位置以及参考基因组序列(ARS-UCD1.2或bosTau9)和注释信息。
1.2 miR-2285f生物信息学分析
在miBase(https:∥www.mirbase.org/cgi-bin/mirna_entry.pl?acc=bta-miR-2285f)中检索猕猴、大猩猩、人、牛、黑猩猩、褐家鼠、小鼠、山羊和家犬的miR-2285f序列,分析其在不同物种间的保守性。
使用NCBI数据库获得前体序列bta-miR-2285f和其宿主基因TBK1的上游序列(2 kb),并使用EMBOSS软件(https:∥www.ebi.ac.uk/Tools/seqstats/emboss_cpgplot/)和UCSC软件(https:∥genome.ucsc.edu/)进行CpG岛分析。
通过Promoter2.0(https:∥services.healthtech.dtu.dk/service.php/Promoter-2.0)和Neural Network Promoter Prediction(https:∥fruitfly.org/seq_tools/promoter.html)预测启动子区域。
1.3 miR-2285f靶基因预测以及功能分析
通过靶基因预测软件TargetScan(https:∥www.targetscan.org/vert_71/)和miWalk(http:∥mirwalk.umm.uni-heidelberg.de/)对miR-2285f靶基因预测并取其交集进行miRNA-mRNA关联分析,并通过Cytoscape (https:∥cytoscape.org/)软件可视化。使用DAVID软件(https:∥david.ncifcrf.gov/)对互作候选靶基因集进行GO和KEGG分析。
1.4 bta-miR-2285f在中国荷斯坦奶牛组织中的表达谱分析
本研究所用组织样本均由实验室前期保存。利用Trizol法提取中国荷斯坦奶牛小肠、肝脏、乳腺、心脏、肾脏和卵巢组织的总RNA,按照HiScript Ⅲ 1 st Strand cDNA Synthesis kit转录试剂盒的说明反转录合成cDNA。茎环法设计miRNA的引物,Primer Premier 6设计mRNA的引物(表1)。miRNA和mRNA分别以U6和GAPDH为内参,每个样本3个重复,使用SYBR Green Pro Taq HS 预混型qPCR试剂盒进行实时荧光定量PCR检测。
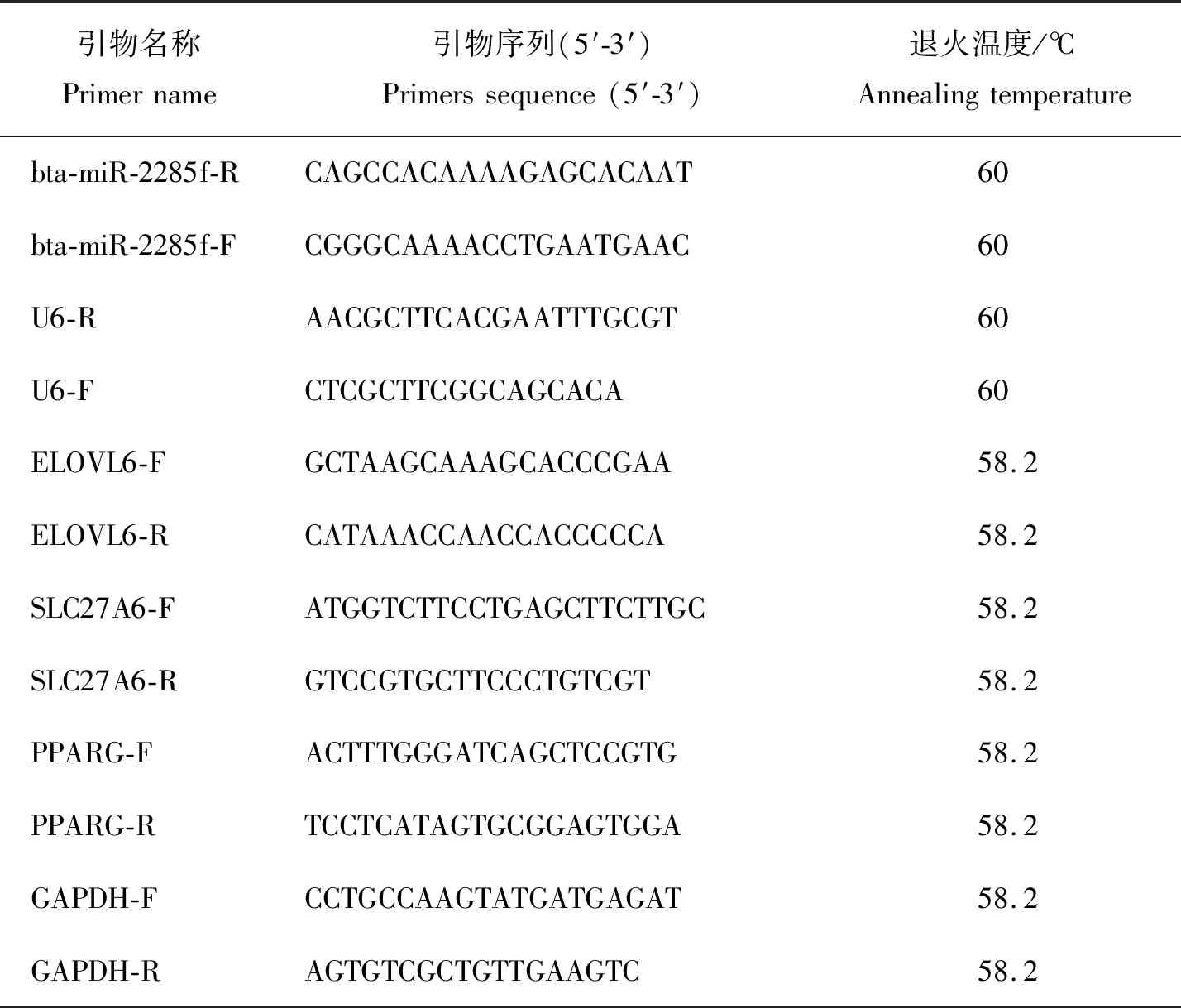
表1 本研究中用到的引物信息Table 1 Information of primers in the study

表2 CAB39基因野生型突变型载体序列信息Table 2 Sequence information of CAB39 wild-type mutant vector
反应体系:上下游引物各0.5 μL,cDNA 2 μL,2x QuantiNova SYBR Green PCR Master Mix 10 μL,RNAase Free ddH2O补至20 μL。反应条件:95 ℃预变性 30 s;95 ℃变性5 s,退火30 s,40个循环,每个样品3个重复。利用2-ΔΔCt计算 miRNA和mRNA 的相对表达量,结果表示为“平均值±标准误”。
1.5 bta-miR-2285f mimic和inhibitor转染乳腺上皮细胞系
1.5.1乳腺上皮细胞系的培养
试验所采用的乳腺上皮细胞系由宁夏大学王兴平教授惠赠。将乳腺上皮细胞系复苏后培养并接种到6孔板中,添加10% FBS的DMEM/F12培养基,置于37 ℃、5%CO2的细胞培养箱中培养。
1.5.2bta-miR-2285f mimic和inhibitor转染BMECs
bta-miR-2285f模拟物(5′-AAAACCUGAA-UGAACUUUUUGG-3′)和抑制剂(5′-CCAAAA-AGUUCAUUCAGGUUUU-3′)均由广州锐博生物科技有限公司所合成。当细胞密度达到70%时,用去血清培养基Opti-MEM代替生长培养基孵育细胞。按照制造商的说明使用脂质体LipofectamineTM3 000混合Opti-MEM,在混合物中加入bta-miR-2285f mimic和mimic NC(Negative control)、bta-miR-2285f inhibitor和inhibitor NC(Negative control),室温孵育20 min后添加到6孔板细胞中。37 ℃、5%CO2的培养箱中孵育6 h,去除细胞原有培养基加入2 mL 10%FBS的DMEM/F12生长培养基继续培养。
1.6 牛CAB39基因荧光素酶报告载体构建及活性检测
1.6.1牛CAB393′UTR双荧光素野生型和突变型载体构建
将bta-miR-2285f对应结合位点的CAB39基因3′UTR 序列克隆到pmirGLO载体上,构建野生型表达质粒;突变型表达质粒是将bta-miR-2285f对应CAB39基因(Calciumbindingprotein39)3′UTR靶位点的碱基突变序列后构建入pmirGLO载体产生,由北京擎科生物科技有限公司合成。
1.6.2双荧光素酶活性分析
在双荧光素酶报告分析中,293T细胞由湖南丰晖生物科技有限公司购买。将293T细胞复苏并接种于24孔板,当细胞密度达到70%~80%时,加入400 μL无血清DMEM高糖培养基孵育4 h,随后用LipofectamineTM3 000将bta-miR-2285f mimic或mimic NC和CAB393′UTR报告质粒共转染细胞。转染48 h后用PBS清洗细胞,加入100 μL细胞裂解液裂解后收集细胞。在96孔板每孔加入50 μL AR Ⅱ后加入20 μL细胞裂解液轻轻混匀后用酶标仪(OMG Labtech)测定萤火虫荧光酶值。最后每孔加入50 μL Stop and Glo Reagent,轻轻混匀后使用酶标仪测海肾荧光素酶数值。
1.7 数据分析
使用SAS2.0软件对数据进行单因素方差(One way ANOVA)分析,P<0.05表示差异显著,P<0.01表示差异极显著,P>0.05表示差异不显著。
2 结果与分析
2.1 miR-2285f来源及保守性分析
通过牛基因组分析发现,miR-2285f-1位于牛5号染色体的49 300 832~49 300 853位置,来源于宿主基因TBK1的第16内含子,属于内含子miRNA(图1(a));而miR-2285f-2位于牛20号染色体的21 823 443至21 823 464位置,来源于基因肌动蛋白样蛋白2(Actin beta like 2,ACTBL2)和富含GC启动子的结合蛋白(GC-rich promoter binding protein 1,GPBP1)基因之间(图1(b))。采用Reciprocal BLAST检测多个动物基因组上miR-2285f的直系同源物,结果仅在牛基因组定位到bta-miR-2285f序列,并在其他物种中未发现bta-miR-2285f的相似序列,表明bta-miR-2285f是牛特异性miRNA。

图1 bta-miR-2285f-1(a) bta-miR-2285f-2(b)在牛基因组中的位置Fig.1 Location of bta-miR-2285f in the bovine genome
2.2 miR-2285的CPG岛和启动子分析
软件预测发现bta-miR-2285f-1、bta-miR-2285f-2和TBK1基因均不含有CPG岛(图2),表明其不是典型的GC-rich启动子,且极易被激活。
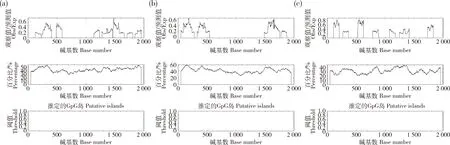
图2 bta-miR-2285f-1(a)、bta-miR-2285f-2(b)和TBK1(c)的CPG岛分析Fig.2 Analysis of CPG islands for bta-miR-2285f-1 (a),bta-miR-2285f-2 (b) and TBK1 (c)
软件PROMOTER2.0预测结果表明,bta-miR-2285f-1的上游序列(2 kb)存在1个转录起始位点,并且在软件Neural Network Promoter Prediction 预测的2个转录起始位点中相近位置586 bp处存在一个转录起始位点。综上,bta-miR-2285f上游序列586处为TSS,启动子范围为上游序列2 004~1 504 bp左右处。
软件PROMOTER2.0预测结果表明,bta-miR-2285f-2的上游序列(2 kb)存在2个转录起始位点,在1 400 bp处得分为:0.64,但是其可信度较低(Marginal prediction)。软件Neural Network Promoter Prediction 预测1个转录起始位点442 bp处得分为0.83其范围为上游序列2 098~1 598 bp。
软件PROMOTER2.0预测结果表明,TBK1的上游序列(2 kb)存在1个转录起始位点,在100 bp处得分为:0.684。软件Neural Network Promoter Prediction 预测4个转录起始位点,并同样在相近105 bp预测到转录起始位点,并且得分最高为0.95。综上,宿主基因TBK1上游序列105 bp处为TSS,启动子范围区域为2 435~1 935 bp。(表3和表4)。

表3 PROMOTER2.0预测启动子Table 3 Promoter of prediction by PROMOTER2.0
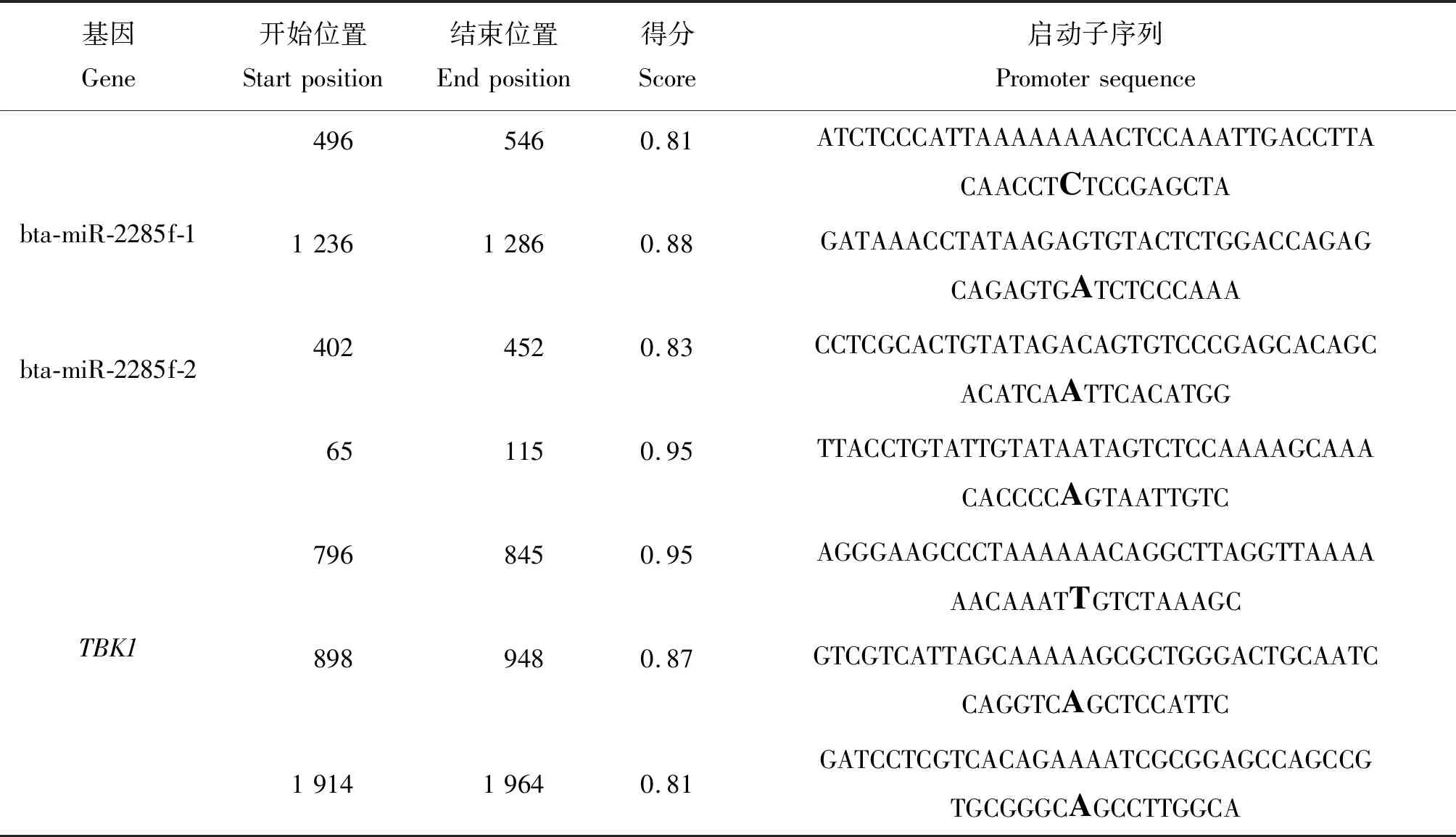
表4 Neural Network Promoter Prediction预测启动子Table 4 Promoter of prediction by Neural Network Promoter Prediction
2.3 过表达bta-miR-2285f转染效率
通过qRT-PCR,使用乳腺上皮细胞系作为体外模型,对bta-miR-2285f的过表达组以及NC组转录水平的相对表达量进行检测。结果显示,bta-miR-2285f过表达量是NC组的5 000多倍。bta-miR-2285f表达量极显著高于NC组(P<0.01)(图3)。结果表明,通过转染bta-miR-2285f mimic可显著增加bta-miR-2285f在乳腺上皮细胞系中的表达水平。
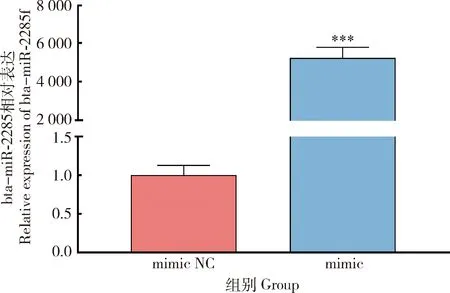
**表示差异显著(P<0.05),***表示差异极显著(P<0.01)。下同。** indicates significant difference (P<0.05),*** indicates extremely significant difference (P<0.01).The same below.
2.4 bta-miR-2285f的组织表达谱分析
通过qRT-PCR检测bta-miR-2285f在中国荷斯坦奶牛各个组织中的表达水平,由图4所示,bta-miR-2285f在奶牛乳腺、心脏、卵巢、肾脏、小肠和肝脏组织中广泛表达其中在乳腺组织中表达量最高,在心脏和卵巢的表达量次之,在肝脏中的表达量最低(P<0.05)。
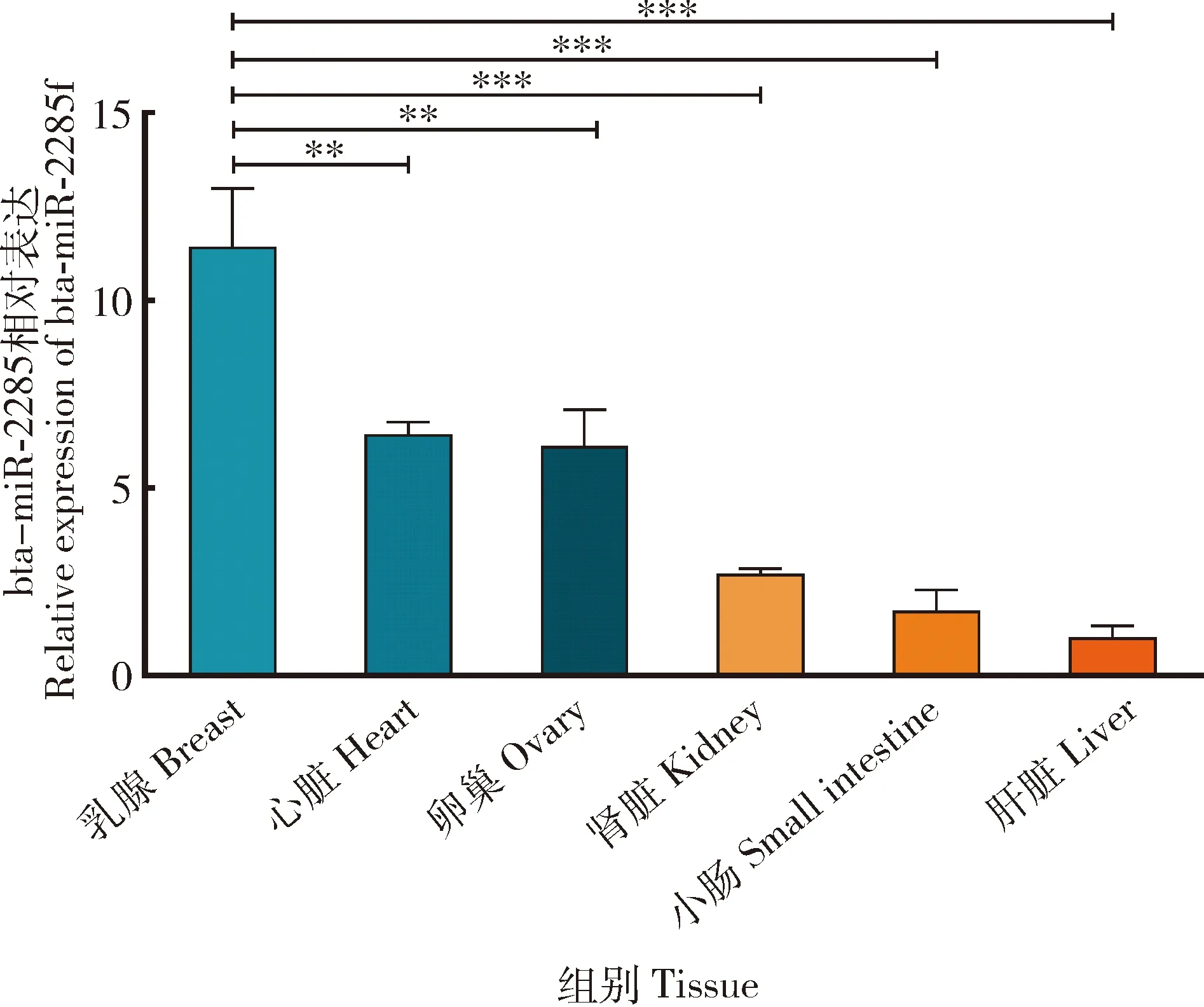
图4 bta-miR-2285f在中国荷斯坦奶牛不同组织的表达水平Fig.4 Expression levels of bta-miR-2285f in different tissues of Chinese Holstein dairy cows
2.5 bta-miR-2285f的靶基因预测、GO和KEGG分析
采用TargetScan和miRWalk分别预测bta-miR-2285f的靶基因(图5),后取其交集后获得172个靶基因集进行KEGG通路注释(图6)和GO功能(图7)。结果显示,KEGG通路富集结果显示,bta-miR-2285f的靶基因共注释到172个信号通路。重点关注乳脂合成代谢相关通路,包括mTOR信号通路、PI3K-AKT信号通路、ErbB信号通路,Hippo信号通路以及甘油酯代谢通路。靶基因集合显著富集的GO条目有854个。靶基因多处于细胞质中、细胞器内和细胞膜中,具有转录辅助因子活性和传递信息的分子作用。生物学过程中主要参与生物新陈代谢、维持细胞的动态性稳定、调节细胞代谢、细胞间信息传递以及细胞增殖、凋亡、免疫等。
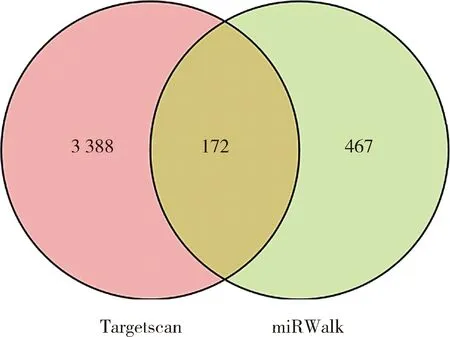
图5 Targetscanhuman7.2和miRWalk2.0数据库对靶基因的预测Fig.5 Prediction of the target genes by Targetscanhuman7.2 and miRWalk2.0 database

(a)靶基因富集相关通路注释;(b)靶基因富集前20信号通路。(a) Annotation of target gene enrichment related pathways;(b) Target genes were enriched in the top 20 signaling pathways.

图7 bta-miR-2285f靶基因的GO分析Fig.7 GO analysis of bta-miR-2285f target genes
2.6 bta-miR-2285f和靶基因CAB39关联分析
将bta-miR-2285f靶基因集合提交到STRING数据库进行蛋白互作网络分析,结果如图8所示,乳脂相关的多个靶基因相互作用并共享功能。功能分析发现,通过在mTOR信号通路和PI3K-Akt信号通路中显著富集的基因CAB39与10个基因具有互作关系(图9(a)),并且参与的信号通路主要在AMPK信号通路、mTOR信号通路、脂肪细胞因子信号通路、FoxO信号通路、自噬、胰岛素信号通路、催产素信号通路、PI3K-Akt信号通路。同时参与脂肪酸生物合成代谢、胆固醇生物合成以及多种氨基酸激酶活性调节过程,具有能量代谢和分子活性调节等多种功能(图9(b))。因此选择基因CAB39为后续进行与bta-miR-2285f的靶向验证。
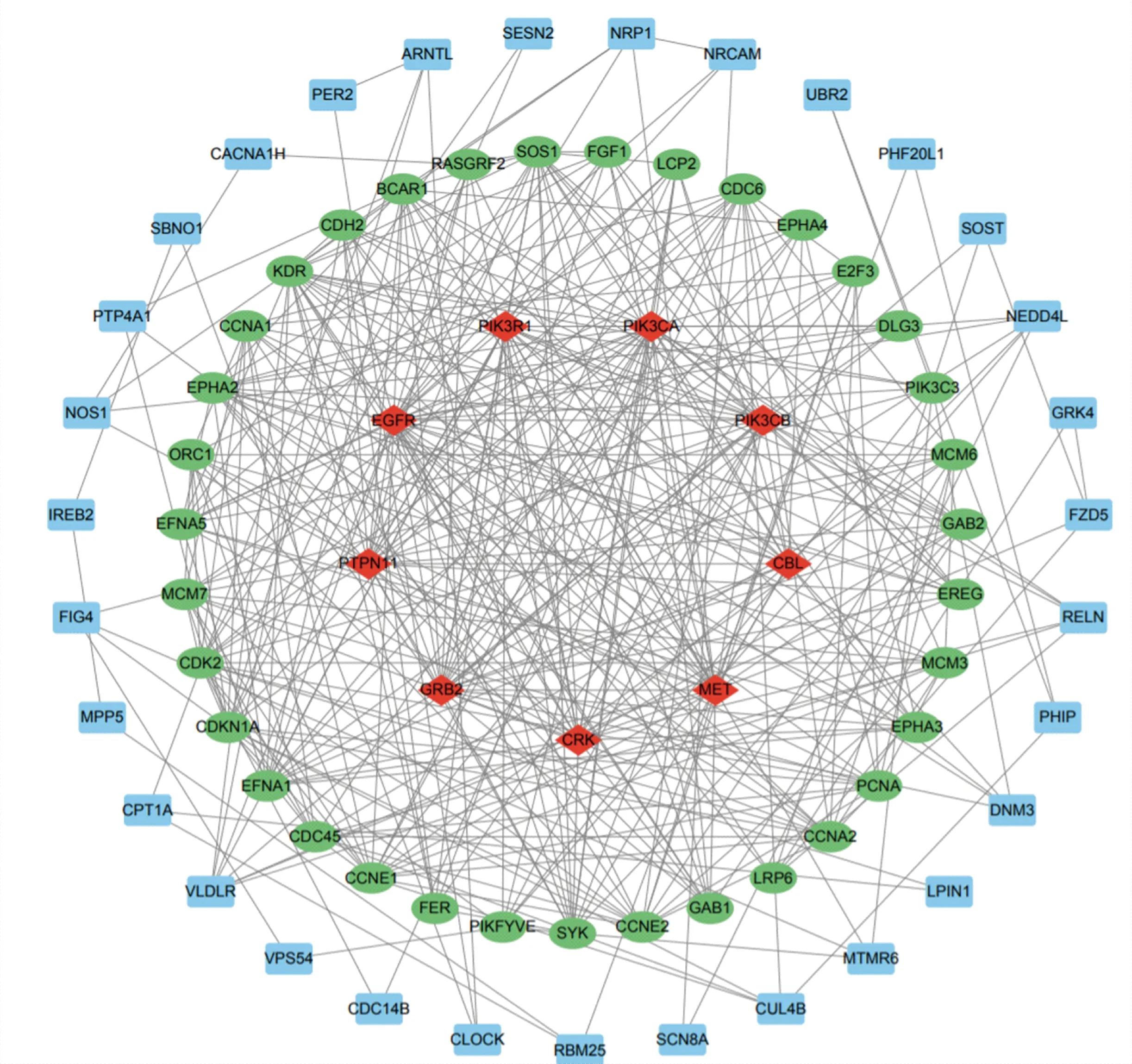
图8 bta-miR-2285f靶基因蛋白互作网络分析Fig.8 Protein interaction network analysis of bta-miR-2285f target gene

(a)靶基因CAB39互作基因网络;(b)靶基因CAB39靶基因生物学功能(BP)和分子功能分析(MF)。(a) Target gene CAB39 interaction gene network;(b) Biological function and molecular function analysis of target gene CAB39.
2.7 bta-miR-2285f与CAB39的靶向关系验证
由图10(a)知,将CAB39基因的野生型和突变型重组质粒经过PCR分析,野生型重组质粒CAB39-3′UTR-WT质粒和突变型重组质粒CAB39-3′UTR-Mut质粒均构建成功。
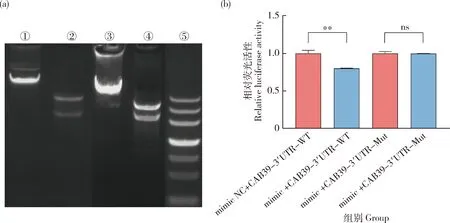
(a)重组质粒酶切鉴定。其中①为添加野生型目的基因的pmirGLO载体,②为野生型重组质粒对照组,③添加突变型目的基因的pmirGLO载体,④为突变型重组质粒对照组,⑤为DNA maker。(b)双荧光素酶活性检测。mimic NC+CAB39-3′UTR-WT:共转染bta-miR-2285f mimic NC和基因CAB39野生型重组质粒。mimic+CAB39-3′UTR-WT:共转染bta-miR-2285f mimic和基因CAB39野生型重组质粒。mimic NC+CAB39-3′UTR-Mut:共转染bta-miR-2285f mimic NC和基因CAB39突变型重组质粒。mimic+CAB39-3′UTR-Mut:共转染bta-miR-2285f mimic和基因CAB39突变型重组质粒。(a) Identification of restriction enzyme products of recombinant plasmids.Among them,① is pmirGLO vector with wild-type target gene added,② is wild type recombinant plasmid control group,③ is pmirGLO vector with mutant target gene added,④ is mutant recombinant plasmid control group,and ⑤ is DNA maker.(b) Dual luciferase activity assay.mimic NC+CAB39-3′utr-wt:bta-miR-2285 fmimic NC and gene CAB39 wild type recombinant plasmid are co-transfected.mimic+CAB39-3′utr-wt:bta-miR-2285f mimic and CAB39 wild-type recombinant plasmid are co-transfected.mimic NC+CAB39-3′utr-mut:bta-miR-2285f mimic NC and CAB39 mutant recombinant plasmid were co-transfected.mimic+CAB39-3′utr-mut:bta-miR-2285f mimic and CAB39 mutant recombinant plasmid are co-transfected.
为验证bta-miR-2285f是否直接靶向调节CAB39基因,将CAB39基因的野生型和突变型重组质粒与bta-miR-2285f mimic、mimic NC分别共转染293T细胞,结果表明,bta-miR-2285f mimic与野生型重组质粒(CAB39-3′UTR-WT)共转染组荧光素酶活性显著低于NC组(P<0.05);而bta-miR-2285f mimic与突变型重组质粒(CAB39-3′ UTR-Mut)共转染组荧光素酶活性与NC组无显著差异(图10(b))(P>0.05)。结果表明,CAB39为bta-miR-2285f的靶基因,bta-miR-2285f可以通过调控CAB39基因表达影响乳脂代谢过程。
2.8 bta-miR-2285f调控乳脂标志基因表达
为探究bta-miR-2285f在奶牛乳脂代谢中的潜在作用,将bta-miR-2285f mimic 和 inhibitor 转染到乳腺上皮细胞中。结果发现,过表达bta-miR-2285f后,乳脂标志基因ELOVL6、SLC27A6、PPARG和FASN的mRNA表达量较对照组(NC)显著增加(图11(a))(P<0.05)。而转染bta-miR-2285f inhibitor后,ELOVL6、SLC27A6、PPARG和FASN的mRNA表达量较对照组显著降低(图11(b))(P<0.05)。
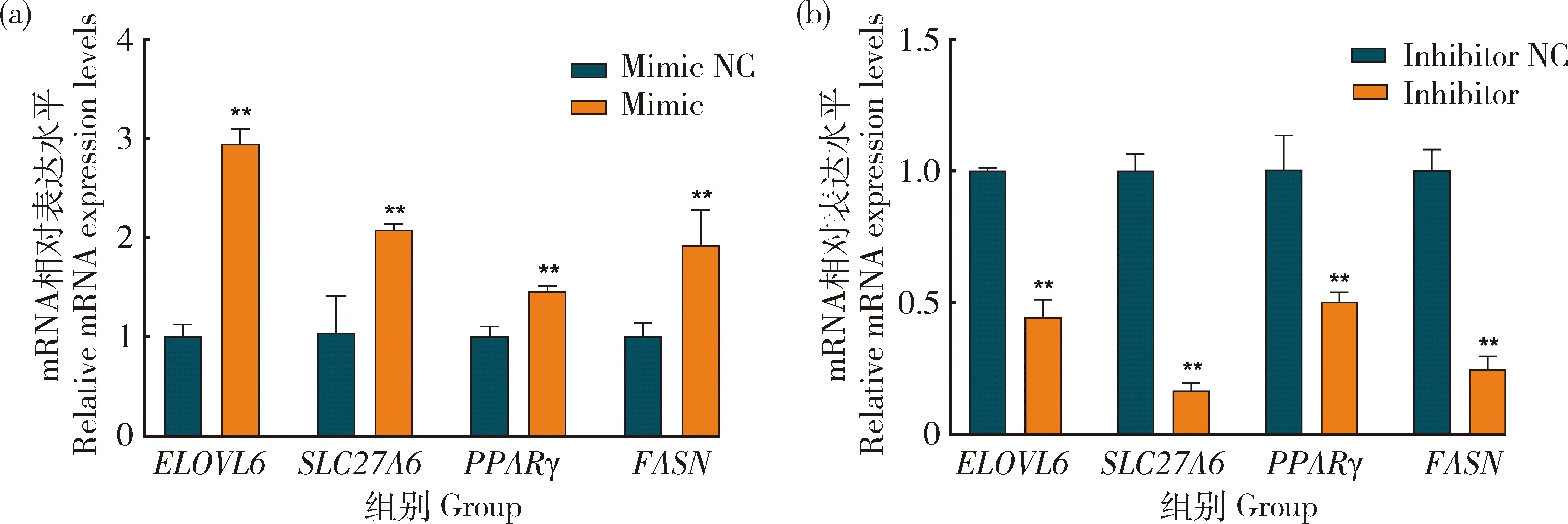
图11 bta-miR-2285f对不同乳脂标志基因的表达水平Fig.11 Expression levels of different milk fat marker genes by Bta-miR-2285f
3 讨 论
哺乳动物中50%的miRNA属于内含子miRNA(Intronic microRNA),其功能是与宿主基因拮抗或协同作用[17]。本研究通过生物信息学分析确定bta-miR-2285f是牛内含子miRNA,其宿主基因为TBK1。虽然TBK1以免疫作用而出名[18],但有研究表明,TBK1可以通过促炎细胞因子激活从而调节小鼠脂肪细胞的生长代谢[19]以及影响细胞凋亡与增殖[20]。TBK1通过影响糖原合成以及在促炎因子与抗炎因子的平衡过程中起关键作用的糖原合成激酶3β(Glycogen synthase kinase 3β)基因从而参与糖原合成和平衡促炎因子与抗炎因子的产生过程[21]。此外,TBK1在其他通路上也有着重要影响,如在胰岛素信号转导通路(TBK1作用于胰岛素受体994位丝氨酸)[22],脂质代谢信号通路、与脂肪细胞合成有关的mTOR通路[23-24]等,因此,bta-miR-2285f对乳脂代谢调节可能反馈调节宿主基因TBK1的表达有关。
启动子CpG岛的DNA甲基化情况与基因的表达调控密切相关[25]。DNA甲基化是真核生物中一种重要的表观遗传修饰,它只发生在CpG双核苷酸的胞嘧啶上[26]。DNA甲基化通过抑制转录因子在基因启动子区域的结合,从而导致基因转录水平的下降甚至停止转录[27],以达到调控基因转录的目的。本研究通过对bta-miR-2285f上游区域(2 kb)和宿主基因上游(2 kb)分析表明不含有明显的CpG岛以及启动子序列,表明其基因可能不是经典的CG-rich启动子[28],说明该区域可能不受甲基化的影响。
内含子miRNA随着宿主基因的启动子共同转录,有共同的调节元件[29]。但是也有研究表明,内含子miRNA不随着宿主基因的启动子转录,有自己独立的转录因子[30]。启动子属于DNA序列的一段,必须与相应的转录因子结合才能激活基因转录,不同的转录因子对基因的表达调控起着不同的作用,基因的表达是这些转录因子共同作用的结果[31]。本研究通过不同软件对bta-miR-2285f-1、bta-miR-2285f-2和TBK1上游(2 kb)启动子序列以及转录结合位点进行分析,发现多个启动子以及转录因子结合位点。研究表明,鳕鱼TBK1基因的基础启动子和阳性转录调节转录起始位点在上游-875 至-425 bp和-245至+28 bp的序列,与本试验预测中国荷斯坦奶牛TBK1基因预测结果相接近[32]。miRNA也可以通过影响基因启动子的CpG岛甲基化,从而在转录水平上直接调控靶基因的表达变化[33]。所以通过对miRNA及其宿主基因CPG岛并结合启动子、转录因子结合位点分析,有助于了解miRNA如何反馈调节宿主基因表达,为后续试验提供理论依据。
MicroRNA(miRNA)在转录水平后与靶基因结合从而发挥调控作用。所以通过预测靶基因并对其所富集通路进行分析可以对研究miRNA的功能机制有着指向作用。通过TargetScan数据库和miWalK数据库对bta-miR-2285f预测并取交集获得靶基因,对其显著富集的前20个通路进行分析。发现候选靶基因主要参与脂质代谢相关的通路,其中mTOR信号通路能够促进脂肪合成[34]、甘油酯代谢通路、PI3K-Akt信号通路主要与脂肪代谢有关[35]、ErbB信号通路可以激活Akt信号通路和MAPK信号通路从而调控脂肪代谢[36]、Hippo信号通路能够抑制成熟脂肪细胞的生成[37]。同时,通过对bta-miR-2285f富集通路进行分类注释可以发现,bta-miR-2285f 还显著富集于免疫疾病相关通路中,其可能原因可能与宿主基因TBK1的免疫调控有关。
双荧光素酶试验表明,bta-miR-2285f mimic+CAB393′-UTR野生型组与bta-miR-2285f mimic NC组相比,野生型组的相对荧光素酶活性显著降低(P<0.05)。然而,在突变组中没有观察到荧光活性的变化,这表明CAB39是bta-miR-2285f的一个靶基因。有研究表明,小鼠中CAB39可以激活AMPK通路从而控制脂肪细胞的脂肪生成和脂肪溶解[38]。CAB39基因通过AMPK/Akt信号通路抑制脂肪酸诱导促炎细胞因子的产生[39]。Wang等[340]研究表明,CAB39基因可以作用于PI3K-Akt通路调控人机体葡萄糖摄取代谢。CAB39基因通过激活mTOR信号通路影响细胞的增殖和凋亡[41]。通过对CAB39蛋白互助网络分析,可以关注到基因CAB39与脂质代谢密切相关。通过对CAB39互作基因的KEGG和GO分析可知CAB39互作基因可以调控蛋白质、葡萄糖、甘油酯的代谢,由此推断bta-miR-2285f可通过与CAB393′-UTR结合来调控基因CAB39的表达,从而发挥对乳代谢调控作用。通过对miRNA靶基因预测及其功能分析,靶向关系验证对miRNA调控乳脂代谢提供了参考。
bta-miR-2285f对乳脂标志基因的表达含量的检测结果表明,bta-miR-2285f能够显著调控ELOVL6、PPARG、FASN和SLC27A6基因。ELOVL6是一些哺乳动物内源性合成饱和和单不饱和长链脂肪酸的重要蛋白质[42]。ELOVL6同时也是影响乳脂表达的标志基因。PPARG能够促进奶牛乳腺上皮细胞甘油三酯的合成[43]。FASN在中国荷斯坦牛乳脂合成和代谢中起着重要作用[44]。同时研究发现,ELOVL6基因表达量上调会促进PPARG、FASN的表达量[45]。SLC27A6基因能够调节脂肪酸在牛乳腺中向大量乳脂合成[46-47]。bta-miR-2285f在中国荷斯坦牛不同组织中乳腺表达水平最高(P<0.05),表明bta-miR-2285f在乳腺上皮细胞中高表达可能影响乳脂标志基因的表达从而促进乳脂的分泌。为后续bta-miR-2285f对乳中甘油三酯,脂滴的影响提供了一定的依据。
4 结 论
综上所述,牛特异性bta-miR-2285f在乳腺组织中的表达水平最高,其靶基因集合在mTOR信号通路、PI3K-AKT信号通路、ErbB信号通路、Hippo信号通路以及甘油酯代谢等乳脂代谢相关的信号通路中显著富集。构建了可能影响乳脂代谢bta-miR-2285f-mRNA调控网络,并证明了bta-miR-2285f可以直接靶向CAB39的3′UTR。此外,bta-miR-2285f mimic可以显著上调乳腺上皮细胞中乳脂标志基因ELOVL6、SLC27A6、PPARG和FASN的表达。该研究结果为进一步探究bta-miR-2285f调节中国荷斯坦奶牛乳脂合成代谢的潜在机制提供理论基础。

