小麦光温敏雄性不育系BS237ω-黑麦碱基因克隆及序列分析
侯起岭 杨卫兵 娄红耀 杜冰 高建刚 赵昌平 张风廷 秦志列


关键词:小麦:光温敏雄性不育系:ω-黑麦碱:基因克隆:序列分析:乳糜泻
中图分类号:S512.1:Q785 文献标识号:A 文章编号:1001-4942(2023) 03-0001-08
黑麦(Secale cereale L.)1R染色体短臂(1RS)取代普通小麦1B染色体短臂(1BS)形成的1BL/1RS易位系携带了抗叶锈、条锈和白粉病等优良基因,具有抗性好、丰产性好、适应性广等优点,在育成的小麦品种中占有30%以上的比例。黑麦种子中的醇溶性储藏蛋白被称作黑麦碱(seca-lin),1RS的引入使编码γ-黑麦碱和ω-黑麦碱的Sec-I位点进入小麦基因组中,但同时也使其丧失了低分子量谷蛋白Clu-B3位点和小麦醇溶蛋白Gfi-B1位点,而黑麦碱不能补偿因丧失低分子量谷蛋白亚基引起的谷蛋白聚合体数量减少,从而导致面团吸水性增大、稳定性和延伸性降低,小麦面团加工品质下降。1RS染色体上的ω-黑麦碱基因是导致小麦面团加工品质下降的重要原因之一。因此,充分了解小麦1BL/1RS易位系的ω-黑麦碱基因和蛋白的序列特征对改良小麦1BL/1RS易位系的加工品质有重要意义。
ω-黑麦碱由1R染色体臂上的Sec-1位点控制,该位点由紧密连锁的多个基因组成,ω-黑麦碱基因家族包含大约15个成员。ω-黑麦碱是与ω-醇溶蛋白相关的一类小单体蛋白(45~ 50kD),富含谷氨酰胺,几乎不含有半胱氨酸,无法参与链内或链间二硫键的形成。目前,许多ω-黑麦碱基因已经陆续从黑麦、小黑麦和小麦1BL/1RS易位系中克隆出来,这些ω-黑麦碱基因的氨基酸序列均是保守的,由信号肽、N端区域、重复结构域和C端区域组成。柴建芳等发现沉默ω-黑麦碱基因可以在不影响产量的前提下提高小麦1B/1R易位系的加工品质。张士昌等利用低能N+离子束诱变1BL/1RS易位系小麦种子,筛选出了9个ω-黑麦碱基因Sec-1位点表达缺失的新的1BL/1RS易位系种质。这些研究为改良小麦1BL/1RS易位系的加工品质和创制新的小麦1BL/1RS易位系种质提供了很好的思路。
乳糜泻(celiac disease,CD)是一种由T-细胞介导的自身免疫性疾病,是因摄入小麦、大麦或黑麦中的面筋蛋白而引发的慢性小肠炎症性疾病,具有基因易感性。目前,已鉴定出的能够诱发乳糜泻疾病的小麦蛋白肽段主要分布在醇溶蛋白中,主要类型是α-、β-和γ-醇溶蛋白,ω-醇溶蛋白中也含有部分肽段。小麦上报道的与CD相关的表位有5个来自α-醇溶蛋白,8个来自γ-醇溶蛋白,3个来自ω-醇溶蛋白.还有2个来自LMW-GS,1个来自HMW-GS。四肽序列PSQQ、QQPY、SPQQ、QQQP和PQQP与一系列腹腔活性肽共同存在,是潜在的毒性抗原表位,其中PQQP在谷物ω-黑麦碱氨基酸序列的可变重复区分布广泛,PSQQ和QQQP目前在贫硫醇溶蛋白中还没有被发现。
BS237是北京市农林科学院杂交小麦研究所选育的骨干不育系,具有育性彻底、配合力高、抗性好、农艺性状优良等特点,但其ω-黑麦碱基因的详细信息尚未有研究。鉴于此,本研究以BS237为材料,通过PCR法获得具有完整开放阅读框的ω-黑麦碱基因的编码区序列,并进行序列比对和进化分析,同时对ω-黑麦碱中可能存在的T-细胞免疫肽段识别分析,以期为杂交小麦1BL/1RS类型不育系加工品质的分子改良和乳糜泻疾病预防提供理论依据和参考。
1材料与方法
1.1试验材料
本试验所用小麦不育系BS237由北京市农林科学院杂交小麦研究所提供,2022年6月从北京市农林科学院杂交小麦研究所顺义基地(40°08′N,116°39′E)不育系繁殖圃收获适量的BS237种子,供后续实验分析使用。
1.2试验方法
1.2.1 DNA提取 将小麦种子置于铺有滤纸的培养皿中培养,1周后取适量叶片,用CTAB法提取基因组DNA。
1.2.2 引物设计及扩增 利用1RS特异引物Bmac0213-F/Bmac0213-R检测BS237是否含有1RS片段,根据已注册的ω-黑麦碱基因序列的保守区设计1对可扩增完整编码区的引物ω-F/ω-R,详见表1。
PCR反应体系:2×Taq Plus Master Mix 10μL,引物(10μmol.L-1)各1μL,模板DNA 2μL,ddH,0补充至20 μL。PCR扩增程序为:98℃预变性30 s;98℃变性10 s,60℃退火20 s,72℃延伸30 s,35个循环;720C延伸10 min。所有引物均由北京睿博兴科生物技术有限公司合成。采用诺唯赞生物(Vazyme Biotech)公司的2xTaq Plus Mas-ter Mix高保真酶,按说明书进行PCR扩增。
1.2.3 PCR产物纯化及克隆测序 将扩增产物在1%琼脂糖凝胶上进行分离,用琼脂糖凝胶回收试剂盒[天根生化科技(北京)有限公司]回收目的条带,与pEasy-Blunt Zero载体(北京全式金生物技术有限公司)连接,将连接产物转化大肠杆菌Trans-Tl感受态(北京全式金生物技术有限公司),涂布于含Kan抗生素的LB培養基上,37℃倒置培养过夜。挑取一定数量的单克隆,用M13引物进行菌落PCR检测,将阳性克隆送北京擎科生物科技有限公司测序。
1.2.4序列分析与进化分析 采用NCBI(ht-tps://www.ncbi. nlm. nih. gov/)和DNAMAN软件对测序结果进行比对、拼接及翻译。利用MegaX软件(https://www. megasoftware. net/)构建neigh-bor-joining系统发育树。
2结果与分析
2.1 BS237的ω-黑麦碱基因的检测、克隆及核苷酸序列分析
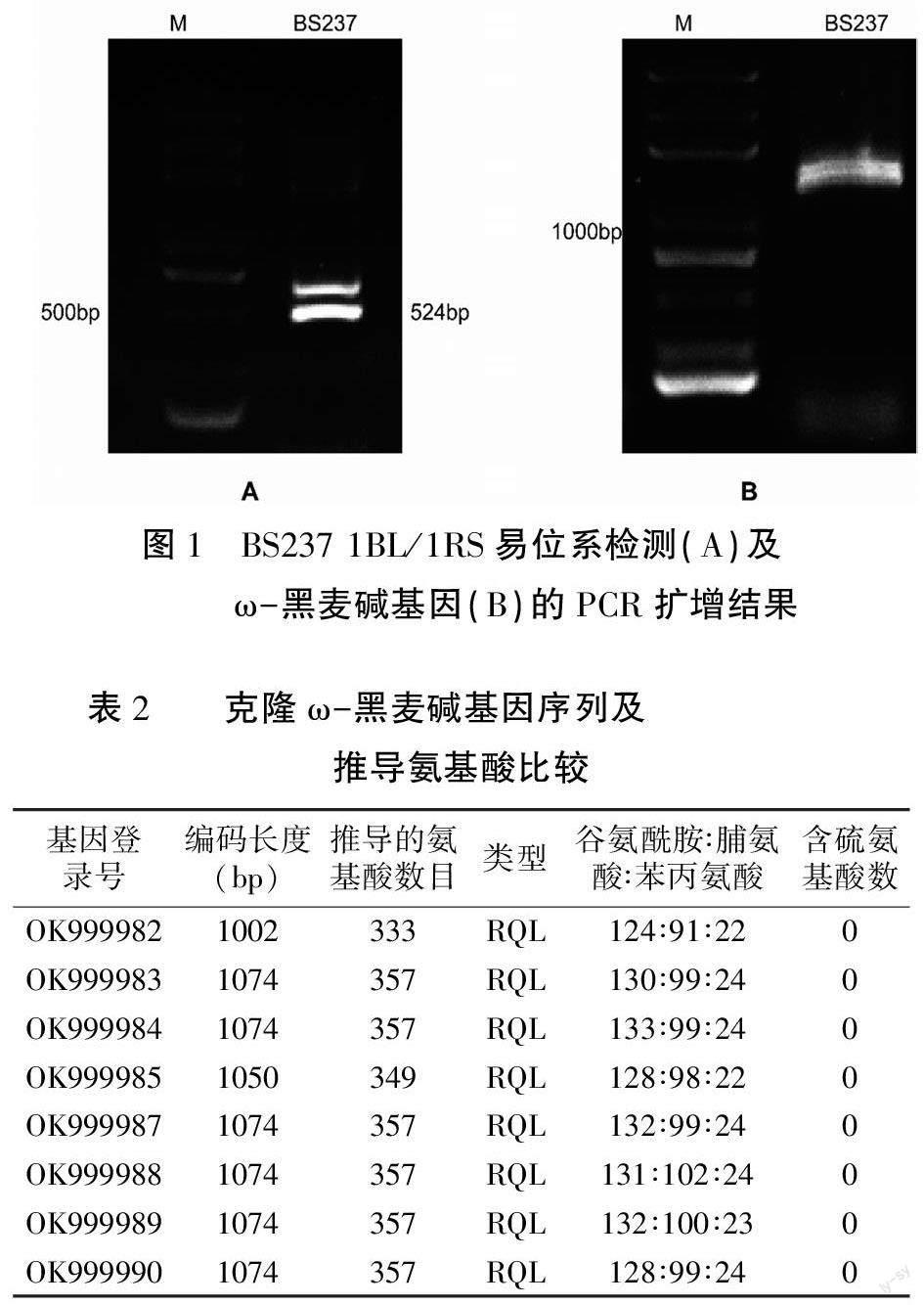
利用1RS特异引物Bmac0213-F/Bmac0213-R扩增BS237的DNA,得到524 bp的特异性条带(图1A),说明BS237是1BL/1RS易位系。利用引物ω-F/ω-R对BS237的ω-黑麦碱基因进行PCR扩增得到单一条带(图1B),对目的条带进行纯化和克隆测序,共得到8个具有完整开放阅读框的不同基因序列(由TBtools软件预测),可编码333~357个氨基酸残基。NCBI BLAST分析表明:克隆获得的8个序列与已知的ω-黑麦碱基因序列的相似度在91%~100%之间,推断其为ω-黑麦碱基因家族。将这8个序列提交GenBank,获得的登录号分别为OK999982~OK999985和OK999987~OK999990,其中,OK999982的长度为1002 bp,OK999985的长度为1050 bp,其余6个序列的长度均为1074 bp(表2)。
2.2克隆基因的推导氨基酸序列分析
本研究把克隆出来的8个ω-黑麦碱蛋白序列与已知的小麦1BL/1RS易位系(AMD09626.1)、六倍体小黑麦(ACQ83629.1)和黑麦(ACQ83628.1)的ω-黑麦碱蛋白序列比对,发现这8个蛋白的氨基酸序列结构均具有ω-黑麦碱典型的分子结构特征(图2),即包含信号肽区(19个氨基酸)、N-端非重复区(12个氨基酸)、可变重复区和C-端非重复区(6个氨基酸)。与已知的ω-黑麦碱的核苷酸序列类似,本研究发现的8个序列在可变重复区富含谷氨酰胺( glutamine,Q)、脯氨酸(pro-line,P)和苯丙氨酸(phenylalanine,F),但缺乏如半胱氨酸(cysteine,C)和甲硫氨酸(methionine,M)的含硫氨基酸:根据N-端非重复区起始3个氨基酸残基的特性,这8个基因均属于RQL型的ω-黑麦碱基因(表2)。
8个序列在N-端非重复区和C-端非重复区很保守,在信号肽区仅OK999984、OK999985和OK999987各存在1个氨基酸的替换,主要差异体现在可变重复区,且表现在氨基酸的替换和插入上(图2):OK999982与普通小麦1BL/1RS易位系中的ω-黑麦碱氨基酸序列(AMD0926.1)高度相似,只存在1个位点的氨基酸插入。其他7个序列与六倍体小黑麦的ω-黑麦碱氨基酸序列(ACQ83629.1)高度相似,其中,OK999983在1个位点存在氨基酸的插入,9个位点存在氨基酸的替换;OK999984在1个位点存在氨基酸的插入,4个位点存在氨基酸的替换:OK999985在1个位点存在氨基酸的插入,4个位点存在氨基酸的替换;OK999987在5个位点存在氨基酸的替换,在1个位点存在氨基酸的插入:OK999988在1个位点存在氨基酸的插入,在4个位点存在氨基酸的替换:OK999989在9个位点存在氨基酸的替换,在1个位点存在氨基酸的插入:OK999990在10个位点存在氨基酸的替换,在1个位点存在氨基酸的插入。
2.3ω-黑麦碱基因的进化分析
将克隆的ω-黑麦碱蛋白序列在NR数据库中进行Blast,与GenBank中33个来自于9个物种具有完整编码框的ω-黑麦碱蛋白、ω-醇溶蛋白和C-醇溶蛋白序列(表3)进行多序列比对并构建最大似然(maximum likelihood)系统发生树(图3)。根据ω-醇溶蛋白和ω-黑麦碱蛋白序列的相似程度分成3类:Clade 1、Clade 2和Clade 3(图3)。Clade 1包含12个普通小麦及其祖先种的ω-醇溶蛋白,其中5个为普通小麦(Triticum,aestivum, L.)ω-醇溶蛋白(ATY51568,AAG17702,AG220259,ATY51554,AG220255),1个为乌拉尔图小麦(T.urartu)ω-醇溶蛋白(AKB95614),4个为尾状山羊草(Aegilops markgrafii)ω-醇溶蛋白(AFV52225,AFV52226,AFV52227,AFV52228),2个为野生二粒小麦(T.dicoccoides)ω-醇溶蛋白(AKA88516,AKA88509)。Clade 2中除了本研究克隆的8个小麦ω-黑麦碱蛋白还包含17个来自小麦、小黑麦和黑麦的ω-黑麦碱蛋白,其中5个为小麦1BL/1RS易位系ω-黑麦碱蛋白(AMD09626, ACQ83636, ACQ83642, ACQ83637,AMD09620),4个为六倍体小黑麦(AABBRR)ω-黑麦碱蛋白(ACQ83629,ACQ83630,ACQ83631,ACQ83632),3个为八倍体小黑麦(AABBDDRR)ω-黑麦碱蛋白(ACQ83633,ACQ83634,ACQ83635),5个为黑麦(Secale cereale L.)ω-黑麦碱蛋白(ACQ83624,ACQ83625,ACQ83626,ACQ83627,ACQ83628)。Clade 3包含了4个大麦(Hordeum vulgare L.)C-醇溶蛋白(AAB28161,AFM77749,CAA42642,AAA92333)。
根據相似度高低,Clade 2可以进一步分为4个亚组:Clade 2-1、Clade 2-2、Clade 2-3和Clade2-4。其中,Clade 2-1包含3个黑麦ω-黑麦碱蛋白(ACQ83626,ACQ83627,ACQ83628)和2个八倍体小黑麦ω-黑麦碱蛋白(ACQ83633,ACQ83635)。Clade 2-2包含3个本研究中克隆的ω-黑麦碱蛋白(OK999985,OK999988,OK999989),2个黑麦ω-黑麦碱蛋白(ACQ83624,ACQ83625)和1个六倍体小黑麦ω-黑麦碱蛋白(ACQ83630);OK999985与黑麦ω-黑麦碱蛋白(ACQ83624,ACQ83625)聚类在一起,OK999988和OK999989与六倍体小黑麦ω-黑麦碱蛋白(ACQ83630)聚类在一起。Clade 2-3包含2个本研究中克隆的∞-黑麦碱蛋白(OK999983,OK999990),3个六倍体小黑麦ω-黑麦碱蛋白(ACQ83629,ACQ83631,ACQ83632),1个八倍体小黑麦ω-黑麦碱蛋白(ACQ83634)和3个小麦1BL/1RS易位系ω-黑麦碱蛋白(ACQ83636,ACQ83637,AMD09620)。Clade 2-4包含了3个本研究中克隆的ω-黑麦碱蛋白(OK999982,OK999984,OK999987),2个小麦1BL/1RS易位系ω-黑麦碱蛋白(AMD09626,ACQ83642)。
本研究克隆的8个ω-黑麦碱蛋白全部聚类在Clade 2中,与Clade 1中的普通小麦及其祖先种的ω-醇溶蛋白和Clade 3中的大麦C-醇溶蛋白不在同一类中,说明ω-黑麦碱基因与小麦族ω-醇溶蛋白基因和大麦C-醇溶蛋白基因亲缘关系较远,存在明显的进化关系,具有基因组特异性。通过进化分析发现不同物种的ω-黑麦碱基因具有较高的同源性,8个ω-黑麦碱基因大部分与小麦1BL/1RS易位系、六倍体小黑麦的ω-黑麦碱基因具有较高的同源性,OK999985与黑麦的ω-黑麦碱基因具有较高的同源性。
2.4克隆基因的T-细胞免疫肽段识别分析
CD毒性肽的识别分析结果(图2,表4)表明,ω-醇溶蛋白中已知的T-细胞免疫肽Gli-ωt(PQQPFPQQ)在克隆的8个ω-黑麦碱蛋白中均有4~6个拷贝的分布,Gli-ω1( PFPQPQQPF)在除OK999984和OK999987外的6个(1)-黑麦碱蛋白中各有1个拷贝的分布,Gli-ω2(PQPQQPF-PW)在克隆的8个ω-黑麦碱蛋白中均没有分布。γ-醇溶蛋白中已知的T-细胞免疫肽DQ2.5-glia-γ5(QQPFPQQPQ)在8个ω-黑麦碱蛋白中有2~3个拷贝的分布;DQ2.5-glia-γ4c(QQPQQPFPQ)除了在OK999990中没有分布,在其余7个ω-黑麦碱蛋白中均有1个拷贝的分布。3个四肽T-细胞免疫肽(PQQP,QQPY,SPQQ)在8个ω-黑麦碱蛋白中均有分布,其中QQPY只有1个拷贝的分布,SPQQ有1~2个拷贝的分布,PQQP在8个ω-黑麦碱蛋白中分布数量较多,高达27~34个拷贝。
3讨论与结论
通过对克隆的8个ω-黑麦碱基因推导的氨基酸序列比对分析得出,8条序列在可变重复区富含谷氨酰胺、脯氨酸和苯丙氨酸,且谷氨酰胺:脯氨酸:苯丙氨酸大于5:4:1,这与小麦的ω-醇溶蛋白基因推导的氨基酸序列中的谷氨酰胺、脯氨酸和苯丙氨酸的比例不一样(4:3:1)。谷氨酰胺的数量最多(124~133),在不同序列间变化较大;脯氨酸的数量次之(91~102),除OK999982数量较少(91)外,其他序列间数量变化较小;苯丙氨酸的数量最少(22~24),8个序列间差异不大,比较稳定。8条序列中均不含有含硫氨基酸,没有半胱氨酸和甲硫氨酸的分布,这与前人的研究结果一致,这种特有的序列特征表明ω-黑麦碱蛋白不能在自身内部和其他蛋白质形成链间连接,可能具有独特的功能。
根据系统进化树发现克隆的8个ω-黑麦碱基因在小麦1BL/1RS易位系、六倍体小黑麦、八倍体小黑麦和黑麦间存在高度同源性,表明该基因家族在黑麦R染色体或整个黑麦基因组与小麦或小黑麦基因组整合后变异较少,具有基因组特异性。
CD毒性肽的识别分析表明,有3个四肽T-细胞免疫肽(PQQP,QQPY,SPQQ)分布在8个ω-黑麦碱基因中,其中PQQP在每个基因的分布数量高达27~34個拷贝,这说明ω-黑麦碱基因可能是导致乳糜泻症状发生的主要基因之一。在未来小麦育种中研究小麦1BL/1RS易位系的T-细胞免疫肽段类型、活性特点以及ω-黑麦碱与人类乳糜泻症状之间的关系对优质小麦和优质杂交小麦选育具有重大意义。
可以通过操纵ω-黑麦碱基因已知的遗传信息来降低其基因表达量,进一步改善小麦1BL/1RS易位系的品质。任燕等认为反义RNA虽然可降低黑麦碱的表达量,但利用反义RNA去除由Sec-1位点产生的所有RNA来降低黑麦碱含量的方法可能难以实施。研究表明,可以通过降低或沉默ω-黑麦碱基因Sec-1位点的表达,创制Sec-1位点缺失的突变体或者利用RNA干扰或基因编辑技术降低甚至沉默∞-黑麦碱基因的表达来实现。同时,在1RS末端导入Glu-B3和Gli-B1位点,可以改善1BL/1RS小麦面团的加工品质。育种过程中配置杂交组合时,如果亲本有一个为非1BL/1RS易位系,含有1RS的杂交后代品质受到的负面影响也可被掩盖,用1BL/1RS易位系含有高分子量谷蛋白亚基5+10的品系制作的面包体积最大,面团强度也最大。因此,往小麦1BL/1RS易位系导入优质高分子量谷蛋白亚基或者与非1BL/1RS易位系杂交也可以改善小麦1BL/1RS易位系的加工品质。
在杂交小麦优质不育系的选育过程中,应该进一步明确亲本材料的遗传背景,注重早代材料的鉴定,加强非1BL/1RS小麦易位系种质资源的使用,还可以利用骨干不育系与优质小麦种质资源多配置组合,逐步改良杂交小麦不育系的品质特性。此外还可以通过创制Sec-1位点缺失的1BL/1RS小麦不育系来改良杂交小麦的加工品质。
本研究从BS237中克隆了8个具有完整开放阅读框的ω-黑麦碱基因,与前人研究结果相符。根据其N-末端序列的前3个氨基酸残基的特点,这8个基因均属于RQL类型;与已知基因的比对分析表明,不同ω-黑麦碱基因在N-端非重复区和C-端非重复区序列相对保守,在中间可变重复区存在氨基酸的替换和插入;进化分析发现,这8个ω-黑麦碱基因很保守,在黑麦R染色体或整个黑麦基因组与小麦或小黑麦基因组整合后变异较少,与小麦1BL/1RS易位系、六倍体小黑麦、八倍体小黑麦和黑麦间存在高度同源性,与小麦及其祖先种的ω-醇溶蛋白基因和大麦C-醇溶蛋白基因亲缘关系较远。CD毒性肽的识别分析表明,已知的分布于ω-醇溶蛋白中的T-细胞免疫肽Gli-ωt(PQQPFPQQ)、分布于γ-醇溶蛋白中的T-细胞免疫肽DQ2.5-glia-γ5(QQPF-PQQPQ)、3个四肽T-细胞免疫肽(PQQP,QQPY,SPQQ)在8个基因中均有分布,其中PQQP在每个基因分布的拷贝数高达27及以上,可能导致潜在的人类乳糜泻毒性。

