类鼻疽伯克霍尔德sRNA 敲除株的构建及生物学功能初步评价
宋心怡,厉安洋,李艳梅,徐淑慧,夏乾峰,
(1.海南医学院公共卫生与全健康国际学院,海南 海口 571199;2.海南医学院热带医学院,海南 海口 571199)
类鼻疽伯克霍尔德菌(burkholderia pseudomallei,B.pseudomallei)是一种兼性的细胞内革兰氏阴性杆菌,是类鼻疽(Melioidosis)的病原体[1]。B.pseudomallei多流行于热带,亚热带地区,特别是东南亚和北澳大利亚多见,我国的疫源地为海南、广东、广西、福建、台湾等地,以海南省的发病率最高[2]。主要通过受污染的水源和土壤以及破损的皮肤入侵人体,病例以肺部感染和败血症为主,是本地居民或外来旅游人群感染的主要风险,未经治疗的类鼻疽患者致死率可高达40%[3,4]。目前类鼻疽伯克霍尔德菌致病机理和宿主的免疫保护机制尚不完全清楚,且无有效疫苗[5]。
细菌小RNA(sRNA)是一类具有重要调控功能的非编码RNA,一般长度在50~500 nt 之间,不编码蛋白,通过与靶标基因 mRNA 碱基互补配对,在转录水平影响对基因的表达进行调节,与细菌的生长、代谢、应激反应和致病性等密切相关[6]。在前期工作中,课题组表达出促进sRNA 表达的分子伴侣蛋白Hfq,通过Hfq 蛋白为基础的免疫共沉淀技术和RNA-seq 测序分析,发现并鉴定了9 种新的类鼻疽伯克霍尔德菌sRNA[7]。本研究选定了其中一种sRNA,位于HNBP001 的2 号染色体上sRNA 基因片段(Sequence ID:CP038806.1,556 924 bp~557 397 bp),暂将其命名为sRNA9,通过生物学实验对该sRNA 在B.pseudomallei致病过程中的重要生物学功能和作用机制进行初步探索,为抗B.pseudomallei感染提供新的分子靶标,同时也对类鼻疽的防治和诊断提供新的思路。
1 材料与方法
1.1 材料
1.1.1 主要菌株、质粒及细胞 类鼻疽伯克霍尔德菌野生株HNBP001 来源于课题组分离保存菌株[8]、质粒TPR-pK18mobSacB 由课题组改造增加甲氧苄啶抗性片段,本实验室保存[9]、大肠杆菌S17-λpair 感受态细胞、DH5ɑ 感受态细胞购于北京庄盟国际生物基因科技有限公司。
1.1.2 主要试剂和仪器 细菌基因组DNA 提取试剂盒、PCR 产物纯化试剂盒、胶回收试剂盒及质粒提取试剂盒,天根;限制性内切酶(BamHⅠ、XbalⅠ、HindⅢ)和T4DNA 连接酶(Biolabs 公司);甲氧苄啶、庆大霉素、卡那霉素,Biosharp;普通PCR 仪,Biorad;多功能酶标仪,Bioteck;凝胶成像分析系统,君意。
1.1.3 培养基 LB 固体培养基(g/L):胰蛋白胨10 g、酵母5 g、氯化钠10 g、琼脂粉16 g;15%蔗糖固体培养基(g/L):胰蛋白胨10 g、酵母5 g、蔗糖150 g、琼脂粉16 g。
1.2 方法
1.2.1 引物设计 根据本实验室上传至NCBI 中类鼻疽伯克霍尔德菌HNBP001 的基因序列,针对编码sRNA9 的上下游同源臂序列设计引物:9sF/9sR、9xF/9xR(图A),用于扩增目的sRNA 的同源臂,R1/F1 引物用来验证基因敲除株。所有引物均由生工生物工程公司合成,引物序列详见表1。

表1 PCR 引物序列Tab 1 PCR primer sequence
1.2.2 载体构建 提取HNBP001 的基因组,使用引物9sF/9sR、9xF/9xR 分别进行上、下游同源臂PCR 扩增,酶切后连接。以质粒TPR-pK18mob-SacB 为模板,使用HindⅢ和BamHⅠ进行双酶切,纯化,酶切体系见表2。使用T4 连接酶对上、下游同源臂与质粒的酶切产物于16 ℃连接数小时。将连接后产物加入DH5ɑ 感受态细胞,按照标准程序进行热激转化,涂布含抗生素的平板。培养12 h后,挑取单菌落于含抗生素的LB 液体培养基,培养过夜后使用Plasmid min kit 试剂盒进行质粒提取,随后将质粒进行酶切验证。

表2 酶切体系Tab 2 System of enzyme digestion
1.2.3 ΔsRNA9 敲除株的构建与鉴定 取适量酶切验证后的质粒稀释,转化S17-1 感受态细胞,感受态细胞经复苏后,涂布抗生素LB 固体平板,培养12 h。挑取平板上的单菌落于含抗生素的LB 液体培养基,同时挑取HNBP001 的单菌落于无抗LB 培养基,培养过夜。转化后S17-1 和HNBP001 菌液各取1 mL,离心取上清,取500 μL 培养基重悬沉淀物,取重悬后的菌液混匀滴加于滤膜上,培养数小时。吸取适量LB 液体培养基清洗菌体,稀释不同浓度梯度(10-1、10-2)后涂布含抗生素的平板,培养36~48 h。挑取抗生素LB 固体平板上单菌落于含抗生素的LB 液体培养基,培养过夜。提取菌液基因组,PCR 验证,获得结合成功的菌液。将结合成功的菌液取50 μL 加入无抗LB 培养基中,培养过夜。将菌液稀释涂布于LB 固体筛选培养基。挑取平板上单菌落,以引物R1/F1 验证HNBP001 是否敲除成功,HNBP001 作为阳性对照,扩增纯化后送测序,比对测序序列,将敲除成功的菌株暂命名为sRNA9。
1.2.4 生长曲线测定 提前一晚挑取野生株HNBP001 和敲除株sRNA9 的单菌落加入新鲜LB 培养基中,将细菌培养至生长对数期。将生长对数期的菌液稀释至OD600=0.1,按照200 μL/孔的液体量加入96 孔板,每个样本设置5 个重复,使用多功能酶标仪程序设置为37°C、双轨道震荡、间隔时间2 h,连续检测72 h 在OD600的吸光度,保存实验结果,重复3 次实验,使用GraphPad 8.0.2 软件绘制生长曲线。
1.2.5 细菌的泳动性实验 配置3%的LB 软琼脂培养基,提前一晚准备好新鲜野生株HNBP001 和敲除株sRNA9 单菌落平板,挑取单菌落在LB 培养基中培养至生长对数期。将菌液稀释至OD600=0.1,吸取2.5 μL 稀释后菌液悬空滴至3%LB 软琼脂培养基,设置3 个重复,晾干10~15 min,放入37 ℃培养箱培养24 h,测量细菌抑菌圈的直径。
1.2.6 生物被膜实验 细菌培养方法同前,将野生株HNBP001、敲除株sRNA9 的菌液浓度调整至OD600=0.1,取5 mL 玻璃试管分别加入3 mL 稀释后的HNBP001、sRNA9 菌液,设置3 个重复,将玻璃试管放入37 ℃培养箱培养48 h,观察到明显区别。移液器吸出玻璃管中菌液,1 × PBS 清洗残余杂质,加入4 mL 1%的结晶紫染色30 min。吸出结晶紫溶液,1 × PBS 再次清洗试管,加入3 mL 30% 乙酸反应20 min,测OD595吸光度值。
1.2.7 药敏实验 使用珠海迪尔非发酵菌/氧化酶阳性革兰氏阴性杆菌鉴定药敏试剂板,严格按照说明书操作。提前准备好新鲜单菌落平板,挑取适宜大小单菌落加入稀释液瓶内研磨混匀制备成细菌悬液,吸取细菌悬液50 μL,加入M-H 肉汤培养基,混匀后加入抗菌药物孔中,100 μL/孔,18 h 后判读抗菌药物MIC 测定结果。
1.3 统计学处理
使用GraphPad 8.0.2 软件进行绘图及统计分析,运用Wilcoxon 符号秩检验对配对样本进行差异性分析,运用Mann-WhitneyU检验对非配对样本进行差异性分析,P<0.05 为差异有统计学意义。
2 结果与分析
2.1 敲除株ΔsRNA9 的构建及验证
2.1.1 目的基因上下臂PCR 扩增结果 以HNBP001 的DNA 为模板,使用引物9sF/R、9xF/R 分别PCR 扩增上下臂片段,得到682 bp,793 bp 长度条带,电泳结果见图1。
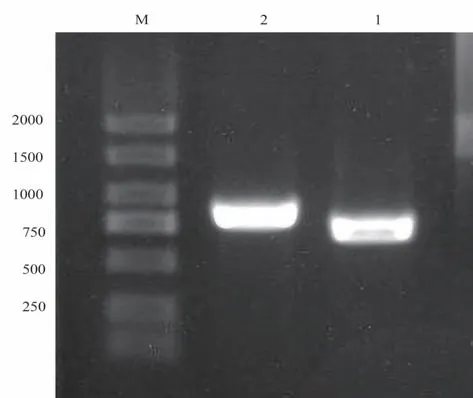
图1 目的基因PCR 扩增结果Fig 1 PCR amplification results of the up and down stream sequences of the target gene
2.1.2 重组载体TPR-pK18mobSacB-sRNA9 的酶切验证 将sRNA9 上、下臂片段连接到质粒TPR-pK18mobSacB 上,连接产物转化至DH5ɑ,涂布至含甲氧苄啶的平板,挑取单菌落提取质粒后用HindⅢ、BamHⅠ双酶切验证,结果见图2。
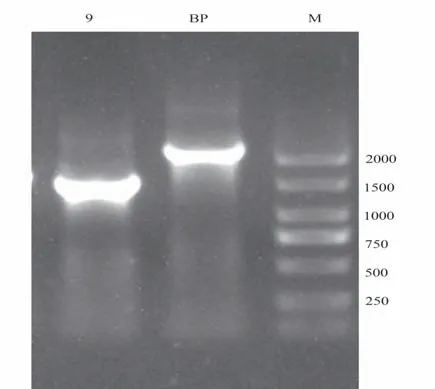
图2 重组载体TPR-pK18mobSacB-sRNA9 的双酶切验证Fig 2 Verification of recombinant plasmid TPR-pK18mobSacB-sRNA9 by double enzyme digestion
2.1.3 类鼻疽伯克霍尔德菌sRNA9 敲除株的验证 将 构 建 好 的 重 组 质 粒TPR-pK18mobSacB-sRNA9 转化S17-1 感受态细胞,与HNBP001 进行双交换,通过甲氧苄啶和庆大霉素双抗板和15%蔗糖板进行筛选,若双抗板不生长,15%蔗糖板生长,则挑取单菌落PCR 验证,得到类鼻疽伯克霍尔德菌sRNA9 敲除株,并提取基因组送往测序公司进一步验证。野生株HNBP001 和敲除株△sRNA9 使用验证引物R1/F1 进行PCR 扩增,HNBP001 包含sRNA9敲除目的片段,扩增大小为2 227 bp,△sRNA9不含sRNA9敲除目的片段,扩增产物大小为1 753 bp,凝胶电泳结果见图3,敲除株构建成功。
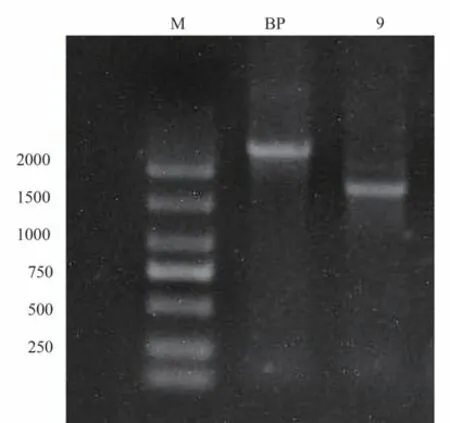
图3 sRNA9 敲除株验证Fig 3 Validation of gene sRNA9 knock-out mutant
2.2 生长曲线
提前准备好单菌落平板,挑取单菌落过夜培养至生长对数期,将菌液稀释至OD600=0.1,多功能酶标仪的程序设置为每间隔4 h 测定OD600吸光度。生长曲线在不同时间段重复3 次,每组设置3 个重复样本。以HNBP001 菌株作为对照,绘制生长曲线图,结果见图4。野生株HNBP001 和敲除株△sRNA9 均在8 h 进入生长对数期,生长对数期后敲除株△sRNA9 生长速率显著低于野生株HNBP001。结果表明,敲除sRNA9 片段会影响B.pseudomallei在LB 培养基中的生长。
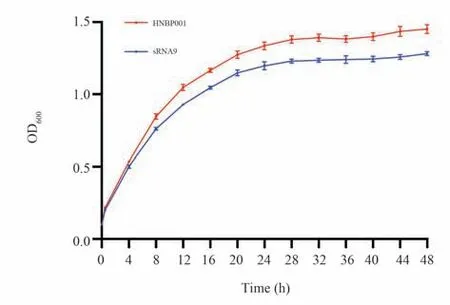
图4 类鼻疽伯克霍尔德菌野生株HNBP001 与敲除株△sRNA9 生长曲线Fig 4 Growth curve of B.pseudomallei HNBP001 WT and△sRNA9
2.3 细菌的泳动实验
在类鼻疽伯克霍尔德菌获取营养和侵袭宿主细胞时,其泳动能力起重要作用。本实验通过观察细菌在3%的软琼脂培养基的泳动环直径大小来判断敲除sRNA9 基因后的泳动能力。以野生株HNBP001 作为参照对象,发现△sRNA9 泳动环直径减小,表明sRNA9 基因敲除后泳动能力减弱,结果见图5。
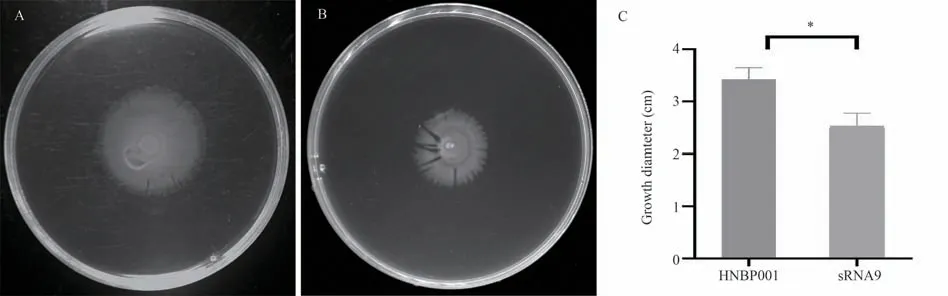
图5 类鼻疽伯克霍尔德菌野生株HNBP001 与敲除株△sRNA9 泳动性Fig 5 Mobility of B.pseudomallei HNBP001 WT and △sRNA9
2.4 生物被膜形成实验
生物被膜的形成对细菌起着重要的自我防护作用,保护细菌抵御极端的外界环境。将类鼻疽伯克霍尔德菌野生株HNBP001 与敲除株△sRNA9 菌液加入试管中培养48 h,观察其生物被膜厚度,经过PBS 洗涤、结晶紫染色、乙酸溶解等步骤,吸取200 μL 液体加入96 孔板进行OD595 吸光度检测,得到生物被膜结果。肉眼观察,敲除株△sRNA9 相较于野生株HNBP001 生物被膜更为稀疏。染色后检测OD595吸光度,敲除株△sRNA9 显著低于野生株HNBP001,结果见图6。实验结果表明,敲除sRNA9 影响其生物被膜的形成。

图6 类鼻疽伯克霍尔德菌野生株HNBP001 与敲除株△sRNA9 生物被膜形成实验Fig 6 Biofilm of B.pseudomallei HNBP001 WT and △sRNA9
2.5 药敏实验
通过抗菌药物MIC 测定实验分析野生株HNBP001 与敲除株△sRNA9 对20 种抗生素的敏感程度,△sRNA9 对所有抗生素MIC 无变化,结果见表3。实验结果表明敲除sRNA9 对B.pseudomallei的耐药性无明显改变。
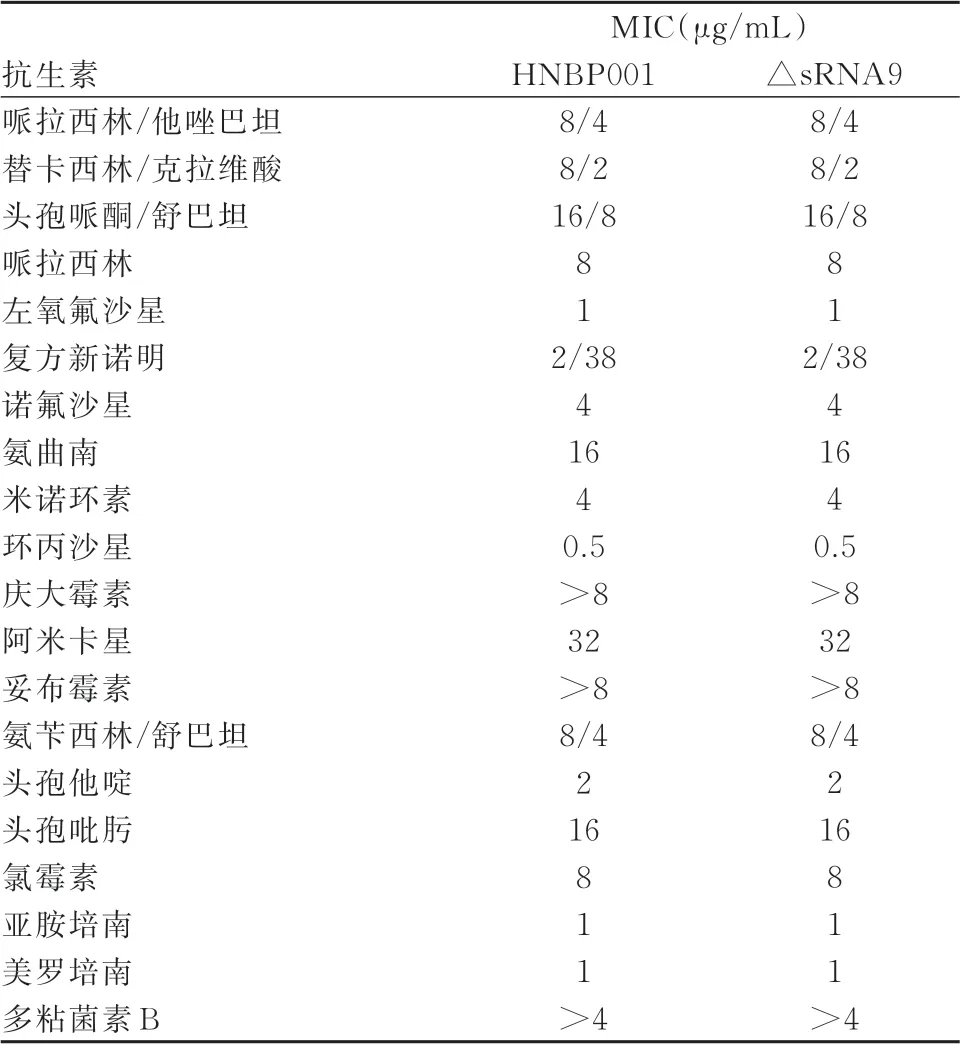
表3 野生株HNBP001 与敲除株△sRNA9 的MIC 值Tab 3 MIC of B.pseudomallei HNBP001 WT and △sRNA9
3 讨论
细菌的基因表达由多种分子进行调控,sRNA通过修饰核糖体和mRNA 的稳定性起到抑制或促进作用,从而介导调控功能的多样性[10]。大量研究证明sRNA 与细菌毒力、宿主-病原体信息传递、抗菌能力和耐药性等密切相关[11]。包括调节细胞膜的蛋白质组成[12],生物膜形成[12],含氮化合物的代谢[13],糖代谢[14],染色体外DNA 元件的调节[15],转录和RNA 降解的控制[16],以及在感染期间细菌毒力的调节和宿主生物的相互作用[17],sRNA 过与靶基因的mRNA 结合,被证实参与转录后的细菌毒力水平和环境压力适应的调节
sRNA 已经在多种细菌中鉴定出来,并发现在许多过程中起着关键的调节作用。法国学者发现一种名为SprD 的sRNA 在金黄色葡萄球菌中发挥着毒力调节作用,并找到其分子靶标Sbi 蛋白,表明sRNA 对细菌的致病性起重要作用[18]。在大肠杆菌中,SgrS 通过下调PtsG 转运蛋白参与细菌磷酸葡萄糖代谢的应激过程,抵抗细菌内葡萄糖磷酸化的堆积[19]。目前关于类鼻疽伯克霍尔德菌中sRNA 的机制研究较少,泰国学者预测了1 306 个B.pseudomallei的sRNA 基因,通过实验筛选出8 个新型sRNA,未进一步验证其功能[20]。马来西亚学者通过模拟不同营养环境,预测了12 种差异表达的顺式编码sRNA 的表达谱,该12 种sRNA 可能与B.pseudomallei适应营养缺乏环境有关[21]。
既往研究证实了sRNA 在其他细菌中的调控作用,如SmsR4 促进含山梨糖醇培养基中的变形链球菌生长[22],ArcZ、CsrB 和OxyS 等sRNA 通过调控鞭毛调节因子的表达影响生物膜的形成[23],DicF 通过调节肠细胞脱落位点基因和志贺毒素表达调控大肠杆菌的毒力[24]。本研究以课题组前期表达纯化的sRNA 伴侣蛋白Hfq 蛋白为基础,通过免疫共沉淀的方法筛选出与Hfq 互作的9 种sRNA,运用TPR-pK18mobSacB 自杀性质粒载体系统构建sRNA9 敲除株。敲除目的sRNA9 基因片段后,细菌平台期的生长速度低于野生株,但泳动能力和生物被膜形成能力均下降,耐药性与野生株相比较无差异,以上实验结果表明sRNA9可能在类鼻疽伯克霍尔德菌HNBP001的生长和毒力中扮演了重要角色。
综上所述,通过构建类鼻疽伯克霍尔德菌sRNA 敲除株,对其生物学功能进行初步评价,为后续研究sRNA 在B.pseudomallei致病过程中的分子机制提供了重要基础。

