青海湖裸鲤实时荧光定量PCR内参基因筛选与验证
刘思嘉,祁得林,祁洪芳,韩步鹰,牛依萌,赵 凯,田 菲
( 1.中国科学院 西北高原生物研究所,青海省动物生态基因组学重点实验室,青海 西宁 810008; 2.青海大学,省部共建三江源生态与高原农牧业国家重点实验室,青海 西宁 810016; 3.青海湖裸鲤救护中心,青海湖裸鲤繁育与保护重点实验室,青海 西宁 810016 )
青海湖裸鲤(Gymnocyprisprzewalskiiprzewalskii)属鲤科裂腹鱼亚科裸鲤属,仅分布于青海湖流域,是我国特有的珍稀高原鱼类。青海湖裸鲤的物种形成与青藏高原的抬升密切相关[1],在长期的适应进化中,能够很好地适应该地区严寒、低氧、高盐碱、强紫外线等极端自然条件,无疑是一种研究生物耐逆的理想模型[2]。已有研究表明,青海湖裸鲤在高盐环境下通过改变鳃和肾脏的组织形态进行渗透和离子调节,维持机体渗透压稳态[3];通过提高肝胰脏、肾脏组织中超氧化物歧化酶基因、酸性磷酸酶基因和碱性磷酸酶基因表达水平,来适应高碱度环境[4]。由于青海湖裸鲤极端环境适应的机制十分复杂,其背后的分子机制需要进一步研究。解析青海湖裸鲤极端环境适应过程中关键基因的表达变化,是探究青海湖裸鲤抗逆分子机制的前提。
实时荧光定量PCR(qRT-PCR),是将聚合酶链式反应与荧光信号检测结合,对DNA或cDNA模板进行定量检测,从而对靶基因在某组织、时间转录丰度或基因拷贝数定量的方法[5]。在鱼类研究中,qRT-PCR常用于鱼类基因功能研究[6],如抗逆分子机理研究[7]、鱼类病理学[8]、病原学检测[9]、免疫应答[10]、环境毒理学[11]及代谢通路调控[12]等方面。除了斑马鱼(Daniorerio)[13]、青鳉(Oryziaslatipes)[14]等模式生物外,qRT-PCR技术也广泛应用于多种经济鱼类和野生鱼类的研究中,如鲤(Cyprinuscarpio)、异育银鲫(Carassiusauratusgibelio)[15]、非洲丽鱼(Astatotilapiaburtoni)[16]等。
在qRT-PCR中,最常用的方法是利用一个已知的、表达稳定的基因来校正目标基因的Ct值,用于校正数据的基因称作内参基因或管家基因(HKGs)[17]。内参基因是矫正基因表达的必要参照,筛选合适的内参基因能够减少试验误差,提高结果可靠性。常用的内参基因主要包括肌动蛋白β(Act-β)基因[18-19]、3-磷酸甘油醛脱氢酶(Gapdh)基因[19]、真核生物延伸因子(Ef-1α)基因[20-21]、核糖体蛋白L13(Rpl13)基因[22]、琥珀酸脱氢酶亚基A(Sdha)基因[23]、次黄嘌呤鸟嘌呤磷酸核糖转移酶1(HPRT1)基因[24]等,这些常用的内参基因,或参与细胞的基本代谢,或作为细胞的组成成分,在不同器官、组织中的表达较稳定。然而,没有一个基因能在所有条件下的表达量恒定不变,因此,对于不同的物种、组织、细胞及试验处理选择合适的内参基因是十分必要的[18-26]。笔者基于青海湖裸鲤多组织RNA-seq数据,筛选表达稳定的候选内参基因,并利用GeNorm、NormFinder和BestKepper 3个程序评估上述基因在0 ℃和17 ℃时,脑、肝胰脏和肌肉组织中qRT-PCR检测结果的稳定性,筛选出适于青海湖裸鲤qRT-PCR分析的内参基因,并验证其校正目标基因表达量的可靠性,以期为高原鱼类极端环境适应的研究提供内参基因。
1 材料与方法
1.1 材料
试验鱼来自青海省裸鲤救护中心。选择体质量(8.5±1.2) g、体长(10.3±2.6) cm的青海湖裸鲤120日龄的幼鱼为试验对象,每组18尾,随机分成低温组和对照组,各组处理设3个平行。对照组温度为17 ℃恒温,低温组在17 ℃恒温饲养7 d后,以0.5 ℃/h速度降温至0 ℃,并恒温在0 ℃饲养15 d,其间除了温度差异,饲养密度、氧气供给、饲喂、水体循环过滤、光照节律等均与对照组一致。处理15 d后,麻醉处理(间氨基苯甲酸乙酯甲磺酸盐),取脑、肝胰脏、肌肉等组织。将所取组织置于冻存管中,液氮中速冻后-80 ℃保存备用。
1.2 方法
1.2.1 候选内参基因挖掘
利用本课题组未发表的青海湖裸鲤多组织低温适应的RNA-seq定量数据,筛选出最符合内参基因标准的基因。理想的内参基因,应该具备以下2个基本条件:(1)在目标组织、细胞中稳定表达;(2)具有高于背景的表达水平[27]。因此,筛选条件为:目标基因在各温度处理、各组织间的FPKM值(每1 000 000单位比对片段中,比对到目的基因1000碱基单位的片段数)>1,组内变异系数<0.2。符合标准的基因利用R语言中NormFinder软件包(https://www.moma.dk/files/r.NormOldStab5.txt)进行进一步筛选,参数设置如下:Groups=TRUE,ctVal=FALSE,pStabLim=0.25。
1.2.2 总RNA提取
剪取约50 mg冻存组织样,放入无RNase的2 mL离心管中,加入钢珠和800 μL组织裂解液(Trizol),在高通道组织匀浆机中以40 Hz、180 s匀浆,4 ℃、12 000 r/min (离心半径 6.5 cm)离心2 min,保留上清液进行总RNA提取试验。总RNA提取按照康为世纪Ultrapure RNA Kit (DNase I)(货号CW0597S)说明书进行操作。
1.2.3 总RNA 的检测
利用超微量核酸蛋白测定仪Nano drop和1%琼脂糖凝胶电泳检测RNA 样品质量和完整性。测定总RNA的浓度和纯度;电泳检测若28S、18S条带清晰,则表明总RNA完整性较好,无DNA及蛋白污染,可作为反转录模板。
1.2.4 反转录cDNA
反转录使用北京全式金生物技术有限公司的EasyScript One-Step gDNA Removal and cDNA Synthesis SuperMix 反转录试剂盒。20 μL反转录体系,各组分如下:随机引物(10 μmol/L) 1 μL,总RNA 5 μL,2×ES Reaction Mix 10 μL,EasyScript®RT/RI Enzyme 1 μL,gDNA酶1 μL,最后用无RNA酶ddH2O将体系补充至20 μL。42 ℃水浴孵育1 h,合成第一链cDNA,95 ℃灭活逆转录酶3 min,然后12 000 r/min (离心半径 6.5 cm)离心1 min,用5倍体积去离子水稀释后,进行后续qRT-PCR反应。
1.2.5 内参基因的选择和引物设计
对Abhd18、Actb、B2m、Bad、Dazap2、Eloa、Fabp3和Gapdh共8个候选基因进行qRT-PCR验证。根据青海湖裸鲤基因库(未发表),利用Primer Premier 6.0软件对上述候选基因进行引物设计,标准如下:引物Tm(55±5) ℃,碱基长度18~22 bp,GC 含量为40%~60%。扩增产物长度100~200 bp,引物信息见表1。另外,为探讨低温下青海湖裸鲤的脂类代谢变化,对Acsl1、Cpt1a、Elovl1和Slc27a4等关键基因的表达丰度进行检测。

表1 基因引物序列Tab.1 Primers used in this study
1.2.6 qRT-PCR
本试验采用SYBR Green I法qRT-PCR(相对定量)进行mRNA表达量分析;定量PCR试剂盒为:北京全式金TransStart Green qPCR SuperMix;检测机器为LightCycler96 RealTime PCR仪(Roche,瑞士)。20 μL反应体系优化如下:2×SYBR Premix EX-Taq Mix 10 μL,QF (10 μmol/L) 0.5 μL,QR (10 μmol/L) 0.5 μL,模板cDNA 2 μL,无RNA酶dH2O 7 μL。每个基因均设3个平行孔。优化后的反应条件如下:95 ℃预变性1 min;95 ℃变性5 s ,61 ℃退火35 s,40个循环。延伸阶段收集荧光信号,反应结束后,97 ℃ 1 min获得熔解曲线。
1.2.7 数据分析
基因相对表达量分析数据处理采用2-△△Ct法处理[28],处理软件利用R语言ddCt包完成。分别采用R语言中NormqPCR软件包[29]、Normfinder软件包及endoGenes.R (https://github.com/hanielcedraz/endoGenes) 脚本文件完成GeNorm、Normfinder及BestKeeper等3种方法对候选内参基因的表达稳定性进行分析。采用独立性t检验对两组间的相对表达量做显著性检验,P<0.05为差异显著,P<0.01为差异极显著。统计作图由R语言ggplot2软件包(https://cran.rproject.org/web/packages/ggplot2/index.html)完成。
2 结 果
2.1 内参基因筛选
根据多组织(脑、鳃、心、肾脏、肠道、肝胰脏、肌肉、皮肤等8个组织)RNA-seq的基因定量结果,在青海湖裸鲤所有表达的编码基因中进行候选内参基因筛选。参考文献[27]的方法,选择各组织中平均log(FPKM)>1,组内FPKM值变异系数<0.2的基因。结果共获得7个候选内参基因,分别是脱水酶(Abhd18)基因、肌动蛋白(Actb)基因、微球蛋白(B2m)基因、延伸蛋白A(Eloa)基因、DAZ相关蛋白(Dazap2)基因、BCL细胞死亡激动因子(Bad)基因和脂肪酸结合蛋白3(Fabp3)基因,各基因在各组织中的RNA-seq表达定量结果见图1。在候选内参基因中,B2m和Actb基因也是其他鱼类常用的内参基因,而Fabp3基因在心脏中高表达,在其他组织中表达量较稳定,因此选作候选内参基因拟验证其在脑、肝胰脏和肌肉组织中的表达稳定性;甘油醛-3-磷酸脱氢酶(Gapdh)基因是鱼类最常用内参基因,但因其在组内的变异系数为0.23,大于0.2,未达到本试验中筛选内参基因的标准。但是,考虑到Gapdh基因作为内参基因应用的广泛性,同时,为了更好地比较分析内参基因对定量结果的影响,笔者也把Gapdh基因作为候选内参基因。因此最终确定8个候选内参基因,进行下游分析。利用NormFinder方法对候选基因按表达稳定值排序,表达稳定值排序见表2,数值越小则表达稳定性越高。
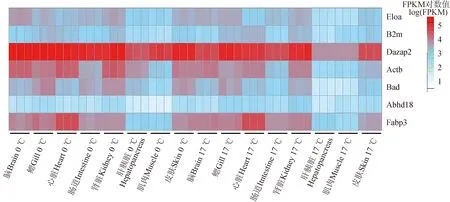
图1 候选基因表达量热图Fig.1 Heatmap of expression level of candidate genes

表2 利用NormFinder方法基于RNA-seq定量结果分析候选基因表达稳定性Tab.2 Stability analysis of candidate genes based on RNA-seq by NormFinder
2.2 RNA质量检测
利用Nano drop测定总RNA的质量浓度均在150 ng/μL以上,纯度D(260 nm)/D(280 nm)及D(260 nm)/D(230 nm)均在2.1~2.0;随机选取5个RNA样品进行核酸琼脂糖凝胶电泳检测,结果显示,总RNA完整性较好,28S、18S条带清晰,无明显的DNA及蛋白污染,可作为反转录模板(图2)。
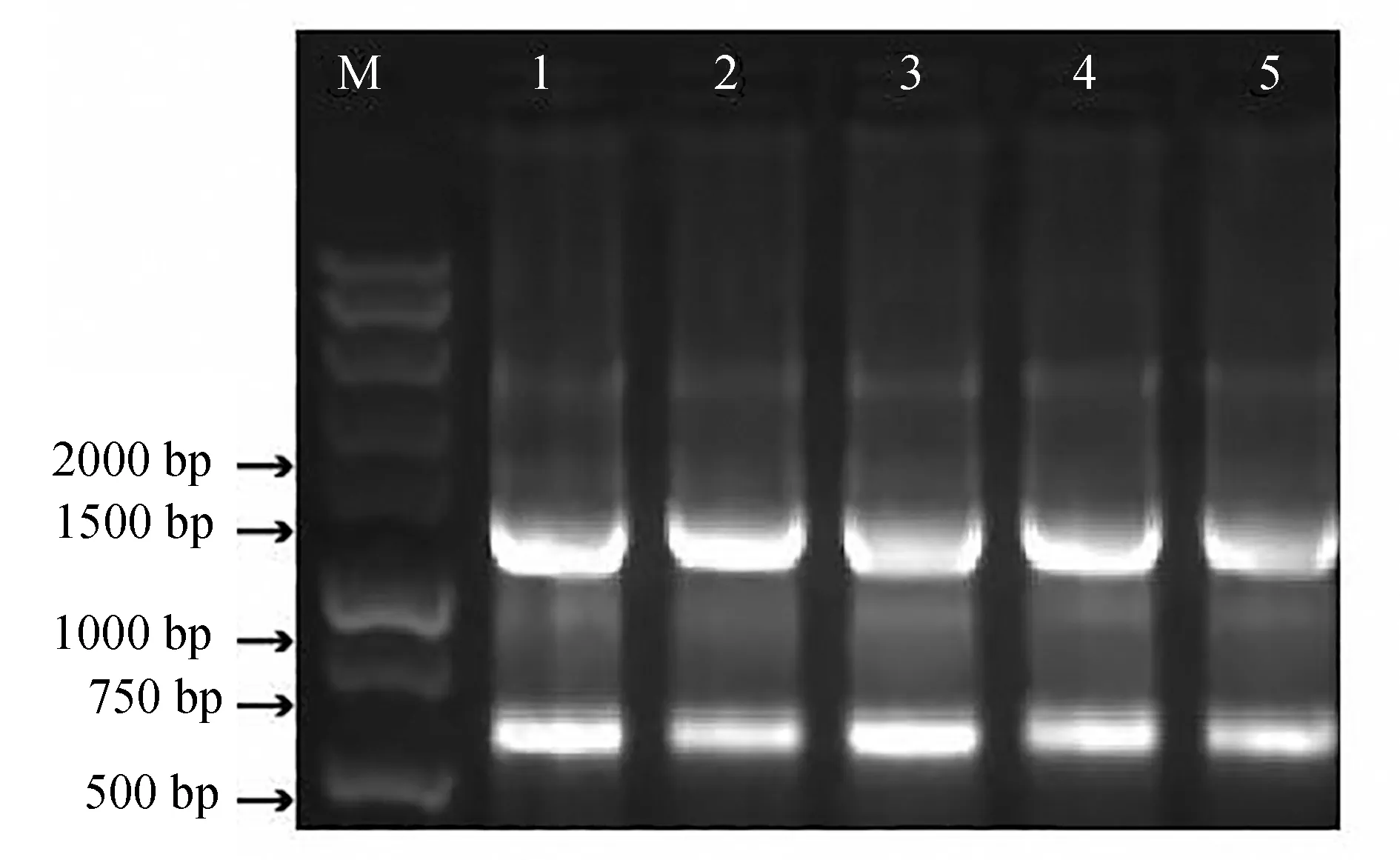
图2 总RNA电泳Fig.2 The agarose gel electrophoresis of total RNAM.标准分子量; 1.0 ℃脑; 2.17 ℃脑; 3.0 ℃肝胰脏; 4.17 ℃肝胰脏; 5.17 ℃肌肉.M.marker; 1.brain at 0 ℃; 2.brain at 17 ℃; 3.hepatopancreas at 0 ℃; 4.hepatopancreas at 17 ℃; 5.muscle at 17 ℃.
2.3 内参基因引物特异性验证
对设计引物的特异性进行验证,结果见图3。由图3可见,各基因的扩增片段熔解曲线呈单一峰,表明引物特异性良好,可用于定量PCR分析。
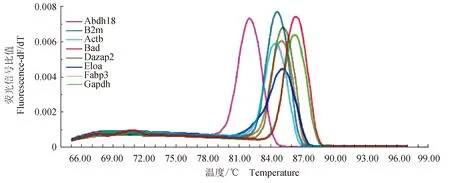
图3 候选内参基因熔解曲线Fig.3 Melting curves of candidate genes
基因的表达丰度用Ct值表示,其数值越高,则表达量越低。理想的内参基因Ct值应大于10,且趋近于20。对青海湖裸鲤不同温度处理下,各候选内参基因的表达丰度统计发现(图4),8个基因在脑、肝胰脏及肌肉组织中的表达水平存在一定变化,其中B2m和Bad基因的表达丰度相对较高,而Abhd18和Gapdh基因表达丰度较低;Fabp3和Gapdh基因在不同温度下组织间的表达量差异较大,B2m和Eloa基因在不同温度下组织间的表达量差异较小。

图4 候选基因Ct值Fig.4 Ct values of candidate genes
2.4 内参基因稳定性分析
为筛选在不同温度和组织间均能稳定表达的内参基因,笔者利用GeNorm、Normfinder和BestKeeper方法对青海湖裸鲤在0、17 ℃条件下脑、肝胰脏及肌肉组织内8个候选内参基因的稳定性进行分析。
2.4.1 GeNorm分析结果
GeNorm是由Vandesompele等编写的专门用于确定qRT-PCR分析中,筛选最适内参基因的程序,该方法已被引入R语言NormqPCR包[29]。GeNorm通过计算每个内参基因稳定性的M值来选择稳定的内参基因。标准是M值越小,内参基因的稳定性越好,反之,内参基因的稳定性越差。分析结果显示,8个候选内参基因在青海湖裸鲤不同温度、不同组织间表达稳定性排序,由高到底依次是:B2m、Eloa、Bad、Dazap2、Actb、Abhd18、Gapdh、Fabp3(图 5)。

图5 GeNorm方法分析候选基因稳定值排序Fig.5 Stability value sort of candidate genes by GeNorm
GeNorm通过计算引入新的内参基因后标准化因子的配对变异V值,并根据(Vn/Vn+1)值确定最优内参基因的数目,以减少使用单一内参基因所引起的偏差。当Vn/Vn+1<0.15时,最优内参基因数为n;如果Vn/Vn+1>0.15,则最优内参基因数为n+1。由图6可知,V2/V3对应变异系数最高且大于0.15,因此确定最合适的内参组合基因数目是3个(图6)。

图6 GeNorm方法检测最适内参基因数目Fig.6 Optimal reference gene analysis by GeNorm
2.4.2 NormFinder 分析结果
NormFinder是Claus等在2004年编写的用于筛选内参基因的一种程序,该程序计算原理与GeNorm程序相似,也是根据表达稳定值筛选最适内参基因,判定标准为表达稳定值最小的基因为最适内参基因[17]。分析结果显示,B2m基因表达最稳定,而Fabp3基因最不稳定(图7)。与GeNorm方法得出的结果相似的是,B2m都是表达最稳定的最适内参基因,而Fabp3基因表达最不稳定。

图7 NormFinder方法对候选基因表达稳定值排序Fig.7 Stability value sort of candidate genes by NormFinder
2.4.3 BestKeeper分析结果
BestKeeper是Michael等编写的针对内参基因和目标基因表达量分析的程序,该程序通过计算基因间相关系数、标准差和变异系数,再比较各值的大小,最终确定最适内参基因[30]。判定原则为相关系数越大、标准偏差和变异系数越小,内参基因稳定性越好,反之,稳定性越差;当标准差>1时,则该内参基因表达不稳定。分析结果(表3)显示,只有B2m、Eloa基因的标准差小于1,且变异系数最小,分别为2.109、2.555,相关系数均大于0.85,综合得出B2m和Eloa基因表达最稳定,最适宜作青海湖裸鲤低温胁迫的内参基因,而Fabp3基因在8个候选基因中表达稳定性最差,不宜选为内参基因。

表3 NormFinder方法分析候选基因表达稳定性Tab.3 Stability analysis of candidate gene by NormFinder
2.5 相对表达量分析结果
综合GeNorm、NormFinder、BestKeeper 3种方法分析的结果,可确定B2m是青海湖裸鲤低温适应过程中多组织转录定量的最适内参基因。为了进一步比较分析内参基因在相对定量分析中的可靠性,利用R语言ddCt包,分别以8个候选内参基因中的1个作内参基因,对其余7个基因在不同温度和不同组织间的表达情况进行相对定量。根据相对定量结果,比较各基因在青海湖裸鲤0 ℃和17 ℃处理后各组织中的表达差异倍数,并与转录组数据进行相关性分析。分析结果显示,以Eloa、B2m做内参基因时,各基因qRT-PCR相对定量的结果的相关性最高,r2分别达到0.95及0.89,表明以Eloa、B2m为内参基因的相对定量结果更可靠,可信度更高(图8)。同时,Eloa、B2m也是GeNorm及BestKeeper方法共同分析获得的最适内参基因。
2.6 低温适应相关基因qRT-PCR分析
根据多种方法最终筛选获得在不同组织中表达最稳定的基因为Eloa和B2m,以上述2个基因作为内参基因,对青海湖裸鲤幼鱼低温适应脂肪酸代谢途径中的关键基因长链脂酰辅酶A合成酶(Acsl1)、肉碱棕榈酰转移酶1a(Cpt1a)、超长链脂肪酸延伸酶(Elovl1)和溶质载体蛋白27a4(Slc27a4)在不同温度下脑、肝胰脏和肌肉组织中的表达量进行qRT-PCR分析。结果表明,以Eloa和B2m作为双内参基因进行2-△△Ct法相对定量分析,4个目标基因在低温条件下肝胰脏和肌肉组织中的表达量均显著上调(P<0.01),在肝胰脏中的表达量最高;在脑组织中Acsl1a基因在低温条件下显著上调(P<0.01),Cpt1a、Elovl1和Slc27a4基因表达量无显著差异(P>0.05)(图9a)。4个目标基因在低温(0 ℃)与适温(17 ℃)的相对表达比值与RNAseq的差异表达倍数的相关性较高,r2均大于0.87(图9b)。
3 讨 论
3.1 候选内参基因的稳定性分析
qRT-PCR技术因其灵敏、高效、精确、廉价的特点,是研究基因表达丰度最常用的方法,已广泛应用于动植物基因功能的研究中。但是,在试验过程中的误差会影响定量结果的准确性,因此,需要一个或多个已知的表达稳定的基因来校正试验误差。水生生物常用的内参基因包括:Act-β[18]、Gapdh[19]、Ef-1α[20-21]、Rpl13[22]、Sdha[23]、HPRT1[24]和18S rRNA[21]等。在功能上,内参基因多参与机体的基础代谢或是细胞结构的基础组成成分,例如Act-β基因编码的肌动蛋白是构成细胞骨架的主要蛋白之一[18],Rpl13基因编码的核糖体蛋白以及18S rRNA基因编码核糖体小亚基RNA[22,25]是构成细胞内核糖体的主要成分之一,Gapdh基因编码的甘油酸-3-磷酸脱氢酶是参与糖酵解活动的关键酶[19],Ef-1α基因编码的延伸因子参与调控蛋白质的翻译、折叠[20],Sdha基因编码的琥珀酸脱氢酶亚基A是线粒体内膜的结合酶,能够调控线粒体的氧化磷酸化和呼吸链的电子传递过程[23],HPRT1基因编码次黄嘌呤鸟嘌呤磷酸核糖转移酶1能够调控细胞内嘌呤核苷酸的合成[24]。然而,基因的表达调控是一个复杂的过程,没有一个基因的表达水平能够在不同物种、器官、组织、细胞,或在不同时间、发育阶段、生理状态、病理过程,或在不同环境和试验处理下保持恒定。Casadei等[18]在对斑马鱼不同发育时期和不同组织的多个候选内参基因的表达稳定性进行分析时发现,bactin2和Ef-1-α在不同发育时期和不同组织中的表达稳定性最高;孙海成等[22]利用实时荧光定量PCR技术比较了尖裸鲤(Oxygymnocyprisstewartii)7个候选内参基因在多个健康组织中的表达量,结果发现,Rpl13在不同组织中的表达较为稳定;费越越等[15]对不同处理条件下异育银鲫组织和尾鳍细胞的4个候选内参基因的表达稳定性进行分析,发现Act-β和Ef-1α基因在健康异育银鲫的多组织中表达较为稳定,而在鲤疱疹病毒Ⅱ型感染条件下,Act-β基因在肾脏和尾鳍细胞中的表达最为稳定,Ef-1α基因在脾脏中的表达最为稳定。
为挖掘适用于青海湖裸鲤低温适应过程中表达稳定的内参基因,笔者以青海湖裸鲤不同温度处理的多组织RNA-seq数据为依据,筛选得到8个表达稳定的候选内参基因Abhd18、Actb、B2m、Bad、Eloa、Dazap2、Fabp3、Gapdh。其中Actb、B2m和Gapdh基因是水生生物常用的内参基因。通过qRT-PCR对其在不同温度条件下,青海湖裸鲤脑、肝胰脏和肌肉组织的表达丰度进行检测。利用Genorm、Normfinder和BestKeeper 3种方法对上述8个候选基因在青海湖裸鲤中的表达稳定性进行分析。根据各软件评价标准[17,24-29],综合3个软件分析的结果,最终筛选出最符合要求的内参基因。3个软件的分析结果表明,在青海湖裸鲤幼鱼不同温度适应下脑、肝胰脏、肌肉组织中表达最稳定的基因是B2m和Eloa基因,3种方法分析结果均显示,Fabp3基因的稳定性较差,说明该基因不适合作为青海湖裸鲤的内参基因。微球蛋白-β2(B2m)是由淋巴细胞、血小板、多形核白细胞产生的一种小分子球蛋白,生理状态下B2m在多种细胞内的含量很稳定,其编码基因B2m是qRT-PCR分子中常用的内参之一,在鳜(Sinipercachuatsi)[31]、花鲈(Lateolabraxmaculatus)[32]等鱼类的分子生物学研究中被用作qRT-PCR分析的内参基因。转录延伸蛋白A(Eloa),是一种转录因子,具有多种生物学活性,广泛存在于多种细胞核基因内[33],目前尚无关于Eloa基因作为内参基因的报道。笔者发现,Eloa基因在青海湖裸鲤不同组织和不同温度处理下的表达量均较稳定,是潜在的新的候选内参基因。另外,Gapdh基因也是动植物常用的内参基因[18],然而在本试验中,通过对RNA-seq数据的筛选及qRT-PCR试验验证,Gapdh基因在青海湖裸鲤幼鱼不同的组织和温度胁迫下的表达差异较大,对低温较为敏感。
3.2 低温对内参基因表达的影响
鱼类是变温动物,由于缺乏体温调控能力,其生存对环境温度的依赖极高。不同于其他环境因素,温度对鱼类生理活动的影响是广泛的[34]。由已有的研究报道可知,温度能显著影响鱼类多组织、多基因的转录活动[7,35-37]。笔者通过分析青海湖裸鲤对低温响应的RNA-seq数据发现:在其他鱼类研究中常用的内参基因,如Ef-1[20-21]、Rpl13[22]、Sdha[23]、HPRT1[24]基因等,在青海湖裸鲤幼鱼0 ℃低温条件下的多组织中的表达量降低,不同温度间(17 ℃和0 ℃)或不同组织间的变异系数>0.2,不能满足内参基因的筛选标准;而Bad、Eloa、Dazap2等基因的表达丰度受温度影响较小,是潜在的候选内参基因。Long等[38]在鲤对低温胁迫下脑、心肌等组织的分子响应中发现,Ef-1基因在不同温度及不同组织间的表达量较为稳定,可作为内参基因;而徐丽华等[7]在研究鲤脑组织低温差异表达候选基因时,选择了18S rRNA基因作内参基因。上述关于鱼类低温适应的分子研究筛选的内参基因与本试验有所差异,可能的原因是,虽然青海湖裸鲤和鲤都属鲤科鱼类,但二者对低温环境的耐受能力不同,对低温胁迫的分子响应机制有差异,同源基因的表达模式具有物种特异性。
除了内参基因的表达活动受到温度的影响外,耐低温鱼类参与脂类代谢过程的基因也普遍受到低温胁迫的调控。例如:在低温条件下,鲤脑组织中的脂肪酸链延伸蛋白、酰基辅酶A脱氢酶、肌醇-1-磷酸合成酶等基因的表达水平显著上调[7];鲤心脏中Acsl1b、Acsl4a、Elovl5以及PPAR信号通路基因表达显著上调[38]。本试验中,对参与脂肪酸代谢过程中的Acsl1、Cpt1a、Elovl1和Slc27a4等4个关键基因表达量进行分析,上述基因在0 ℃时肝胰脏和肌肉组织表达量均显著上调,其结果与徐丽华等[7,38]对鲤的研究一致。Acsl1、Cpt1a、Elovl1和Slc27a4基因分别是参与长链脂肪酸活化、脂肪酸转运、脂肪酸链延伸和脂肪酸β氧化过程的关键基因,其在低温环境下的高表达模式表明青海湖裸鲤幼鱼在低温环境下脂肪酸的代谢活动增强。脂肪酸是鱼类主要能源物质和膜结构组分,耐寒鱼类在低温环境下通过提高脂肪酸氧化获能应对低温下的能量消耗[36];并且通过改变细胞膜组分、增加磷脂不饱和脂肪酸含量,以改善膜流动性及僵化,避免细胞组织受到低温损害[7]。青海湖裸鲤的物种形成与青藏高原海拔的抬升、青海湖的形成密切相关[2],随着栖息地海拔的升高、温度逐渐降低,青海湖裸鲤也演化出复杂的耐低温机制以应对高寒的气候条件。已有研究表明,青海湖裸鲤多种能量代谢相关基因发生了大规模扩张事件[39],这也暗示着青海湖裸鲤可能进化出特殊的能量代谢过程以应对低温环境对能量的需求。
另外,本试验中使用的3种方法均已编译成R语言中的软件包或脚本文件,而相似的研究大多采用的是Excel中相应的3种分析插件。相比于后者,R语言的数据处理能力更强,更适用于处理RNA-seq等体量较大的数据;且R语言软件包均为开源程序,调用更灵活、高效,适于批量自动化的分析工作,具有更广泛的应用前景。
4 结 论
笔者基于GeNorm、NormFinder、BestKeeper 3种方法评估了Abhd18、Actb、B2m、Bad、Eloa、Dazap2、Fabp3和Gapdh共8个候选内参基因在青海湖裸鲤低温胁迫下多个组织中的表达稳定性,筛选并验证得到2个最适内参基因B2m和Eloa。以B2m和Eloa作为双内参基因对青海湖裸鲤脂类代谢调控基因Acsl1、Cpt1a、Elovl1和Slc27a4在不同温度下脑、肝胰脏和肌肉组织中的表达量进行qRT-PCR分析,结果显示,4个目标基因低温与适温的相对表达比值与RNAseq的差异表达倍数高度正相关,说明qRT-PCR定量结果可靠。试验结果可为高原鱼类耐逆分子生物学的研究及内参基因的选择提供科学依据。

