逆转细菌耐药性研究进展
廖燕科 蓝素桂 苏爱秋 彭燕鸿 谭强

摘要:细菌耐药性是治疗细菌感染类疾病失败的主要原因,寻找高效、低毒的逆转细菌耐药性策略成为了治疗细菌感染的研究热点。本文就逆转细菌耐药性机制与策略等进行综述,为解决细菌耐药性问题、开发新型抗生素提供研究方法与思路。
关键词:细菌耐药性;逆转耐药;细菌感染;机制与策略
中图分类号:R978.1文献标志码:A
Abstract The bacterial resistance is the main cause of failure in the treatment of bacterial infection. Effective and low-toxicity strategies to reverse the bacterial resistance have become a popular research field. In this paper, the mechanisms and strategies of reversing the bacterial resistance were reviewed to provide the research methods and ideas for solving the bacterial resistance problem.
Key words Bacterial resistance; Reverse drug resistance; Bacterial infection; Mechanisms and strategies
细菌耐药性是指在常规治疗剂量下细菌对药物的敏感性下降甚至消失,导致药物对病原菌的疗效降低或者无效 [1]。细菌耐药性分为固有耐药性和获得性耐药,固有耐药性即天然耐药性或通过突变产生的耐药性,是细菌染色体遗传基因介导的耐药性;而获得性耐药是指细菌与抗菌药物接触后,自身代谢途径发生改变,使其不易被抗菌药物杀灭或抑制,是细菌产生耐药性的主要原因[2],可通过接合、转化或转导等方式使细菌产生耐药性 [3]。
1928年青霉素首次发现以来[1],各种抗生素相继被开发出来。大量抗生素被用于治疗细菌感染,导致细菌对抗生素产生耐药性[4]。1959年首次发现耐甲氧西林金黄色葡萄球菌(MRSA)以及2010年发现的新德里“超级细菌”,都能抵抗当前人类大部分抗生素[5]。
MRSA和新德里“超级细菌”从发现至今已几乎传播遍及全球,成为院内和社区感染的危险病原菌,严重威胁人类的健康。世界卫生组织(WHO)全球抗生素耐药报告显示,细菌耐药性每年导致70万人死亡;预计到2050年,全球因细菌耐药性感染问题所消耗的医疗成本将达100万亿美元,每年死亡人数将达1000万[6],这将给人类健康和经济发展造成严重威胁。
细菌耐药性的发展速度越来越快,新型抗生素的研发速度已跟不上细菌耐药性的发展速度[7]。为解决不断出现的细菌耐药性问题,科学家们致力于寻找高效、低毒的细菌耐药性逆转策略,恢复菌株对抗生素的敏感性。在20世纪80年代国内学者研究发现某些中草药提取物具有恢复菌株对抗菌药物的敏感性,从此中草药作为细菌耐药逆转剂被广泛研究和临床使用[8]。在2012年日本科学家Hiramatsu等[9]在研究尼博霉素对Mu50(一株耐喹诺酮类的MRSA菌株)的作用机制,发现尼博霉素能恢复菌株对喹诺酮类抗生素的敏感性,提出了逆转细菌耐药性可能是解决细菌耐药性问题的关键措施。除此之外,某类吲哚信号分子也被证实具有逆转细菌固有耐药性的潜力,这一发现为消除细菌耐药性提供新的思路[10]。
1 耐药逆转机制
逆转细菌耐药性的机制主要是通过阻断或抑制细菌耐药性机制。不同微生物对不同抗生素产生的耐药机制不同,因此逆转细菌耐药性的机制也不尽相同。逆转细菌耐药性的机制主要包括消除耐药质粒、抑制主动外排泵、抑制生物被膜形成和抑制耐药基因表达等(图1)。
1.1 消除耐药质粒
细菌对抗生素产生耐药性,可以通过获得耐药性质粒实现的。质粒介导的抗生素耐药性基因可通过接合、转化或转导等方式在不同种属细菌间迅速传播,使得细菌耐药性迅速发展[12]。研究表明,可通过药物消除剂或基因编辑技术抑制耐药质粒复制,从而消除部分或全部耐药质粒,恢复细菌对抗生素的敏感性[11-13]。十二烷基磺硫酸钠(SDS)是常用的质粒消除剂,其消除机制为SDS溶解菌株内膜蛋白,破环细胞膜完整性,改变质粒在细胞膜上的结合位点,导致耐药质粒不能完成复制而达到消除目的;药物消除剂还可以通过水解质粒复制所需的酶来达到消除质粒的目的[12](图1a)。SDS具有消除细菌耐药质粒的作用,但由于其本身对动物机体有较强的毒副作用,故不适于体内应用。
1.2 抑制主动外排泵
外排泵耐药机制是当抗菌药物进入细菌细胞内,会激活细菌内膜的外排蛋白,将药物排出,降低胞内药物浓度[14]。外排泵家族的组成主要包括多药及毒性化合物外排家族(MATE)、易化子超家族(MFS)、小多重耐药家族(SMR)、耐药结节化细胞分化家族(RND)和ATP结合盒超家族(ABC)等。其中,ABC类外排泵是利用ATP水解释放的能量排出膜内药物,其余4类均通过质子进入膜内形成浓度差所产生的势能提供质子驱动力为能量,从而将药物外排至膜外[15]。抑制主动外排泵可通过干扰外排泵的表达和组裝、阻断能量来源以及阻碍底物通过外排通道等机制发挥作用[16](图1b)。若主动外排泵作用减弱,细菌对抗生素的敏感性也随之而增加[16]。
1.3 抑制生物被膜形成
生物被膜是细菌生长繁殖过程中分泌的胞外多糖、脂质和蛋白质等物质,使抗菌药物不能穿透胞外基质,导致细菌对抗菌药物的敏感性降低,是细菌耐药的重要机制[17]。抑制生物被膜形成,对逆转细菌耐药性有极大的增强作用[18];生物被膜的生命周期包含4个阶段:①初始黏附;②形成微菌落;③生长和成熟:刺激群体感应(QS)系统产生群体感应信号分子;④释放:呈浮游菌状态。抑制生物膜的形成机制包括阻断浮游菌的附着、阻断QS系统和降低胞外多聚糖(EPS)基质等(图1c)。浮游菌可通过绒毛或鞭毛使细菌移动到一个新的位置,再次形成生物被膜引起感染;因此,清除细菌绒毛或鞭毛可阻断浮游菌的附着,减少生物被膜的形成[18]。细菌生物被膜形成、细菌耐药性产生和毒力因子都受QS系统的调控,可通过QS抑制剂干扰或抑制该现象。QS抑制剂主要通过抑制QS自体诱导物信号分子的产生、降解QS自体诱导信号分子与QS自体诱导物信号分子竞争受体[19]。EPS基质在生物被膜的形成中发挥重要作用,降低EPS生物被膜基质可通过降解多种分子包括胞外多糖、胞外蛋白质、胞外DNA和脂类等[20]。
1.4 抑制耐药基因表达
耐药基因是编码细菌耐药性的一段核苷酸序列,是细菌产生耐药性的重要因素。耐药基因位于细菌染色体上或位于染色体外的质粒。通过抑制耐药基因表达,可以恢复耐药细菌对相应抗生素的敏感性。另外,通过抑制耐药基因的表达可以达到消除耐药质粒、抑制主动外排泵和生物被膜形成的作用,从而恢复细菌对抗菌药物的敏感性[21]。利用SDS从耐药E. coli中消除质粒介导的耐药基因mcr,消除耐药基因后的菌株对多黏菌素敏感,证明消除耐药基因可以恢复耐药菌株对相应抗生素的敏感性[12]。
除了以上4种逆转耐药机制外,还可通过改变细胞膜通透性、抑制β-内酰胺酶活性、破坏细胞质膜的形成等机制提高细菌对抗生素的敏感性,但对于其逆转机制还有待进一步研究。
2 耐药逆转策略研究
细菌耐药性现象已成为全球面临的一大公共卫生难题,研究人员在研究细菌耐药机制的同时,也聚焦于如何恢复耐药菌对抗菌药物的敏感性。本文总结了逆转细菌耐药的策略,主要包括联合用药、反义疗法、外排泵抑制剂(EPI)和基因敲除等。
2.1 联合用药
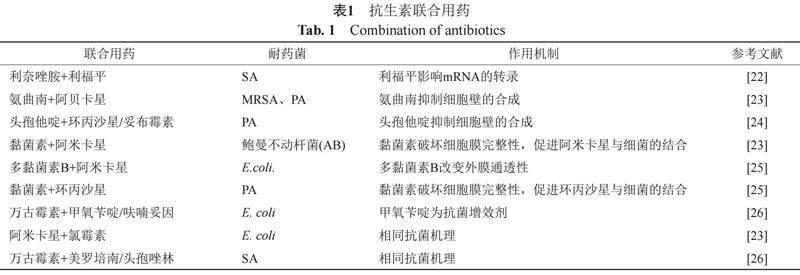
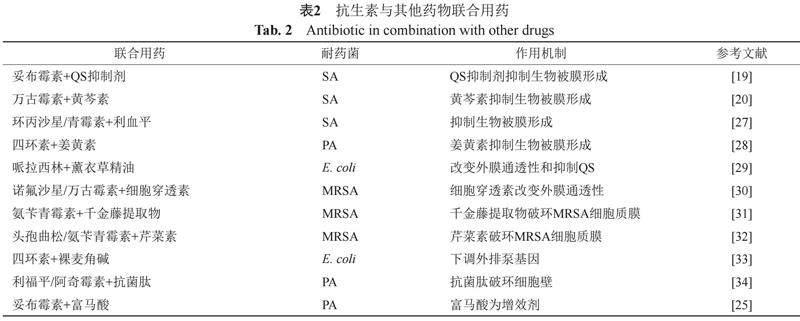
通过两种或两种以上的药物联合使用,发挥药物间的协同或相加作用,从而提高细菌对抗菌药物的敏感性。在临床治疗细菌感染时,联合用药往往优于单方治疗,能够有效地抑制细菌耐药性,增强抗菌药物的抑菌效果。联合用药见表1~2。
抗生素联合治疗是一种用于限制抗生素耐药性传播的有效策略。同时使用两种或两种以上抗生素,由于药物的作用机制,一般会出现3种联合效益:协同、相加和拮抗[27]。协同和相加作用的用药组合会产生以下的用药优势:扩大抗菌谱、增强抗菌作用、减少抗菌药物的用量和降低药物的不良反应等,所以联合用药应选择具有协同作用或相加作用的药物组合[23]。
由表2可知,某些抗菌中药提取物或单体单用时具有抑菌作用,如裸麦角碱、千金藤提取物、芹菜素和黄芩素等,可通过下调外排泵基因、破坏细胞壁合成或抑制生物被膜形成等机制,增强抗生素的抗菌作用。目前,抗菌肽被认为是抗生素类药物的理想替代品,单用或联合抗生素都能很好地杀灭病原体和多重耐药菌,而与利福平或阿奇霉素联用时,能破环细胞壁和抑制细菌生物被膜形成,恢复多重耐药菌对相应抗生素的敏感性[34]。值得注意的是,富马酸单用无抗菌作用,但与妥布霉素联用时,能增强妥布霉素对细胞的杀伤力,其增敏作用机制是通过三羧酸循环激活细胞呼吸并刺激质子动力,而妥布霉素需要依赖质子动力的转运才能穿透细胞并作用于细胞 [25]。
由表1~2可知,不管是抗生素之间的联合用药还是抗生素与其他药物的联合用药,其抗菌作用都比单一抗生素强,这也将成为未来治疗细菌耐药性的一个发展趋势。对于联合用药的未来发展方向,可更多地关注抗菌中药,挖掘更多的中药单体;还可以开发一些增效剂,增强细菌对抗生素的敏感性。联合用药虽能提高抗菌效果,但在使用过程中也应注意药物合理配伍和使用,减少药物间相互作用引起的不良反应或产生新的耐药性。
2.2 反义疗法
反义疗法有望成为逆转细菌耐药性的一种新方法,通过设计反义寡核苷酸的序列,破环脱氧核糖核酸到核糖核酸再到蛋白质的信息流,使细菌部分基因无法表达,恢复菌株对抗生素的敏感性[35]。反义抗菌剂是一类通过碱基互补配对原则识别靶基因mRNA,调控靶基因表达的化学合成类药物。反义抗菌剂的核苷酸序列特异性强、长度短,对人类基因表达的影响风险低。设计反义抗菌剂抑制耐药基因的表达有两种机制(图2所示)[36]。
1997年,White等[37]通过设计反义序列抑制耐药菌E. coli的marRAB操纵子,成功地提高了诺氟沙星对耐药菌的杀菌活性,恢复耐药菌对诺氟沙星的敏感性,首次证实通过设计反义序列能抑制耐药基因表达。使用反义寡核苷酸作为抗菌剂最大障碍即是在没有合适载体的情况下细菌对抗菌剂的摄取有限,特别是在具有药物外排机制的多药耐药菌中。DNA纳米结构(TDN)已被开发为反义寡核苷酸的递送载体,将靶向基因ftsZ的反义肽核酸通过TDN有效运送到MRSA细胞中,能有效抑制细菌基因ftsZ表达[38]。另外,抑制细菌生长的一种方法是阻断保守基因的表达,mecA基因在MRSA中高度保守,这使其成为反义抗菌剂的理想靶点。在mecA的mRNA编码区发现一段对反义抑制剂特别敏感的靶序列,将反义寡核苷酸封装在阴离子脂质体中,经反义序列处理后的MRSA临床耐药菌株表现出对β-内酰胺类抗生素敏感[39]。除了设计反义DNA序列外,还可通过设计反义RNA序列,抑制必要基因的表达。acpP(酰基载体蛋白P)是目前最为常用的抗菌反义靶点,对脂肪酸的生物合成至关重要。通过设计acpP基因的反义RNA序列,可导致E. coli的AcpP蛋白表达减少,从而抑制E. coli的生长,降低细胞活力[40]。
以上研究结果表明,通过反义疗法能中断必要基因的表达,恢复耐药菌对相应抗生素的敏感性和延长现有抗生素的药效。反义抗菌剂几乎针对任何微生物,而且具有靶向性,可通過快速设计和合成,能为药物的开发提供灵活性,这可能是逆转细菌耐药性的一种有前途策略。
2.3 外排泵抑制剂(EPI)
EPI是近年来兴起的研究热点。EPI主要是通过不同的机制失活或抑制细菌的外排泵活性,阻止药物外排,从而增强抗生素的抗菌作用[17],达到逆转细菌耐药性的效果。常见EPI种类见表3。
EPI已广泛在临床上用于逆转细菌耐药性,包括CCCP、维拉帕米、利血平等。CCCP主要通过作用于NorA外排泵的能量源-质子动力,并阻断转运蛋白的能量供应,间接提高细菌对黏菌素的敏感性[41]。维拉帕米属于ABC外排泵抑制剂,主要是通过抑制ATP结合膜糖蛋白[42]。有研究表明,利血平能显著抑制外排基因mefA的mRNA转录水平及其编码蛋白MefA的表达水平,推测利血平可能是通过影响外排蛋白MefA的表达,从而发挥抑制MefA外排泵的形成[43]。溴化乙啶的积累和外排是检测药物作用于外排泵良好指标,当辣椒素作用于SA-1199(野生型)和SA-1199B(过表达的NorA)时,细胞内的溴化乙啶流出量减少,表明辣椒素能抑制NorA外排泵的形成,增加SA对环丙沙星的敏感性[45]。
抑制剂是一种具有阻滞或降低细菌耐药性的物质,除了上述综述的EPI外,可以根据不同细菌耐药性机制开发不同的抑制剂,比如β-内酰胺酶抑制剂、丙氨酸消旋酶抑制剂或QS抑制剂等。因外排泵是细菌正常的生理所需,所以在开发时,应注意避免对正常的生理代谢产生影响。
2.4 基因敲除
基因敲除是自80年代后半期发展起来的一种分子生物学技术[48],目前已广泛应用于细菌基因功能等相关领域研究。基因敲除技术从传统的同源重组技术逐渐发展出许多的新技术,如λRed同源重组系统、Cre/LoxP位点特异性重组、插入突变、CRISPR/Cas9基因编辑系统等[49]。利用基因敲除技术能有效去除细菌部分耐药基因,使其恢复对抗菌药物的敏感性见表4[50-56]。
采用同源重组方法从嗜麦芽寡养单胞菌菌株中敲除phop基因,可增加细菌细胞膜通透性,恢复耐药菌对氨基糖苷类抗生素的敏感性;在进一步研究中,强调以phop基因为靶点,开发抗多药耐药药物的可能性[51]。包膜多糖输出蛋白(WZA)对耐碳青霉烯类AB荚膜具有一定的毒力作用,通过同源重组方法清除耐药菌上的wza基因;结果显示,wza基因敲除后,细菌荚膜缺陷,细胞壁厚度减少,细胞粘附力和生物被膜形成能力减弱,从而能增强碳青霉烯类抗生素的抗菌能力以及能降低耐药菌荚膜的毒力,恢复细菌对碳青霉烯类抗生素的敏感性[52]。自2016年首次报道质粒携带的黏菌素耐药基因mcr-1以来,mcr-1质粒清除和恢复黏菌素敏感性成为了当前的研究热点[12]。利用CRISPR/Cas9基因编辑系统技术能成功敲除E. coli上的耐药基因mcr-1,后续研究发现,敲除菌株能恢复对多黏菌素的敏感性[53]。以上研究表明,敲除细菌部分耐药基因可抑制或阻断细菌耐药性的表达,从而恢复细菌对相应抗生素的敏感性。
基因敲除手段种类繁多,CRISPR/Cas9作为现在常用的基因编辑技术,具有高效、便捷、精确等优点。通过基因敲除技术,敲除相应的耐药基因,深入探讨其耐药机制,从而开发抑制细菌耐药性和增强抗菌作用的药物,达到逆转细菌耐药性目的。
3 结语与展望
细菌耐药性问题是治疗细菌感染的主要障碍,逆转细菌耐药性是克服此类难题的有效方法。致力于寻找疗效好、毒副作用小与成本低的逆转策略,从而达到解决细菌耐药性的目的是当前研究热点。逆转细菌耐药性机制主要包括消除耐药质粒、抑制主动外排泵、抑制生物被膜形成和抑制耐药基因表达等。逆转细菌耐药的策略主要包括联合用药、反义疗法、EPI和基因敲除等,以上部分策略在逆转细菌耐药性的治疗中取得了可喜的成果。
在未来的工作中,可通过分子生物学等技术去研究逆转细菌耐药性的机制,深入探讨逆转机制与耐药机制之间的联系;通过结合逆转细菌耐药性靶点筛选的研究工作,寻找新的药物作用靶点,开发新的细菌耐药逆转剂,同时避免细菌对新的逆转药物产生耐药性;中药作为细菌耐药逆转剂,具有很大的开发潜力,因此筛选具有逆转耐药的中药也将是一种有效途径;还可以寻找新的细菌耐药逆转策略与思路等。
逆转细菌耐药性是控制细菌耐药性快速发展的有效策略,但由于部分策略方法缺乏临床实验数据支撑。因此,我们仍然需要深入探讨逆转细菌耐药性机制与策略等相关问题,争取早日为抑制细菌耐药性的发展和新型逆转剂的研发提供理论依据。
参考文献
蓝素桂, 李治蓉, 苏爱秋, 等. 金黄色葡萄球菌抗生素耐药研究进展[J]. 食品与发酵工业, 2021, 47(13): 310-317.
Coculescu B. Antimicrobial resistance induced by genetic changes[J]. J Med Life, 2009, 2(2): 114-123.
Zhang H, Zhao D, Quan J, et al. mcr-1 facilitated selection of high-level colistin-resistant mutants in Escherichia coli[J]. Clin Microbiol Infect, 2019, 25(4): 517.e1-517.e4.
Kaufman G. Antibiotics: mode of action and mechanisms of resistance[J]. Nurs Stand, 2011, 25(42): 49-55.
Lan S G, Li Z R, Su A Q, et al. Study on the mechanisms of the cross-resistance to TET, PIP, and GEN in Staphylococcus aureus mediated by the Rhizoma Coptidis extracts[J]. J Antibiot (Tokyo). 2021, 74(5): 330-336.
Shawn M M, Catherine S. Emergence of global antibiotic resistance[J]. Prim Care, 2018, 45(3): 467-482.
Wu D M, Lu R C, Chen Y Q, et al. Study of cross-resistance mediated by antibiotics, chlorhexidine and Rhizoma coptidis in Staphylococcus aureus[J]. J Glob Antimicrob Resist. 2016, 7: 61-66.
吳东美, 陆若辰, 陈钰泉, 等. 黄连、氯己定与抗生素介导的细菌交叉耐药[J]. 中国医院药学杂志, 2017, 37(13): 1250-1255.
Hiramatsu K, Igarashi M, Morimoto Y, et al. Curing bacteria of antibiotic resistance: Reverse antibiotics, a novel class of antibiotics in nature[J]. Int J Antimicrobial Agents, 2012, 39(6): 478-485.
Wang Y, Tian T, Zhang J J, et al. Indole reverses intrinsic antibiotic resistance by activating a novel dual-function importer[J]. mBio, 2019, 10(3): e00676-19.
Denap J C B, Thomas J R, Musk D J, et al. Combating drug-resistant bacteria: Small molecule mimics of plasmid incompatibility as antiplasmid compounds[J]. J Am Chem Soc, 2004, 126(47): 15402-15404.
Zhang P, Bai L, Li Y, et al. Loss of mcr genes mediated by plasmid elimination and ISApl1[J]. Antimicrob Agents Chemother, 2019, 63(9): e01002.
Gomaa A A, Klumpe H E, Luo M L, et al. Programmable removal of bacterial strains by use of genome-targeting CRISPR-Cas systems[J]. mBio, 2014, 5(1): e00928-13.
Saier M H, Reddy V S, Tsu B V, et al. The transporter classification database (TCDB): Recent advances[J]. Nucleic Acids Res, 2016, 44(D1): D372-379.
Weston N, Sharma P, Ricci V, et al. Regulation of the AcrAB-TolC efflux pump in enterobacteriaceae[J]. Res Microbiol, 2018, 169(7-8): 425-431.
Opperman T J, Nguyen S T. Recent advances toward a molecular mechanism of efflux pump inhibition[J]. Front Microbiol, 2015, 6: 421.
Le M F. Exopolysaccharides and biofilms[J]. Curr Top Microbiol Immunol, 2020, 425: 225-254.
Kim H S, Lee S H, Byun Y, et al. 6-Gingerol reduces Pseudomonas aeruginosa biofilm formation and virulence via quorum sensing inhibition[J]. Sci Rep, 2015, 5(1): 8656.
Poudyal B, Sauer K. The ABC of biofilm drug tolerance: The MerR-like regulator BrlR is an activator of ABC transport systems, with PA1874-77 contributing to the tolerance of Pseudomonas aeruginosa biofilms to tobramycin[J]. Antimicrob Agents Chemother, 2018, 62(2): e01981-17.
Chen Y, Liu T J, Wang K, et al. Baicalein Inhibits Staphylococcus aureus biofilm formation and the quorum-sensing system in vitro[J]. PLoS One, 2017, 11(4): e0153468.
Tang M R, Wei X, Wan X, et al. The role and relationship with efflux pump of biofilm foration in Klebsiella pneumoniae[J]. Microb Pathog, 2020, 147: 104244.
Zinner S H, Golikova M V, Strukova E N, et al. Predicting antibiotic combination effects on the selection of resistant Staphylococcus aureus: In vitro model studies with linezolid and gentamicin[J]. Int J Antimicrob Agents, 2018,52(6): 854-860.
Oie S, Uematsu T, Sawa A, et al. In vitro effects of combinations of antipseudomonal agents against seven strains of multidrug-resistant Pseudomonas aeruginosa[J]. J Antimicrob Chemother, 2003, 52(6): 911-914.
Vestergaard M, Paulander W, Marvig R L, et al. Antibiotic combination therapy can select for broad-spectrum multidrug resistance in Pseudomonas aeruginosa[J]. Int J Antimicrob Agents, 2016, 47(1): 48-55.
Baek M S, Chung E S, Jung D S, et al. Effect of colistin-based antibiotic combinations on the eradication of persister cells in Pseudomonas aeruginosa[J]. J Antimicrob Chemother, 2020, 75(4): 917-924.
Coates A, Hu Y, Holt J, et al. Antibiotic combination therapy against resistant bacterial infections: Synergy, rejuvenation and resistance reduction[J]. Expert Rev Anti Infect Ther, 2020, 18(1): 5-15.
Randhawa H K, Gautam A, Sharma M, et al. Cell-penetrating peptide and antibiotic combination therapy: A potential alternative to combat drug resistance in methicillin-resistant Staphylococcus aureus[J]. Appl Microbiol Biotechnol, 2016, 100(9): 4073-4083.
Raj Dwivedi G, Khwaja S, Singh Negi A, et al. Design synthesis and drug resistance reversal potential of novel curcumin mimics Van D: Synergy potential of curcumin mimics[J]. Bioorg Chem, 2021, 106: 104454.
Yap P S X, Krishnan T, Yiap B C, et al. Membrane disruption and anti-quorum sensing effects of synergistic interaction between Lavandula angustifolia (lavender oil) in combination with antibiotic against plasmid-conferred multi-drug-resistant Escherichia coli[J]. J Appl Microbiol, 2014, 116(5): 1119-1128.
Liu J, Chen F Y, Wang X F, et al. The synergistic effect of mud crab antimicrobial peptides sphistin and sph12-38 with antibiotics azithromycin and rifampicin enhances bactericidal activity against Pseudomonas aeruginosa[J]. Front Cell Infect Microbiol, 2020, 10: 572849.
Teethaisong Y, Autarkool N, Sirichaiwetchakoon K, et al. Synergistic activity and mechanism of action of stephania suberosa forman extract and ampicillin combination against ampicillin-resistant Staphylococcus aureus[J]. J Biomed Sci, 2014, 21(1): 90.
Akilandeswari K, Ruckmani K. Synergistic antibacterial effect of apigenin with β-lactam antibiotics and modulation of bacterial resistance by a possible membrane effect against methicillin resistant Staphylococcus aureus[J]. Cell Molecul Biol (Noisy-le-grand), 2016, 62(14): 74-82.
Dwivedi G R, Maurya A, Yadav D K, et al. Synergy of clavine alkaloid "chanoclavine" with tetracycline against multi-drug-resistant E. coli[J]. J Biomol Struct Dyn, 2019, 37(5): 1307-1325.
Parai D, Banerjee M, Dey P, et al. Reserpine attenuates biofilm formation and virulence of Staphylococcus aureus[J]. Microb Pathog, 2020, 138: 103790.
Stewart D B. Anti-Sense antibiotic agents as treatment for bacterial infections[J]. Surg Infect, 2018, 19(8): 831-835.
Sully E K, Geller B L. Antisense antimicrobial therapeutics[J]. Curr Opin Microbiol, 2016, 33: 47-55.
Bai H, Xue X, Hou Z, et al. Antisense antibiotics: A brief review of novel target discovery and delivery[J]. Curr Drug Discov Technol, 2010, 7(2): 76-85.
Zhang Y X, Ma W J, Zhu Y, et al. Inhibiting methicillin-resistant Staphylococcus aureus by tetrahedral DNA nanostructure-enabled antisense peptide nucleic acid delivery[J]. Nano Lett, 2018, 18(9): 5652-5659.
Meng J R, He G H, Wang H, et al. Reversion of antibiotic resistance by inhibiting mecA in clinical methicillin-resistant staphylococci by antisense phosphorothioate oligonucleotide[J]. J Antibiot, 2015, 68(3): 158-164.
Suzuki Y, Ishimoto T, Fujita S, et al. Antimicrobial antisense RNA delivery to F-pili producing multidrug-resistant bacteria via a genetically engineered bacteriophage[J]. Biochem Biophys Res Commun, 2020, 530(3): 533-540.
Baron S A, Rolain J M. Efflux pump inhibitor CCCP to rescue colistin susceptibility in mcr-1 plasmid-mediated colistin-resistant strains and Gram-negative bacteria[J]. J Antimicrob Chemother, 2018, 73(7): 1862-1871.
Chen C, Gardete S, Jansen R S, et al. Verapamil targets membrane energetics in mycobacterium tuberculosis[J]. Antimicrob Agents Chemother, 2018, 62(5): e02107-17.
Parai D, Banerjee M, Dey P, et al. Reserpine attenuates biofilm formation and virulence of Staphylococcus aureus[J]. Microb Pathog, 2020, 138: 103790.
Emiliano L, Nicholas C, Gianmarco M, et al. Natural alkaloid berberine ativity against Pseudomonas aeruginosa MexXY-Mediated aminoglycoside resistance: In silico and in vitro studies[J]. J Nat Prod, 2019, 82(7): 1935-1944.
Kalia N P, Mahajan P, Mehra R, et al. Capsaicin, a novel inhibitor of the NorA efflux pump, reduces the intracellular invasion of Staphylococcus aureus[J]. J Antimicrob Chemother, 2012, 67(10): 2401-2408.
Dwivedi G R, Upadhyay H C, Yadav D K, et al. 4-Hydroxy-α-tetralone and its derivative as drug resistance reversal agents in multi drug resistant Escherichia coli[J]. Chem Biol Drug Des, 2014, 83(4): 482-492.
Wang D, Xie K P, Zou D, et al. Inhibitory effects of silybin on the efflux pump of methicillin?resistant Staphylococcus aureus[J]. Mol Med Rep, 2018, 18(1): 827-833.
Tang P W, Chua P S, Chong S K, et al. A review of gene knockout strategies for microbial cells[J]. Recent Pat Biotechnol, 2015, 9(3): 176-197.
Zerbini F, Zanella I, Fraccascia D, et al. Large scale validation of an efficient CRISPR/Cas-based multi gene editing protocol in Escherichia coli[J]. Microb Cell Fact, 2017, 16(1): 68.
Lei X L, Fan Q X, Huang T, et al. Efficient circular gene knockout system for Burkholderiales strain DSM 7029 and Mycobacterium smegmatis mc2 155[J]. Acta Biochim Biophys Sin, 2019, 51(7): 697-706.
Liu M C, Tsai Y L, Huang Y W, et al. Stenotrophomonas maltophilia phop, a two-component response regulator, involved in antimicrobial susceptibilities[J]. Plos One, 2016, 11(5): e0153753.
Niu T S, Guo L H, Luo Q X , et al. wza gene knockout decreases Acinetobacter baumannii virulence and affects Wzy-dependent capsular polysaccharide synthesis[J]. Virulence, 2020, 11(1): 1-13.
Dong H S, Xiang H, Mu D , et al. Exploiting a conjugative CRISPR/Cas9 system to eliminate plasmid harbouring the mcr-1 gene from Escherichia coli[J]. Int J Antimicrob Agents, 2019, 53(1): 1-8.
Synefiaridou D, Veening J W. Harnessing CRISPR-Cas9 for genome editing in Streptococcus pneumoniae D39V[J]. Appl Environ Microbiol, 2021, 87(6): e02762-20.
Kim J S, Cho D H, Park M, et al. CRISPR/Cas9-Mediated Re-sensitization of antibiotic-resistant Escherichia coli harboring extended-spectrum β-lactamases[J]. J Microbiol Biotechnol, 2016, 26(2): 394-401.
Bikard D, Euler C W, Jiang W Y, et al. Exploiting CRISPR-Cas9 nucleases to produce sequence-specific antimicrobials[J]. Nat Biotechnol, 2014, 32(11): 1146-1150.
基金項目:广西区自然资金项目(No. 2014GXNSFAA118176)
作者简介:廖燕科,女,生于1996年,在读硕士研究生,主要研究方向为药物新剂型、新制剂的研究与开发,E-mail: 2895824900@qq.com

