小桐子YABBY全基因组家族成员的鉴定、表达和可变剪接分析
张作胜, 龚明, 吴丹丹, 杨宇, 刘丽宵, 王莎莎
小桐子全基因组家族成员的鉴定、表达和可变剪接分析
张作胜, 龚明, 吴丹丹, 杨宇, 刘丽宵, 王莎莎*
(云南师范大学生命科学学院, 生物能源持续开发利用教育部工程研究中心, 云南省生物质能与环境生物技术重点实验室, 昆明 650500)
为发掘能源植物小桐子()的YABBY转录因子,以最新公布的小桐子基因组序列为参考,在全基因组层面鉴定出5个亚家族的7个基因,同一亚家族的成员具有相似的氨基酸序列、基因结构和保守基序组成。YAB2和FIL/YAB3亚家族的2个旁系同源基因对(/、/)具有良好的共线性关系,表明片段复制或全基因组复制是小桐子YABBY家族扩张的主要方式。纯化选择是进化的主要动力,而YAB2亚家族成员可能在进化中经历了更明显的功能分化。基因表达模式和蛋白互作预测分析表明JcYAB2B和JcYAB3可能在种子的发育过程中起到重要的调控作用;同时,细胞分裂素、干旱或高盐胁迫处理抑制了大多数JcYABs成员的基因表达。此外,转录组测序结合qRT-PCR分析表明,低温处理有效诱导和的基因表达模式发生变化,并伴随着新的、截短的可变剪接转录本的动态积累。因此,推测JcYABs可能通过剪接体的功能竞争或功能互补参与低温响应的调节,这些结果有助于更好地了解YABBY家族成员的功能分化并阐明可变剪接如何调控小桐子低温响应的分子机制。
小桐子; YABBY基因家族; 可变剪接; 生物信息学; 基因表达
YABBY转录因子属锌指蛋白超家族,是植物特有的小基因家族,成员均由2个高度保守的结构域组成,即N-端的锌指结构域(Cys2-Cys2型)和C-端的YABBY结构域(具螺旋-环-螺旋基序),被认为与DNA特异性结合有关[1]。自1999年首次报道以来[2],基因已在多种植物中得到分离和功能研究,被认为广泛参与植物的多项生命进程,如器官发生和极性建立、果实成熟、激素信号和非生物胁迫响应等[3–4]。
模式植物拟南芥()有6个基因,被分为5个亚家族,分别是CRABS CLAW (CRC)、 FILAMENTOUS FLOWER (FIL)/ YABBY3 (YAB3)、INNER NO OUTER (INO)、YABBY2 (YAB2)和YABBY5 (YAB5),在器官发育中起不同作用并存在一定的功能冗余[5]。总的来说,所有AtYABBYs均可通过促进远轴表面的细胞分化参与侧生器官近-远轴极性的建立,其中AtFIL/AtYAB1、AtYAB2、AtYAB3和AtYAB5共同调控营养器官如叶片的发育[5],而AtCRC和AtINO则主要参与花器官的形成,如心皮、蜜腺[2]和胚珠的发育[6]。番茄()有9个基因,具有与拟南芥同源基因相似的组织表达模式但存在明显的功能差异。以YAB2亚家族为例,主要在开花和果实发育中调节心皮数量[7],而则可能影响果皮细胞的发育命运并调控果实的成熟[8]。在单子叶植物中,水稻()的YABBY家族由8个成员构成,其中OsDL和OsYAB1分别作为AtCRC和AtYAB2的直系同源物,前者主要调控叶片主脉形成、心皮发育和花分生组织的决定[9–10],后者可参与赤霉素生物合成的反馈调节[11], 但尚未证明二者功能与侧生器官的极性形成有关。OsYAB2在种子落粒中起关键作用[12],而()、()、()则是维持所有生殖分生组织正确功能所必需的[13]。此外,前人[14–15]通过在拟南芥中异源表达先后证实了小麦()的TaYAB1和TaYAB2均参与叶片的极性建立,Strable等[16]则证实玉米()的2个CRC同源物drooping leaf1 (drl1)和drooping leaf2 (drl2)是花分生组织决定和花器官发育所必需的。
另一方面,表达分析表明干旱、高盐和ABA等处理下棉花() YABBY家族的多数成员均下调表达[17],大豆() GmYABBY3、GmYABBY10和GmYABBY16虽为诱导表达,但在拟南芥中异源表达证实GmYABBY10作为负调因子参与干旱和高盐胁迫响应[18]。荧光素酶活性分析显示番茄SlYABBY2b可直接与(吲哚乙酸酰胺合成酶基因)的启动子结合抑制其转录[19],而VvYABBY4则可作为转录激活因子影响葡萄()的种子发育和果实形态建成[20]。这些均表明YABBY家族在种子植物生长发育过程中的功能相对保守,但参与激素或环境胁迫响应的作用方式及家族成员的功能分化仍有待探索。
可变剪接(alternative splicing, AS)是一种广泛存在于真核生物的mRNA前体(pre-mRNA)加工方式, 通过调节不同mRNA剪接体的稳定性、运输、定位和翻译,维持细胞内功能性和非功能性转录本之间的稳态和平衡[21],或调节编码蛋白的分子特征和功能,是增加转录组和蛋白组结构与功能多样性的重要机制,在昼夜节律、成花转变、表型变异、发育调控以及胁迫响应中起重要作用[22]。但目前有关植物基因的可变剪接研究还未见报道。
小桐子()是大戟科(Euphorbiaceae)麻疯树属具有巨大开发潜力的能源植物树种,其种子含油量平均可达34.4%,是加工生物柴油的优质原料[23]。小桐子属典型的喜温冷敏型植物[24],低温冷害是限制其种子产量、地理分布和产业发展的重要环境因素。基于基因家族在种子植物生长发育和逆境响应的前期研究,本研究根据最新公布的小桐子基因组组装和转录组测序数据[25–26],在全基因组层面对基因家族成员的理化性质、结构特征、进化关系和表达模式进行系统分析,揭示了不同成员在低温胁迫下的转录及可变剪接变化, 为其在小桐子的遗传改良和抗逆分子育种的潜在应用奠定理论基础。
1 材料和方法
1.1 小桐子YABBY家族的成员鉴定和理化性质分析
从NCBI网站提取小桐子基因组和蛋白质序列用于构建本地数据库(RJC1_Hi-C, BioProject: PRJ NA601606),采用2种方法从小桐子全基因组中查找基因。首先自Pfam数据库获取YABBY基因(PF04690.15)的隐马尔科夫模型(hidden Markov model),用HMMER软件(v3.2.2)的hmmsearch程序在小桐子蛋白质数据库中搜索候选YABBY蛋白(E- value<0.001);同时以拟南芥的全部6个YABBY蛋白(下载自拟南芥数据库TAIR)作为参考序列,对小桐子蛋白数据库进行BLAST检索(E-value<10–5)。经去除冗余序列、过滤可变剪接体获得候选序列, 提交至NCBI CDD数据库(Conserved Domains Database)验证保守结构域,最终获得基因家族成员(s)。ExPASy在线程序被用于蛋白理化性质分析,亚细胞定位和磷酸化修饰位点分别以Plant-mPLoc server (http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/)和GPS 5.0软件进行预测。
1.2 系统发育、蛋白基序组成和基因结构分析
利用Clustal Omega对小桐子、拟南芥、葡萄、水稻和玉米YABBY家族成员的氨基酸序列进行多重比对,并通过MEGA X以邻接法(neighbor-joining, NJ)构建无根系统发育树(参数设置:P-distance模型, complete deletion, bootstrap 1 000次重复),并依据与拟南芥同源物的进化关系对家族成员进行命名。WEB LOGO (http://weblogo.threeplusone.com)和MEME suite (https://meme-suite.org/meme/)分别用于创建保守结构域(C2C2锌指域和YABBY域)的序列标识图和注释保守基序(参数设置:氨基酸残基数6~100 aa,基序数量15),并以MPI Bioinformatics Toolkit (https:// toolkit.tuebingen.mpg.de)的REPPER工具对保守域的二级结构进行预测。最后以Gene Structure Display Server (GSDS, v2.0, http://gsds.gao-lab.org)在线工具绘制基因结构图。为全面展示基因及编码蛋白的结构特征,本文自NCBI、小桐子JCDB数据库[26]提取的全部转录本用于基因结构及蛋白基序分析。
1.3 染色体定位、共线性和选择压力分析
从NCBI基因组数据库下载小桐子基因组gff3注释文件,结合高密度连锁图谱[27]获取基因的染色体位置信息,用MapGene2Chrom (v2.0, http://mg2c.iask.in/ mg2c%5Fv2.0/)绘制基因的染色体分布图。共线性分析基于蛋白序列的本地blast (参数设置: E-value<10–5, num_alignments 5)和基因位置信息,以TBtools内置的MCScanX程序完成[28]。经OrthoFinder程序查找同源基因对,KaKs_Calculator 2.0软件根据自然梯度(natural gradient descent, NG)法计算非同义替换率(non-synonymous substitution rate, Ka)和同义替换率(synonymoussubstitution rate, Ks)值, 以Ka/Ks评估同源基因对的进化选择压力[29]。
1.4 启动子顺式作用元件和蛋白互作网络预测分析
提取各基因编码区起始ATG上游2.0 kb的基因组序列提交至PlantCARE数据库(http://bioinfor matics.psb.ugent.be/webtools/plantcare/html/),用于统计基因启动子区顺式作用元件的数量和种类;以STRING网站(https://string-db.org)预测蛋白间的互作关系,以基因为节点、以连线展示互作关系,最后依据可信度分值选择前20% (90对)互作关系以Cytoscape软件(v3.8.2)加以可视化。
1.5 基于转录组数据的表达模式分析
1.6 植物材料与低温处理
供试小桐子种子采收自云南省元谋县,经消毒后置于浸湿的滤纸上浸种18 h,转入26 ℃、75%相对湿度的人工气候箱中暗萌发7 d,此后转移至营养土中生长至两叶一心期[31]。选取健康均一的幼苗转入12 ℃低温锻炼并分别于0 (对照)、6、12、24和48 h后收获第2片(形态学顶端)真叶,经液氮速冻后于–80 ℃保存,用于总RNA提取。每个处理设置3组生物学重复,每个重复取自3株独立幼苗。
1.7 低温胁迫下的基因表达模式分析
叶片组织总RNA的提取按照RNAprep Pure多糖多酚植物总RNA提取试剂盒(天根生化科技有限公司, 北京)完成。选择高质量的RNA样品1g, 经PrimeScript RT reagent Kit with gDNA Eraser for RealTime试剂盒反转录(宝日医, 大连)获得第一链cDNA。qRT-PCR反应采用TB Green®™ II试剂盒(宝日医, 大连),每10L反应体系中包含5L TB GreenII (2×)、上/下游引物(10mol/L)各0.4L、cDNA模板稀释液1L和ddH2O 3.2L。反应由Roche LightCycler®96 System实时荧光定量PCR仪完成。反应程序为: 95 ℃预变性30 s;然后95 ℃变性10 s,57 ℃退火10 s,72 ℃延伸10 s,共45个循环;最后95 ℃ 5 s,60 ℃ 60 s,97 ℃15 s。以2–△△CT法计算相对表达量,以基因(GenBank登录号XM_012230660.3)作为内参。每组实验均设置3个技术重复、2~3组生物学重复, 以SPSS软件(v26.0)进行显著性分析(ANOVA,单因素分析),以<0.05表示显著差异。
1.8 转录组测序和可变剪接分析
小桐子幼苗的第2片真叶被用于RNA-seq测序(Illumina HiSeq 4000平台,NCBI登录号: PRJNA66 1688),用rMATS软件(v4.0.1)检测可变剪接事件[32]。以SnapGene软件分析同一基因不同mRNA剪接体的结构,根据RNA-seq预测的AS位点两端设计特异性引物,经高保真酶(LA Taq, 宝日医, 大连)扩增和测序验证后,以半定量RT-PCR (Green Taq Mix, 诺唯赞,南京)同时检测全长转录本和可变剪接体在低温胁迫下的丰度变化。以基因(GenBank登录号XM_ 012230243.3)为内参对cDNA样本均一化,以电泳条带的灰度分析评估转录变化。
2 结果和分析
2.1 YABBY基因家族成员的鉴定
综合利用本地BLAST和hmmsearch工具,首先在小桐子全基因组中共查找到12条候选YABBY蛋白。经去除冗余序列、可变剪接体过滤和保守结构域确认,最终鉴定出7个基因(s), 并依据其在进化上与模式植物拟南芥中同源蛋白的亲缘关系进行命名。从表2可见,JcYAB2B是最小的成员,仅由162个氨基酸残基组成,分子量为18.32 kD, 而最大的JcINO则包含284个氨基酸残基,分子量为32.18 kD。JcYABs蛋白的等电点为6.83~9.34,亚细胞定位预测所有成员均定位于细胞核。
2.2 JcYABs的系统进化、蛋白基序及其基因结构分析
选择模式植物拟南芥、玉米、水稻和葡萄的YABBY蛋白用于构建系统发育树(NJ法),结果表(图1)明,5种植物的41个YABBY成员可分为5个亚家族,其中YAB5亚家族仅由双子叶植物来源的YABBY蛋白构成。YAB5、INO、CRC亚家族各包含1个JcYAB成员,这与其他双子叶植物一致。此外,JcYAB1、JcYAB3同属FIL/YAB3亚家族, 而YAB2亚家族也包含2个成员JcYAB2A、JcYAB2B,但值得注意的是,大多数JcYABs与葡萄的同源蛋白聚在同一支,而JcYAB2B似乎与单子叶植物的同源蛋白有更近的系统发育关系。

表1 本研究所用引物信息
对小桐子与拟南芥的YABBY同源蛋白进行多重序列比对,结果表明(图2: A), 位于N-端的锌指结构域保守度相对较低,且各成员在二级结构组成上也存在差异,主要的保守结构包括3个较小的片层,中间以1个短-螺旋间隔存在(, 图2: B, C)。位于C-端约40个氨基酸残基构成的结构域存在于所有成员中,具高的保守性,不仅包括潜在的核定位信号(nuclear localization signal, NLS),二级结构预测也显示该区域存在2个高度保守、较长的-螺旋元件(图2: B, C)。
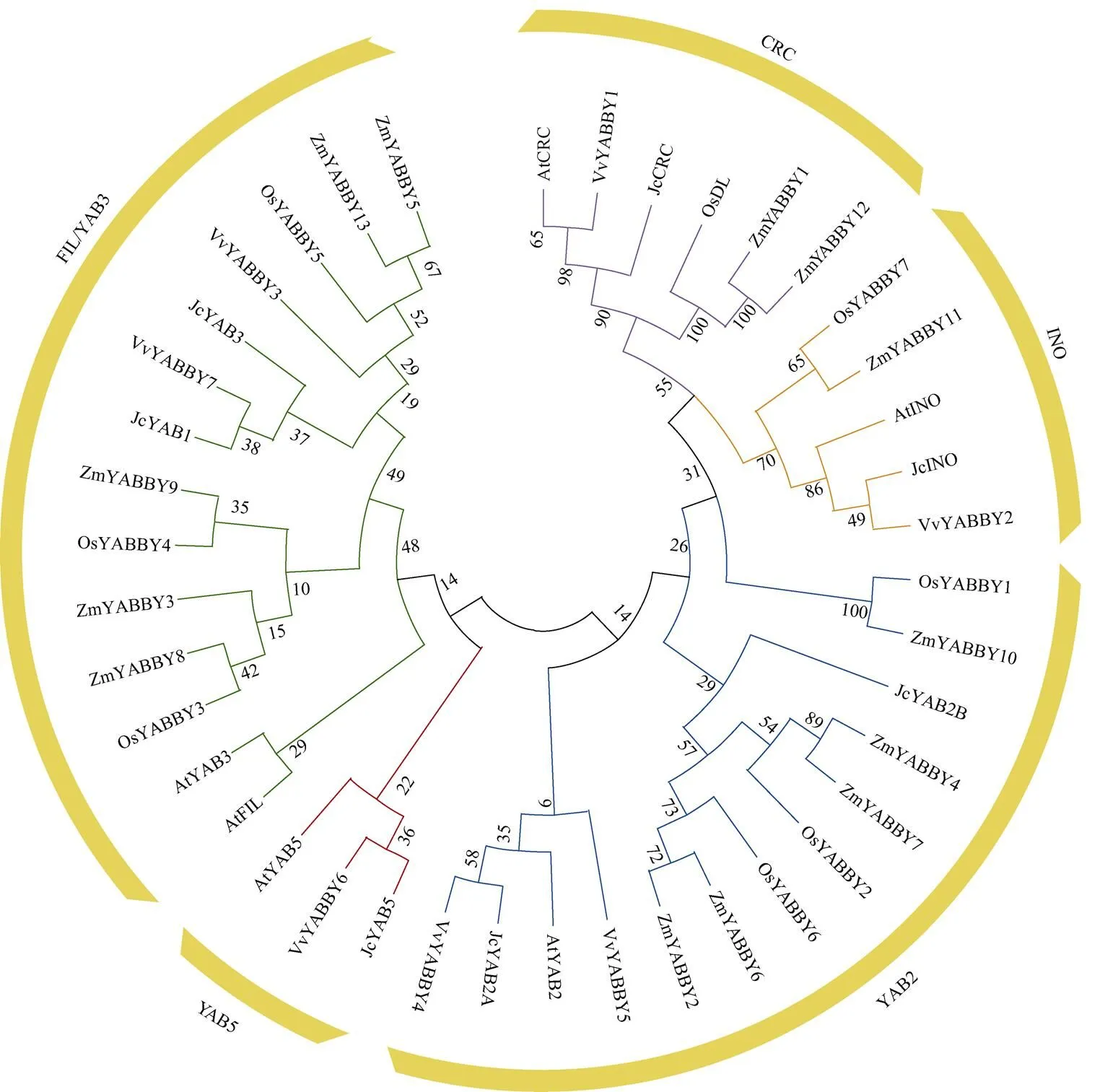
图1 小桐子YABBY家族成员的系统进化分析。Jc: 小桐子; Os: 水稻; At: 拟南芥; Zm: 玉米; Vv: 葡萄。下同
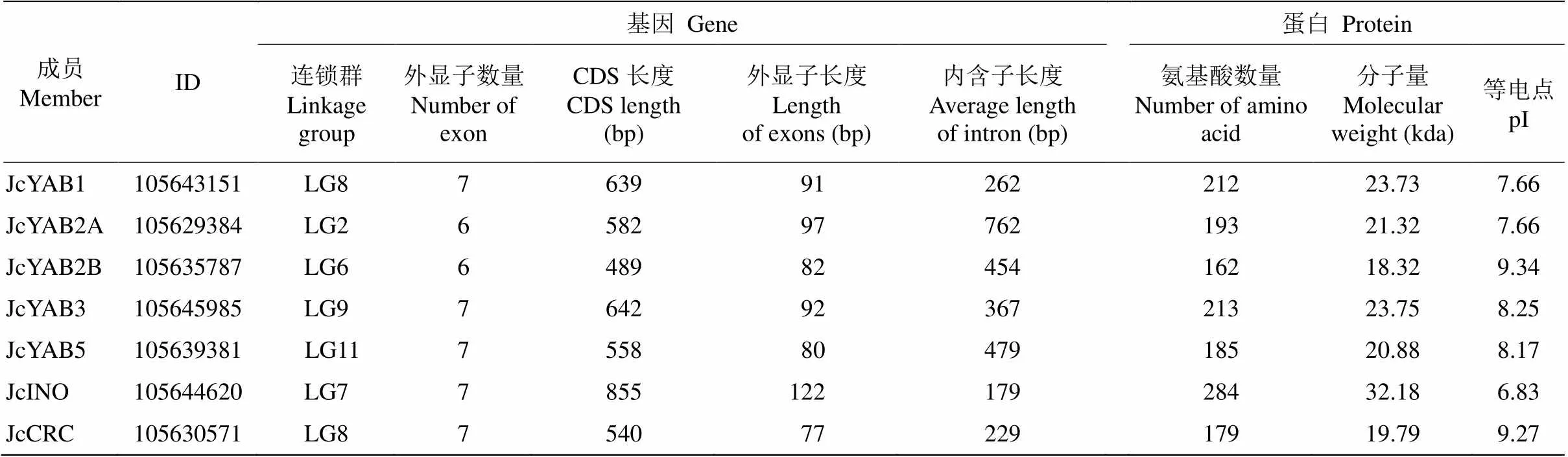
表2 小桐子YABBY家族成员的理化性质和基因结构
从图2: D可见,在注释到的15个基序中,Motif 1、Motif 3 (YABBY结构域)及Motif 2 (锌指域)存在于所有JcYAB蛋白(含可变剪接体)中,且同一亚家族成员的基序构成高度一致,如Motif 6、Motif 10和Motif 11为CRC亚家族所特有,而INO亚家族的2个成员均含有特异性的Motif 9和Motif 12。唯一的例外是JcYAB2B,在进化上属YAB2亚家族,但保守基序组成却与YAB5亚家族成员完全一致,并缺失了JcYAB2A与AtYAB2共有的、紧邻YABBY结构域的特征性小基序Motif 14 (LKLDGNN/KK), 这与我们的系统发育分析结果一致。
基因结构分析表明,s基因长度为2 271 ()~5 365 bp (),由6~7个内含子组成,外显子平均长度为91.60 bp,与拟南芥(92.63 bp)十分接近。YABBY结构域的编码区均位于第3~6个外显子,尤其外显子4和5在所有成员中的长度完全一致,分别为49、76 bp。JcCRC的3个可变剪接转录本(~)在外显子3的5′端存在6~33 nt的缺失,而JcINO.2仅在外显子5的5′端缺失3 nt,但这均不影响编码蛋白的保守基序组成(图2: D)。另一方面,基因内含子长度差异极大,其中内含子4的相对最小(84~179 bp),而位于N-端锌指结构域编码区间隔的内含子1长度差异最大(118~2 448 bp),暗示内含子对JcYABs家族进化和成员功能分化的潜在意义。
2.3 JcYABs的染色体定位、共线性和选择压力分析
根据Wu等[27]公布的小桐子高密度基因连锁图谱, 7个s基因定位至11个连锁群(linkage group, LG)中的6个(表2),呈明显的不均匀分布,其中和均定位于LG8,而其余基因分别定位于LG2、LG6、LG7、LG9和LG11。MCScanX共线性分析表明,小桐子基因家族中不存在串联复制事件,、和属散在复制, 其余4个均来自片段复制或全基因组复制(segmental duplication/WGD),其中//为共线性基因对(图3: A)。基于OrthoFinder的同源性分析也证实//分别为来自FIL/YAB3和YAB2正交群的2个旁系同源基因对,其Ka/Ks分别为0.17、0.22 (均远小于1,表3),表明这2个亚家族在进化中受到强烈的纯净选择。
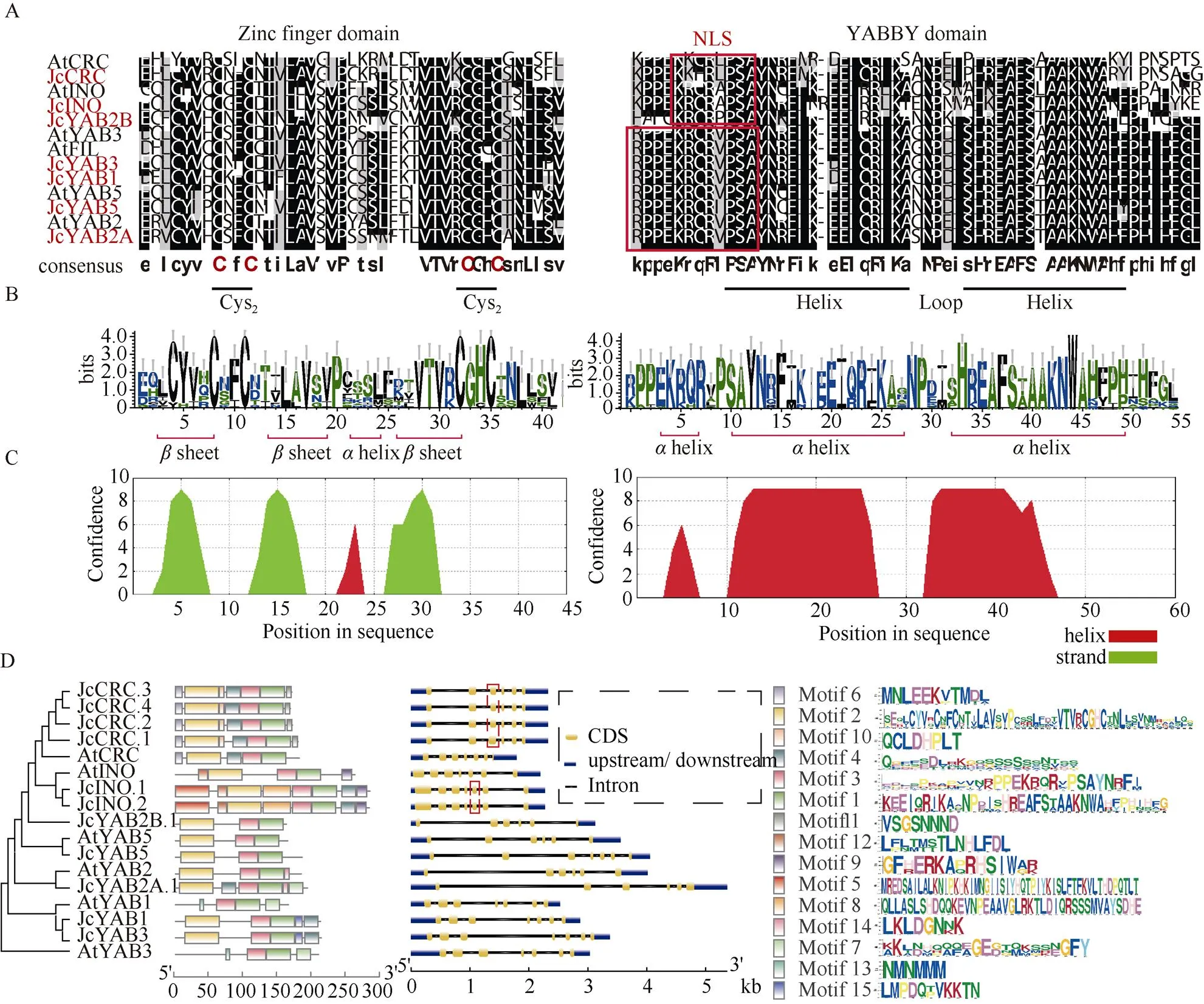
图2 小桐子与拟南芥YABBY蛋白的序列比对(A)、保守度(B)、保守结构域的二级结构预测(C)和基因结构(D, 红色方框示不同转录本的差异)

表3 小桐子YABBY家族同源基因对的Ka/Ks
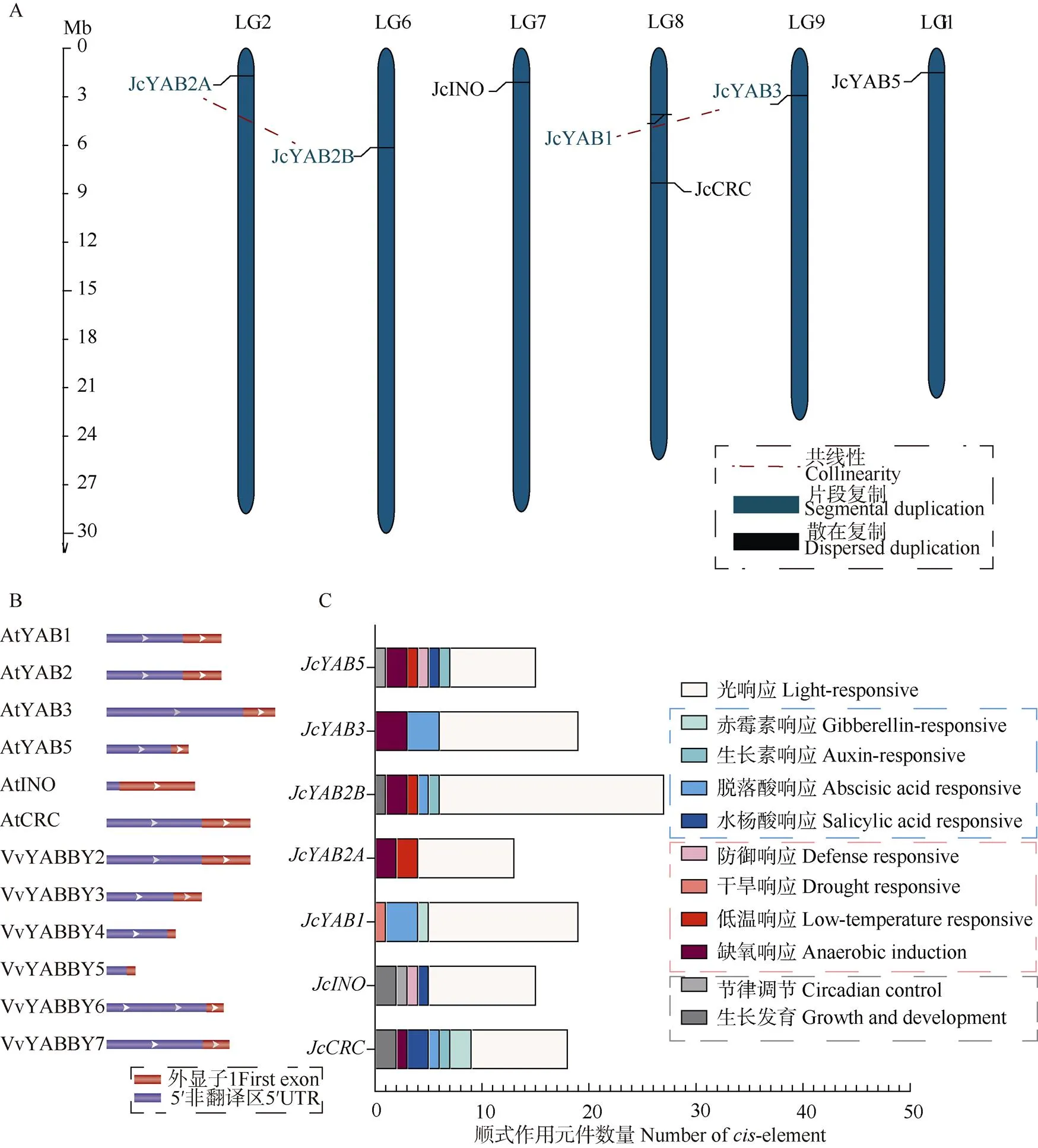
图3 JcYABs基因的染色体定位和启动子顺式作用元件。A: 染色体定位,2个共线性基因对以红色虚线相连; B: 拟南芥、葡萄的YABBY同源基因的5′ UTR结构; C: 顺式作用元件。
2.4 JcYABs基因启动子顺式作用元件分析
首先从NCBI基因组数据库获取拟南芥、葡萄的YABBY同源基因的基因组DNA和全长mRNA序列, 其5′ UTR均无内含子(图3: B),基于此对s基因潜在启动子区域(起始密码子上游2.0 kb)的顺式作用元件进行查找, 其顺式作用元件可归为4类(图3: C),其中光响应元件的丰度最高且分布于全部s基因的启动子区域(8~21个),其次是激素(包括脱落酸、水杨酸、赤霉素和生长素)和防御胁迫响应元件。值得注意的是,HD-Zip 1和GCN4 Motif分别作为参与叶肉细胞或内皮细胞分化的重要调控元件,仅存在于2个“生殖型”基因和的启动子中。含较多的激素响应元件,包括2个TCA-element (水杨酸响应)、2个P-box (赤霉素响应)、1个TGA-element (生长素响应)和1个ABRE (脱落酸响应),暗示其在小桐子生长发育调控中的潜在功能。典型的防御胁迫响应元件,如低温响应元件LTR (low-temperature responseveness)、干旱诱导元件MBS (MYB binding site involved in drought-inducibility)则主要存在于、和,表明这些“营养型”基因可能是小桐子非生物胁迫响应中的重要调节因子。
2.5 JcYABs的表达模式
基因的表达模式是功能预测的重要参考,从图4: A可见,和分别仅在花器官(花序、花芽)和幼叶中表达,具明显的组织表达特异性(T值分别为0.96、0.93)。其余s的组织表达特异性则较低(T值0.41~0.62),几乎在所有的组织器官中均可表达,其中和在至少3种组织(如茎尖、幼叶、花芽)中具有远高于其他成员的转录丰度,可能是参与营养器官形成与发育的重要成员。这些差异化的表达模式暗示s基因在小桐子生长发育过程中的功能多样性。
图4: B为在小桐子种子发育阶段(14~45 DPA)的表达模式变化,在整个阶段维持相对稳定的表达,而和多数时候几乎不表达,其剧烈的表达上调主要归因于二者极低的本底表达。在关键的胚乳干物质积累期(29~41 DPA)强烈诱导,在35 DPA时可达发育初期(14 DPA)的20.1倍。与此相反,和的转录水平则分别在29和35 DPA后持续下降至对照(14 DPA)的7.7%、8.4%,而的转录水平则随种子发育持续下降,到41 DPA后几乎检测不到,我们推测和可能分别是小桐子种子发育的正调节和负调节因子。
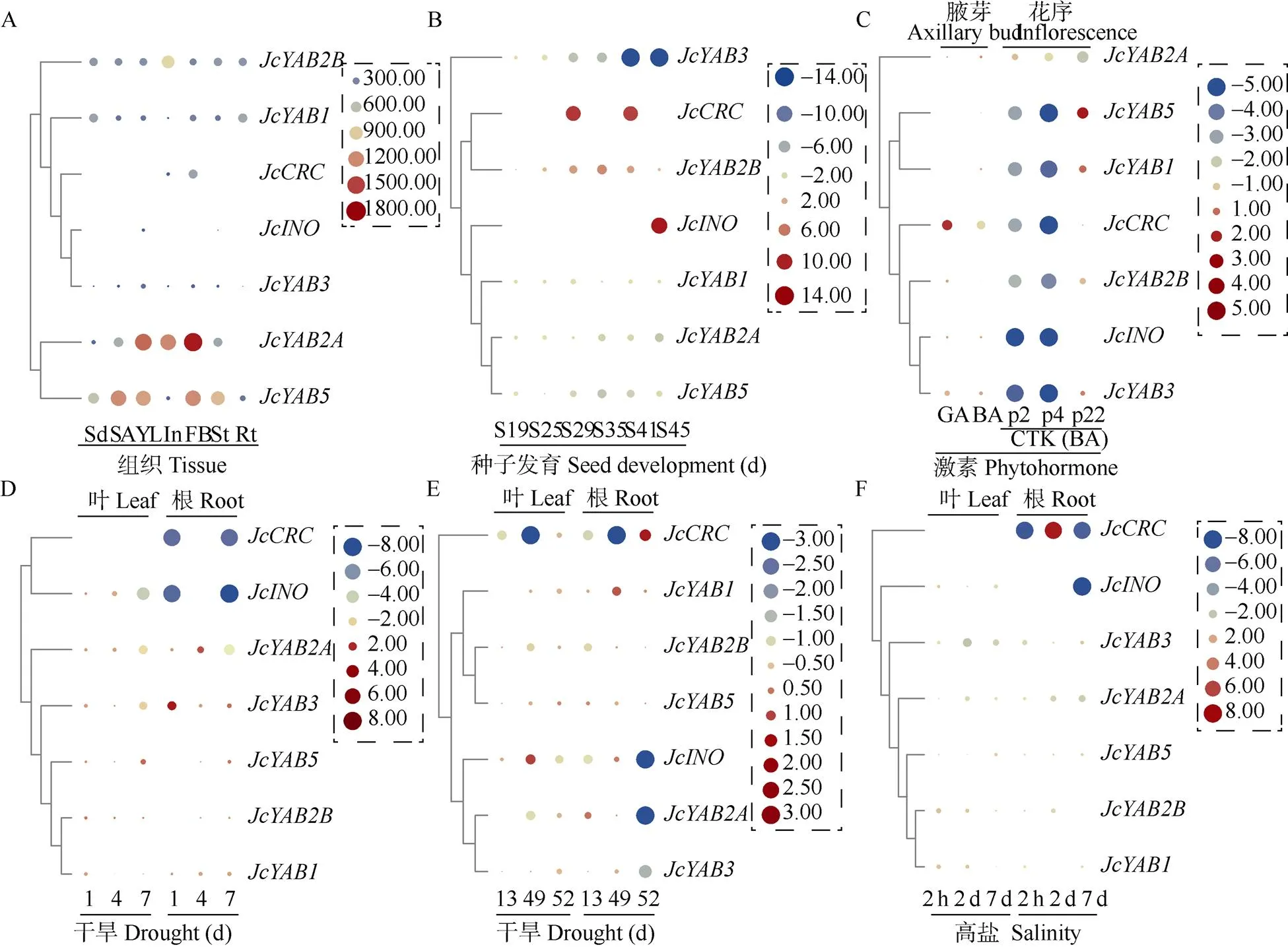
图4 JcYABs基因的表达模式(公开的RNA-seq数据)。A: 组织器官; Sd: 种子; SA: 茎尖; YL: 幼叶; In: 花序; FB: 花芽; St: 茎; Rt: 根; B: 种子发育阶段(授粉后14~45 d); C: 激素处理; GA: 赤霉素; BA: 6-苄基腺嘌呤; CTK: 细胞分裂素; p2, p4, p22: CTK分别处理2、4、22 h; D, E: 干旱胁迫; F: 高盐胁迫。
2.6 植物激素与非生物胁迫下的表达模式
顺式作用元件分析表明,s基因对植物激素和非生物胁迫存在差异性的转录响应。对小桐子腋芽(茎部节间)的转录组数据分析可知,仅的转录受到赤霉素GA3或6-BA的显著影响但效应相反,显示2种激素调节表达的拮抗性作用(图4: C)。另一方面,细胞分裂素对JcYABs家族的转录抑制效应在花序分生组织中普遍存在。经1 mmol/L 6-BA处理后,除持续下调外,其余s 在处理2 h后明显下调(-16.88~-2.63倍)并持续至处理后4 h (p4),至处理后22 h,此抑制效应基本消失甚至在个别成员转为诱导表达,如(2.02倍)和(4.19倍),这暗示JcYABs可能作为小桐子茎部分支和成花诱导的负调节因子起作用。
从图4: D, F可见,对短期干旱(1~7 d)的响应模式与高盐胁迫(100 mmol/L NaCl,2 h~7 d)类似,根系和叶片组织中的、和均无显著响应。2种胁迫分别抑制和在叶片中的转录,而2个“生殖型”YABBY成员和的抑制表达主要发生在根系(约为对照的5%)。另一方面,长期干旱胁迫下、和在根系和叶片中呈相反的响应模式, 但显著的胁迫诱导表达效应仅出现在极个别根系样品中(图4: E)。
2.7 JcYABs在低温胁迫下的转录和可变剪接变化模式
小桐子是典型的冷敏植物,低温是限制其生长发育、地理分布和种子产量的主要环境因子。对经12 ℃处理的叶片进行qRT-PCR分析(图5),、、和均不同程度地受到低温的诱导,其中的诱导效应最为明显且持续至48 h, 达对照的2.25倍。相反,和则在低温处理24 h内持续下调,分别下降至对照的15.91%、50.21%,之后有不同程度的恢复。此外, 虽同为共线性同源基因对,/和/表达量的皮尔逊相关系数却分别为-0.44和0.77,这表明JcYABs家族的不同成员在小桐子的低温响应中存在一定的功能分化。
综合Hi-seq高通量转录组测序和半定量RT-PCR分析,初步揭示s基因在低温处理小桐子叶片中的可变剪接(AS)变化模式,结果表明和均可通过可变剪接产生1条新的转录本(图6: A, B)。根据二者在NCBI数据库已有的注释信息,我们将新AS型转录本分别命名为。对新转录本及其编码区的结构分析可知,属“第1个外显子可变剪接(alternative firstexon, AFE)”类型,其推测的蛋白产物与相比缺失N-末端的第1~30个氨基酸,从而引起motif2和C2C2锌指域的部分缺失(图6: C, 图2: D)。则为“外显子跳跃(exon skipping, ES)”型可变剪接事件,原转录本的第5外显子缺失引起编码序列中第123位Lys突变为Asn及之后的移码突变,造成motif 1和YABBY结构域的大段缺失(图6: C, 图2: D)。2个新转录本均随着低温处理时间的延长在小桐子叶片中显著积累,且在24 h时相对丰度达到最高, 随后有所下降,且低温对的转录诱导更为明显(图6: D),这暗示低温胁迫可能通过不同剪接体间的相对丰度变化调节小桐子的低温抗性。
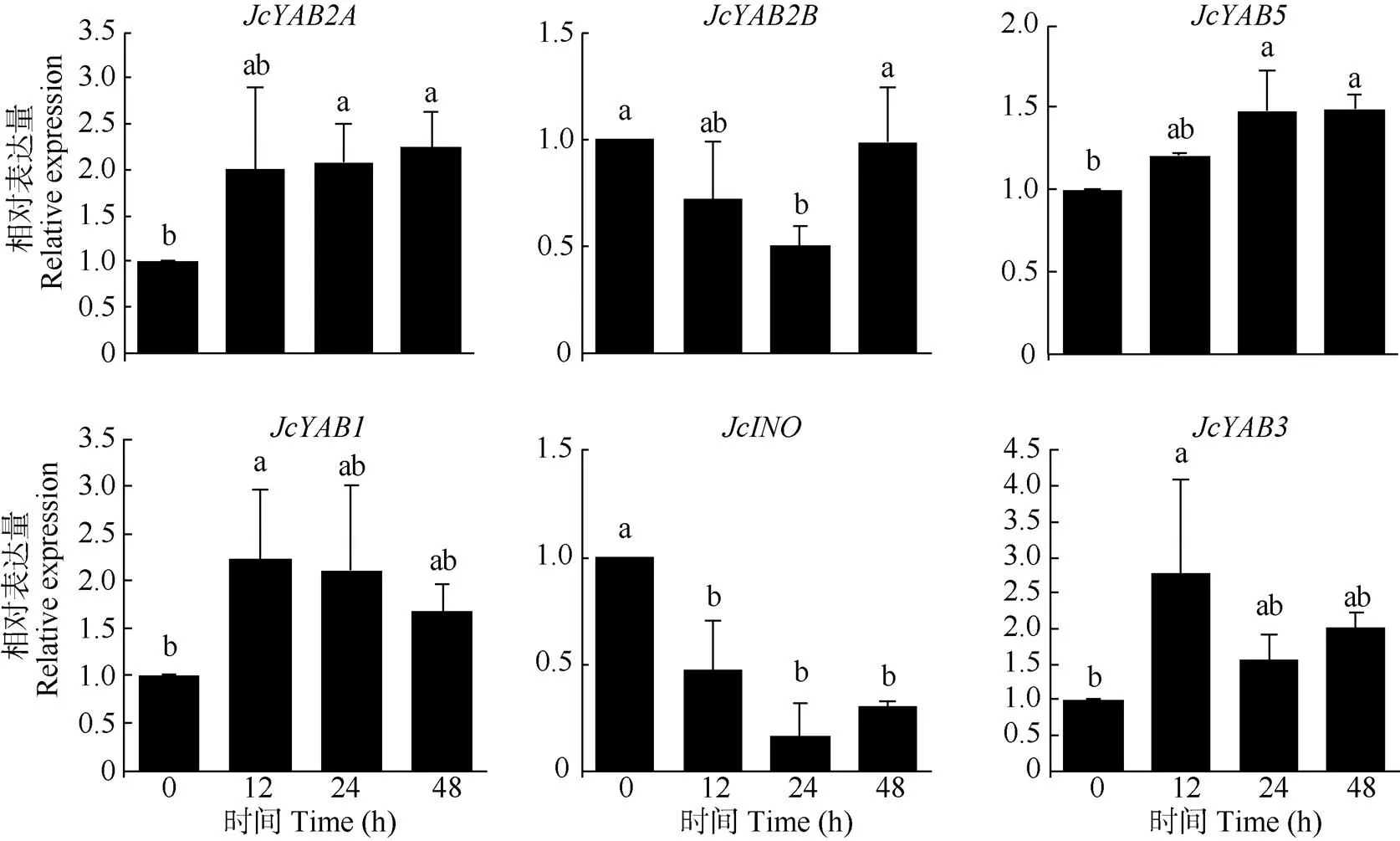
图5 JcYABs基因在12 ℃低温胁迫下的表达模式(qRT-PCR)
2.8 JcYABs蛋白的互作网络预测分析
蛋白互作对转录因子的活性调节及作用机制十分重要,基于STRING数据库中的互作关系预测了JcYABs蛋白的互作网络(图7),转录因子ANT、KANADI似乎是小桐子YABBY蛋白互作的中心, 暗示他们可能通过形成大的转录因子复合体共同调控下游基因的表达。一个与MAPK途径密切相关的LRR型受体ERECTA可与JcINO、JcCRC和JcYAB JcYAB1发生互作,而磷酸化修饰位点预测分析也表明这3个JcYAB蛋白均含有至少5个MAPK潜在作用位点。此外,6个生长素相关蛋白(如SAUR20、ARG7)仅与JcYAB5存在互作,而JcYAB2B的互作调节相对独立于其他成员,表明该成员在作用机制上也存在特异性。这可为深入探索小桐子YABBY家族转录因子的分子功能提供新的思路。
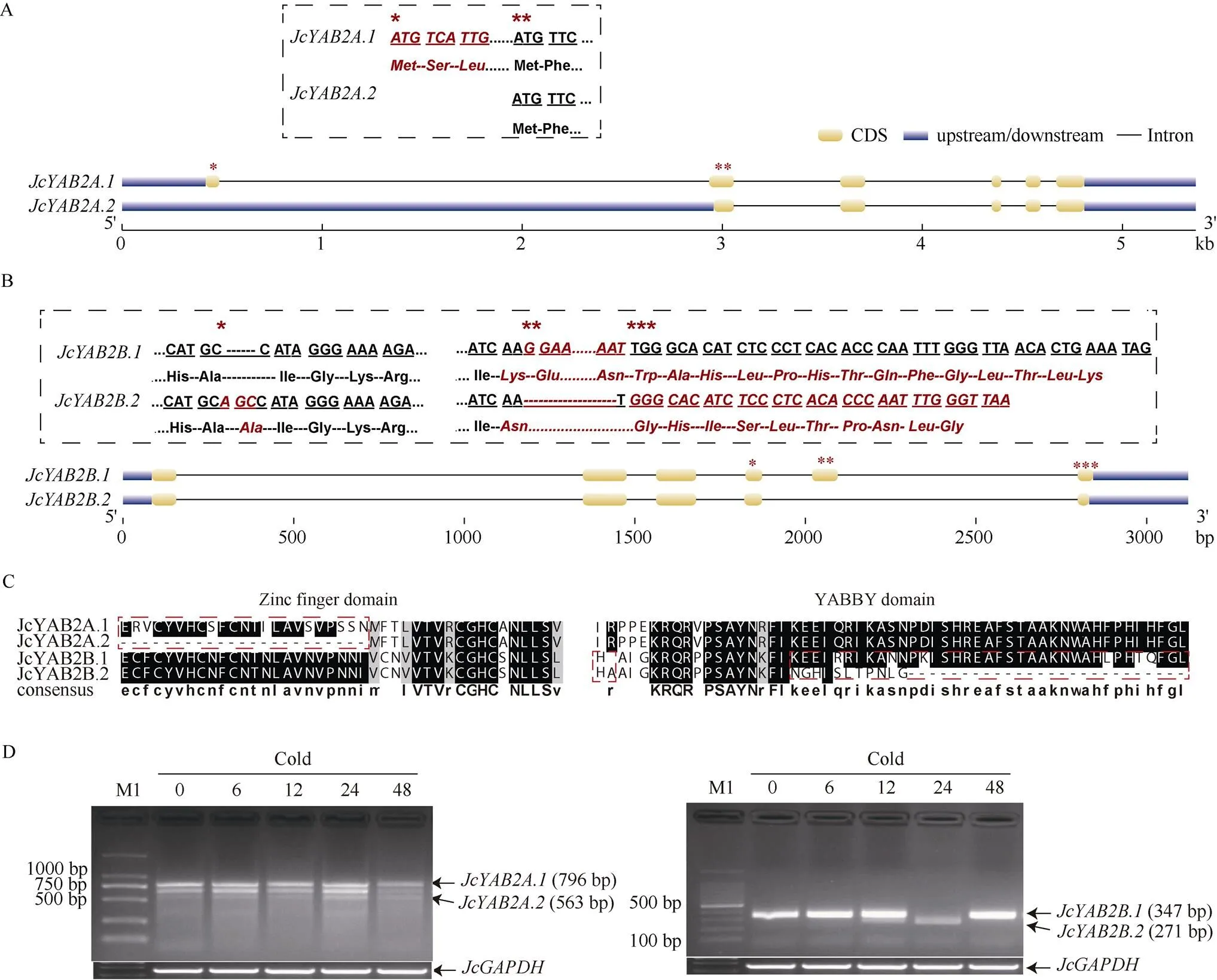
图6 JcYABs基因可变剪接体的鉴定和低温响应。A: 可变剪接事件引起JcYAB2A转录本和推定的蛋白序列变化; B: 可变剪接事件引起JcYAB2B转录本和推定的蛋白序列变化; C: 可变剪接事件对编码蛋白保守结构域的影响; D: JcYAB2A/2B本底与新可变剪接转录本在低温下的积累模式;*和红色虚线框均表示不同转录本和蛋白产物的差异。
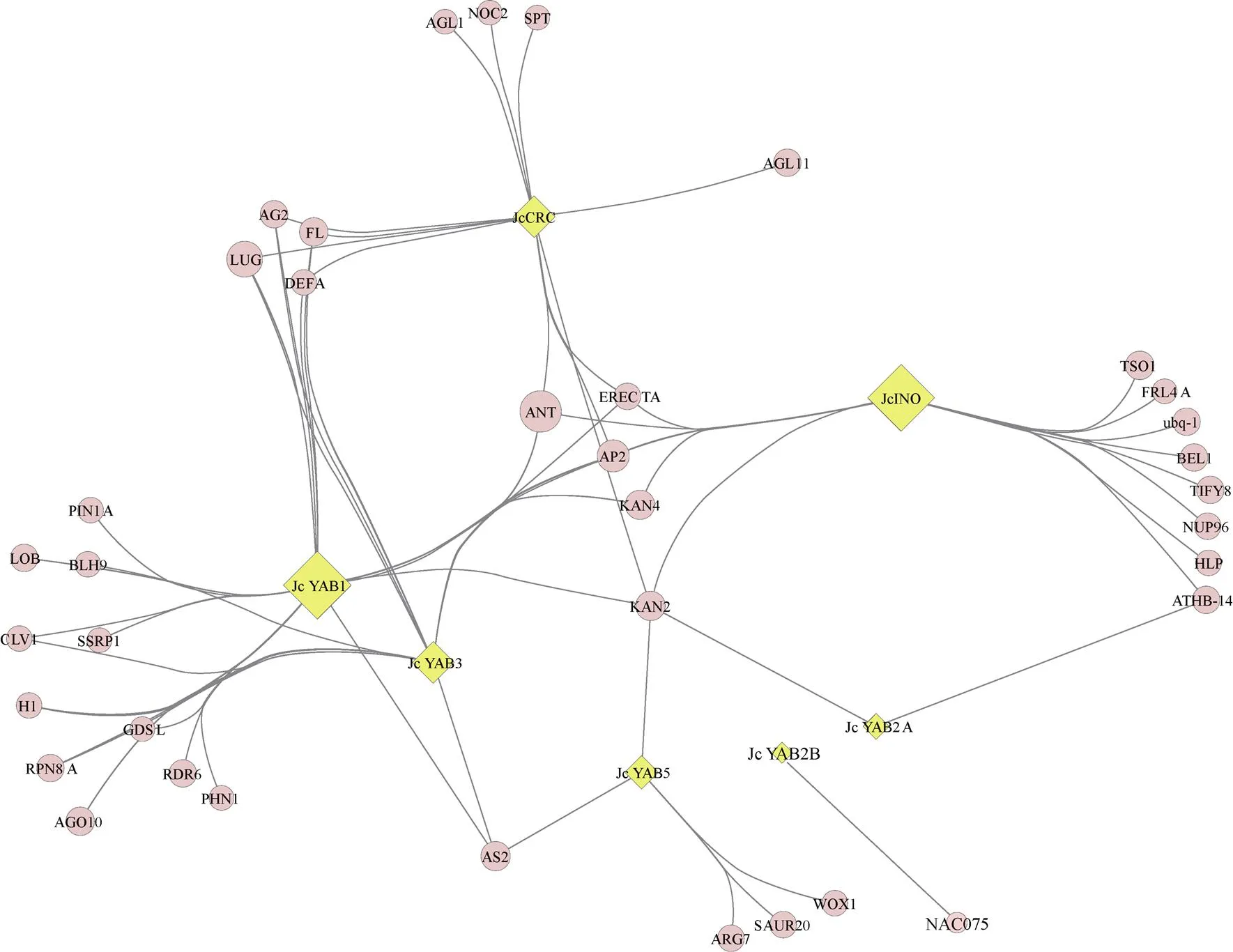
图7 JcYABs蛋白互作网络预测
3 结论和讨论
基因编码一组植物特异性转录因子,主要参与叶片、茎和花器官的分化与发育,目前其功能研究已在拟南芥、番茄、水稻等植物中广泛开展,但在能源植物小桐子中还未见报道。本研究在小桐子基因组中鉴定出7个基因,在5个亚家族中的成员分布与拟南芥基本一致。同一亚家族的JcYABs成员具有相似的结构特征,包括2个保守结构域(锌指域、YABBY结构域)的氨基酸组成、蛋白基序排列、基因结构、外显子数量乃至部分外显子长度都是高度保守的。已有研究表明C-X2-C-X9- P-X11-C-X2-C基序是形成C2C2型锌指结构域的核心元件,在小桐子和棉花的YABBY蛋白中均存在[17]。此外JcYAB蛋白的YABBY结构域在序列保守度高于N-端的锌指域,含有2个位置上相邻、较长的-螺旋结构,这与Siegfried等[33]的报道一致。这表明同一亚家族的成员可能来自一个共同的祖先并具有保守的生物学功能。
基因复制是基因家族扩张的主要推动力,其中片段复制、串联复制和转座事件被认为是植物主要的进化模式[34]。基因共线性分析表明小桐子的FIL/YAB3和YAB2亚家族似乎保留了基因复制产生的旁系同源基因,/基因对较高的Ks值(1.62)显示其复制与分化时间远早于/[35], 可能来自于小桐子基因组经历的古老的全基因复制事件(同源基因对Ks=1.56)[27],结合二者在蛋白基序组成上的差异,我们推测JcYAB2亚家族成员在进化中经历了更为明显的功能分化。此外,系统发育分析表明FIL/YAB3和YAB2具有明显高于其余亚家族的基因数量,主要归因于单子叶植物玉米和水稻中的亚家族扩张[36]。YAB5亚家族仅包含双子叶植物来源的YABBY成员,这与之前的报道[3,17]一致, 而该亚家族是在单子叶植物进化过程中发生丢失还是在单子叶、双子叶植物分化后才出现的,还有待进一步研究。
小桐子是一种生产种子油的重要能源植物,种子产量是其产业发展的重要保障,其种子发育阶段的转录分析可为胚胎发生、种子成熟与品质评价提供重要信息。施用外源性植物生长调节剂(如6-BA)增加雌花和/或两性花的数量是提升小桐子种子产量的可选措施[37]。本研究表明6-BA对JcYAB家族的转录抑制效应在花序分生组织中普遍存在,推测JcYABs可能作为负调节因子参与花器官发育、两性花诱导和雌雄花比例控制[38]。已有研究表明发育后期(授粉29 d后)是小桐子种子贮存物质(三酰甘油为主)合成与积累的关键时期[39],尽管本研究中近一半基因的转录(、和)随种子发育持续下降,但却在发育后期显著上调,可能是通过转录调控网络调节胚胎发生和代谢转换的重要环节。
对开花植物而言,种子是来自胚珠的最重要的生殖器官,Kelley等[40]指出()和基因家族是拟南芥胚珠珠被发育和远轴细胞命运决定的核心调节因子。尽管目前尚无直接证据证实KAN与YABBY家族的协同功能源自蛋白的直接互作[41],但KAN家族编码含GRAP结构域的MYB转录因子,对拟南芥突变株(/)的功能研究表明,其不同成员间(~)存在功能冗余, 可能是基因的上游转录抑制因子[42]。Stahle等[43]以酵母双杂交证实AtFIL、AtYAB3和AtYAB5均可与LEUNIG (LUG)发生互作,后者编码1个富含WD重复的转录抑制因子,可通过抑制花同源异型基因()的转录参与花器官的发育[44]。本研究的蛋白互作预测分析表明KAN可能是小桐子YABBY蛋白互作的中心,KAN4与JcINO、JcYAB1和JcYAB3均可能发生直接互作,而LUG则特异性地与JcYAB1和JcCRC存在互作,显示这3个JcYAB成员在小桐子生殖发育中的潜在功能。
除在植物形态发生和器官发育的保守功能外,近年对棉花、葡萄等植物的研究均证实YABBY家族成员具有显著胁迫响应特异性,Peng等[45]指出3个基因的转录受4 ℃低温诱导,并可与生长素或其他激素的互作参与构树()在低温下的生长发育调节。本研究表明小桐子基因在干旱和高盐胁迫下多为下调表达,却不同程度受低温诱导,尤以的诱导效应最为明显,而其旁系同源基因则呈现低温下的转录抑制,这在菠萝()的家族中同样存在[46]。此外,蛋白互作预测显示虽同为YAB2亚家族成员但仅JcYAB2A可与转录因子KAN2互作,这与我们对该亚家族成员的功能分化预测相符,未来可借助酵母双杂交、双荧光素酶等系统进一步验证YABBY转录因子形成转录抑制复合物调节下游靶基因的分子机制。
可变剪接是增加真核生物转录组和蛋白组多样性的重要转录后基因表达调控机制[47],尽管有关基因可变剪接的研究还未见报道,但本研究综合RNA-seq查找和qRT-PCR验证,表明和均可产生1条新的、受低温显著诱导积累的可变剪接转录本。低温对的转录诱导更为明显,与该基因在低温胁迫下的转录抑制正相反,这与Li等[48]对茶树()低温适应下可变剪接的研究结果一致。我们推测JcYABs可能存在全长和剪接体的功能互补或功能竞争,维持功能型和非功能型转录本间的稳态和平衡[21,49], 调节小桐子的低温抗性。然而,此类“截短的”翻译产物是否产生且活性如何,或转录本是否在翻译初期即被无义介导的mRNA降解(nonsense-mediated mRNA decay, NMD)机制清除,还有待深入探索。
综上所述,尽管YABBYs被报道能够参与植物的多种生长发育和胁迫响应过程,但有关其在植物低温胁迫响应中的功能目前仍知之甚少。本研究在能源植物小桐子基因组中鉴定出分属5个亚家族的7个基因,在基因结构、蛋白组成、进化分析等生物学分析的基础上,通过转录响应、可变剪接模式和蛋白互作预测,揭示了不同成员在生长发育和胁迫响应等方面的潜在功能差异,并初步明确了YAB2亚家族的2个成员JcYAB2A和JcYAB2B的明显功能分化,为今后基因的功能解析和小桐子抗逆性基因资源的挖掘利用提供参考信息。
[1] KANAYA E, NAKAJIMA N, OKADA K. Non-sequence-specific DNA binding by the FILAMENTOUS FLOWER protein fromis reduced by EDTA [J]. J Biol Chem, 2002, 277(14): 11957– 11964. doi: 10.1074/jbc.M108889200.
[2] BOWMAN J L, SMYTH D R., a gene that regulates carpel and nectary development in, encodes a novel protein with zinc finger and helix-loop-helix domains [J]. Development, 1999, 126(11): 2387–2396. doi: 10.1242/dev.126.11.2387.
[3] ROMANOVA M A, MAKSIMOVA A I, PAWLOWSKI K, et al.genes in the development and evolution of land plants [J]. Int J Mol Sci, 2021, 22(8): 4139. doi: 10.3390/ijms22084139.
[4] ZHANG T P, LI C Y, LI D X, et al. Roles of YABBY transcription factors in the modulation of morphogenesis, development, and phyto- hormone and stress responses in plants [J]. J Plant Res, 2020, 133(6): 751–763. doi: 10.1007/s10265-020-01227-7.
[5] ECKARDT N A.genes and the development and origin of seed plant leaves [J]. Plant Cell, 2010, 22(7): 2103. doi: 10.1105/tpc.110. 220710.
[6] GALLAGHER T L, GASSER C S. Independence and interaction of regions of the INNER NO OUTER protein in growth control during ovule development [J]. Plant Physiol, 2008, 147(1): 306–315. doi: 10. 1104/pp.107.114603.
[7] CONG B, BARRERO L S, TANKSLEY S D. Regulatory change in YABBY-like transcription factor led to evolution of extreme fruit size during tomato domestication [J]. Nat Genet, 2008, 40(6): 800–804. doi: 10.1038/ng.144.
[8] BARTLEY G E, ISHIDA B K. Ethylene-sensitive and insensitive regu- lation of transcription factor expression duringtomato sepal ripening [J]. J Exp Bot, 2007, 58(8): 2043–2051. doi: 10.1093/jxb/erm075.
[9] NAGASAWA N, MIYOSHI M, SANO Y, et al.andgenes control floral organ identity in rice [J]. Development, 2003, 130(4): 705–718. doi: 10.1242/dev.00294.
[10] YAMAGUCHI T, NAGASAWA N, KAWASAKI S, et al. Thegeneregulates carpel specification and midrib development in[J]. Plant Cell, 2004, 16(2): 500–509. doi: 10.1105/tpc.018044.
[11] DAI M Q, ZHAO Y, MA Q, et al. The ricegene is involved in the feedback regulation of gibberellin metabolism [J]. Plant Physiol, 2007, 144(1): 121–133. doi: 10.1104/pp.107.096586.
[12] LIN Z W, LI X R, SHANNON L M, et al. Parallel domestication of thegenes in cereals [J]. Nat Genet, 2012, 44(6): 720–724. doi: 10.1038/ng.2281.
[13] TANAKA W, TORIBA T, HIRANO H Y. Three-relatedgenes are required to maintain proper function of the spikelet and branch meristems in rice [J]. New Phytol, 2017, 215(2): 825–839. doi: 10.1111/nph.14617.
[14] ZHAO X Y, XIE H T, CHEN X B, et al. Ectopic expression of, a member ofgene family in wheat, causes partial abaxialization of adaxial epidermises of leaves in[J]. Acta Agron Sin, 2012, 38(11): 2042–2051. [赵翔宇, 谢洪涛, 陈祥彬, 等. 小麦基因的过量表达造成转基因拟南芥叶片近轴面特征趋向远轴面 [J]. 作物学报, 2012, 38(11): 2042–2051. doi: 10.3724/SP.J. 1006.2012.02042.]
[15] ZHAO W, SU H Y, SONG J, et al. Ectopic expression of, a member ofgene family in wheat, causes the partial abaxia- lization of the adaxial epidermises of leaves and arrests the develop- ment of shoot apical meristem in[J]Plant Sci, 2006, 170 (2): 364–371. doi: 10.1016/j.plantsci.2005.09.008.
[16] STRABLE J, VOLLBRECHT E. Maizegenesandregulate floret development and floral meristem determinacy [J]. Development, 2019, 146(6): dev171181. doi: 10.1242/ dev.171181.
[17] YANG Z E, GONG Q, WANG L L, et al. Genome-wide study of YABBY genes in upland cotton and their expression patterns under different stresses [J]Front Genet, 2018, 9: 33. doi: 10.3389/fgene.2018.00033.
[18] ZHAO S P, LU D, YU T F, et al. Genome-wide analysis of the YABBY family in soybean and functional identification ofinvol- vement in high salt and drought stresses [J]. Plant Physiol Biochem, 2017, 119: 132–146. doi: 10.1016/j.plaphy.2017.08.026.
[19] SUN M H, LI H, LI Y B, et al. Tomatocontrols plant height through regulating indole-3-acetic acid-amido synthetase (.) expression [J]. Plant Sci, 2020, 297: 110530. doi: 10.1016/j.plantsci. 2020.110530.
[20] ZHANG S L, WANG L, SUN X M, et al. Genome-wide analysis of the YABBY gene family in Grapevine and functional characterization of[J]. Front Plant Sci, 2019, 10: 1207. doi: 10.3389/fpls.2019. 01207.
[21] REDDY A S N, MARQUEZ Y, KALYNA M, et al. Complexity of the alternative splicing landscape in plants [J]. Plant Cell, 2013, 25(10): 3657–3683. doi: 10.1105/tpc.113.117523.
[22] LALOUM T, MARTÍN G, DUQUE P. Alternative splicing control of abiotic stress responses [J]. Trends Plant Sci, 2018, 23(2): 140–150. doi: 10.1016/j.tplants.2017.09.019.
[23] KAVITHA K R, BEEMKUMAR N, RAJASEKAR R. Experimental investigation of diesel engine performance fuelled with the blends of, ethanol, and diesel [J]. Environ Sci Pollut Res Int, 2019, 26(9): 8633–8639. doi: 10.1007/s11356-019-04288-x.
[24] CONTRAN N, CHESSA L, LUBINO M, et al. State-of-the-art of theproductive chain: From sowing to biodiesel and by- products [J]. Ind Crops Prod, 2013, 42: 202–215. doi: 10.1016/j.ind crop.2012.05.037.
[25] JALALI S, KANCHARLA N, YEPURI V, et al. Exploitation of Hi-C sequencing for improvement of genome assembly andvali- dation of differentially expressing genes inL. [J]. 3 Biotech, 2020, 10(3): 91. doi: 10.1007/s13205-020-2082-0.
[26] ZHANG X, PAN B Z, CHEN M S, et al. JCDB: A comprehensive knowledge base for, an emerging model for woody energy plants [J]. BMC Genomics, 2019, 20(S9): 958. doi: 10.1186/s 12864-019-6356-z.
[27] WU P Z, ZHOU C P, CHENG S F, et al. Integrated genome sequence and linkage map of physic nut (L.), a biodiesel plant [J]. Plant J, 2015, 81(5): 810–821. doi: 10.1111/tpj.12761.
[28] CHEN C J, CHEN H, ZHANG Y, et al. TBtools: An integrative toolkit developed for interactive analyses of big biological data [J]. Mol Plant, 2020, 13(8): 1194–1202. doi: 10.1016/j.molp.2020.06.009.
[29] HURST L D. Thea/s ratio: Diagnosing the form of sequence evolution [J]. Trends Genet, 2002, 18(9): 486–487. doi: 10.1016/S 0168-9525(02)02722-1.
[30] XU W J, ZHAO Y Y, CHEN S S, et al. Evolution and functional divergence of the fructokinase gene family in[J]. Front Plant Sci, 2020, 11: 484. doi: 10.3389/fpls.2020.00484.
[31] WANG S S, ZHAO H C, DENG F F, et al. Isolation and functional characterization of a new ERF transcription factorfrom[J]. Plant Physiol Commun, 2017, 53(8): 1399–1408. [王莎莎, 赵洪春, 邓凤飞, 等. 小桐子中一个新的ERF家族转录因子基因的克隆及功能分析 [J]. 植物生理学报, 2017, 53(8): 1399–1408. doi: 10.13592/j.cnki.ppj.2017.1030.]
[32] SHEN S H, PARK J W, LU Z X, et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data [J]. Proc Natl Acad Sci USA, 2014, 111(51): E5593–E5601. doi: 10.1073/pnas.1419161111.
[33] SIEGFRIED K R, ESHED Y, BAUM S F, et al. Members of thegene family specify abaxial cell fate in[J]. Development, 1999, 126(18): 4117–4128. doi: 10.1242/dev.126.18.4117.
[34] CANNON S B, MITRA A, BAUMGARTEN A, et al. The roles of segmental and tandem gene duplication in the evolution of large gene families in[J]. BMC Plant Biol, 2004, 4: 10. doi: 10.1186/1471-2229-4-10.
[35] LYNCH M, CONERY J S. The evolutionary fate and consequences of duplicate genes [J]. Science, 2000, 290(5494): 1151–1155. doi: 10.1126/ science.290.5494.1151.
[36] TORIBA T, HARADA K, TAKAMURA A, et al. Molecular charac- terization thegene family inand expression analysis of[J]. Mol Genet Genom, 2007, 277(5): 457–468. doi: 10.1007/s00438-006-0202-0.
[37] PAN B Z, XU Z F. Benzyladenine treatment significantly increases the seed yield of the biofuel plant[J]. J Plant Growth Regul, 2011, 30(2): 166–174. doi: 10.1007/s00344-010-9179-3.
[38] PAN B Z, CHEN M S, NI J, et al. Transcriptome of the inflorescence meristems of the biofuel planttreated with cytokinin [J]BMC Genom, 2014, 15(1): 974. doi: 10.1186/1471-2164-15-974.
[39] JIANG H W, WU P Z, ZHANG S, et al. Global analysis of gene expression profiles in developing physic nut (L.) seeds [J]PLoS One, 2012, 7(5): e36522. doi: 10.1371/journal.pone.0036522.
[40] KELLEY D R, SKINNER D J, GASSER C S. Roles of polarity deter- minants in ovule development [J]. Plant J, 2009, 57(6): 1054–1064. doi: 10.1111/j.1365-313X.2008.03752.x.
[41] MANUELA D, XU M L. Patterning a leaf by establishing polarities [J]Front Plant Sci, 2020, 11: 568730. doi: 10.3389/fpls.2020.568730.
[42] ESHED Y, IZHAKI A, BAUM S F, et al. Asymmetric leaf development and blade expansion inare mediated by KANADI and YABBY activities [J]Development, 2004, 131(12): 2997–3006. doi: 10.1242/dev.01186.
[43] STAHLE M I, KUEHLICH J, STARON L, et al. YABBYs and the transcriptional corepressors LEUNIG and LEUNIG_HOMOLOG maintain leaf polarity and meristem activity in[J]Plant Cell, 2009, 21(10): 3105–3118. doi: 10.1105/tpc.109.070458.
[44] CONNER J, LIU Z C., a putative transcriptional corepressor that regulatesexpression during flower development [J]Proc Natl Acad Sci USA, 2000, 97(23): 12902–12907. doi: 10.1073/ pnas.230352397.
[45] PENG X J, WU Q Q, TENG L H, et al. Transcriptional regulation of the paper mulberry under cold stress as revealed by a comprehensive analysis of transcription factors [J]. BMC Plant Biol, 2015, 15: 108. doi: 10.1186/s12870-015-0489-2.
[46] LI Z Y, LI G, CAI M X, et al. Genome-wide analysis of the YABBY transcription factor family in pineapple and functional identification ofinvolvement in salt stress [J]. Int J Mol Sci, 2019, 20(23): 5863. doi: 10.3390/ijms20235863.
[47] MARQUEZ Y, BROWN J W S, SIMPSON C, et al. Transcriptome survey reveals increased complexity of the alternative splicing land- scape in[J]Genome Res, 2012, 22(6): 1184–1195. doi: 10.1101/gr.134106.111.
[48] LI Y Y, MI X Z, ZHAO S Q, et al. Comprehensive profiling of alternative splicing landscape during cold acclimation in tea plant [J]BMC Genom, 2020, 21(1): 65. doi: 10.1186/s12864-020-6491-6.
[49] KALYNA M, SIMPSON C G, SYED N H, et al. Alternative splicing and nonsense-mediated decay modulate expression of important regu- latory genes in[J]Nucl Acids Res, 2012, 40(6): 2454– 2469. doi: 10.1093/nar/gkr932.
Genome-wide Characterization, Expression Profiles and Alternative Splicing Events ofFamily Genes in
ZHANG Zuosheng, GONG Ming, WU Dandan, YANG Yu, LIU Lixiao, WANG Shasha*
(Key Laboratory of Biomass Energy and Environmental Biotechnology of Yunnan Province, Engineering Research Center of Sustainable Development and Utilization of Biomass Energy, Ministry of Education, School of Life Sciences, Yunnan Normal University,Kunming 650500, China)
In order to explore the YABBY transcription factor of, sevengenes from five subfamilies were identified at the genome-wide level based on the newly published genome sequence of. Members of the same subfamily had similar amino acid sequence, gene structure and conserved motifs. Two pairs of paralogs from YAB2 (/) and FIL/YAB3 (/) subfamily showed good collinearity, indicating that segmental duplication/whole genome duplication was the primary cause ofgene family expansion. Purifying selection might be the main impetus during evolution, while candidates of YAB2 subgroup underwent more significant functional divergence. The expression patterns together with protein-protein interaction prediction suggested that JcYAB2B and JcYAB3 might play crucial roles in seed development, meanwhile the transcription of mostwere remarkably repressed under exogenous cytokinin, drought or salinity stress. In addition, RNA sequencing and qRT-PCR analysis confirmed that the paralogous gene pair/exhibited differential cold-responsive transcription in leaves, and their newly identified transcripts were both dynamically accumulated during cold treatment. Therefore, it was speculated thatmight participate in the regulation of low temperature response through functional competition or functional complementarity of spliceosome. These would help to understand the evolution of YABBY family, and elucidate how AS events play a crucial regulatory role in cold response of.
; YABBY family gene; Alternative splicing; Bioinformatics; Gene expression
10.11926/jtsb.4570
2021-11-17
2022-03-11
国家自然科学基金项目(31860062, 31460182)资助
This work was supported by the National Natural Science Foundation of China (Grant No. 31860062, 31460182).
张作胜,男,硕士研究生。E-mail: 3048937351@qq.com
通讯作者Corresponding author.E-mail: sasha_wng@163.com

