黄瓜DIR家族基因的全基因组鉴定及其表达分析
张开京,何帅帅,贾利,胡玉超,杨德坤,陆晓民,张其安,严从生✉
1安徽科技学院农学院,安徽凤阳 233100;2安徽省农业科学院园艺研究所/农业农村部园艺作物种质创制与利用重点实验室(部省共建)/园艺作物种质创制及生理生态安徽省重点实验室,合肥 230031
0 引言
【研究意义】Dirigent(DIR)基因家族广泛存在于维管植物中,是参与木脂素及木质素生物合成的一类关键基因,在植物防御反应、次生代谢和抗病性等方面具有重要的作用[1-2]。黄瓜(Cucumis sativusL.)作为第一个完成基因组测序工作的蔬菜作物[3],在国民经济发展中占据着重要地位。通过在黄瓜全基因组范围内进行DIR基因家族的鉴定,并分析其在不同组织器官和胁迫响应过程中的表达模式,以初步探明DIR基因家族在黄瓜应答生物和非生物胁迫过程中所发挥的相关作用,为后续深入研究DIR基因在黄瓜中的功能奠定重要基础,并为黄瓜抗性分子育种提供参考。【前人研究进展】DIR基因家族蛋白结构均含有一个典型的 dirigent结构域,但少数成员含有两个串联的dirigent结构域[4]。植物DIR家族通常分为DIR-a、DIR-b/d、DIR-c、DIR-e、DIR-f和 DIR-g等 6个亚族[5],研究表明,DIR-a亚族的一些成员直接参与了木脂素和木质素的合成,参与细胞壁修饰,而关于其他DIR亚族的基因功能研究较少[6-7]。DIR蛋白最先在连翘中发现[8],目前,已在许多重要植物中进行了DIR蛋白家族的鉴定研究,如拟南芥[9]、水稻[10-11]、云杉[5]、白梨[12]、杨树[13]、白菜[14]、辣椒[15]、草莓[16]、陆地棉[17]等,并发现DIR基因家族在响应非生物胁迫和生物胁迫中发挥重要作用,如高温[11,18]、冷害[11,14]、干旱[11,14,18-21]、盐害[11,19,21]、重金属[11]、H2O2[18-19]和病虫害[1,14,21-26]等,还受乙烯、JA、SA、ABA等信号分子的调控[14,18,21]。【本研究切入点】利用黄瓜的高质量基因组信息,已经在黄瓜全基因组范围内开展了大量的基因家族鉴定工作,如WRKY[27]、MADS-box[28]、NBS[29]、bZIP[30]等基因家族的鉴定。虽然有个别研究报道了黄瓜DIR家族基因的鉴定[21,31],但其是基于黄瓜ChineseLong_V2版本基因组信息[3]进行的分析,这就导致了黄瓜DIR家族基因的全基因组鉴定不完全,并且之前的研究没有进行黄瓜DIR家族基因在响应生物胁迫和非生物胁迫过程中的表达模式分析,这极大地限制了DIR基因在黄瓜中的生物学功能研究。【拟解决的关键问题】本研究将利用生物信息学手段,在黄瓜 ChineseLong_V3版本基因组[32]中进行全基因组范围内的DIR家族基因鉴定和分析;再基于黄瓜转录组测序大数据,利用黄瓜 ChineseLong_V3版本基因组信息,重新进行转录组分析,完成黄瓜DIR基因家族的组织特异性表达分析以及在胁迫响应过程中的表达模式分析,初步探明DIR家族基因在黄瓜生长发育过程中的生物学功能,为后续黄瓜DIR基因功能的深入研究奠定重要基础,并为黄瓜抗性分子育种提供有利基因。
1 材料与方法
试验于2021年在安徽科技学院进行。
1.1 黄瓜DIR家族基因鉴定与染色体定位
从Pfam数据库(http://pfam.xfam.org/)[33]中下载DIR基因家族的HMM模型文件(PF03018),从葫芦科基因组数据库(http://www.icugi.org/)中下载黄瓜ChineseLong_V3蛋白组序列文件(http://cucurbitgenomics.org/ftp/genome/cucumber/Chinese_lon g/v3/),并构建本地蛋白数据库,利用HMMER软件包[34]的hmmsearch程序从黄瓜蛋白数据库中筛选出可能的DIR基因ID(E<1×10-5),利用Perl脚本提取候选蛋白的序列信息,并利用在线工具Pfam、SMART(http://smart.embl.de/smart/batch.pl)[35]对可能的DIR基因序列进行结构域验证,选择含有Dirigent结构域的序列,最终确定黄瓜中的DIR基因家族成员。根据拟南芥DIR基因家族鉴定研究结果[9],从拟南芥基因组网站(https://www.arabidopsis.org/)中下载拟南芥DIR基因家族成员信息。利用在线工具 ExPASy(https://web.expasy.org/protparam/)分析黄瓜DIR基因家族成员的氨基酸数目、分子量、等电点、不稳定性系数、脂肪系数、亲水性平均值等理化特征。利用在线网站 CELLO(http://cello.life.nctu.edu.tw/)[36]对黄瓜中DIR基因进行亚细胞定位预测。利用TBtools软件[37]绘制黄瓜DIR基因家族在染色体上的分布图。
1.2 黄瓜DIR家族基因特征分析与系统进化分析
利用在线工具GSDS(http://gsds.cbi.pku.edu.cn/)[38]分析黄瓜 DIR基因家族成员的结构。利用在线软件MEME(http://meme-suite.org/)[39]分析黄瓜 DIR 家族蛋白的保守基序(motif),参数设置为:保守基序个数为10,保守基序最佳匹配长度为6—100。利用MEGA 11[40]软件对拟南芥和黄瓜的 DIR家族基因进行系统进化树构建,采用邻接法,参数设置为:Poisson model, pairwise deletion, 1 000 bootstrap replications。利用在线网站 PlantCare(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)[41]分析黄瓜DIR基因家族成员启动子cis元件。
1.3 黄瓜DIR家族成员基因复制类型及共线性分析
利用MCScanX[42]软件分析黄瓜DIR基因家族中的串联重复基因和片段重复基因。为了解黄瓜DIR基因家族与拟南芥、水稻DIR基因家族的种间共线性关系,用MCScanX软件对黄瓜、拟南芥、水稻DIR家族基因进行共线性分析,使用 Circos软件[43]可视化DIR基因家族在种间的共线性关系。
1.4 黄瓜转录组测序数据重新进行RNA-seq分析
从NCBI数据库(https://www.ncbi.nlm.nih.gov/)中检索到黄瓜相关的转录组测序数据,进行下载,然后利用TBtools中的SRA to Fastq插件,将下载的sra数据转换成fastq数据,利用FastQC插件查看数据质量,利用Trimmomatics插件去除接头和低质量序列,清理完原始数据后获得clean data,再利用Hisat2-Build Index插件对黄瓜ChineseLong_V3版本基因组建立索引,利用 Hisat2-Align插件将过滤后的转录组数据比对到黄瓜 ChineseLong_V3版本基因组上,生成 bam文件,接着利用StringTie Quantify插件进行基因表达量分析,最后,基于每个基因的counts数据,利用DESeq2插件完成差异表达基因的分析。
1.5 黄瓜DIR家族基因组织特异性表达分析
在 NCBI数据库(https://www.ncbi.nlm.nih.gov/)中检索黄瓜不同组织间的转录组测序项目PRJNA80169[44],利用黄瓜ChineseLong_V3版本基因组信息,重新进行转录组分析,再利用 TBtools软件绘制黄瓜DIR家族基因在黄瓜不同组织器官中的表达热图。
1.6 黄瓜 DIR家族基因在非生物胁迫和生物胁迫下的表达模式分析
在 NCBI数据库中检索黄瓜在高温胁迫(PRJNA634519)[45]、低温胁迫(PRJNA438923)、盐和硅胁迫(PRJNA477930)[46]、霜霉病(PRJNA285071)[47]、白粉病(PRJNA321023)[48]、根结线虫胁迫(PRJNA419665)[49]处理下的转录组测序数据,基于黄瓜 ChineseLong_V3版本基因组信息,重新进行转录组分析,利用TBtools软件绘制黄瓜DIR家族基因在非生物胁迫和生物胁迫响应过程中的表达热图,并根据不同处理中的差异表达基因结果筛选出每个处理中发生差异表达的黄瓜DIR基因。
2 结果
2.1 黄瓜DIR基因家族成员的基本信息
基于已公布的黄瓜 ChineseLong_V3基因组信息,利用生物信息学方法,在黄瓜全基因组范围内鉴定到23个DIR基因家族成员,基因CDS大小介于237—2 055 bp,编码氨基酸数量在78—684,分子量大小为 8.70—73.82 kD,脂肪系数在 64.37—100.63。23个DIR蛋白的等电点理论值在4.31—10.08。除CsaV3_1G003540.1、CsaV3_1G010290.1、CsaV3_2G015830.1、CsaV3_5G006670.1、CsaV3_7G005610.1、CsaV3_7G005630.1和CsaV3_7G022920.1属于不稳定蛋白外(不稳定性指数大于40),其余DIR基因的不稳定性指数小于40,属于稳定蛋白。在23个DIR基因家族中,14个DIR基因的平均亲水性小于零,表明它们是亲水性蛋白;9个DIR基因的平均亲水性大于零,表明它们是疏水蛋白。通过亚细胞定位预测,发现黄瓜DIR基因主要定位于质膜、细胞外间隙、线粒体和叶绿体中(表1)。
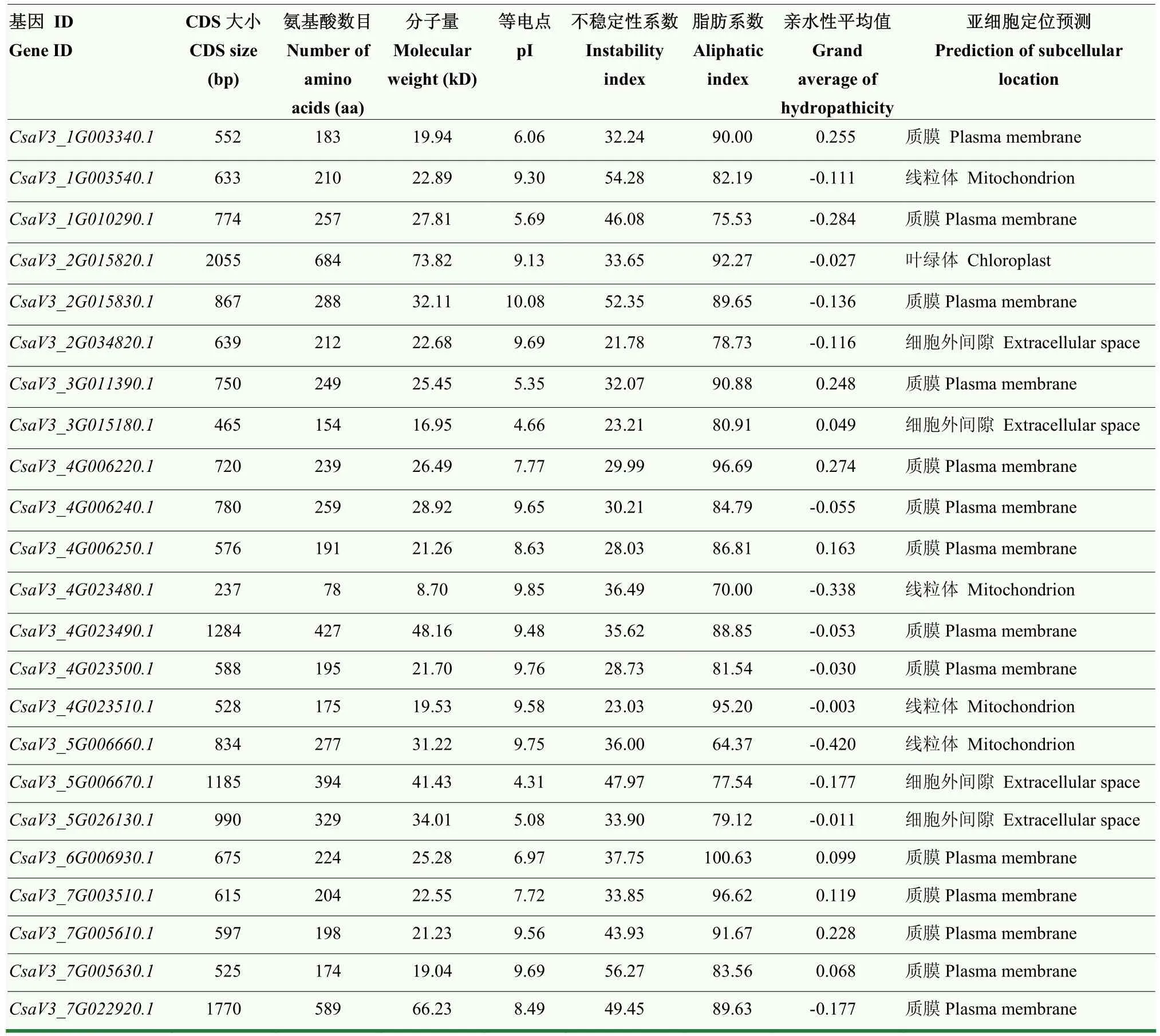
表1 黄瓜23个DIR基因家族成员的理化特征Table 1 The physiochemical characteristics of 23 members in the cucumber DIR gene family
2.2 黄瓜DIR家族基因的染色体定位
根据黄瓜23个DIR家族基因的染色体定位分析,绘制DIR家族基因在黄瓜染色体上的分布图(图1)。黄瓜DIR家族基因在黄瓜的每条染色体上均有分布,其中4号染色体上的DIR基因最多,有7个DIR基因,其中CsaV3_4G006240/CsaV3_4G006250、CsaV3_4G023480/CsaV3_4G023490/CsaV3_4G023500/CsaV3_4G023510为串联重复基因;6号染色体的DIR基因最少,仅有1个DIR基因;3号染色体上有2个DIR基因;7号染色体上有 4个 DIR基因,其中CsaV3_7G003510和2号染色体上的CsaV3_2G034820为片段重复基因;其余染色体上分别各有3个DIR基因,2号染色体上的CsaV3_2G015820/CsaV3_2G015830、5号染色体上的CsaV3_5G006660/CsaV3_5G006670为串联重复基因(图1)。
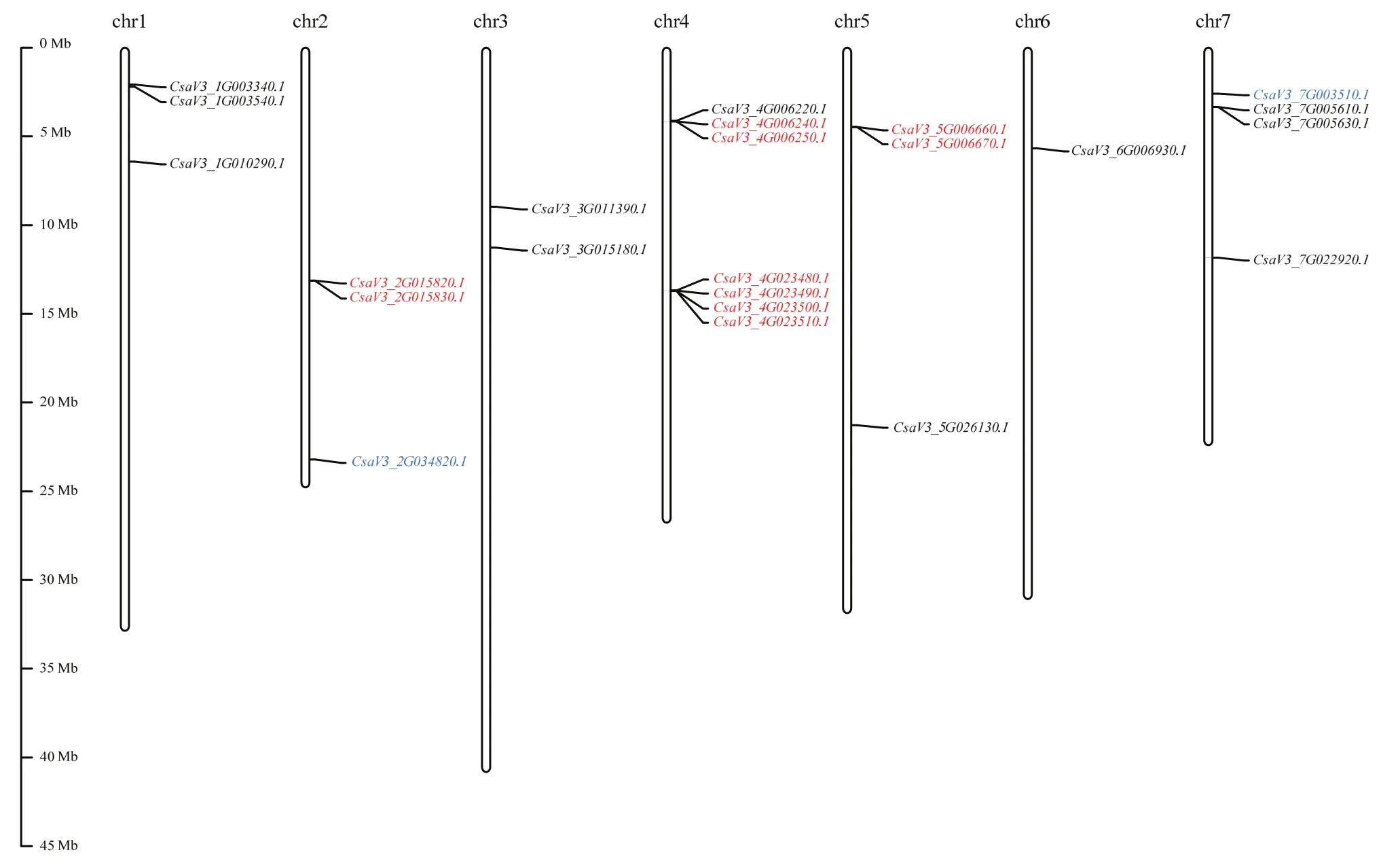
图1 黄瓜DIR家族基因在染色体上的分布Fig.1 The distribution of DIR gene family on cucumber chromosomes
2.3 黄瓜和拟南芥DIR家族基因聚类分析
为充分明确黄瓜DIR家族各基因间亲缘关系与生物学功能,对鉴定到的黄瓜DIR家族基因和模式植物拟南芥的DIR家族基因成员进行多序列比对分析,构建系统进化树(图2)。依据拟南芥DIR家族基因的分类结果,发现进化树共分为3个亚族:a、b/d、e,其中b/d亚族中的DIR基因最多,共有29个;而a亚族中的DIR基因最少,只有10个;e家族中含有11个黄瓜 DIR基因。分析发现黄瓜与拟南芥的 DIR家族基因间存在 3对直系同源基因,即CsaV3_7G022920/AT5G42655、CsaV3_2G015820/AT2G21110和CsaV3_2G015830/AT2G21100,黄瓜DIR基因家族中存在5对旁系同源基因,分别是CsaV3_1G003540/CsaV3_4G023510、CsaV3_2G034820/CsaV3_7G003510、CsaV3_4G006240/CsaV3_4G006250、CsaV3_4G023480/CsaV3_4G023490、CsaV3_7G005610/CsaV3_3G005630,在进化关系上比较相近的一类DIR基因在结构和功能上均比较相似,可以根据模式植物拟南芥中相似基因的研究结果推测黄瓜中DIR基因的生物学功能。
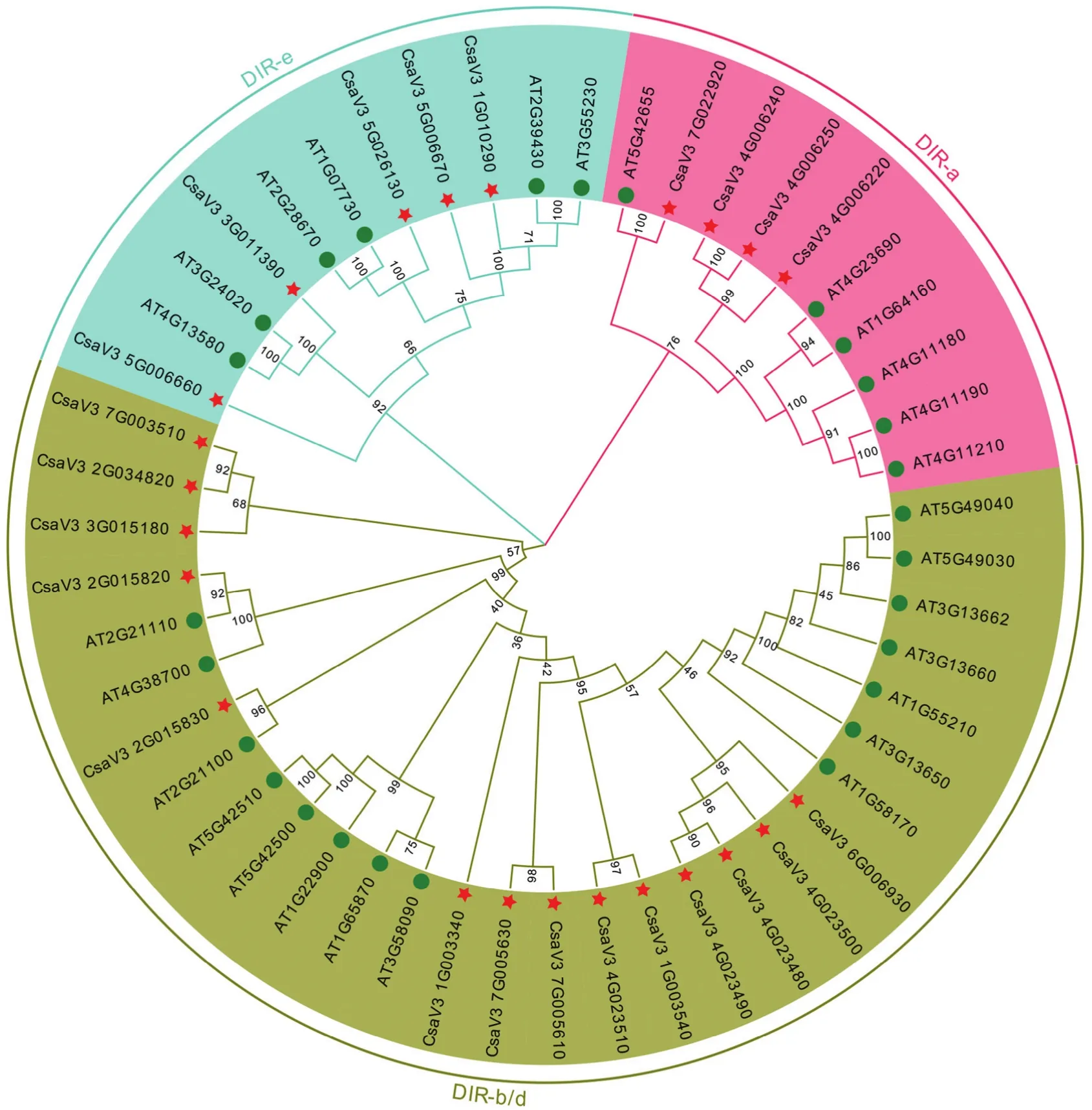
图2 黄瓜、拟南芥DIR家族基因聚类分析Fig.2 Phylogenetic analysis of DIR genes from cucumber and Arabidopsis
2.4 黄瓜DIR家族基因结构与保守序列
利用TBtools软件绘制黄瓜DIR家族基因聚类分析图和结构示意图,发现黄瓜DIR基因家族可以分为3个亚族,即a、b/d、e亚族(图3),与黄瓜、拟南芥 DIR基因聚类结果一致(图 2),其中 b/d亚族中的黄瓜DIR基因最多,有14个;而a亚族中的DIR基因最少,只有4个;e家族中含有5个黄瓜DIR基因。根据黄瓜DIR家族基因的结构示意图,发现a亚族中的黄瓜DIR基因平均外显子和内含子个数最多,分别是3.75和2.75个,而e亚族中的黄瓜DIR基因平均外显子和内含子个数最少,分别是1.4和0.4个,b/d亚族中的黄瓜DIR基因平均具有 2.1个外显子和 1.1个内含子。利用在线软件MEME对黄瓜中DIR蛋白进行保守motif分析,共获得10条motif(表2)。通过黄瓜DIR家族基因的结构示意图,发现不同亚族中的DIR蛋白保守序列不同,而相同亚族中的DIR蛋白具有相同的保守序列,例如在b/d亚族中,大多数基因都含有motif 3、1、4、2,且具有相同的排列顺序,而在 e亚族中大多数基因都含有motif 3、1、2、10,且具有相同的排列顺序,这说明不同亚族中基序分布的不同导致其可能在进化过程中出现了功能多样性演变。同时,在相同亚族中DIR基因相似的保守基序表明其含有相似的功能。

表2 黄瓜DIR蛋白的基序信息Table 2 The motifs information of cucumber DIR proteins

图3 黄瓜DIR家族基因结构与保守序列示意图Fig.3 Exon-intron structures of DIR genes and a schematic diagram of the amino acid motifs of DIR proteins in cucumber
2.5 黄瓜、拟南芥和水稻DIR家族基因共线性分析
对黄瓜与拟南芥、黄瓜与水稻中的DIR家族基因进行共线性分析,发现黄瓜中有 12个 DIR基因(CsaV3_1G003540、CsaV3_1G010290、CsaV3_2G015820、CsaV3_2G015830、CsaV3_3G011390、CsaV3_4G006220、CsaV3_4G023510、CsaV3_5G006670、CsaV3_5G026130、CsaV3_6G006930、CsaV3_7G005610、CsaV3_7G022920)与拟南芥中的19个DIR基因(AT1G22900、AT1G55210、AT1G56180、AT1G58170、AT1G64160、AT1G65870、AT2G21100、AT2G21110、AT2G28670、AT2G39430、AT3G13650、AT3G13662、AT3G24020、AT3G55230、AT3G58090、AT4G13580、AT4G23690、AT5G42500、AT5G49040)存在27种线性关系;黄瓜中有12个DIR 基因(CsaV3_1G003340、CsaV3_1G003540、CsaV3_2G015830、CsaV3_3G011390、CsaV3_4G006220、CsaV3_4G023500、CsaV3_4G023510、CsaV3_5G006670、CsaV3_5G026130、CsaV3_7G005610、CsaV3_7G005630、CsaV3_7G022920)与水稻中的 11个 DIR基因(Os01g06250、Os02g07250、Os03g05030、Os03g17220、Os07g44450、Os07g44930、Os11g07680、Os11g07830、Os11g27620、Os11g42550、Os12g07580)存在19种线性关系。另外还有8个DIR基因(CsaV3_ 2G034820、CsaV3_3G015180、CsaV3_4G006240、CsaV3_4G006250、CsaV3_4G023480、CsaV3_4G023490、CsaV3_5G006660、CsaV3_7G003510)在黄瓜中较保守,既不与拟南芥中的DIR基因存在共线性,也不与水稻中的DIR基因存在共线性。对黄瓜中的DIR家族基因进行共线性分析,发现仅有1对黄瓜DIR家族基因(CsaV3_2G034820/CsaV3_7G003510)存在线性关系(图4)。
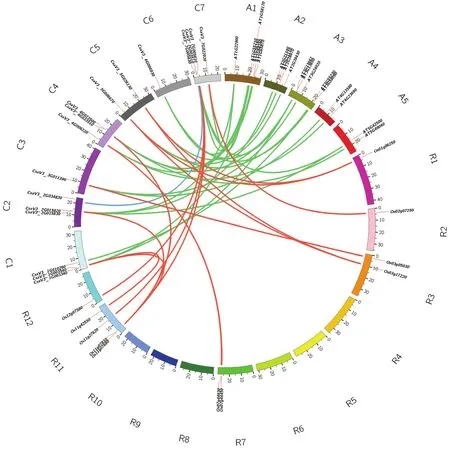
图4 黄瓜、拟南芥、水稻中DIR家族基因的共线性关系Fig.4 Syntenic relationships of DIR gene family in cucumber, Arabidopsis and rice
2.6 黄瓜DIR家族基因启动子序列cis元件
在23个黄瓜DIR家族基因启动子序列中,共鉴定到14种不同的cis元件(图5)。其中,与光响应(lightresponsiveness)相关的cis元件是最多的一类cis元件,包括ACE、Box 4、G-box、I-box等,占总cis元件数目的51%。另外,还鉴定到一些其他cis元件,包括与激素(生长素、赤霉素、水杨酸、脱落酸、MeJA)响应,逆境(干旱、低温)响应,光周期调控,胚乳表达和分生组织表达等相关的cis元件。不同的DIR基因启动子区域存在不同的cis元件成员,表明黄瓜DIR基因在植物生长发育过程中发挥了多种功能。

图5 黄瓜DIR家族基因启动子序列cis元件分析Fig.5 Cis-elements analysis of the promoters of cucumber DIR family genes
2.7 黄瓜DIR家族基因的组织特异性表达
基于已公布的黄瓜不同组织间的转录组测序数据(PRJNA80169)[44],利用黄瓜ChineseLong_V3版本基因组信息,重新进行转录组分析,绘制黄瓜DIR家族基因在不同组织间的表达热图(图 6),发现CsaV3_3G011390、CsaV3_3G015180、CsaV3_1G003540、CsaV3_4G023480、CsaV3_5G006660和CsaV3_1G010290在所有组织中均不表达;CsaV3_2G034820、CsaV3_5G006670、CsaV3_7G003510、CsaV3_4G006220、CsaV3_4G023510、CsaV3_4G006250和CsaV3_4G006240在黄瓜的根中特异性表达,但在其他组织中低表达或者不表达;CsaV3_7G005610、CsaV3_7G005630、CsaV3_7G022920、CsaV3_4G023490、CsaV3_2G015820、CsaV3_5G026130和CsaV3_1G003340在黄瓜的所有组织中均有表达,但表达量较低;CsaV3_4G023500在所有组织中都有表达,且表达量较高,在根、茎、子房和受精子房中表达量最高;CsaV3_6G006930在卷须中高表达,在根中不表达,在其余组织中有表达;CsaV3_6G006930在茎、叶片和卷须中特异性表达,在其余组织中有表达,但表达量较低。

图6 黄瓜DIR家族基因在不同组织器官中的表达热图Fig.6 The expression heatmap of DIR gene family in different tissues of cucumber
2.8 黄瓜DIR家族基因在非生物胁迫处理下的表达模式
基于已公布的黄瓜在高温(PRJNA634519)[45]、低温(PRJNA438923)、盐和硅胁迫(PRJNA477930)[46]处理下的转录组测序数据,利用黄瓜ChineseLong_V3版本基因组信息,进行转录组分析,绘制黄瓜DIR家族基因在不同非生物胁迫响应下的表达热图(图7)。研究发现在高温胁迫处理下,与对照材料相比,仅有CsaV3_4G023490这一个DIR基因发生显著的上调表达,而CsaV3_6G006930、CsaV3_2G015830、CsaV3_4G023500、CsaV3_1G0003340发生显著的下调表达。在低温胁迫处理下,与对照材料相比,发现CsaV3_2G015830和CsaV3_1G003540分别发生显著的下调表达和上调表达,但由于FPKM值小于5,不认为是可靠的差异表达基因,因此,在低温胁迫处理下,仅发现CsaV3_6G006930发生显著的下调表达,CsaV3_4G023490发生上调表达。在盐胁迫处理下,与对照材料相比,发现所有黄瓜DIR基因对盐胁迫处理没有显著的响应调控,但在硅胁迫下,与对照材料相比,CsaV3_4G023490和CsaV3_4G023510发生显著地上调表达。
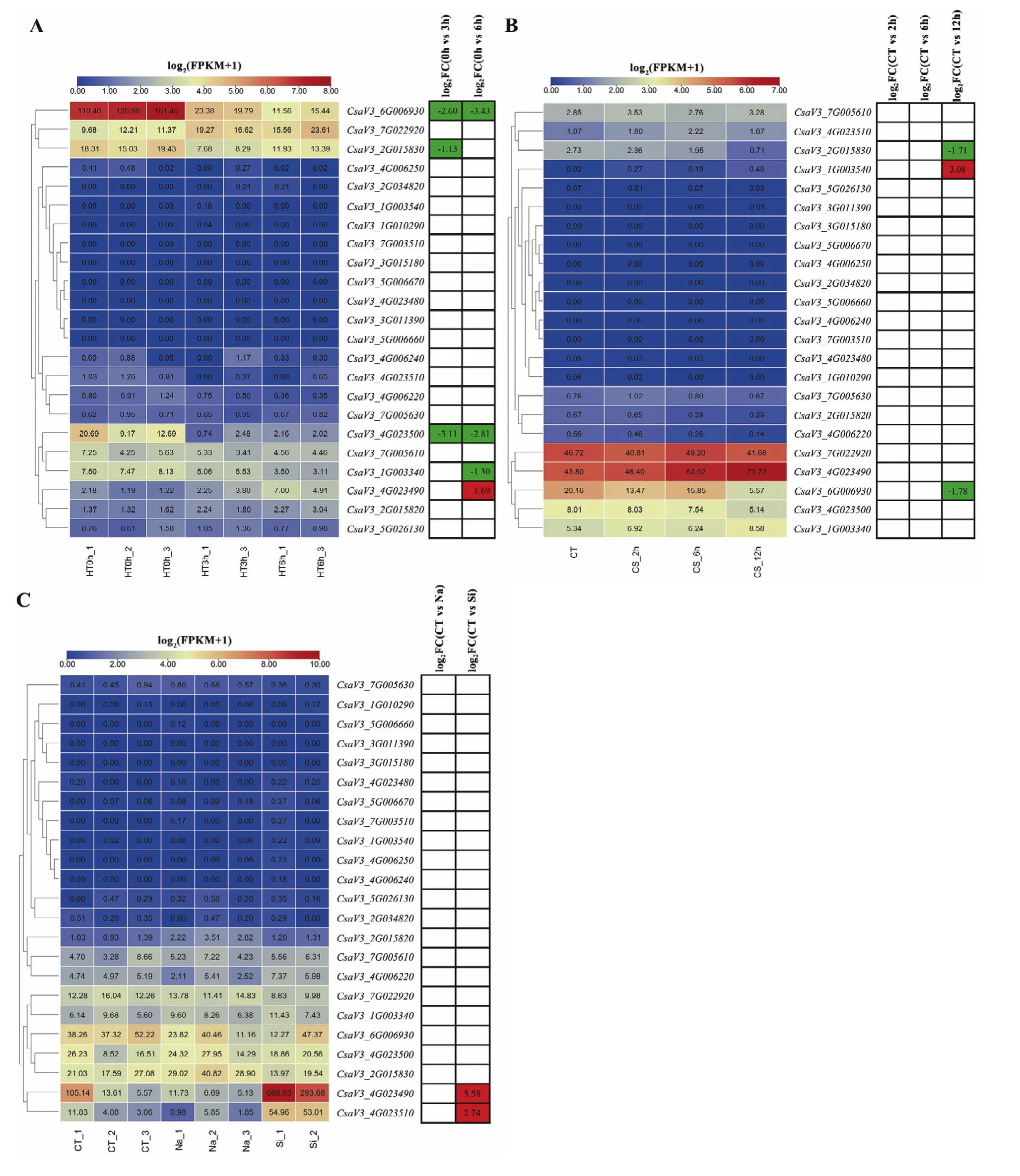
图7 黄瓜DIR家族基因在非生物胁迫处理下的表达热图Fig.7 The expression heatmaps of cucumber DIR gene family under abiotic stress treatments
2.9 黄瓜 DIR家族基因在生物胁迫处理下的表达模式
基于已公布的黄瓜在霜霉病(PRJNA285071)[47]、白粉病(PRJNA321023)[48]、根结线虫胁迫(PRJNA419665)[49]处理下的转录组测序数据,利用黄瓜ChineseLong_V3版本基因组信息,重新进行转录组分析,绘制黄瓜DIR家族基因在生物胁迫响应下的表达热图(图8)。研究发现,在霜霉病胁迫处理下,与对照材料相比,CsaV3_4G023490、CsaV3_2G015820、CsaV3_4G023500、CsaV3_1G003340在抗、感材料中全都发生显著的上调表达,CsaV3_4G006250、CsaV3_4G006240、CsaV3_4G023510仅在感病材料中发生明显的上调表达,而CsaV3_7G005610和CsaV3_2G034820的FPKM值小于5,因此不认为是差异表达基因。在白粉病胁迫处理下,与对照材料相比,仅发现CsaV3_4G023490在抗、感材料中均发生显著地上调表达,而CsaV3_4G023510、CsaV3_2G015830和CsaV3_2G015820的FPKM值小于5,因此不认为是差异表达基因。在根结线虫胁迫处理下,与对照材料相比,CsaV3_6G006930和CsaV3_2G034820在抗、感材料中均发生显著上调表达,CsaV3_4G023510和CsaV3_5G006660在抗、感材料中均发生显著地下调表达;CsaV3_4G023490和CsaV3_4G006240只在感病材料中发生显著上调表达,在抗病材料中也发生上调表达,但不显著,其中CsaV3_4G023490的表达水平随着接种时间的增加而逐渐上调表达,而CsaV3_4G006240的表达水平随着接种时间的增加先增高后下降;CsaV3_4G006250表达水平在感病材料接种1 d后显著上调表达,随后开始缓慢下降,而在抗病材料中是持续地下调表达;CsaV3_7G003510在感病材料中的表达水平随着接种时间增加而逐渐下降,但在抗病材料中的表达水平是先增加后下降;CsaV3_5G006670、CsaV3_5G026130和CsaV3_1G010290在感病材料中发生显著下调表达,在抗病材料中也发生下调表达,但不显著;CsaV3_4G006220在抗病材料中发生显著的下调表达,在感病材料中也发生下调表达,但不显著;CsaV3_1G003340的FPKM值小于5,因此不认为是差异表达基因。

图8 黄瓜DIR家族基因在生物胁迫处理下的表达热图Fig.8 The expression heatmaps of cucumber DIR gene family under biotic stress treatments
2.10 黄瓜DIR家族基因在非生物胁迫和生物胁迫响应下的调控模式分析
通过上述黄瓜DIR家族基因在非生物和生物胁迫下的表达模式分析,将有差异表达的黄瓜DIR基因进行标注,绘制热图(图 9)。发现CsaV3_4G023490在非生物和生物胁迫下均呈现明显上调表达,表明该基因积极参与胁迫响应,可以作为后续研究的重点候选基因;还有一些黄瓜DIR基因,例如CsaV3_4G023500、CsaV3_6G006930、CsaV3_1G003340、CsaV3_4G023510,同时在个别非生物和生物胁迫处理中有差异表达,但表达模式不一样;还有一些黄瓜DIR基因只在非生物胁迫中参与响应表达,例如CsaV3_2G015830只在高温胁迫中下调表达;还有一些黄瓜DIR基因,例如CsaV3_4G006250、CsaV3_7G003510、CsaV3_2G034820、CsaV3_4G006240、CsaV3_5G006670、CsaV3_5G026130、CsaV3_1G010290、CsaV3_2G015820、CsaV3_4G006220、CsaV3_5G006660,只在生物胁迫中参与响应表达。

图9 黄瓜DIR家族基因在非生物和生物胁迫下的表达模式热图Fig.9 The expression patterns heatmap of cucumber DIR gene family under the abiotic and biotic stresses
3 讨论
3.1 黄瓜 DIR基因家族的鉴定与表达分析为后续深入研究提供借鉴
近年来,随着基因组测序技术的不断发展和广泛应用,越来越多的植物基因组序列信息已经公布[50],许多重要的基因家族鉴定工作也已经陆续开展。DIR蛋白几乎存在于所有维管植物中,往往以基因家族的形式出现,DIR基因家族已经在模式植物拟南芥[9]、水稻[10-11]中相继鉴定,随后在一些重要蔬菜作物如白菜[14]、辣椒[15]中也有报道。黄瓜作为在世界范围内广泛栽培的重要蔬菜作物,其栽培面积仅次于番茄和洋葱[51],早在2009年就完成了黄瓜基因组测序工作,获得黄瓜ChineseLong_V2版本基因组信息[3],虽然已有根据黄瓜 ChineseLong_V2版本基因组信息进行DIR家族基因鉴定的报道[21,31],但鉴定的 DIR家族基因数目少于本研究中利用黄瓜ChineseLong_V3版本基因组信息[32]鉴定到的DIR家族基因数目,表明之前研究中的黄瓜DIR家族基因全基因组鉴定不完全,并且之前的研究没有进行黄瓜DIR家族基因在响应生物胁迫和非生物胁迫过程中的表达模式分析,这极大地限制了DIR基因在黄瓜中的生物学功能研究。因此,本研究利用黄瓜ChineseLong_V3版本基因组信息,对黄瓜中DIR家族基因成员进行鉴定,并基于黄瓜ChineseLong_V3版本基因组信息和转录组测序大数据,重新进行转录组分析,探究黄瓜DIR家族基因在不同组织器官和不同胁迫响应过程中的表达模式,可为黄瓜 DIR基因的功能深入研究提供参考,并为黄瓜抗性分子育种提供理论依据。
3.2 DIR基因家族在不同物种间具有保守性
不同植物中DIR家族基因的数目变化很大,本研究共鉴定到23个黄瓜DIR家族基因成员,而拟南芥[9]、水稻[10-11]、云杉[5]、白梨[12]、杨树[13]、白菜[14]、辣椒[15]、草莓[16]、陆地棉[17]中鉴定的DIR基因数目分别是25个、61个、35个、35个、40个、29个、24个、33个和12个。系统进化树分析将黄瓜中的23个DIR基因分为3个亚族,即a、b/d、e,这与拟南芥[9]、杨树[13]、辣椒[15]和陆地棉[17]中DIR家族基因的系统进化分析结果类似,也与之前利用黄瓜 ChineseLong_V2版本基因组信息进行DIR家族基因分析的聚类结果一致[21],3个亚族成员间的基因结构存在明显差异,每个亚族中的基因结构和 motif基本一致。通过对黄瓜中的DIR家族基因进行重复基因分析,发现黄瓜DIR基因家族有5对串联重复基因对,仅有1对片段重复基因对,可见黄瓜中DIR基因的扩张主要来自于串联重复。通过对拟南芥、水稻和黄瓜中DIR家族基因的共线性分析,发现黄瓜中有8个DIR基因比较保守,其与拟南芥和水稻中的DIR基因都不存在共线性,而另外15个黄瓜DIR基因与拟南芥和水稻中的DIR基因存在多种共线性关系,表明各物种以特异的方式进行了基因扩张,这种现象也普遍出现在其他植物基因家族研究中[52-53]。
3.3 CsaV3_4G023490积极参与黄瓜对生物和非生物胁迫响应的调控
由于近年来高通量测序技术的发展,转录组测序成本逐渐下降[54],有很多研究者进行了大量的黄瓜转录组测序,形成了黄瓜转录组测序大数据。因此,利用好这些转录组测序大数据,不仅能降低研究成本,还能深入挖掘这些数据,并且可以结合不同处理的黄瓜数据来研究黄瓜不同基因家族的分子生物学功能。本研究发现23个黄瓜DIR家族基因中有些基因在所有组织中低表达甚至不表达,有些基因在所有组织中均有表达,有些基因只在部分组织中特异表达,正是由于这些DIR家族基因在不同组织中的特异表达,协同调控了黄瓜的生长发育。通过分析胁迫响应过程中黄瓜 DIR家族基因表达模式,发现黄瓜CsaV3_4G023490在生物和非生物胁迫中均发生上调表达,表明该基因积极响应黄瓜胁迫过程中的抗性调控,这与前人报道的CsDIR16(黄瓜ChineseLong_V2版本基因组信息中命名为Csa4G280630)功能一致,在响应霜霉威(杀菌剂 Propamocarb)、JA、ABA、PEG、NaCl、黄瓜褐斑病胁迫过程中均呈现上调表达[21],可见该基因在抵御生物和非生物胁迫过程中积极响应,可进行后续深入研究。另外,本研究还发现黄瓜DIR家族基因在生物胁迫和非生物胁迫中的表达模式也不一样,在非生物胁迫响应过程中,发生差异表达的DIR家族基因数目较少,除了CsaV3_4G023490上调表达,其余大部分基因下调表达;而在生物胁迫响应过程中,发生差异表达的 DIR家族基因数目较多,且大多数差异表达的 DIR基因上调表达,这表明相对于非生物胁迫,在病原菌侵染黄瓜后,较多的DIR家族基因会协同上调表达,以积累更多的木质素,从而抵御病原菌的侵染。这证实了植株在病原菌侵染过程中,侵染部位或侵染点周围的植物细胞会积累木质素,从而达到抵抗病菌侵染和扩展的目的[55]。很多研究已经证明 DIR基因在植株与病原菌互作过程中的重要性[56-59]。此外,还有一些黄瓜DIR基因在个别的非生物胁迫中也发挥着重要的作用,特别是在低温、干旱、激素信号等胁迫下,这与启动子中的cis元件类型相符,其他作物中的DIR家族基因也具有非生物胁迫响应调控功能[11,14,18-21],但黄瓜中大多数非生物胁迫响应调控基因为下调表达,这也有待进一步探讨。
4 结论
在黄瓜全基因组范围内共鉴定到23个DIR家族基因,分为3个亚族。黄瓜DIR家族基因在不同组织器官和胁迫响应下的表达模式具有差异性,协同调控了黄瓜的生长发育,其中CsaV3_4G023490在非生物和生物胁迫下均呈现出明显的上调表达,表明该基因积极参与逆境胁迫响应调控。

