日本沼虾体重和出肉率联合GWAS分析
刘 帅,蒋 丽
(1.上海海洋大学水产科学国家级实验教学示范中心,上海 201306;2.中国水产科学研究院农业农村部水生动物基因组学重点实验室,北京 100141)
日本沼虾(Macrobrachiumnipponense)俗称青虾、河虾,属于节肢动物门(Arthropoda)甲壳纲(Crustacea)十足目(Decapoda)长臂虾科(Palaemonidae)沼虾属(Macrobrachium),广泛分布于我国及东南亚大部分地区,由于其肉质鲜美,营养丰富,是我国重要的淡水养殖虾种之一。雌雄差异及群体均匀度低是制约集约化养殖产量的主要因素,因而,从基因组水平对其体重相关的基因进行解析,进而培育具有较高均匀度且增重快的新品种对其养殖业发展具有战略意义。
全基因组关联分析(genome-wide association study,GWAS)是现代分子育种的重要辅助手段,通过单核苷酸多态性(single nucleotide polymorphism,SNP)与目标性状之间的关联检验,实现相关基因的定位,在水产动物育种中发挥着重要作用。经典的GWAS只针对单一性状进行分析,但当不同性状间存在遗传相关时,考虑性状间协方差的多性状分析相较于单性状分析具有更高的检验功效[1]和参数估计的精度[2]。已有研究证明,多性状联合 GWAS分析对显著遗传变异的检测能力更强[3],且更具生物学意义[4]。基于此,笔者对日本沼虾体重和出肉率性状进行联合分析,实现对相关基因的定位和筛选,以期为日本沼虾生长增重的基因表达解析提供理论支撑,进而促进其养殖业发展。
1 材料与方法
1.1 试验群体及表型测量试验用虾养殖于中国水产科学研究院淡水渔业研究中心宜兴大浦基地,收集日龄52 d的成熟虾,随机挑选200尾健康虾(雌雄各100尾)编号,记录体重,收集虾尾部的肌肉并称重,尾部肌肉与体重的比值为出肉率。此外,收集的肌肉需转入冻存管中于-80 ℃保存,以便进行DNA提取。
1.2 DNA提取、测序及基因型数据的获取以收集的肌肉组织为材料,根据酚-氯仿法[5]提取DNA,使用1%琼脂糖凝胶评估提取DNA质量,并将其浓度调整至2.5 μg/μL,最终在北京华大基因完成序列测定。原始测序数据经工具包NGS QC Toolkit[6]质控过滤后,通过软件BWA[7]与日本沼虾参考基因组(GCA_015104395.1)进行对比分析,通过软件Picard(https://github.com/broadinstitute/picard)去除对比结果中的重复序列,最终由软件GATK(https://github.com/broadinstitute/gatk)实现SNP位点的筛选,从而完成基因分型。获得的分型数据需要通过PLINK[8]软件进一步质控和过滤,去除样本分型成功率≤90%,最小等位基因频率≤5%的位点并生成二进制基因型数据。
1.3 全基因组关联分析分别对体重、出肉率性状进行单性状的全基因组关联分析,并对2个性状进行多性状联合分析,其中单性状分析使用GEMMA[9]软件中的单性状混合模型分析,多性状联合分析采用GEMMA软件中的多性状线性混合模型。以Bonferroni校正阈值[10]为界限筛选与性状显著相关的SNP位点,并通过R语言绘制曼哈顿和Q-Q图。
1.4 基因注释根据检验显著SNP位点于参考基因组上的物理位置,提取其上下游区域250 kb范围内的序列,通过软件KOBAS(http://kobas.cbi.pku.edu.cn/kobas3)调整参数“-e 1e-5”实现对提取序列的注释,根据NCBI数据库及文献检索实现注释的功能注释。
2 结果与分析
2.1 表型及基因型描述统计剔除体重、出肉率表型数据中的缺失项后共获得181尾虾的表型数据(表1),性状间的相关关系及分布见图1~3。基于181尾虾分型获得的基因型数据中剔除分型成功率≤90%及最小等位基因频率≤5%的位点后,共获得7 793个高质量SNP位点。

表1 表型描述性统计Table 1 Descriptive statistics of phenotype
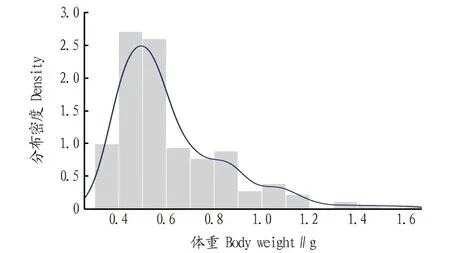
图1 日本沼虾体重性状分布Fig.1 Distribution in traits of body weight for Macrobrachium nipponense
2.2 全基因组关联分析对体重、出肉率进行单一性状的全基因组关联分析及两性状的联合分析,结果如图4所示。观察图4 d~图4f,根据统计检验概率P值拟合的实际曲线均与理论曲线有较高的一致性,且基因组控制值(λ)位于1.00附近[11],说明关联检验的统计准确性较高,进而可说明曼哈顿图中检测到的SNP具有较高的可信度(图4a~c),水平线表示Bonferroni校正阈值(0.05/7793,6.42e-06)分布于水平线上方的位点具有与性状的显著相关性。由图4a可知,在体重单性状关联分析中,仅检测到1个SNP位点与之相关;在出肉率的单性状关联分析中(图4b),并未检查测到显著SNP位点;在两性状的联合分析中(图4c),共检测到6个SNP位点,且体重单性状分析检测到的SNP位点也被包含在内,这证明多性状联合分析对SNP检测的效率具有显著提升作用。
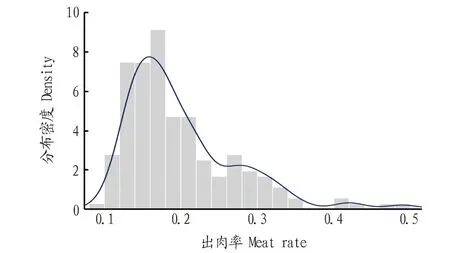
图2 日本沼虾出肉率性状分布Fig.2 Distribution in traits of meat rate for Macrobrachium nipponense
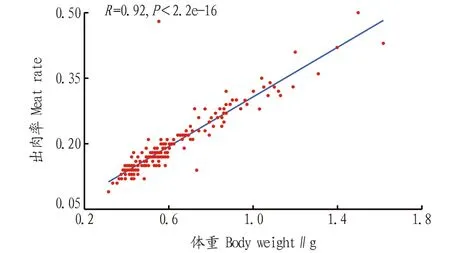
图3 日本沼虾体重和出肉率性状相关关系Fig.3 The correlation between trait of body weight and meat rate in Macrobrachium nipponense
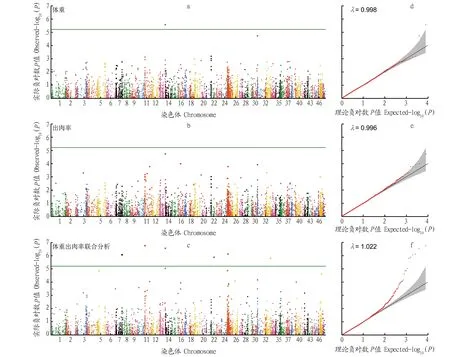
图4 日本沼虾体重、出肉率单一分析及联合分析的曼哈顿和Q-Q图Fig.4 The manhattan and Q-Q plot of single and combine analysis for body weight,meat rate in Macrobrachium nipponense
2.3 候选基因根据日本沼虾体重和出肉率联合分析获得的显著SNP 位点为探针,提取其所在日本沼虾全基因组位置上下游250 bp区域内的序列,通过数据库kobas完成候选基因的在线注释与鉴定,共鉴定出5个候选基因(表 2),分别为Jrkl(JRK like)、Tep2(Thioester-containing protein 2)、ZNF84(zinc finger protein 84)、CG12299和swm(second mitotic wave missing)。通过KEGG通路注释分析,这些候选基因主要参与基因表达调控、转录调控、细胞信号转导、蛋白酶解、内肽酶分析及先天性免疫系统发育等生物学过程,在机体分化发育方面发挥着重要作用。

表2 日本沼虾体重和出肉率两性状联合分析的显著位点与基因Table 2 Significant loci and genes of joint analysis for body weight and meat yield in Macrobrachium nipponense
3 讨论
随着测序成本的下降及多种水产动物基因组全测序的完成,GWAS技术逐渐应用于水产动物的基因定位及育种领域。吴碧银等[12]通过GWAS分析技术发现了影响低氧环境下鲤耐受性的23个候选基因。陈小明等[13]利用GWAS检测到26个与大黄鱼耐高温相关的基因。徐鸿飞等[14]对红罗非鱼低温体色变异现象进行GWAS分析,发现了19个与之相关的功能基因。
尽管GWAS在水产动物已有诸多应用,但大多数都是针对单一表型的关联分析,而水产动物的表型如体重、体长、性别之间存在显著相关性,因而对表型进行单一性状关联分析时,通常会忽略某个SNP位点对多个性状的共同影响,致使检验SNP位点的个数变少,筛选到的候选基因精度降低。相较于单一性状关联分析,多个性状联合分析方法在显著SNP的定位上表现出更高的检测能力和精确度。通过对比日本沼虾体重和出肉率两性状 GWAS 与两性状分别进行单一性状 GWAS 的结果发现,在相同的显著水准下,前者检测到 6个显著SNP 远多于后两者共检测到的1个与体重相关的SNP。基于联合分析检测到的6个显著SNP位点筛选到的5个功能基因中,Jrkl为功能未知的新基因;Tep2编码含硫蛋白2,参与代谢、细胞程序性死亡及免疫应激等有关生物学活动的调控[15];ZNF84和CG12299[16]分别编码锌指蛋白84和锌指蛋白135,作为锌指蛋白家族的成员,可通过其锌指结构域与转录因子结合进而调控基因表达[17],参与细胞增殖、分化和凋亡等多种生理过程,维持组织稳态;swm编码的蛋白是Hh信号的负调节因子[18],对细胞极性至关重要。该研究定位到的候选基因都与机体发育相关,可能在日本沼虾体重和出肉率性状的表现上有重要影响,因而可对日本沼虾体重和出肉率的解析提供理论支撑。

