陆地棉动态株高与SSR标记的关联分析
文 佳,黄陈珏,嵇子涵,李黎贝,冯 震,喻树迅
(浙江农林大学现代农学院,浙江临安 311300)
0 引 言
【研究意义】棉花是重要的经济作物之一[1-3]。棉花株高与棉花产量、抗倒伏和机械化等密切相关[4]。株高过高会增加作物倒伏的风险,过低则影响生物量,降低光合产物的积累。因此,培育合适株高的棉花品种对棉花高产稳产具有重要意义。【前人研究进展】Wang等[5]较早利用重组自交系群体,对株高等性状进行数量性状位点(quantitative trait locus,QTL)定位,获得了3个与株高性状相关的QTL位点。王新坤等[6]以矮秆突变体为亲本,构建分离群体,采用复合区间作图法进行QTL定位,共检测到4个与株高相关的QTL,其中qPH-LG6-1表型贡献率最大,为47.98%,显著大于前人所得株高QTL的贡献率。何蕊等[7]通过对408个单株进行SSR鉴定,发现了16个与株高性状相关的QTL,其中有7个在2个及以上环境中被检测到,这些在多环境检测到的QTL表现出很好的稳定性。Shang等[8]以重组自交系以及后代作为材料对两个环境的棉花株高以及多个发育阶段的杂种优势进行了动态分析,在2个环境中分别获得41和23个株高相关的QTL位点,可以更综合、更全面地了解控制该性状位点的动态表达。Ma等[9]探索了不同发育阶段下棉花株高相关的动态QTL,通过部分显性和超显性及上位效应解释了陆地棉株高的遗传基础。全基因组关联分析(genome-wide association study,GWAS)是复杂数量性状研究的主要方法,能以较高的分辨率和灵敏度识别出目标性状关联的候选基因[10-11]。相比于常规QTL,GWAS具有无需构建作图群体、可以同时对多个等位基因进行分析、利用群体长期进化中的重组信息使定位分辨率更高等优点。Su等[12]对355份陆地棉组成的自然群体在6个环境下的株高性状进行了调查,通过GWAS得到8个与株高性状显著相关的SNP位点,最终定位到与株高相关的候选基因Gh_D03G0922。Ji等[13]发现在棉花矮化突变体AS98中HDPA-D12位点拷贝变异编码了一个反应元件结合蛋白转录因子GhDREB1B,研究表明GhDREB1B过表达会使棉花株高显著降低。【本研究切入点】植物株高是一个动态发育性状,受多个主效基因和微效基因控制[14]。虽然前人通过GWAS方法开展了棉花株高的遗传研究已有较多报道,但关于研究棉花动态株高方面的报道较少[10-11,15]。需利用棉花自然群体对动态株高性状进行关联分析。【拟解决的关键问题】以185份陆地棉品种组成自然群体,对PH-ST1和PH-ST2进行表型鉴定,结合137对SSR多态性引物扩增出的355个等位变异,利用GLM和MLM模型对开花期和吐絮期的株高及标记进行关联分析,发掘不同时期与株高显著相关的SSR标记,为培育棉花株型新品种提供理论基础。
1 材料与方法
1.1 材 料
选取185份陆地棉品种(系),其中170份为中国品种(分属于我国长江流域棉区、黄河流域棉区、西北内陆棉区和北部特早熟棉区);15份材料来自于美国、阿塞拜疆等国家。
1.2 方 法
1.2.1 试验设计
分别在中国海南三亚(18°29′N,109°52′E)(2020年和2021年)种植2个重复;中国湖北黄冈(30°57′N ,114°92′E)(2021年)、中国山东临清(36°48′N ,115°41′E)(2021年)种植3个重复,每重复选取9株长势一致的植株测量株高。田间试验均采取随机区组试验设计,单行根据当地栽培方式种植。中国海南三亚:行长3 m, 行距为0.5 m,株距0.2 m ;中国湖北黄冈:行长6 m,行距为0.8 m, 株距 0.4 m ;中国山东临清:行长5 m,行距为0.6 m ,株距0.3 m 。
1.2.2 SSR标记及基因型分型
采用改良CTAB法[16]提取棉花叶片基因组,所用试剂从南京诺唯赞生物科技有限公司购买。对185份陆地棉品种(系)自然群体进行基因型检测,检测到355个等位变异[17]。137对SSR引物由实验室选取,均匀覆盖陆地棉26条染色体上[17],所有SSR引物由生工生物工程(上海)股份有限公司合成。
1.2.3 关联分析
群体结构分析参照文献[19]的数据,用矩阵“Q”作为协变量来降低假阳性率。采用Tassel 3.0软件计算亲缘关系矩阵(K),通过一般线性模型(general linear model,Q)和混合线性模型(mixed linear model,Q+K)2种方法分别对5个环境下不同时期株高的表型数据与SSR标记进行关联分析。参考Bresghellohe等[18]提出的无效等位变异(null allele)计算方法,分析棉花株高表型效应值。参照Evanno等[19]方法,计算ΔK来确定适宜的亚群数。
1.3 数据处理
使用软件R对开花期和吐絮期株高数据进行描述性统计分析,包括平均值、极大值、极小值、标准差和变异系数。参照Knapp等[20]方法计算广义遗传力(h2)。采用R语言中lme4包[21]计算出多环境下不同时期株高的最佳线性无偏预测(best linear unbiased prediction,BLUP)分析4个环境下的表型数据,获得2个时期BLUP值作为第5个环境下的陆地棉表型数据。
2 结果与分析
2.1 棉花动态株高表型差异
研究表明,4个环境中,PH-ST1平均值分别为58.45、46.93、60.02和59.92 cm,标准差分别为8.72 、5.84 、7.95 和7.49 ,变异系数介于12.45%~14.56%;PH-ST2平均值分别为66.21、98.05、71.66和88.30 cm,标准差分别为11.14、18.07 、10.61 和15.78 ,变异系数介于14.81%~18.43%。不同时期株高的基因型、基因型与环境互作变异方差均达到极显著水平;PH-ST1广义遗传率为63.67%,PH-ST2广义遗传率为85.66%。PH-ST1和PH-ST2的遗传力稳定且比较高,主要受基因型的影响,适合关联分析。表1
2.2 群体结构差异
研究表明,K=2时,ΔK出现最大值,即185份材料从遗传结构上可被分为2类亚群。其中,一个亚群包含了95份材料,约占总材料的51%;另一个亚群包含了90份材料,约占总材料的49%。在Structure软件中,当K=2时,获得了每个材料的Q值,将得到的Q矩阵作为关联分析的协变量,消除群体结构对关联分析结果的影响。图1
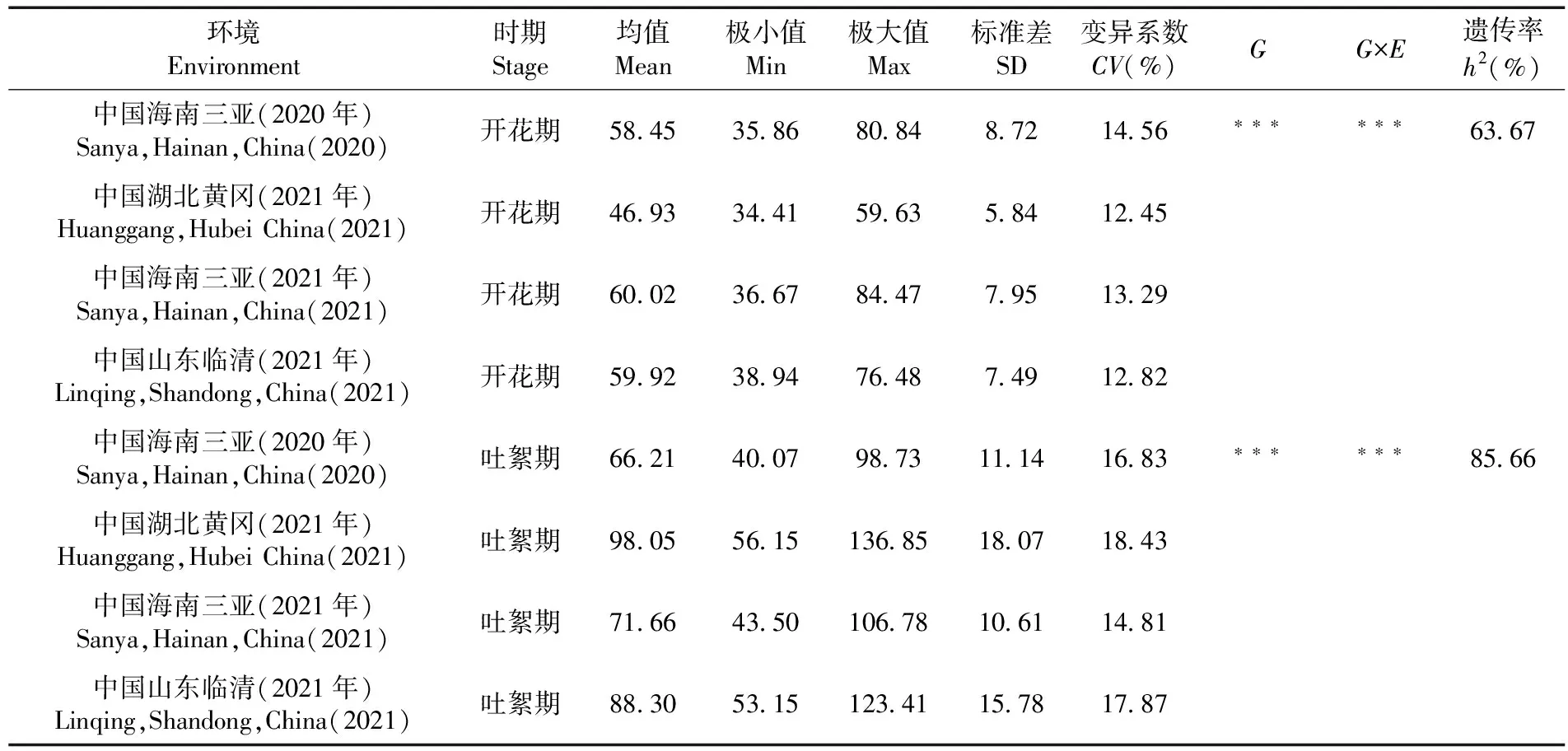
表1 185份陆地棉材料在不同环境下株高性状
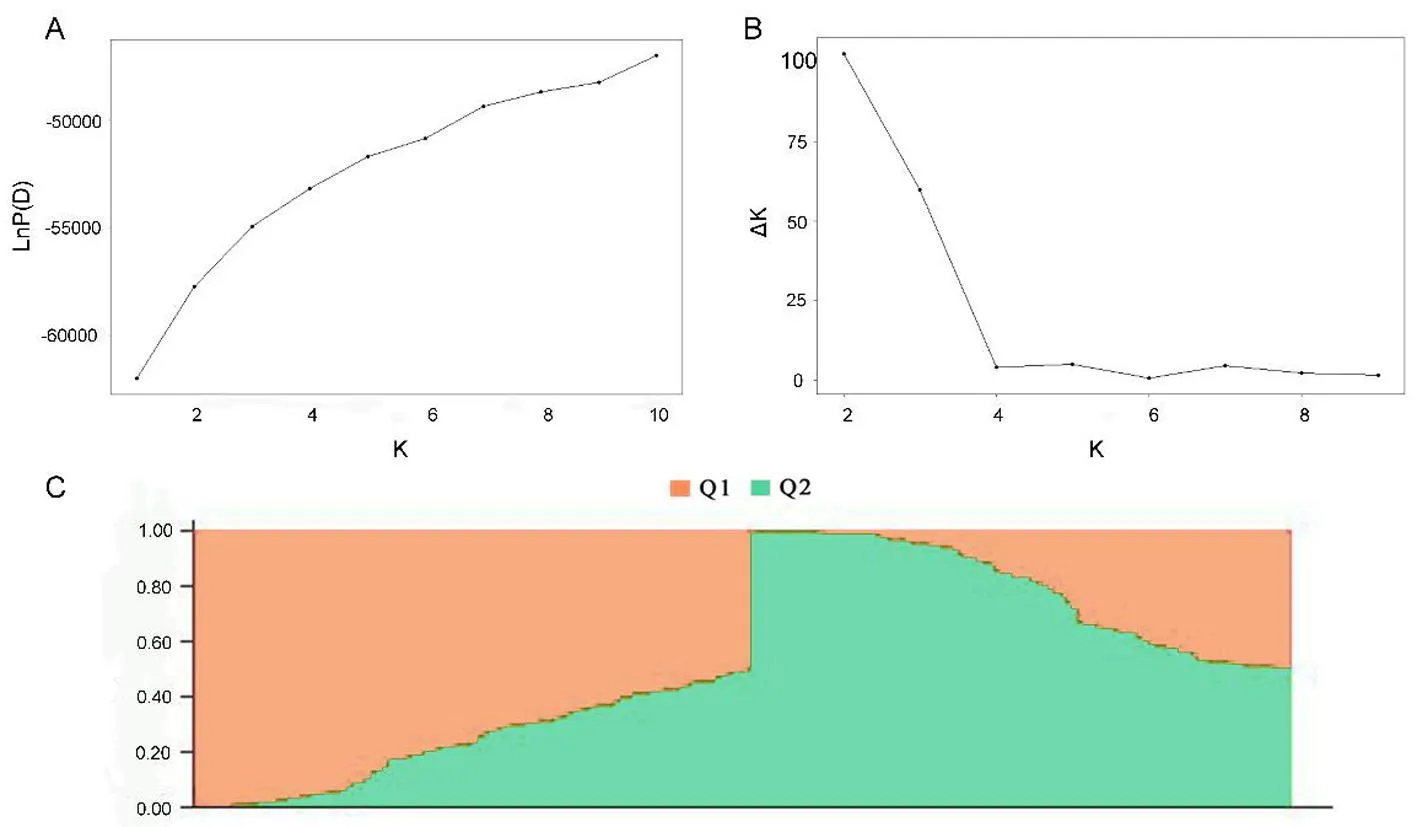
图1 185份陆地棉材料的群体结构Fig.1 The population structure of 185 upland cotton accessions
2.3 基于GLM模型下动态株高的关联性
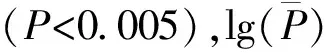
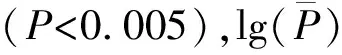
2.4 基于MLM模型下动态株高的关联性
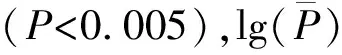
表2 基于GLM模型在3个及以上环境的PH-ST1关联性
2.5 显著位点表型贡献率及效应
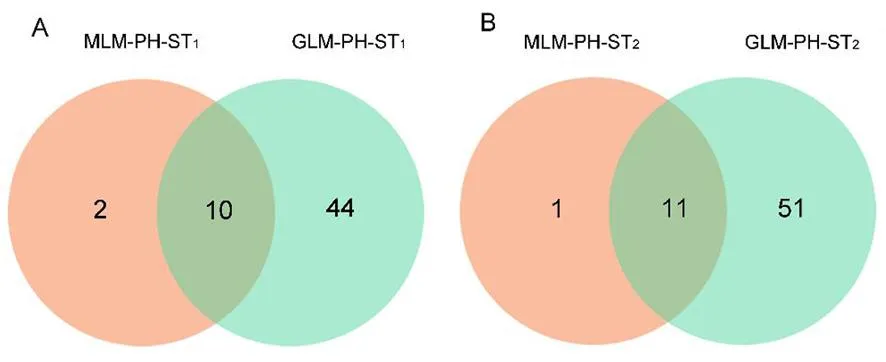
图2 在GLM和MLM模型下不同时期株高的SSR位点韦恩图Fig.2 Venn diagrams of SSR locus associated with plant height at different stages based on GLM and MLM model
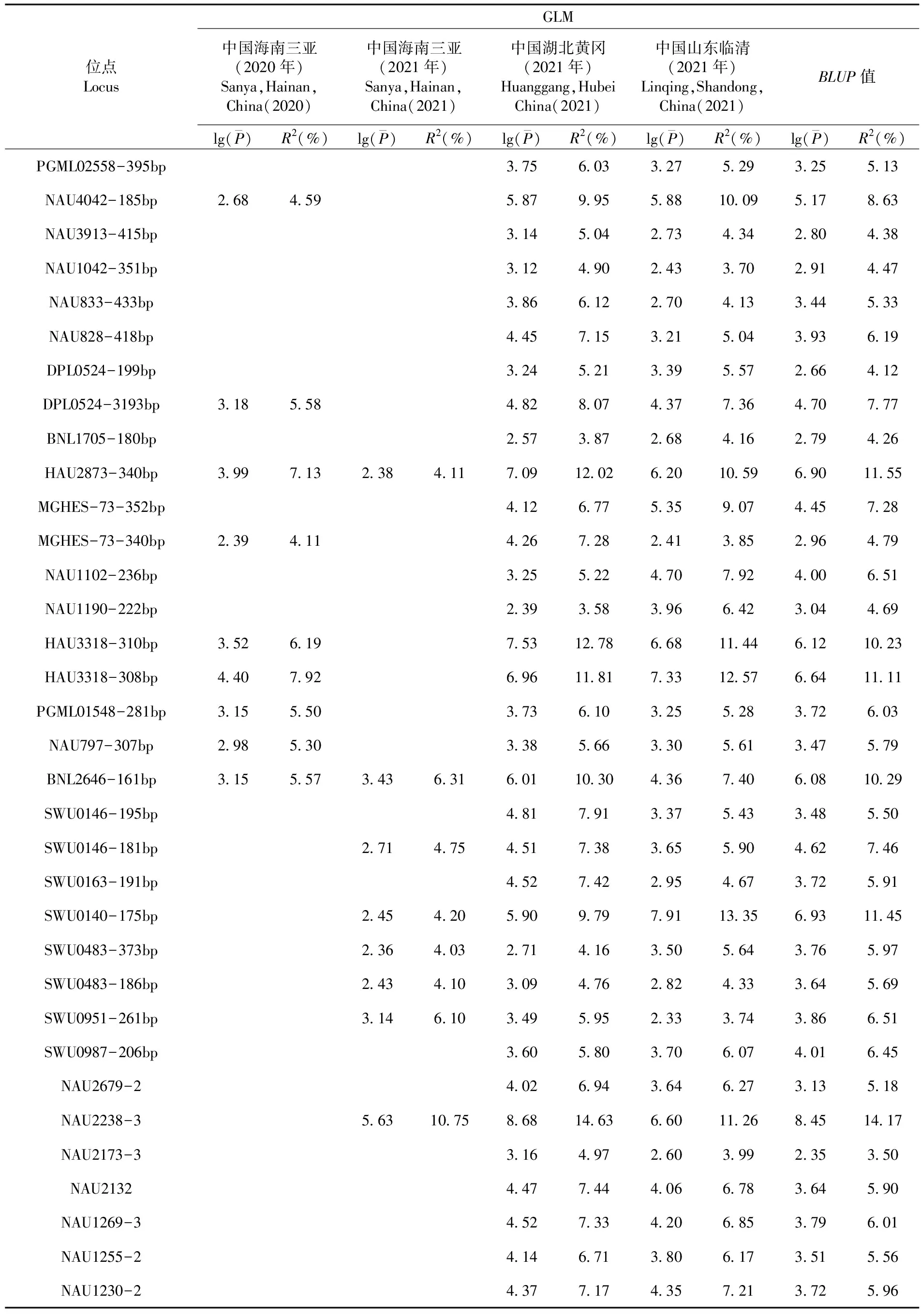
表3 基于GLM模型在3个及以上环境的PH-ST2的关联性
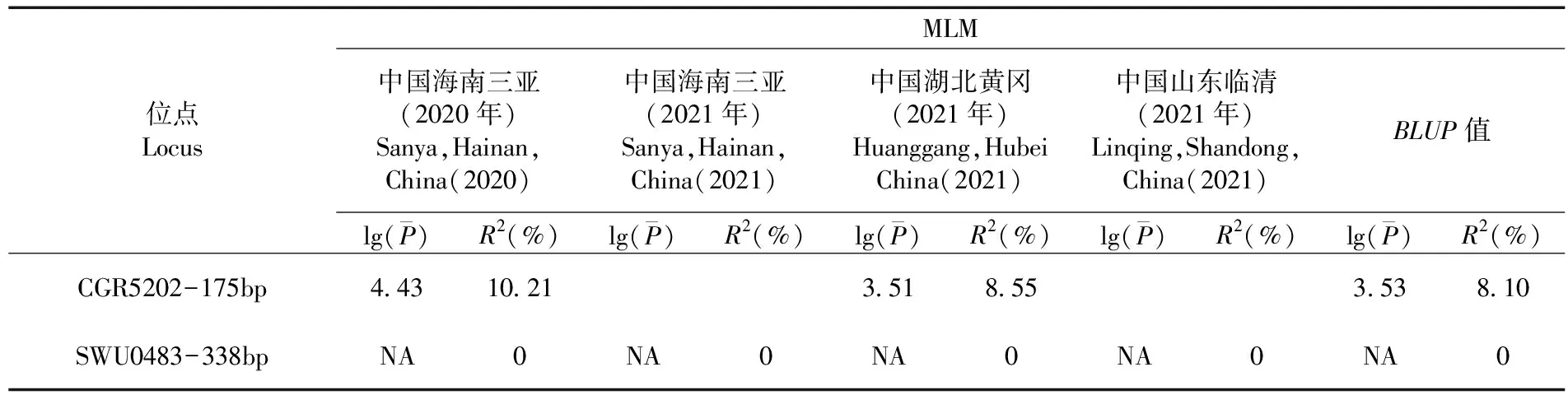
表4 基于MLM模型在3个及以上环境的PH-ST1关联性

表5 基于MLM模型在3个及以上的环境的PH-ST2的关联性

注:Hap 1:不含有CGR5202-175bp位点的材料;Hap 2:含CGR5202-175bp位点的材料
贡献率最高的位点CGR5202-175bp在2020年中国海南三亚和2021年中国湖北黄冈环境下都能与PH-ST1表型显著关联到,其中125份材料含有该位点,47份材料不含该位点,含该位点的材料的株高平均值要显著低于不含有该位点的材料的株高平均值。与PH-ST2相关的贡献率最高的位点为NAU2238-3,且在2021年中国海南三亚、中国湖北黄冈、中国山东临清环境下能显著关联到。其中,153份材料不含该位点,27份材料含该位点,含该位点的材料的株高平均值要显著低于不含有该位点的材料的株高平均值。图3,图4,图5
3 讨 论
3.1 分子标记在作物育种中的作用
随着分子标记技术的迅速发展,不仅加快了育种进程,同时也为复杂的数量性状的遗传分析提供了基础[22]。传统分子标记主要有以下几种:限制性长度多态性(restriction fragment length polymorphism,RFLP)、扩增片段长度多态性 (amplified fragment length polymorphism,AFLP)、SSR等。
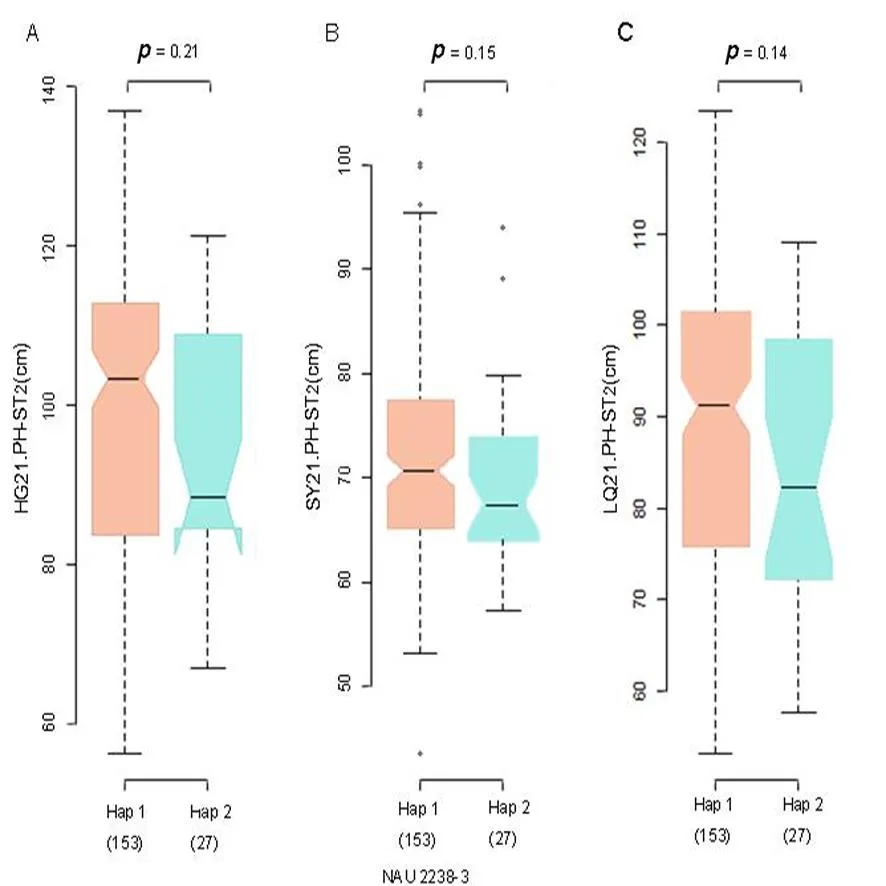
Hap 1:不含有NAU2238-3位点的材料;Hap 2:含NAU2238-3位点的材料

注:蓝色代表前人定位到的QTL,红色代表位于前人定位QTL 1Mb范围内的SSR位点
RFLP和AFLP已成功应用于许多物种的遗传多样性分析,但对棉花多倍体基因组等大型基因组进行基因分型时效率较低[23]。相比于RFLP和AFLP,SSR标记具有多态性高、可重复性强、易于转化和稳定可靠等优点[24-25]。与前人研究与株高性状相近显著关联的位点:位点NAU833-433bp、染色体A05、物理位置15535921.15536120[26],位点NAU828-418bp、染色体A05、物理位置15535921.15536120[26],位点NAU1042-351bp、染色体A05、物理位置15535848.15536094[26],位点MGHES-73-352bp、染色体A09、物理位置72905477.72905691[9],位点HAU2873-340bp、染色体A10、物理位置12349682.12350027等[8],位点NAU797-307bp、染色体D05、物理位置14213616.14213809等[9],位点NAU1255-2、染色体D05、物理位置14213543.14213783[8,9]。研究利用185份陆地棉品种(系)构成的自然群体为材料,选取了137对SSR多态性引物,对5个环境的PH-ST1和PH-ST2进行关联分析。由于研究筛选的SSR引物密度较低,导致标记之间的遗传区域较大,难以快速挖掘目标位点和基因。因此,后续可根据结果划定关联区域,在关联区域结合基因功能注释开发高密度的单核苷酸多态性(single nucleotide polymorphism,SNP)标记,得到与目标性状紧密连锁的标记位点,并应用到相关性状的辅助选择中,加快育种进程。
3.2 动态株高的分析在作物育种中的作用
研究结果与前人研究共同定位到株高性状相关的位点有7个,新定位到与株高性状相关的位点有24个。shang等[8]通过构建遗传图谱并结合不同时期株高的表型数据进行QTL定位分析,检测到的qPH-chr10-3和qPH-chr19-4分别与研究鉴定的显著关联位点HAU2873-340bp 和NAU1255-2位置相近;NAU797-307bp、MGHES-73-352bp 位点与Ma等[9]QTL定位得到的株高QTL位点qPH-Chr19-4、qPH-Chr9-1的位置相近,NAU1255-2位点与qPH-Chr19-4中NAU3437标记重合;Li等[26]研究发现了与赤霉素生物合成途径相关的基因GhGA2Ox8,在改良植物株型方面显示出良好的潜力,能够使冠层更加致密,适合高密度种植,提高棉花产量,定位到该基因的 SNP位点与研究NAU833-433bp、NAU828-418bp、NAU1042-351bp位点物理位置小于1 Mb。该7个位点可能是调控棉花株高的关键位点,但还需后续进一步验证。
3.3 利用GLM和MLM模型鉴定动态株高的主要位点
全基因组关联分析是解析作物复杂数量性状遗传结构的一个重要的工具[27-30]。研究同时采用 GLM模型和MLM模型进行关联分析[31-32]。研究结果表明无论是单个环境还是多个环境下所关联出来的结果,MLM模型所关联到的SSR位点始终少于GLM模型,刘其宝等[17]、王龙等[33]也得出相似的结论,MLM模型的假阳性率低于GLM模型。研究中发现与PH-ST1相关联的CGR5202-175bp位点和与PH-ST2相关联的NAU2238-3位点能在多个环境中同时被检测到且贡献率大于10%,且在前人研究结果中尚未出现。今后可通过构建分离群体对CGR5202-175bp和NAU2238-3位点进一步验证,提高结论的准确性。
4 结 论