基于靶扫描的3D卷积网络及基因检测在肺癌筛查中的运用
徐存来,曹 卓,,尹章勇 ,陈 璇 ,李雨玲 ,龚易莎 ,蒋奕薇 ,潘炯伟
(1.丽水市人民医院 呼吸与危重症科,浙江 丽水 323000;2.温州医科大学 第一临床医学院,浙江 温州 325035)
癌症筛查的目的就是尽量在没有任何症状的情况下,通过各类检测手段,包括但不限于血液学检查和影像学检查,来发现潜在的肿瘤或疾病。如何准确地发现早期肺癌并进行干预是预防肺癌主要的任务之一。与胸部X线相比,低剂量计算机断层扫描可以将肺癌死亡率降低20%以上[1]。然而,每天用肉眼筛选大量CT图像对于放射科医师来说是巨大的压力,且单纯的影像学筛查早期肺癌准确性和特异性仍需提高[1]。近年来发展起来的人工智能信息挖掘技术,在解决大量数据带来的多参数统计分析问题上具有独到的优势,因此,利用掌握的涉及不同机制的分子生物标志,同时联合影像学数据建立的数据分类模型,将对提高肺癌的早期判别准确率有巨大的促进作用。为此,本文拟以基于靶扫描的3D卷积神经网络(3D-CNN)技术联合基因检测建立模型并运用于早期肺癌筛查。
1 资料与方法
1.1 一般资料 本研究为前瞻性巢式病例对照研究。选取2017年12月—2020年12月经丽水市人民医院府前院区呼吸与危重症科因“肺结节”入院患者作为研究队列,收集其基线数据、外周血及CT影像学,并随访90天。队列中经手术病理活检确诊的Ⅰ/Ⅱ期非小细胞肺癌患者80例作为肺癌组,术前未经任何抗癌治疗、无职业致癌物接触史。选取队列中和病例组同一个月在我科经手术病理活检确诊为感染性病变且无肿瘤病史的肺结节患者80例作为良性组。两组的年龄、性别差异无统计学意义(P>0.05),本研究方案经医院伦理学委员会批准,研究对象均知情同意。
1.2 DNA提取及甲基化检测 所有研究对象均入组次日抽取空腹外周静脉血样本2 mL,置于4 ℃保存不超过1周,在新鲜状态下提取DNA备用,选择受试者外周血中pl6、RASSFIA基因启动子区甲基化水平作为观察指标,严格按照说明书操作。主要试剂:DNA提取试剂盒(TIANamp),人类DNA甲基化试剂盒(Zymo Research),人类基因组甲基化p16和RASSF1A基因检测试剂盒(上海透景)。p16基因:上游引物 5′-GAAGAAAGAGGAGGGGCTG-3′,下游引物 5′-GCGCTACCTGATTCCAATTC-3′;RASSF1A基因:上游引物5′-GGGTTTTGCGAGAGCGCG-3′,下游引物 5′-GCTAACAAACGCGAACCG-3′。基因扩增条件:Taq 酶0.5 μL,模板DNA 2.5 ng,上游引物(20 μmol/L)1 μL,下游引物(20 μmol/L)1 μL,GC buffer125 μL,dNTP(含Mg2+)8 μL,灭菌蒸馏水加至50 μL。PCR反应条件:95 ℃热启动15 min;95 ℃30 s,62 ℃30 s,72 ℃30 s,共35个循环;最后72 ℃8 min。
1.3 影像学处理 所有入组对象进行常规的低剂量CT扫描,加上我院的人工智能系统(飞图智能软件),发现肺部结节后进行靶扫描+二三维度重建,靶扫描采用较高剂量进行扫描(120 kV,408 mA),且针对病灶部位进行扫描,执行ROI重建并且图像重建大小为1 024×1 024,层厚为1 mm,层数为56层。参与的读片医师均为高年资医师,由3人以上一起读片。
1.4 Fisher判别分析 Fisher判别分析模型的基本思想是先投影后判别。投影是指将原来的P维(X空间)的输入变量投影到K维(Y空间)上。本文按3∶1的比例将数据随机分成训练集和预测集两组,在SPSS Modeler 18.0中实现。
1.5 C5.0决策树模型 决策树最上面的节点是根节点,从上到下每个节点都会遇到一次测试,不同的测试结果会输出不同的分支。其在SPSS Modeler 18.0中实现对整理好的数据建立预测模型。
1.6 3D-CNN方法 以患者靶扫描的图像作为数据来源,由3名高年资影像医师进行标注,选择最能够体现肺结节特征的3层图像。针对每一个具有金标准的病灶,用矩形框标注出病灶的每一层结构。其中,肺癌设置标签为1,肺良性病变设置标签为2。160例数据里面总共标注了80个肺癌结节和80个肺良性病变结节。本课题组按5∶3的比例将数据随机分成训练集和预测集两组,在SPSS Modeler 18.0中实现。研究采用神经网络算法,在SPSS Modeler 18.0软件中参数如下:使用分割数据=否;方法=Quick;避免过度训练=50%;设置随机种子数=否;按条件停止=Times1 min;优化=memory;继续训练存在的模型=否;使用二进制元集合编码=是;显示反馈图=是。
1.7 统计学方法 应用SPSS 21.0软件处理数据。2组pl6和RASSFIA基因甲基化比较采用Z检验,基于靶扫描的3D-CNN网络模型分类结果采用直接卡方检验。设定的检验水准为0.05。
2 结果
2.1 甲基化检测 肺癌组外周血pl6、RASSFIA基因启动子甲基化水平均高于良性组,差异有统计学意义(P<0.05),见表1。

表1 外周血肺癌组和良性组pl6和RASSFIA基因甲基化比较
2.2 基于靶扫描的3D-CNN网络模型分类 采用3D-CNN网络模型后,预测集分类准确率(83.33%)比训练集(72.00%)提高,但二者差异无统计学意义(χ2=0.602,P=0.438)。
2.3 预测模型的诊断效果 从Fisher、决策树、3D-CNN模型对预测集的诊断结果中可见,3D-CNN模式的诊断灵敏度(79.52%)、特异度(89.24%)、准确度(85.14%)均最高。见表2。
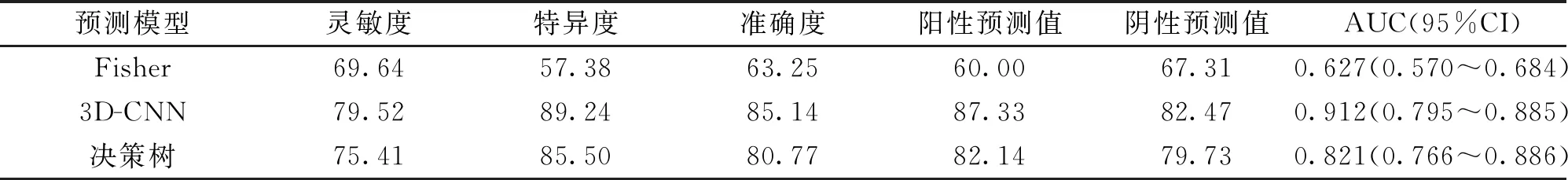
表2 三种预测模型对预测集的诊断结果
3 讨论
3D-CNN模型在医学领域的运用仍以科学研究为主,尤以结肠癌领域最为多见。Hildebrand等[2]开发了一种基于无症状的癌症筛查方法:通过血液学检查、年龄和性别等特征,基于GBDT和随机森林算法来鉴定受试者是否具有结肠直肠癌的风险,其模型检测结肠直肠癌的AUC达到0.82。Zhou等[3]使用多层3D-CNN用于肺结节检测可使诊断模型假阳性减少,所提出的算法在LUNA比赛中获奖[4-5]。本次研究采用肺癌组及良性组训练集,训练基于靶扫描的3D-CNN网络模型后,其分类准确率较前提高、误分率减少。但经卡方检验,训练集及预测集准确率差异无统计学意义,可能与本研究训练集及预测集样本量过少有关,该模型预测集仅为单中心60人次,还需要多中心大样本的研究加强对模型的训练。
另一方面,国外有学者在3 000例受试者的痰液中发现其中6个基因(p16、MGMT、DAPK、 RASSFIA、 PAX513及GATA5)与早期肺癌的风险增高有关[6]。本次研究发现肺癌组和良性组外周血pl6、 RASSFIA基因启动子甲基化水平有显著性差异,证实了上述观点。
然而,由于早期肺癌的发生发展极其复杂,近年来发展起来的多肿瘤标志联合检测也存在相互作用等难以解决的统计学问题[7]。结直肠癌领域的拓荒者和实践者郑树教授[8]将神经网络联合多种肿瘤标志物用于鉴别消化道恶性肿瘤,建立的人工智能消化道肿瘤模型能较好地鉴别肝癌、肠癌和胃癌等消化道肿瘤。本文在我院小样本研究中利用p16和RASSFIA两种基因组合及受试者影像学特征建立了基于Fisher判别及决策树及3D-CNN等三种模型的早期肺结节智能诊断模型,其中3D-CNN模型对早期肺癌及肺良性疾病的判别敏感性及特异性较高(分别为79.52%和89.24%)。
综上所述,本研究从肺癌遗传易感标志和早期效应标志2个方面为切入点,筛选了p16和RASSFIA基因甲基化水平以及应用数据挖掘技术中的3D人工神经网络检测初步构建了肺癌-肺良性疾病辅助诊断模型,为高危人群的筛查和临床肺癌的早期诊断提供有价值的参考资料,对于提高肺恶性肿瘤患者总体的生存率有积极的意义。但该模型预测集仅为单中心60人次,还需要多中心大样本的研究,且未进行全基因组测序明确甲基化结果,有待进一步研究加以证实。

