基于Transformer的强泛化苹果叶片病害识别模型
徐艳蕾,孔朔琳,陈清源,高志远,李陈孝
基于Transformer的强泛化苹果叶片病害识别模型
徐艳蕾,孔朔琳,陈清源,高志远,李陈孝※
(吉林农业大学信息技术学院,长春 130118)
模型泛化能力是病害识别模型多场景应用的关键,该研究针对不同环境下的苹果叶片病害数据,提出一种可以提取多类型特征的强泛化苹果叶片病害识别模型CaTNet。该模型采用双分支结构,首先设计了一种卷积神经网络分支,负责提取苹果叶片图像的局部特征,其次构建了具有挤压和扩充功能的视觉Transformer分支,该分支能够提取苹果叶片图像的全局特征,最后将两种特征进行融合,使Transformer分支可以学习局部特征,使卷积神经网络分支学习全局特征。与多种卷积神经网络模型和Transformer模型相比,该模型具有更好的泛化能力,仅需学习实验室环境叶片数据,即可在自然环境数据下达到80%的识别精度,相较卷积神经网络EfficientNetV2的72.14%精度和Transformer网络PVT的52.72%精度均有较大提升,能够有效提升对不同环境数据的识别精度,解决了深度学习模型训练成本高,泛化能力弱的问题。
图像识别; 农业;卷积神经网络;苹果叶片病害;Transformer模型;强泛化性;特征融合
0 引 言
苹果是重要的农业经济作物,具有很高的营养价值和经济价值。然而,苹果叶片极易感染锈病、炭疽病等病害,导致苹果产量和质量严重下降。因此,对苹果病害的高效、精准识别是防治苹果病害的关键。
近年来,基于卷积神经网络(Convolutional Neural Networks)的苹果病害识别成为代替人工识别和传统机器学习识别的方法。很多研究人员针对苹果叶片的实验室数据进行了相应的研究,取得了较好的识别结果。郝菁等[1]以4种苹果叶片病害图像作为研究对象,对Resnet 50、Mobilenet v2、Vgg16、Vgg19、Inception v3进行迁移学习,其中识别效果最佳的模型正确率达97.40%。Zhong等[2]在DenseNet-121网络的基础上,分别使用Focal损失函数、逻辑回归和多标签分类方法对不平衡的苹果叶片病害进行识别,达到93.51%、93.31%和93.71%识别准确率。刘斌等[3]以Inception- ResNet V2为基础模型,引入CBAM模块和焦点损失函数,实现苹果黑星病和锈病程度90.82%的平均识别精准度。
这些研究虽然识别准确率高,但不适合应用在实际生产中。针对复杂自然环境下的苹果病害识别,很多研究人员也进行了深入的研究。Singh等[4]提出一种仅有8层深度的CNN网络,以田间拍摄的两种苹果病害叶片图像作为试验数据,获得99.2%的准确率。Chao等[5]以SENet为基础,设计出的SE-DEEPBlock与Xception模型结合,在复杂背景的苹果叶片病害上得到99.40%的识别准确率。Li等[6]使用NAS搜索算法设计出轻量CNN网络RegNet,在具有复杂背景的苹果叶片病害上实现了99.23%的识别准确率。Fu等[7]使用多尺度模块、空洞卷积对AlexNet进行轻量化改进,并应用于五类复杂背景的苹果叶片病害识别任务中,获得97.36%的准确率。
以上研究无论是针对实验室数据还是自然环境数据,识别准确率都很高,然而很多研究证明以数据驱动的深度学习模型在训练与识别数据为不同地点的图像时,会造成模型识别精度的衰退。Mohanty等[8]发现,使用卷积神经网络GoogLeNet模型分类病害图像,能获得99.34%的极高准确率,但对不同条件下拍摄的图像进行识别时,模型的准确性大幅降低,仅略高于31%。Ferentinos等[9]使用PlantVillage数据和自然环境数据,分别对模型进行训练,试验表明AlexNetOWTBn与VGG模型使用实验室条件图像进行训练并识别自然图像时准确度仅为32.23%和33.27%。因此,目前迫切需要研究一种泛化性强的深度学习模型,对不同背景、不同采集地点的苹果叶片病害都能够进行高效精准的识别。
最近,受Transformer模型在自然语言处理领域成功应用的启发,Dosovitskiy等[10]提出一种适用于计算机视觉的Transformer模型ViT。该模型是一种提取全局特征的非局部(Non-local)模型[11],在不使用卷积核的情况下能够达到极高的识别精度,并且具有较强泛化能力[12-14]。此外,该模型在图像上加入了可学习的位置信息,解决了CNN中没有可用的空间信息[15]以及在图像中融入不同的纹理会导致CNN识别出现极大偏差[16]的问题,相较于CNN模型,ViT模型具有更好的稳健性。PVT模型[17]是对ViT模型的进一步改进,通过卷积的方式,以卷积核的尺寸控制数据块的尺寸,降低了Transformer模型的计算量。此外,Transformer模型具有更高的鲁棒精度,更科学的特征学习方式[18-20]。视觉Transformer模型作为一种Non-local模型,虽然依靠弱偏置归纳、易并行结构成为计算机视觉(Computational Vision)领域中的新热点,但目前并没有研究证明全局信息绝对优于局部信息。并且因为Transformer模型几乎没有偏置归纳,导致在小数据上有着更高的过拟合风险以及训练成本高的问题[21]。
为了提升深度学习模型泛化能力,使苹果病害识别模型可以在更多场景达到较高识别精准度。该研究以5中苹果叶片为研究对象,从多尺度特征融合模型得到启发[22-24],将CNN和Transformer模型进行改进并结合,设计了一种通过双分支结构提取全局特征和局部特征的强泛化深度学习模型CaTNet。该研究有效降低了深度学习模型训练成本,为设计泛化能力强的深度学习模型提供了思路。
1 材料与方法
1.1 苹果叶片病害数据采集
为验证模型的泛化能力,不仅需要简单背景的实验室数据还需要复杂背景的自然数据。该研究数据集由3个公开数据具和自建数据构成,涉及苹果健康叶片、锈病、炭疽病、斑点落叶病和花叶病,公开数据集来自PlantVillage[25]、Plant pathology[26]、Ai Studio[27]。自建数据集为自然环境与实验室数据混合的5种苹果叶片病害数据。其中自然环境数据拍摄于吉林省延边朝鲜族自治州安图县苹果园。首先,在自然环境中直接拍摄苹果叶片获取自然环境苹果叶片数据。其次,采摘各类别苹果叶片,放置于实验室桌面上,叶片下铺垫白色PVC板进行拍摄。拍摄工具为智能手机,型号为Oneplus8pro,共拍摄973张RGB图像,图像像素3 000×3 000。PlantVillage数据集为多种作物病害实验室数据,该研究采用PlantVillage数据集中苹果健康叶片和炭疽病叶片图像数据。Plant pathology数据集为4种苹果叶片病害自然环境数据,Ai Studio数据集为自然环境与实验室数据混合的5种苹果叶片病害数据。为更客观验证模型泛化能力,该研究数据集中,除了使用自建数据中实验室数据构建训练数据集,使用自然环境数据构建测试数据集。还在健康叶片训练数据中加入PlantVillage数据,测试数据中加入Plant pathology数据;在锈病训练数据中加入Ai Studio数据,测试数据中加入Plant pathology数据;在炭疽病训练数据中加入PlantVillage数据,测试数据中加入Plant pathology自然环境数据;在斑点落叶病的训练数据和测试数据中加入Ai Studio数据;在花叶病训练数据和测试数据中加入Ai Studio数据。苹果叶片病害数据集中,部分实验室数据如图1a所示,部分自然数据如图1b所示。从图中可以看出,不同条件下的苹果叶片图像中,苹果叶片摆放姿态不同,特征差异较大,证明以该数据集为训练数据将有效证明模型泛化能力。

图1 苹果叶片病害图像
1.2 数据预处理
经过整理后的每类病害以及数据来源和数据量如表 1所示,其中锈病、斑点落叶病和花叶病数据过多,炭疽病数据过少。数据不平衡会导致模型对稀疏样本识别能力下降,因此该研究对数据进行预处理来平衡各数据数量,提高模型识别精度。

表1 苹果叶片数据详细信息
首先对图像数据尺寸统一至224×224像素。其次对取自Ai Studio数据集的锈病训练数据,斑点落叶病测试数据,花叶病测试数据中部分相似图像进行删减。然后,对炭疽病和健康叶片数据进行扩充,扩充操作为随机色彩抖动和添加噪点。最后,对自然环境数据进行裁剪,保留大部分病害特征,减少复杂背景面积。通过对数据的预处理得到最终数据集,总共15 567张RGB图像数据。苹果叶片数据信息如表1所示,其中训练数据均为实验室数据,测试数据均为自然环境数据,训练集数据处理后总量达12 567张,测试集数据处理后总量达3 728张,训练集与测试集比约7∶3。
2 CaTNet网络模型
CaTNet模型由CNN分支和Transformer分支构成,两分支输出的特征进行融合促使深度学习模型同时关注全局与局部特征,增强了模型的泛化能力,而且CNN网络解决了Transformer网络计算速度慢,计算量大的问题。CaTNet模型结构如图2所示,其主体结构由多个Block、降采样、自适应池化和全连接层构成。Block由CNN分支与Transformer分支组成。CNN分支中包含多个CNN块,CNN块由深度卷积层和点卷积层构成。其中深度卷积层由尺寸3×3大小,分组数为输入特征图通道数的卷积核、批归一化和激活函数构成。点卷积层由尺寸1×1大小的卷积核、批归一化、激活函数构成。两分支通过融合操作将特征进行融合,在输入下一个Block前进行降采样减少特征图尺寸,因此模型具有特征金字塔结构。最后一个Block将不再进行降采样操作,经过融合的特征将直接输入1×1自适应池化层,然后输入全连接层得到苹果叶片图像的病害类型。

注:Block为多个CaTNet Block堆叠结构,Conv(Ks=3 G=Input)表示以卷积核大小为3×3,分组数为输入通道的卷积层(Convolution),其中Ks为卷积核大小(Kernel size),G为分组数(Group),Conv(Ks=1)表示以卷积核大小为1×1的卷积层,BN表示批归一化层,ReLU表示ReLU激活函数,Full connection layer表示全连接层,Multi-head attention layer表示多头注意力层。
CaTNet模型不同层中特征图尺寸、分支中块的重复次数和不同分支间融合方式如表2所示,模型在Block1-4采用维度融合(Concat),在Block5采用数值融合(Add)。维度融合利于双分支学习不同特征,数值融合利于分类操作。CNN分支与Transformer分支中块的重复次数相同,如表2中块重复次数所示。

表2 CaTNet网络参数
2.1 构建CaTNet中Transformer分支
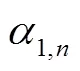


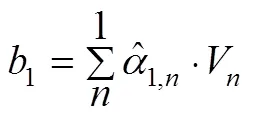
为了解决上述问题,提高Transformer模型计算速度,降低训练成本,本文首先对Transformer分支进行改进,其次对分支中Transformer块(TR块)进行改进。
针对Transformer分支部分,本文设计出一种具有挤压功能的挤压模块,将输入特征图通道维度进行缩减,挤压模块由卷积层、归一化层构成,卷积层由1×1大小的卷积核组成,使用卷积对通道维度进行缩减,将尽可能避免因通道维度缩减导致的信息流失,解决Softmax激活函数易造成显存溢出的问题。其次,在Transformer分支最后添加了通道扩充模块,对通道维度进行扩充,与来自卷积神经网络分支的特征图通道维度匹配。扩充模块与挤压模块结构相似,通过卷积层对特征通道维度进行扩充,在归一化操作后使用激活函数ReLU6对特征进行非线性化处理。ReLU6相较ReLU激活函数,最大输出值为6,对多数以Float16作为计算单位的移动设备更加友好,模型更易于移植到小型移动设备上。具有通道挤压和通道扩充模块的Transformer分支结构如图3所示,其中TR块为Transformer模型。
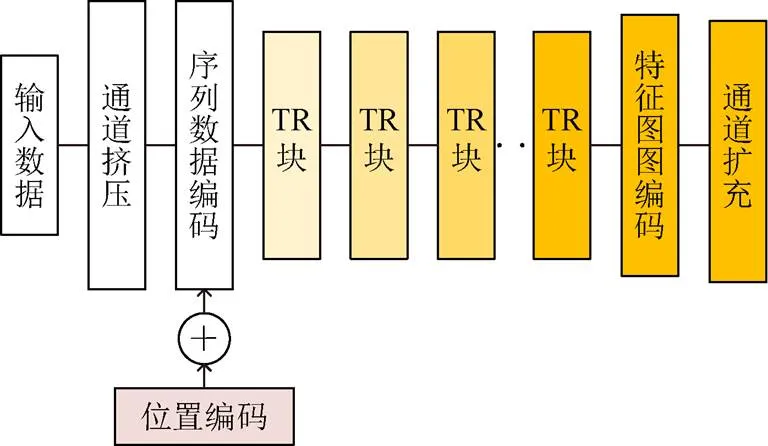
图3 CaTNet中Transformer分支结构
针对Transformer块部分,Transformer模型结构过于复杂,本文对Transformer分支中每个负责提取全局信息的TR块进行优化。首先对Transformer模型结构进行优化,仅使用一个多头注意力和两个全连接神经网络搭建,大幅简化传统Transformer模型,确保Transformer块仅提取序列数据间的序列关系特征。其次,使用全连接网络替换传统前馈神经网络。TR块结构如图4所示,首先将数据输入全连接层得到、和向量,然后进入多头注意力层得到全局特征,之后将特征输入全连接神经网络得到最终序列特征。在全连接网络后添加Dropout层对低权重神经元进行删减,降低Transformer模型在小数据上的过拟合风险。

图4 Transformer块结构
Transformer分支中设定的参数也会影响模型的性能与训练成本。为了保持Transformer分支提取全局信息的能力并提高计算速度,本文设置序列数据为特征图尺寸的1/8,多头注意力中头初始数量设置为2并从第二个Block后每次增加2,从而保证模型具有较高计算精度的同时,尽可能减少模型算力消耗。Transformer分支中,每个Transformer模块中头的数量,序列数据尺寸以及通过通道缩减模块后输出的通道数如表3所示。
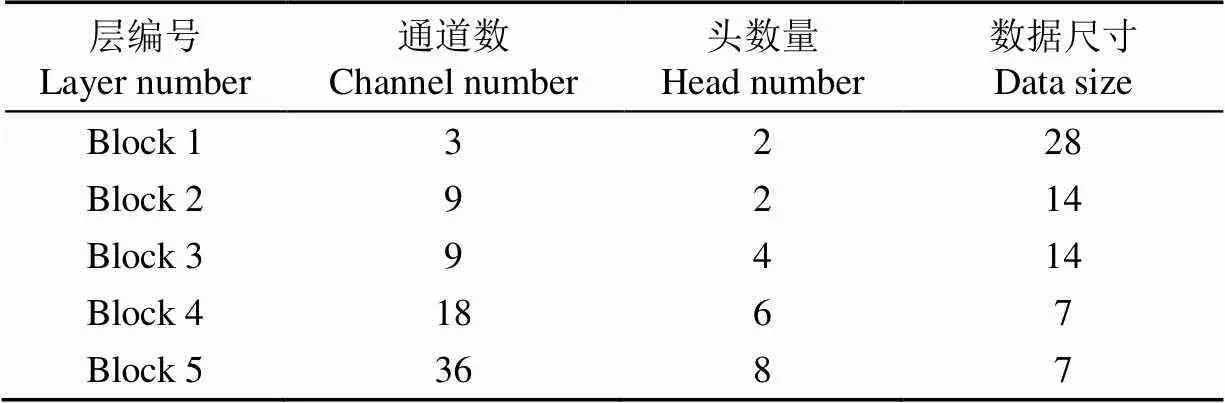
表3 Transformer分支在各Block中输入的超参数
2.2 构建CaTNet中CNN分支
目前多数CNN网络如MobileNet系列[28-30],EfficientNet系列[31-32]均以轻量化为前提对网络进行设计,为匹配计算量较大的Transformer分支并提供局部特征,该研究同样以轻量化为前提设计CaTNet的CNN分支。CNN分支中由多个CNN块构成,CNN块结构如图 5所示,由深度卷积层、归一化、激活函数和点卷积层构成。相较多种CNN网络,本文首先在深度卷积层同时进行特证信息扩充和局部特证信息提取,避免计算冗余。其次,点卷积层并未对通道数进行缩减,避免了信息的流失,提高特征信息流动性。最后将输出数据和输入该块的原始数据进行相加,构成短路连接并输入激活函数,使模型学习不同层次特征,解决网络退化问题。

图5 CaTNet中CNN块结构
2.3 CaTNet模型的4种变体
降采样使得深度学习模型具有金字塔结构,帮助深度学习模型提取不同尺寸的特征信息,提高模型性能,扩大模型应用场景。过去,深度学习模型多以不可学习的最大池化和平均池化操作进行降采样[33-34]。随着CNN模型的发展,模型开始使用卷积核进行降采样。CaTNet模型中Transformer模型负责提取全局信息,如果使用卷积核进行降采样操作,存在破坏全局特征的可能。由于目前没有研究证明卷积核对全局特征具有破坏性,因此该研究针对CaTNet模型的每个Block中的降采样层,设计了多种使用不同降采样操作的CaTNet模型变体,并在后续(3.3节)进行大量试验充分验证各种降采样层对全局特征的影响。
这些变体分别是,使用3×3尺寸卷积核进行降采样的CaTNet(conv)模型,使用1×1尺寸卷积核进行降采样的CaTNet(pconv)模型,使用最大池化进行降采样的CaTNet(mp)模型,使用平均池化进行降采样的CaTNet(ap)模型。
2.4 试验环境与参数配置
试验基于Pytorch1.9.1深度学习框架和Python3.9.0搭建,使用TitanX GPU对模型进行训练。在训练过程中,使用SGD优化器[35]更新模型参数,使用交叉熵损失函数计算模型输出与真实值的差异,SGD优化器学习率每10轮训练衰退0.1倍,计算动量0.7。模型共经历40轮训练,每批次输入16张图片。
2.5 评价指标
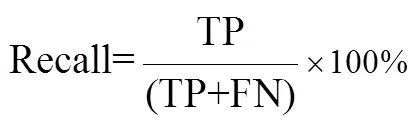
该研究使用准确率(Accuracy),F1值(F1score),精准率(Precision, P)和召回率(Recall, R)衡量模型识别性能。准确率计算如式(4)所示。TP(True Positive)为正确分类的正样本数,TN(True Negative)为正确分类为负样本数,FP(False Positive)为错误分类的正样本数,FN(False Negative)为错误分类的负样本数。训练准确率为使用实验室数据对模型训练得到的准确率,测试准确率为模型对自然环境数据识别得到的准确率。F1值均衡了精准率与召回率,同时使用F1值和准确率可以更客观地衡量模型性能,F1值计算如式(5)所示。为了避免出现F1值相差较小导致无法衡量模型性能的情况发生,该研究还使用精准率和召回率对模型进行评价。精准率代表预测为正的结果中,有多少是真实为正,如式(6)所示。召回率代表模型对正样本预测效果,即预测了多少真实为正,如式(7)所示。



3 试验与分析
3.1 不同CaTNet模型变体性能比较及选择
使用不同池化操作的CaTNet模型各变体在实验室数据(训练)与自然数据(测试)的准确率如表4所示。可以看出,所有模型在实验室数据下都达到理想准确率,约99%。但各模型在自然数据下的识别效果参差不齐。CaTNet(ap)与CaTNet(mp)模型识别准确率达近80%,远大于使用卷积进行降采样的CaTNet(conv)与CaTNet(pconv)模型74.06%与67.95%的准确率,这证明了使用卷积核进行降采样对特征具有一定的破坏性,导致模型精度下降。

表4 不同降采样的CaTNet训练结果
CaTNet模型4种变体的F1值、精准率和召回率,如表5所示。其中,使用卷积核进行降采样的CaTNet(conv)和CaTNet(pconv)模型在4种病害及健康叶片的F1值均低于使用池化进行降采样的CaTNet(mp)和CaTNet(ap)模型,进一步证明使用卷积核进行降采样会降低模型识别精度。其次,使用最大池化的CaTNet(mp)模型虽然在健康叶片,炭疽病获得78.52%和71.96%的F1值,高于CaTNet(ap)模型但相差不大。CaTNet(mp)模型在锈病、炭疽病和斑点落叶病的精准率为90.41%、70.96%和72.16%,对比CaTNet(ap)模型精确率更高,说明CaTNet(mp)模型判定为这3类病害中真正为这3类病害的比重更大,可信度更高。此外CaTNet(ap)模型在锈病,炭疽病,斑点落叶病和花叶病的召回率为82.21%、75.30%、89.15%和63.97%,对比CaTNet(mp)模型召回率更高,证明CaTNet(ap)模型可以将更多上述3类病害图像识别出来,对正例的查全能力更强。
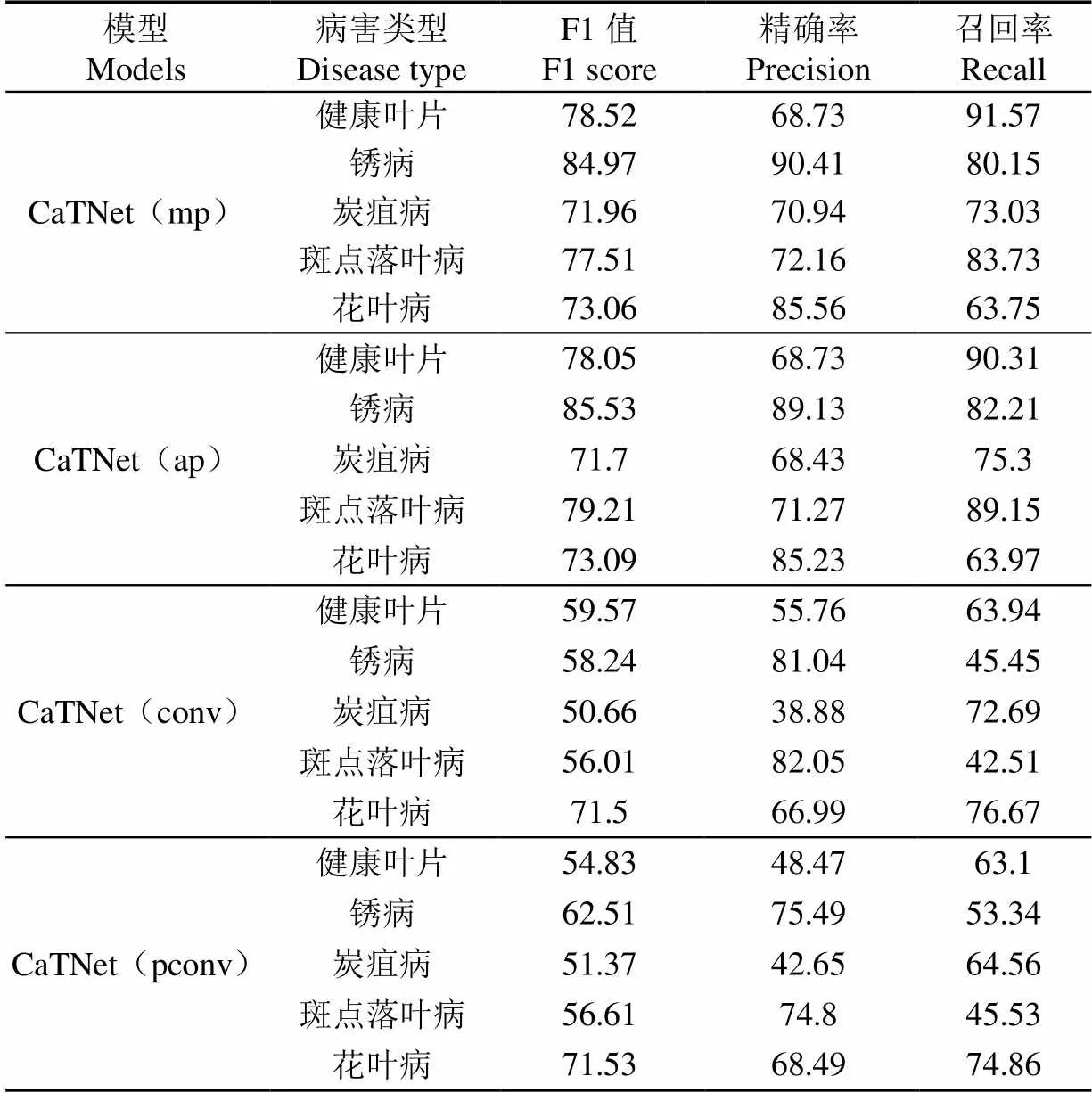
表5 CaTNet各模型在5种叶片类型的识别F1、精准率和召回率
CaTNet模型4种变体训练过程中,模型每个步长的准确率曲线(Accuracy)和损失值曲线(Loss)如图6所示。图6a、6b为使用4种降采样的CaTNet变体模型在实验室数据中40次迭代下的准确率与损失曲线,图6c、6d为这些变体对自然环境数据进行识别得到的准确率与损失曲线。结合这些信息可以发现,这些变体在实验室数据下,不论是准确率曲线还是损失曲线都非常平滑,但这些模型在实际自然数据上的识别准确率与损失曲线却有着较大波动,说明虽然在数值上各模型在实验室数据上已经达到拟合,但实际上,这些模型经过每一次迭代后,所学习的具体特征有着较大差距。
CaTNet变体模型中,使用平均池化进行降采样的CaTNet(ap)模型准确率和损失曲线相对其余3种模型更加平滑,如图6a、6b所示。此外,CaTNet(ap)模型相较CaTNet(mp)召回率更高,在实际作业中较高的召回率还有助于模型进行检测与分割等任务,因此选择CaTNet(ap)模型进行后续试验。

a. 实验室数据准确率曲线a. Accuracy curve of laboratory datab. 实验室数据损失曲线b. Loss curve of laboratory datac. 自然环境数据准确率曲线c. Accuracy curve of natural datad. 自然环境数据损失曲线d. Loss curve of natural data
3.2 模型有效性分析
为验证CaTNet模型有效性,该研究选择CNN模型和Transformer模型同时与CaTNet(ap)模型进行对比,其中CNN模型包括EfficientNetV2、ResNext[36]、ShuffleNetV2[37]。Transformer模型包括ViT与PVT。各模型训练指标如表6所示,所有模型在实验室数据上的表现较好,均能达到98%以上。而对自然数据的测试准确率中,本文设计的CaTNet(ap)模型的自然数据测试准确率达到79.35%,F1值达到78.33%,远远高于其他模型。

表6 多种模型苹果叶片病害识别结果
从计算速度上看,CaTNet(ap)计算单张图片时间为0.108 2 s,仅慢于ShuffleNetV2 0.5和ShuffleNetV2 1.0。但ShuffleNetV2为单分支网络,并且测试准确率极低(58.77%),因此证明了CaTNet(ap)模型很好的平衡了计算效率和计算精度。相较CNN中精度最高的EfficientNetV2 s 模型计算速度更快,证明并行的双分支结构可以在更短时间内调动更多神经元进行计算,具有高效计算能力的模型能够适用于更多场景,极大地拓宽了深度学习的使用范围。
从该表中还可以发现,CNN模型在自然数据准确率普遍比Transformer模型高,CNN模型中表现最佳的EfficientNetV2 s准确率为72.14%,F1值为73.76%,大幅超越Transformer模型中表现最佳的PVT m模型,验证了Transformer模型在小数据集中不易提取正确信息特征,而使用双分支结构的CaTNet模型解决了CNN的泛化能力弱和Transformer模型在小数据集表现不佳的问题。
为了进一步对比CNN模型、Transformer模型和CaTNet模型的性能,该研究对CaTNet(ap)、EfficientNetV2 s、ResNext50、ShuffleNetV2 1.0、ViT和PVT m模型的准确率和损失曲线进行对比,结果如图7所示。由图7a、7b中各模型在实验室数据准确率、损失曲线可以看出,这些模型的训练曲线非常平滑,且都在最后达到了拟合。而对自然数据进行识别的准确度曲线,如图7d、7c所示,CNN模型(EfficientNetV2 s,ResNext50,ShuffleNetV2 1.0)与Transformer模型(ViT,PVT m)有较大不同。相对CNN模型,Transformer模型的自然数据曲线不论是准确率还是损失曲线,都随着训练趋近平滑,即模型对特征的提取稳定,但由于Transformer弱偏置归纳的缺点导致其准确率较低,损失较高,并产生了过拟合现象。而CNN模型,包括本文设计的CaTNet(ap)模型,它们的自然数据准确率和损失曲线在后期都有一定程度的振荡,但CaTNet(ap)曲线相对两种CNN模型更平滑,没有出现每轮学习后,模型准确率值变化较大的情况。这证明通过Transformer分支提取全局特征有效的缓解了CNN分支中局部特征造成的特征偏离,使CaTNet(ap)模型的精度相较CNN模型和Transformer模型更高。同时CaTNet(ap)模型的CNN分支也使CaTNet模型获得偏置归纳能力,有效解决了Transformer模型在小数据集上识别精度低,易过拟合的缺点。

a. 实验室数据准确率曲线a. Accuracy curve of laboratory datab. 实验室数据损失曲线b. Loss curve of laboratory datac. 自然环境数据准确率曲线c. Accuracy curve of natural datad. 自然环境数据损失曲线d. Loss curve of natural data
3.3 模型先进性分析
为验证该研究提出模型CaTNet先进性,本文首先和对作物叶片病害识别模型泛化能力的研究进行对比。由于各研究试验所用硬件环境不同,为了更客观地对比各研究中给出的计算速度,本文对各研究中给出的目标模型计算速度进行标准化,即使用对比研究中给出的某一易复现的模型,计算出该模型在本文环境下的计算速度S并记录该模型在对比研究中计算的速度S。之后通过二者的比值与需要对比的模型计算速度S相乘,得到对比模型在本文实验环境中的近似计算速度,标准化速度计算过程如式(8)所示。

如表7所示,本文提出的CaTNet(ap)测试准确率为79.35%,相较Mohanty等[8]、Ferentinos等[9]测试准确率(31%,32.23%,33.27%)有极大提升,计算速度相较Ferentinos等[9]研究中使用的模型计算速度更快。

表7 作物病害识别模型泛化能力对比
其次,目前多数苹果叶片病害识别研究,使用的训练数据集和测试数据集为同源数据。为了进一步验证模型先进性,和同源数据病害识别研究进行对比,本文对CaTNet模型重新进行了训练。与针对实验室数据研究进行对比中,本文使用1.1节中采集的实验室数据数据对模型进行训练。与针对自然环境数据研究进行对比中,使用本文采集的自然环境数据对模型进行训练。这些试验中所用数据以7∶3比例进行训练并测试数据集划分并进行2倍随机角度旋转扩充。
表8中的各项研究数据可以看出,本文模型在实验室环境数据与自然环境数据上的准确率均高于其他6种研究,相较Fu等[7]以轻量化为主题的模型计算速度更快,充分证明了CaTNet模型先进性。
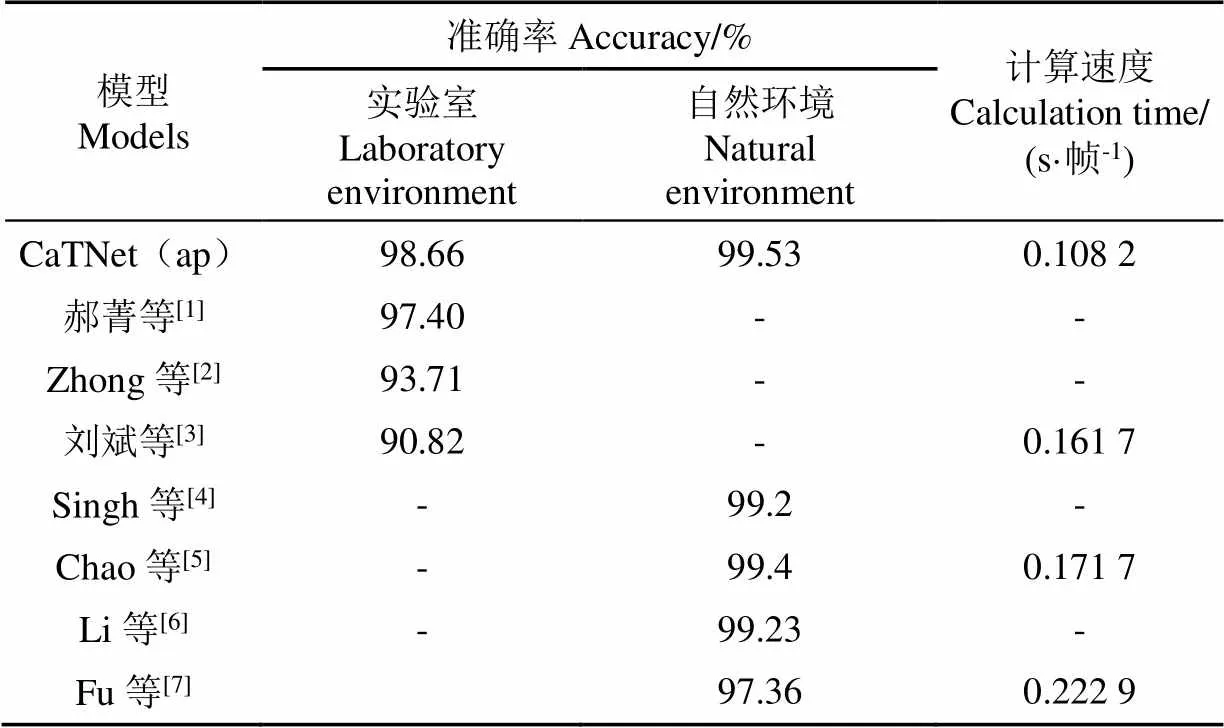
表8 多种苹果叶片病害模型对比
4 结 论
本文基于CNN模型与Transformer模型,研究开发了可以增强模型泛化能力的双分支深度学习模型CaTNet,并用于对苹果健康和患病的叶片图像进行识别。通过对降采样层、CNN模型、Transformer模型以及模型结构对模型的影响进行了分析,并得出以下结论:
1)使用卷积进行降采样的CaTNet(conv)获得74.06%测试准确率,远低于使用池化进行降采样的CaTNet(ap)测试准确率为79.35%,证明使用卷积进行降采样会破坏来自Transformer的全局特征信息。
2)CNN模型EfficientNetV2 s的测试准确率为72.14%,远高于Transformer模型PVT m的52.72%,说明Transformer模型在小型数据集中更难拟合。
3)CaTNet(ap)获得79.35%的测试准确率,相较CNN模型72.14%和Transformer模型52.72%的测试准确率大幅提升。相较文献[8-9]的研究,准确率从30%提升至79%,为提升模型泛化能力提供了更细致和科学的方法。
4)具有双分支结构的CaTNet模型虽然集成了两套模型,但训练成本并未增加,计算速度0.108 2 s/帧,证明本文提出的双分支结构以及各种优化方法可以降低训练成本,提高神经元利用效率。
[1] 郝菁,贾宗维. 基于图像识别的苹果叶片病害识别模型对比研究[J]. 中国农学通报,2022,38(12):153-158.
Hao Qing, Jia Zongwei. Comparative study on apple leaf disease identification models based on image recognition[J]. Chinese Agricultural Science Bulletin, 2022, 38(12): 153-158. (in Chinese with English abstract)
[2] Zhong Y, Zhao M. Research on deep learning in apple leaf disease recognition[J]. Computers and Electronics in Agriculture, 2020, 168: 105146.
[3] 刘斌,徐皓玮,李承泽,等. 基于快照集成卷积神经网络的苹果叶部病害程度识别[J]. 农业机械学报,2022,53(6):286-294.
Liu Bin, Xu Haowei, Li Chengze, et al. Identification of apple leaf disease degree based on snapshot integrated convolutional neural network[J]. Transactions of the Chinese Society of Agricultural Machinery, 2022, 53(6): 286-294. (in Chinese with English abstract)
[4] Singh S, Gupta I, Gupta S, et al. Deep learning based automated detection of diseases from apple leaf images[J]. CMC-Computers, Materials & Continua, 2022, 71(1): 1849-1866.
[5] Chao F, Hu X, Feng Z, et al. Construction of apple leaf diseases identification networks based on xception fused by SE module[J]. Applied Sciences, 2021, 11(10): 4614.
[6] Li L, Zhang J, Wang B. Apple leaf disease identification with a small and Imbalanced dataset based on lightweight convolutional networks[J]. Sensors, 2021, 22(1): 173.
[7] Fu L, Li S, Sun Y, et al. Lightweight-convolutional neural network for apple leaf disease identification[J]. Frontiers in Plant Science, 2022, 13: 831219.
[8] Mohanty S P, Hughes D P, Salathé M. Using deep learning for image-based plant disease detection[J]. Frontiers in Plant Science, 2016, 7: 1419.
[9] Ferentinos K P. Deep learning models for plant disease detection and diagnosis[J]. Computers and electronics in agriculture, 2018, 145: 311-318.
[10] Dosovitskiy A, Beyer L, Kolesnikov A, et al. An image is worth 16x16 words: Transformers for image recognition at scale[EB/OL]. (2020-06-03)[2022-04-18]. org/abs/2010. 11929.
[11] Wang L, Girshick R B, Gupta A, et al. Non-local neural networks[EB/OL]. (2018-04-13)[2022-04-18]. https://arxiv. org/abs/1711. 07971.
[12] Zhang C, Zhang M, Zhang S, et al. Delving deep into the generalization of vision transformers under distribution shifts[C]/IEEE Conference on Computer Vision and Pattern Recognition (CVPR). New Orleans: IEEE, 2022: 7277-7286.
[13] Fang Y, Liao B, Wang X, et al. You Only Look at One Sequence: Rethinking Transformer in Vision through Object Detection[EB/OL]. (2021-10-27)[2022-04-18]. https://arxiv. org/abs/2106. 00666.
[14] Nasser M, Ranasinghe K, Khan S, et al. Intriguing Properties of Vision Transformers[EB/OL]. (2021-3-21)[2022-04-18]. https://arxiv.org/abs/2105. 10497.
[15] Sabour S, Frosst N, Hinton G E. Dynamic routing between capsules[J]. Advances in Neural Information Processing Systems, 2017, 30: 3859-3869.
[16] Geirhos R, Rubisch P, Michaelis C, et al. ImageNet-trained CNNs are biased towards texture; increasing shape bias improves accuracy and robustness[EB/OL]. (2019-01-14)[2022-04-18]. https://arxiv.org/abs/1811. 12231.
[17] Wang H, Xie E, Fan P, et al. Pyramid vision Transformer: A versatile backbone for dense prediction without convolutions[C]//International Conference on Computer Vision (ICCV). Online: IEEE, 2021: 568-578.
[18] 贾伟宽,孟虎,马晓慧,等. 基于优化Transformer网络的绿色目标果实高效检测模型[J]. 农业工程学报,2021,37(14):163-170.
Jia Weikuan, Meng Hu, Ma Xiaohui, et al. Efficient detection model of green target fruit based on optimized Transformer network[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2021, 37(14): 163-170. (in Chinese with English abstract)
[19] Tuli S, Dasgupta I, Grant E, et al. Are Convolutional Neural Networks or Transformers more like human vision?[EB/OL]. (2021-07-01)[2022-04-18]. https://arxiv.org/abs/2105. 07197.
[20] Landau B, Smith L B, Jones S. Syntactic context and the shape bias in children's and adults' lexical learning[J]. Journal of Memory and Language, 1992, 31(6): 807-825.
[21] Lin Y, Wang X, Liu Y, et al. A survey of Transformers[EB/OL]. (2021-06-15)[2022-04-18]. https:// arxiv.org/abs/2106. 04554.
[22] Gong J, Shen H, Zhang M, et al. Highly Efficient 8-bit Low Precision Inference of Convolutional Neural Networks with Intelcaffe[M]. NewYork: IEEE, 2018.
[23] Wang F, Wang J, Li R, et al. T-CNN: Trilinear convolutional neural networks model for visual detection of plant diseases[J]. Computers and Electronics in Agriculture, 2021, 190: 106468.
[24] Gao M, Yang Y, Chen H, et al. A multiscale dual-branch feature fusion and attention network for hyperspectral images classification[J]. IEEE Journal of Selected Topics in Applied Earth Observations and Remote Sensing, 2021, 14: 8180-8192.
[25] Hughes D, Salathé M. An open access repository of images on plant health to enable the development of mobile disease diagnostics[EB/OL]. (2016-04-12)[2022-04-18]. https://arxiv. org/abs/1511. 08060.
[26] Thapa R, Snavely N, Belongie S, et al. The Plant Pathology 2020 challenge dataset to classify foliar disease of apples[EB/OL]. (2020-04-24)[2022-04-18]. https://arxiv.org/abs/2004. 11958.
[27] AiStudio. Pathological image of apple leaves[EB/OL]. (2019-11-17)[2022-04-18]. https: //aistudio. baidu. com/aistudio/datasetdetail/11591/0.
[28] 孙俊,朱伟栋,罗元秋,等. 基于改进MobileNet-V2的田间农作物叶片病害识别[J]. 农业工程学报,2021,37(22):161-169.
Sun Jun, Zhu Weidong, Luo Yuanqiu, et al. Recognizing the diseases of crop leaves in fields using improved Mobilenet-V2[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2021, 37(22): 161-169. (in Chinese with English abstract)
[29] Sandler M, Howard A, Zhu M, et al. Mobilenetv2: Inverted residuals and linear bottlenecks[C]//IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Salt Lack: IEEE, 2018: 4510-4520.
[30] Howard A, Sandler M, Chu G, et al. Searching for mobilenetv3[C]/IEEE/CVF International Conference on Computer Vision (ICCV). Seoul: IEEE, 2019: 1314-1324.
[31] 甘雨,郭庆文,王春桃,等. 基于改进EfficientNet模型的作物害虫识别[J]. 农业工程学报,2022,38(1):203-211.
Gan Yu, Guo Qingwen, Wang Chuntao, et al. Recognizing crop pests using an improved EfficientNet model[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2022, 38(1): 203-211. (in Chinese with English abstract)
[32] Tan Mingxing, Le Q. Efficientnetv2: Smaller models and faster training[C]//International Conference on Machine Learning (ICML). Online: IMLS, 2021: 10096-10106.
[33] Simonyan K, Zisserman A. Very deep convolutional networks for large-scale image recognition[EB/OL]. (2014-09-04)[2022-04-18]. https: //arxiv. org/abs/1409. 1556.
[34] Krizhevsky A, Sutskever I, Hinton G E. Imagenet ilassification with deep convolutional neural networks[J]. Advances in Neural Information Processing Systems, 2012, 25: 84-90.
[35] Bottou L. Stochastic Gradient Descent Tricks[M]. Berlin: Springer, 2012.
[36] 宋磊,李嵘,焦义涛,等. 基于ResNeXt单目深度估计的幼苗植株高度测量方法[J]. 农业工程学报,2022,38(3):155-163.
Song Lei, Li Rong, Jiao Yitao, et al. Method for measuring seedling height based on ResNeXt monocular depth estimation[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2022, 38(3): 155-163. (in Chinese with English abstract)
[37] Ma N, Zhang X, Zheng H T, et al. Shufflenet v2: Practical guidelines for efficient cnn architecture design[C]//15th European Conference on Computer Vision (ECCV). Munich: Springer, 2018: 116-131.
Model for identifying strong generalization apple leaf disease using Transformer
Xu Yanlei, Kong Shuolin, Chen Qingyuan, Gao Zhiyuan, Li Chenxiao※
(,,130118,)
Apple diseases have pose a serious risk on the income of orchards in recent years. An accurate and rapid identification of apple diseases can be great benefit to better prevent and control diseases. Most effort has been made in the laboratory to train the identification model, due mainly to the limited condition for the deliberately infect apples in the real orchard. However, most models cannot fully meet the requirement of the disease detection in the large-scale production. In this study, a deep learning model (called CaTNet) was proposed to extract both the global and local information from the diseases of apple leaf. The image data of disease was collected from the apple orchards in the Jilin Province of China. A total of 16,464 images were obtained from the several publicly available datasets with the laboratory and natural environmental data collected from the field. Firstly, a model structure was constructed with both Transformer and convolutional neural network (CNN). Global and local information was extracted from the original images using the two branches. The strong generalization ability of the model was improved to learn a wider variety of features. Meanwhile, the global features were acquired to improve the resistance of the model to interference. Secondly, the Transformer block in the Transformer branch was optimized to make the structure simpler. In addition, a channel compression and expansion module was designed in the Transformer branch, in order to reduce the training cost of CaTNet for the less channel dimension of the input features. Afterwards, the multiple multilayer perceptrons were replaced by the grouped convolutional layers to further improve the computational speed of the model. Thirdly, the lightweight CNN branch was constructed with an inverse residual structure to fuse the point convolution of the expanded channels with the 3×3 convolution of the extracted information. The CNN branch was utilized to extract the local features of the image. As such, the model was more sensitive to the fine-grained features. Finally, the concat operation was implemented to fuse the different output of features from the two branches. After that, the CNN branch was selected to extract the local features from the global ones, whereas, the Transformer branch was extracted the global from the local. The multiple features to be cycled were also improved the generalization of the model. A comparison was made to clarify the effect of different down-sampling on the two-branch network. Specifically, an accuracy rate of 79.35%, 74.06% and 67.95% were obtained using pooling, 3×3 size convolution kernel, and 1×1 size convolution kernel for the down-sampling, respectively. The CaTNet model with two branches showed a computational speed of 0.108 2 s/Frame), which was faster than the various deep learning models, such as the EfficientNetV2 s (0.383 2 s/Frame) and PVT t (0.177 8 s/Frame). Consequently, the two-branch structure can be expected to accommodate more computation for the much higher computational speed. This finding can provide a design approach to build the deep learning models with the high generalization capability, particularly on the training with the high accuracy under only easily accessible data.
image identification;agriculture; convolutional neural networks; apple leaf disease; Transformer model; strong generalization ability; feature fusion
10.11975/j.issn.1002-6819.2022.16.022
TP391.4
A
1002-6819(2022)-16-0198-09
徐艳蕾,孔朔琳,陈清源,等. 基于Transformer的强泛化苹果叶片病害识别模型[J]. 农业工程学报,2022,38(16):198-206.doi:10.11975/j.issn.1002-6819.2022.16.022 http://www.tcsae.org
Xu Yanlei, Kong Shuolin, Chen Qingyuan, et al. Model for identifying strong generalization apple leaf disease using Transformer[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2022, 38(16): 198-206. (in Chinese with English abstract) doi:10.11975/j.issn.1002-6819.2022.16.022 http://www.tcsae.org
2022-04-18
2022-08-01
吉林省科技厅国际科技合作项目(20200801014GH);长春市科技局重点科技攻关项目(21ZGN28)
徐艳蕾,博士,教授,博士生导师,研究方向为农业信息化。Email:yanleixu@jlau.edu.cn
李陈孝,博士,讲师,硕士生导师,研究方向为智能传感器及农业信息化。Email: licx@jlau.edu.cn

