多重耐药UPEC细菌毒力基因分布特征及进化分型分析
刘 斌,余子琦,闫 萌,戴彩华,张 玮,叶紫辰,傅 强,2
多重耐药UPEC细菌毒力基因分布特征及进化分型分析
刘 斌1,余子琦1,闫 萌1,戴彩华1,张 玮1,叶紫辰1,*傅 强1,2
(1.井冈山大学医学部,江西,吉安 343009;2.井冈山大学脊柱疾病研究所,江西,吉安 343009)
本研究对临床尿路感染病例病原菌耐药情况、毒力基因分布及其进化分型特征,以期为耐药性尿路感染病原防控提供参考。通过对收集的尿样进行细菌分离培养,结合革兰氏染色镜检、16S rDNA对细菌进行鉴定,利用K-B纸片扩散法对细菌进行药敏试验,并通过PCR扩增进行毒力基因及系统发育和进化分型分析。本研究成功检测出20株尿路致病性大肠杆菌(Uropathogenic, UPEC),药敏试验显示多数菌株具有多重耐药特征,该批UPEC对青霉素、红霉素、万古霉素耐药率达100%;对羧苄西林、氨苄西林、阿奇霉素耐药率达90%以上;对四环素、诺氟沙星、链霉素、耐药率达80%以上;对环丙沙星、庆大霉素、卡那霉素、氟氧沙星、头孢曲松耐药率达70%以上;对头孢噻肟、呋喃妥因、氯霉素耐药率在35%以上;对阿莫西林与多粘菌素B耐药率低,分别仅为10%与5%。六个毒力基因()检出率分别为85%、95%、85%、75%、25%、10%。其系统发育分型中,归属B2群的细菌最多,占比40%(n=8);D群次之,占比35%(n=7);其次为A群,占比25%(n=5);未检测到B1群菌株。通过MLST分型分析,20株大肠杆菌涉及10个ST型,其中ST131、ST648、ST744、ST1193型别较多。CRISPR序列扩增结果显示其中6株细菌含有CRISPR序列,提示不同菌株的进化差异。本研究通过对临床多重耐药UPEC进行毒力和分型特征分析,为临床有效防控耐药菌导致的尿路感染提供重要依据。
UPEC;多重耐药,毒力因子;分布特征;进化分型
大肠杆菌(,)属革兰氏阴性短杆菌,是构成肠道正常菌群的主要细菌。通常在肠道栖居条件下不致病,但当其侵入人体脏器时则会引起肠道外感染,其中以尿路感染为主[1-2]。尿路感染(Urinary Tract Infection,UTI)是由于尿路上皮细胞被大量病原菌入侵、繁殖而引发的炎症反应,是一种常见的细菌感染性疾病。95%以上的尿路感染是由单一细菌引起的,而大肠杆菌是引发UTI的主要致病菌,其占比超过80%,称为尿路致病性大肠杆菌(Uropathogenic, UPEC)[3]。根据UTI发病部位差异,可导致上尿路及下尿路感染,前者多为膀胱炎,后者则为肾盂肾炎,严重尿路感染患者甚至可能会因此引发菌血症、败血症,从而危及患者生命,而慢性肾盂肾炎最终可致发展为尿毒症[4-5]。
目前,临床上普遍采用抗生素疗法治疗UTI,但由于抗生素的滥用,以及耐药基因在菌群中的水平转移,导致许多病原菌逐渐出现多重耐药特征(Multi-drug-resistant, MDR)[6-7]。多重耐药是指对细菌对3类或3类以上临床使用的抗菌药物同时耐药。病原菌株多重耐药性的逐年增加,不仅导致尿路感染具有易再感染,易复发、难治疗的特点,给临床治疗带来了极大挑战[8-9]。而且会迫使抗生素使用量加大,增加药物不良反应,提高二重感染风险,给公共卫生安全带来巨大隐患。UPEC携带有多种毒力基因,如毒素因子、黏附因子和致病岛。黏附因子包含P型菌毛、 I型菌毛、FIC菌毛及Dr菌毛。研究表明,这类毒力因子与UPEC 的高致病性密切相关[10]。不同毒力因子彼此间相互作用,借助细菌突破宿主的防御系统,定植于泌尿道或入侵上皮细胞,引发对宿主有害的炎症[11]。因此,分析UPEC菌株毒力因子的分布情况,对研究其多重耐药性及致病性具有重要意义。
多位点序列分型(Multilocus Sequence Typing,MLST)技术通过扩增多个管家基因并进行测序,将每一组的不同等位基因排列组合成为一个基因型,根据被发现的时间顺序给每个位点的基因序列等位基因编号,细菌的等位基因编号按规定的顺序排列后可以得到等位基因谱,称为该菌的序列型(Sequence Type,ST),通过比较菌株的ST可判定不同样本的相关性以及多样性。该方法只需扩增细菌的管家基因后,将数据提交至MLST网站,即可比较不同菌株信息,具有较好的重复性和较高的分辨率,且操作方法简单快捷、数据准确。同时,在古细菌和细菌中广泛存在的CRISPR-Cas适应性免疫系统,即成簇的规律间隔的短回文重复序列(Clustered regularly interspaced short palindromic repeats, CRISPRs)也被广泛应用于多种致病微生物的进化分型研究[12-13]。CRISPR序列包括重复序列(Direct Repeat,DR)以及插入到重复序列中的间隔序列(Spacer)。Spacer在亲缘关系很近的细菌中也可以快速进化、高度可变,其在不同种属细菌,同一种属不同菌株间或不同血清型间具有明显差异。此外,间隔序列的不同间隔序列数量和排列方式以及多态性通常与CRISPR位点的活跃水平密切相关。因此,可以利用多态性极高的CRISPR位点对大肠杆菌进行分型[14-16]。CRISPR分型方法在分析数据方面优于其他分型方法,作为细菌分型方法之一被广泛应用。
UPEC作为尿路感染最常见的病原体,其不断出现的MDR特征严重危害人类健康。由于其自身携带的毒力基因及其所属的系统进化群、ST型与菌株的致病性有着密不可分的关系,本研究拟进行UPEC分离株的系统进化群、MLST、CRISPR分型和毒力基因的分布,以揭示细菌毒力与基因型的关系,为进一步研究多重耐药UPEC的致病机理和防治提供一定的科学依据。
1 材料与方法
1.1 菌株
本研究中分离的细菌均来源于吉安市某医院尿路感染病例的尿液标本,根据编号依次命名为U5002,U4909,U4863,U4789,U4647,U5433, U4528,U4469,U4275,U4228,U4196,U4059,U3945,U3626,U3520,U3519,U129,U612,U616,U4469,经LB平板划线培养分离,由本实验室保存。
1.2 实验仪器
恒温培养振荡器ZWYR-240购置于上海智城分析仪器制造有限公司,实验室专用超纯水机WP-UP-YJ-20购置于四川沃特水处理设备有限公司,电泳仪EPS300购置于上海天能科技有限公司,基因扩增仪ETC811-384型购置于苏州东胜兴业科学仪器有限公司,凝胶成像分析系统ZF-28型购置于上海嘉鹏科技有限公司。
1.3 实验试剂
琼脂粉(Agar)、氯化钠(NaCl)、酵母提取物(Yeast Extract)、胰蛋白胨(Tryptone)购置于生工生物工程(上海)股份有限公司,DM2000 DNA Marker、2*Taq PCR Master Mix(Dye)购置于康为世纪,引物由上海擎科生物科技有限公司合成,药敏纸片购置于北京天坛药物生物技术开发公司。液体LB培养基按照配方:NaCl 10.0 g,Yeast Extract 5.0 g,Tryptone 10.0 g,加超纯水SW定容至1.0 L,调节pH至7.4,121℃,20 min高压蒸汽法灭菌(配置固体培养基需在调节pH后加入15 g琼脂粉;半固体培养基加入7.5 g琼脂粉)。
1.4 细菌鉴定
1.4.1 形态学鉴定
用接种环沾取少量菌液均匀涂布至清洁的玻片中央,固定标本后,结晶紫染色1.0 min,随后用细水流缓慢冲洗干净;卢戈氏碘液媒染1.0 min,冲洗干净;然后95%酒精脱色30 s,冲洗;番红溶液复染30 s,随后用细水流缓慢冲洗干净;最后用吸水纸吸净玻片上的水分,待标本干燥后置于油镜下观察。
1.4.2 16S rDNA鉴定
采用煮沸法提取细菌DNA,吸取1.5 mL的菌液于1.5 mL Eppendorf 离心管中,于7000 rpm 离心10 min,倒掉上清液,瞬时离心后吸去残余上清液;用500 µL超纯水洗涤一次,于7000 rpm离心10 min,同法去上清液;再加入100 µL超纯水,悬浮于水浴锅中煮沸10 min,后于7000 rpm离心10 min,吸取上清液保存至-20 ℃中。选用16S rDNA通用引物357F和1115R(见表1.A),PCR体系(25 µL)为2*Taq PCR Master Mix,12.5 µL;模板,2.0 µL;357F,1.0 µL;1115R,1.0 µL;超纯水,8.5 µL。将该体系置于PCR仪中进行基因扩增,调节PCR反应程序为:94 ℃ 5 min,94 ℃40 s,56 ℃40 s,72 ℃45 s,30个循环,72 ℃5 min。配制1%的琼脂糖凝胶,凝胶放入电泳仪中,将Marker D2000、PCR产物加入1%的琼脂糖凝胶孔内,调节电泳仪电压100 V,电流100 A,电泳40 min后取出凝胶。在紫外光分析仪下可见有800 bp左右位置的亮条带,将该亮条带切下,进行胶回收。最后将回收产物送于上海擎科生物科技有限公司测序,并将序列结果上传至NCBI BLAST网站(https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行16S rDNA同源性比对,并保存比对结果。

表1 PCR扩增相关引物
1.5 药敏试验
对1.4中鉴定为大肠杆菌的细菌用Kirby-Bauer(K-B)纸片琼脂扩散法进行药物敏感试验。将19种常用抗生素药敏纸片贴在已经接种好测试菌的平板上,置于恒温箱12 h后,测量其抑菌直径大小。根据临床实验标准化委员会(CLSI)2016版文件标准(如表2),判定菌株为耐药(Resistance,R) 、中介(Intermediary,I) 或敏感(Sensibility,S)。
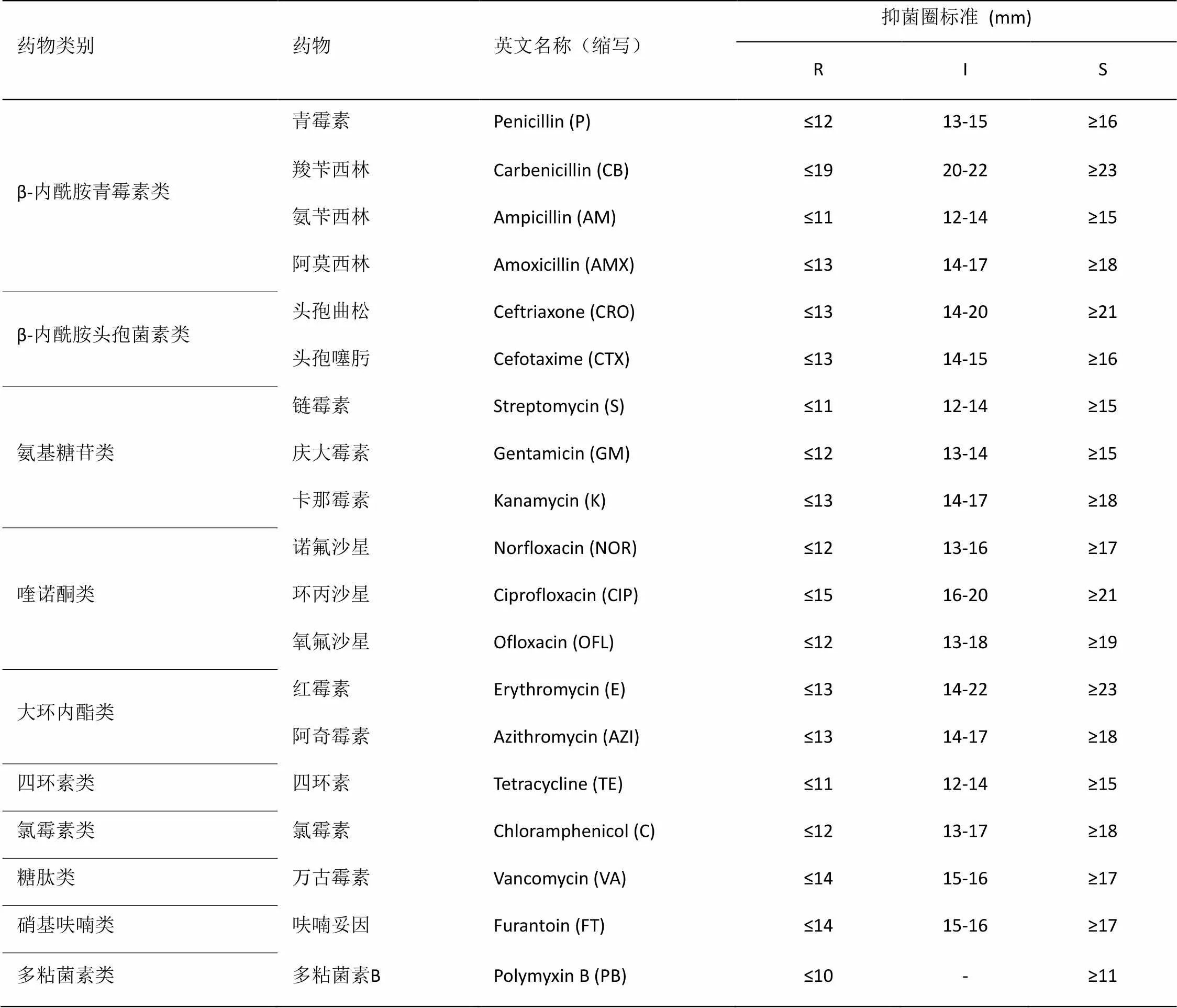
表2 细菌药物敏感性判断标准
1.6 毒力基因的检测
以1.4.2中提取的DNA作为模板,通过PCR扩增对大肠杆菌毒力因子进行检测,毒力因子引物见表1B。反应体系为20 µL(2*Taq PCR Master Mix液10 µL,上下游引物各1.0 µL,模板1.0 µL,补足SW至20 μL);反应条件与1.4.2中相同。将PCR产物进行1%琼脂糖凝胶电泳鉴定。
1.7 大肠杆菌系统发育分型
通过多重PCR对分离的细菌进行系统发育分型鉴定,涉及的三对引物见(表1C)。反应体系为25 µL(其中2*Taq PCR Master Mix液12.5 µL,三对引物上下游各1.0 µL,模板1.0 µL,补足SW至25 μL),将PCR产物进行电泳分析,根据图1判定分群结果。
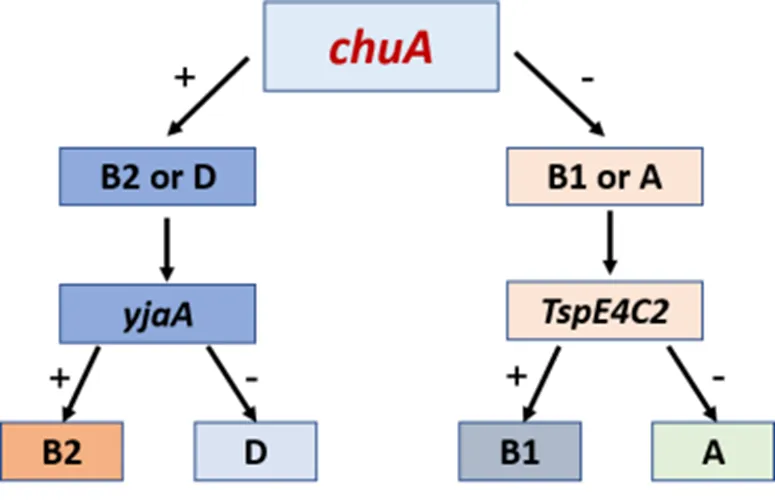
图1 大肠杆菌系统发育分型鉴定图示
1.8 细菌的MLST分型
MLST分型参照MLST数据库提供的 7 对管家基因序列进行(https://www.shigatox.net/ecmlst/cgi-bin/scheme)[17]。引物见表1D,扩增反应体系与反应条件与1.4.2中相同。取10 μL 产物进行电泳,并将扩增条带大小相符的胶回收产物送测序。测序结果上传至(https://pubmlst.org/bigsdb?db=pubmlst_escherichia_seqdef)网站中进行比对,获得各菌株等位基因值,并将7组等位基因值对比,从而得出该菌株ST型。
1.9 基于CRISPR序列的分型
实验中使用CRISPR引物(表1E)与1.4.1中模板进行PCR扩增,扩增反应体系以及反应条件同上。取10 μL PCR产物进行电泳,将扩增合格的产物送至上海擎科测序。测序结果上传(https://crispr:i2bc.paris-saclay.fr/Server/)网站进行序列分析。
2 结果
2.1 细菌的形态学观察与16S rDNA鉴定
分离菌株在LB平板上的菌落形态皆呈现为圆形凸起、半透明、光滑、湿润、灰白色,符合大肠杆菌菌落形态特征;分离株革兰氏染色形态呈红色,短杆状,两端圆形,单个或多个排列,提示为革兰氏阴性菌。选用16S rDNA通用引物57F,1115R 进行PCR鉴定,产物测序结果与NCBI中已报道的序列进行BLAST比对,同源性均达到97%以上,进一步证实为大肠杆菌。
2.2 药敏实验结果
药敏试验结果显示(如表3),20株大肠杆菌对青霉素、红霉素、万古霉素耐药率达100%;对羧苄西林、氨苄西林、阿奇霉素耐药率达90%以上;对四环素、诺氟沙星、 链霉素、耐药率达80%以上;对环丙沙星、 庆大霉素 、卡那霉素、氟氧沙星、头孢曲松耐药率达70%以上;对头孢噻肟、呋喃妥因、氯霉素耐药率在35%以上;对阿莫西林及多粘菌素B耐药率低,仅为10%和5%。
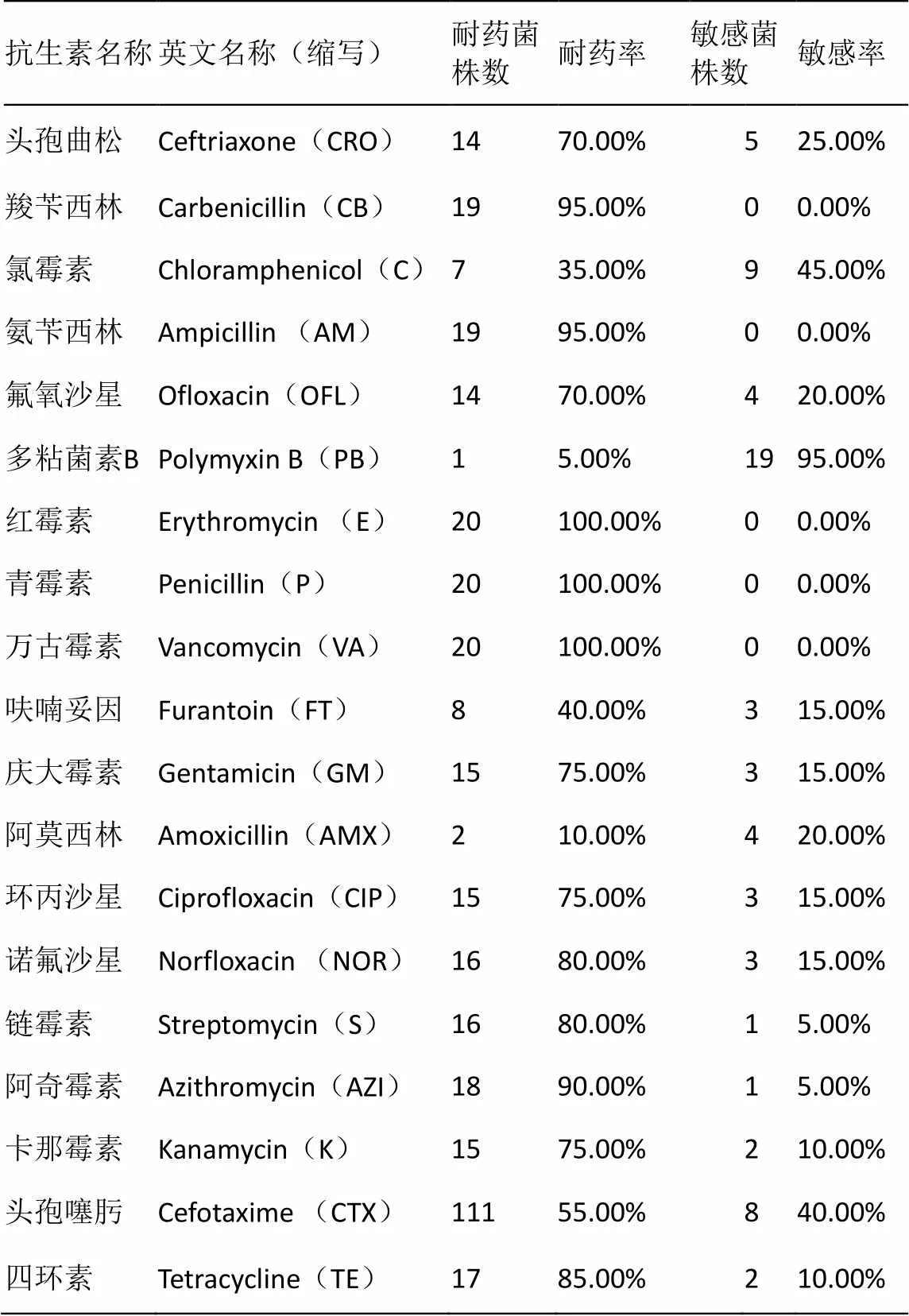
表3 尿路感染病例分离株的耐药性分析
2.3 毒力基因分布情况
毒力因子PCR扩增产物经凝胶电泳鉴定,结果显示毒力因子的检出率均较高,分别为85%、95%、85%、75%;与的检出率较低,为25%和10%(图2),各分离株所携带毒力因子情况见表4。
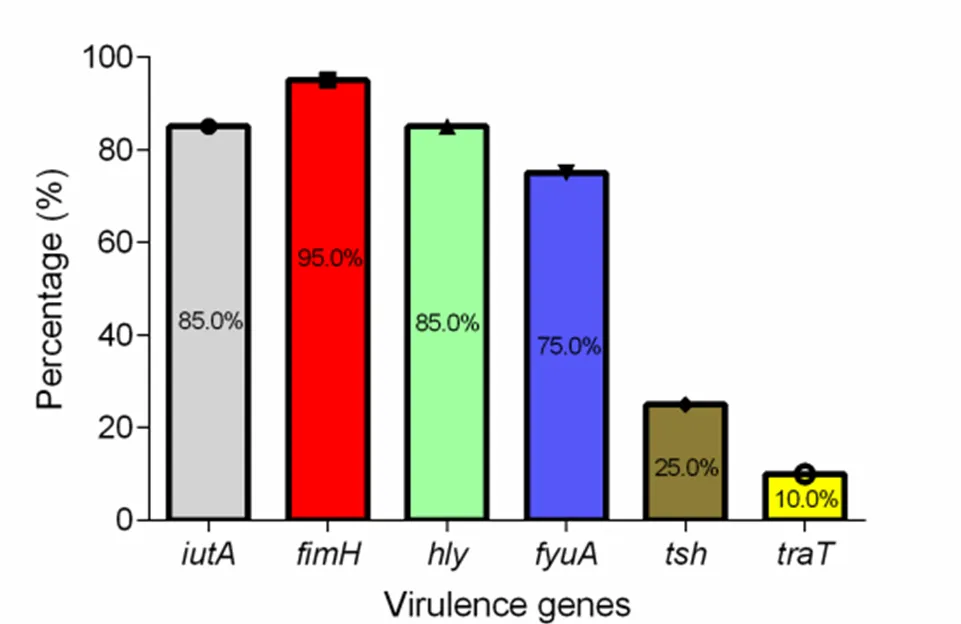
图2 分离株毒力基因检出率

表4 分离株毒力因子分布情况
2.4 系统发育进化分群
系统发育分析表明,根据携带标记基因不同,大肠杆菌可分为4个主要的群别(A、B1、B2和D),其中A和B1群常存在于共生群,少见致病性,而B2和D群为携带毒力相关基因的肠外病原菌,属于高致病群[18]。20株大肠杆菌中,B2群较多,有8株,占比40%; D群次之,有7株,占比35%;其次为A群有5株,占比25%;无B1群(图3);即所分离的20株菌株中有15株属于高致病群,提示临床分离株高致病性流行特征。
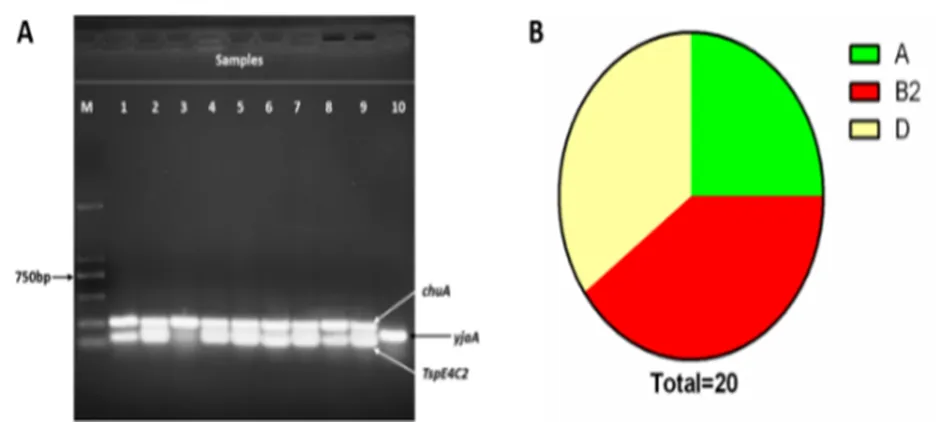
图3 部分菌株系统发育分群基因扩增结果(A);系统发育分群比例(B)
2.5 多位点序列分型结果
PCR扩增7对MLST分型基因(如图4),电泳鉴定后进行胶回收,送测序。测序结果在MLST数据库中进行比对,得到各菌株的ST型。结果显示(如表5),分离株归属10种ST型,其中ST131较多,有7株;其次为ST648、ST744、ST1193,分别有3株、2株、2株;ST569、ST450、ST88、ST73、ST44、ST10均有1株。ST744与ST10、ST44与ST10均只有一对等位基因的差异,分别为和。
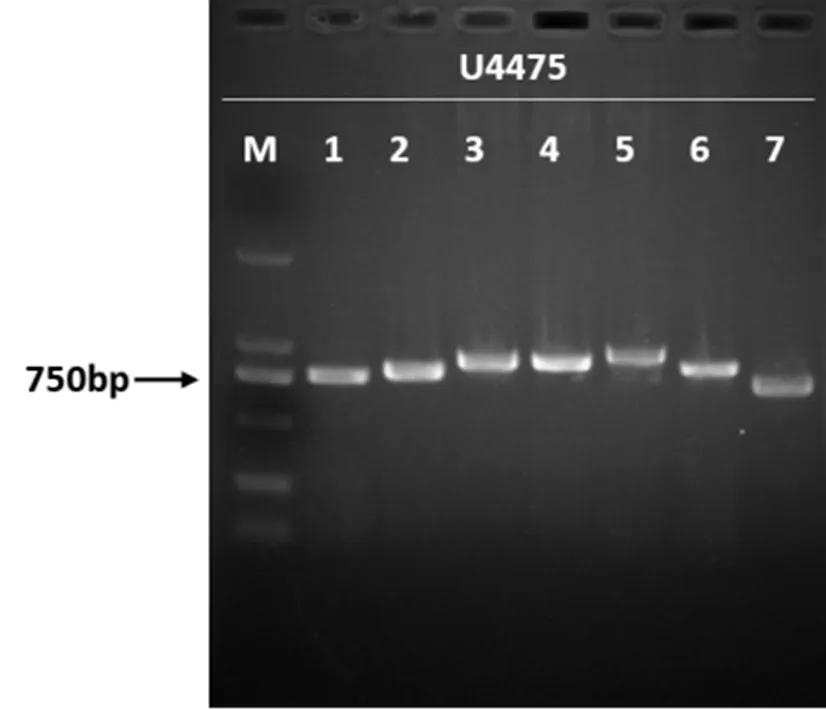
1-7泳道依次为adk、fumc、gyrB、icd、mdh、purA、recA基因
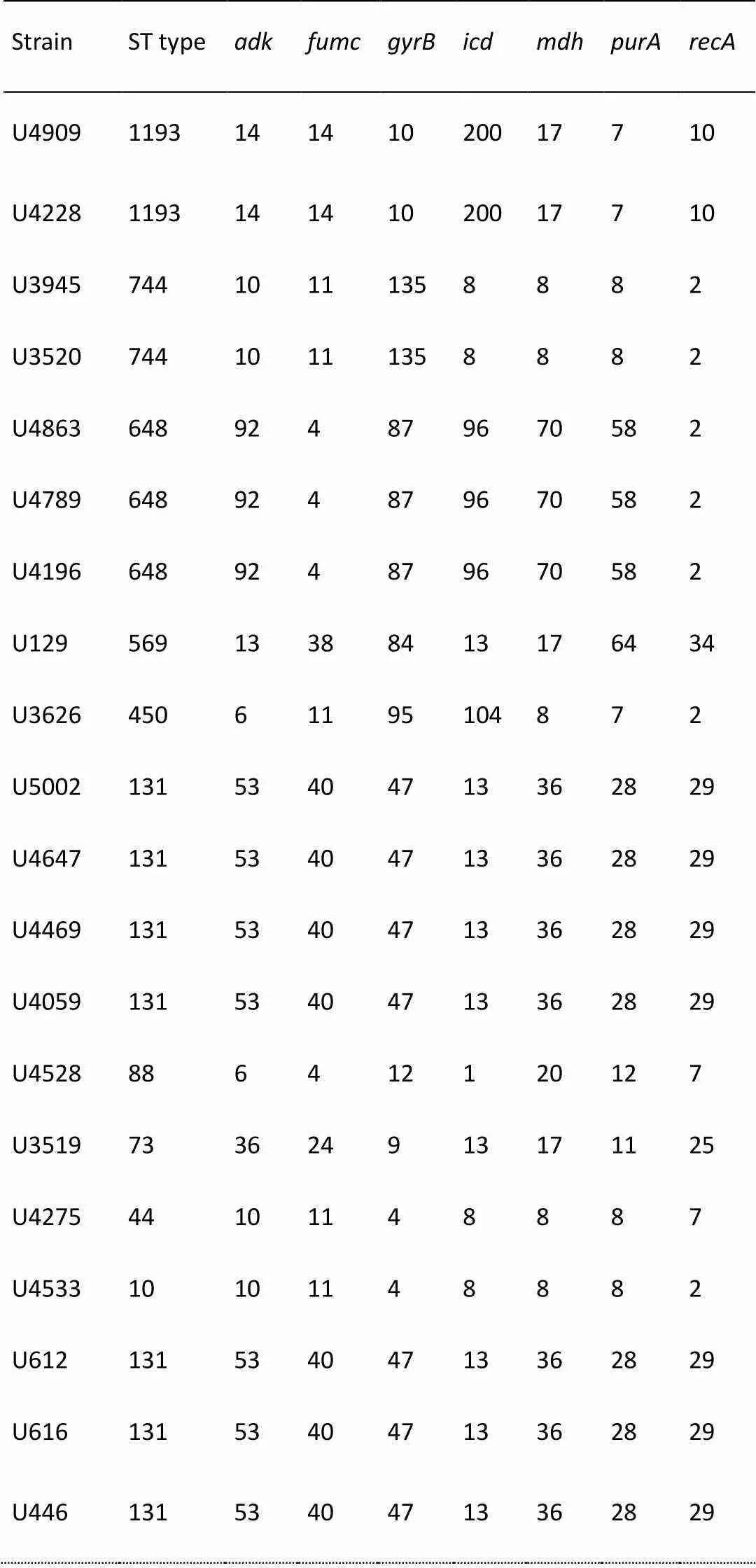
表5 UPEC菌株MLST分型特征
2.6 CRISPR分型结果
CRISPR序列扩增结果显示,分离株中有6株细菌含有CRISPR1序列,分别为U3520、U3626、U4275、U4533、U4789和U4863。所获得的CRISPR序列经CRISPRtionary(fr/CRISPRcompar/Dict/Dict.php)比对分析后发现,CRISPR重复序列DR主要为“CGGTTTATCCCCGC TGGCGCGGGGAACAC”和“CGGTTTATCCCCG CTGGCGCGGGGAACTC”两种,不同菌株携带54种不同的间隔序列Spacer(如图5),提示菌株的进化差异。其中U4789和U4863菌株的不同Spacer的顺序排列提示两者进化过程中的相似特征,在细菌分型上具有参考意义。

图5 分离株CRISPR序列特征图谱
3 讨论
UPEC广泛存在于自然界中,是最为常见的革兰氏阴性致病菌。近年来,随着抗生素滥用,耐药病原菌株不断产生,给尿路感染治疗带来了极大挑战,对于UPEC的耐药性分析及其进化特征的研究分析将有助于临床尿路感染的有效防控。药敏实验显示,本研究中所分离的20株临床UPEC菌株对青霉素、红霉素、万古霉素耐药率达100%;对羧苄西林、氨苄西林、阿奇霉素耐药率达90%以上;对四环素、诺氟沙星、链霉素、耐药率达80%以上;对环丙沙星、庆大霉素、卡那霉素、氟氧沙星、头孢曲松耐药率达70%以上;对头孢噻肟、呋喃妥因、氯霉素耐药率在35%以上;对阿莫西林和多粘菌素B耐药率较低,分别仅为10%和5%,提示该类抗生素适用于临床UPEC的感染治疗。
相关研究表明大肠杆菌的致病性主要与其毒力因子相关,如荚膜、铁摄取系统、脂多糖、毒素、I型菌毛、P菌毛等[19]。其致病性依赖于细菌的局部黏附定植,进而侵入机体产生毒素,毒力的强弱与其携带的毒力因子联系密切。本研究通过对分离菌株进行等6种毒力基因检测,结果显示分离株中均携带较多的毒力因子,其中、的检出率均在70%以上,这可能是造成其临床感染发病的主要原因。其中,的基因表达产物为黏附素的I型菌毛,黏附素具有黏附作用,是使病原菌可以在感染部位黏附而不被清除的重要原因,同时基因对细菌在感染部位能否产生趋化运动起关键作用[20-21]。基因编码的产物是溶血素,能够在红细胞膜表面形成穿孔能力,并使得红细胞溶解,溶血素对多种细胞比如红细胞、内皮细胞、粒细胞、单核细胞等均有细胞毒性[22]。与属于铁摄取系统毒力因子,可为大肠杆菌在低铁环境下摄取游离铁,为细菌在缺铁环境中增殖提供保障,前者与气杆菌素有关,后者则是耶尔森菌强毒力岛的标志基因之一[23-24]。通过对分离菌株的系统发育分型分析,结果显示分离株中归属B2群的菌株较多,占比41.18%;其次为D群与A群,占比29.41%。据报道,致病性大肠杆菌多为B2 群或D 群,A 和 B1 群致病性较低[25],本研究中有15株UPEC归属于B2或D群,进一步证实了其高致病特征,在免疫力低下的机体容易引发肠道外感染。
然而,随着研究深入,传统的分型方法已经不能满足研究者对病原微生物的分型、鉴定,以及菌株的溯源需求,MLST以其较好的重复性和较高的分辨率,被广泛应用于多种致病微生物的进化研究中,且操作方法简单快捷、数据标准、实验结果具有较高可比性。本研究中分离的UPEC菌株共检测到10种ST型, ST131有7株,ST648有3株,是主要流行型。同为ST1193和同为ST744的菌株分别具有相同的毒力基因,但同为ST648和ST131的菌株的毒力基因检测结果并不相同,说明ST型相同的菌株其毒力仍存在差异,可能与其他因素相关。
CRISPR系统通过适应性免疫功能从而使细菌可以逃避外源噬菌体的侵袭,并促进噬菌体与细菌的共进化。CRISPR序列中的间隔序列即使在亲缘关系很近的菌株中也可以迅速进化,是进化最快的基因组元件,其间隔序列的多样性为细菌的分型提供了具有高灵敏度和强分辨率的可靠方法。本实验检测到6株细菌含有CRISPR1序列,其重复序列包括两种,两种碱基序列仅有一个碱基差异。间隔序列差异性较大,数量与碱基序列均不相同。CRISPR 系统会因为外源噬菌体以及质粒的侵袭而改变,不同的菌株间的亲缘关系可以从其CRISPR间隔序列反映出来,亲缘关系越近的菌株,其间隔序列越相似,反之,亲缘关系较远的菌株之间间隔序列的差异性较大[14,26]。有学者研究发现,除间隔序列外,CRISPR的重复序列也参与细菌的进化过程,在大肠杆菌中CRISPR的重复序列会引起与细菌耐热性相关的基因的染色体变化,其染色体区域的复制,会使大肠杆菌的耐热性有所升高[27]。而本研究中分离株CRISPR序列检出率较其他类型大肠杆菌低[14],且仅具有单一的CRISPR位点,这可能与该类细菌经历的水平基因转移或可移动遗传元件的侵袭程度不同有关。
总之,本研究发现临床分离的UPEC菌株具有多重耐药特征,建议临床治疗使用多粘菌素B或阿莫西林等敏感抗菌药物。UPEC菌株毒力基因的检出率较高,与其高致病性存在密切联系。MLST分型发现分离株具有10种ST型,其中ST131、ST648、ST744、ST1193型菌株较为流行。本研究针对临床多重耐药UPEC进行了毒力和分型特征分析,为临床有效防控耐药菌导致的尿路感染提供重要依据。
[1] 戴彩华,张玮,刘斌,等.一株尿路致病性大肠杆菌噬菌体的分离鉴定及生物学特性分析[J].井冈山大学学报:自然科学版,2021,42(3):52-58.
[2] Lagha R, Ben Abdallah F, Al-Sarhan B O, et al. Antibacterial and biofilm inhibitory activity of medicinal plant Essential oils against escherichia coli isolated from UTI patients [J]. Molecules, 2019, 24(6):1161.
[3] Flores-Mireles A L, Walker J N, Caparon M, et al. Urinary tract infections: epidemiology, mechanisms of infection and treatment options [J]. Nat Rev Microbiol, 2015, 13: 269-284.
[4] Czajkowski K, Bros-Konopielko M, Teliga-Czajkowska J. Urinary tract infection in women [J]. Prz Menopauzalny, 2021, 20: 40-47.
[5] 周宏伟.肠杆菌科细菌耐药机制以及UPEC对HeLa细胞的致病性研究[D].杭州:浙江大学,2009.
[6] Ballesteros-Monrreal M G, Arenas-Hernandez M M, Enciso-Martinez Y, et al. Virulence and resistance determinants of uropathogenic escherichia coli strains isolated from pregnant and non-pregnant women from two states in mexico [J]. Infect Drug Resist, 2020, 13: 295-310.
[7] Madrazo M, Esparcia A, Lopez-Cruz I, et al. Clinical impact of multidrug-resistant bacteria in older hospitalized patients with community-acquired urinary tract infection [J]. BMC Infect Dis, 2021, 21: 1232.
[8] Mazzariol A, Bazaj A, Cornaglia G. Multi-drug-resistant Gram-negative bacteria causing urinary tract infections: a review [J]. J Chemother, 2017, 29: 2-9.
[9] 常国伟,周航,胡文,等. 2013 ~ 2017年医院尿路感染住院患者抗菌药物应用分析[J]. 井冈山大学学报:自然科学版,2019,40(4):103-106.
[10] Ribic R, Mestrovic T, Neuberg M, et al. Effective anti-adhesives of uropathogenic Escherichia coli [J]. Acta Pharm,2018, 68: 1-18.
[11] Pakbin B, Bruck W M, Rossen J W A. Virulence factors of enteric pathogenic escherichia coli: A review [J]. Int J Mol Sci, 2021, 22.
[12] 黄涛,山珊,黄艳梅,等.大肠埃希氏菌的分型方法及其研究进展[J]. 微生物学通报,2020,47(3):892-902.
[13] Kubelova M, Kolackova I, Gelbicova T, et al. Virulence properties of mcr-1-positive escherichia coli isolated from retail poultry meat [J]. Microorganisms, 2021, 9(2):308.
[14] Fu Q, Su Z, Cheng Y, et al. Clustered, regularly interspaced short palindromic repeat (CRISPR) diversity and virulence factor distribution in avian escherichia coli [J]. Res Microbiol, 2017, 168: 147-156.
[15] Dong H, Cui Y, Zhang D. CRISPR/Cas technologies and their applications in escherichia coli [J]. Front Bioeng Biotechnol, 2021, 9: 762676.
[16] 王建,邱少富,宋宏彬,等. CRISPR在细菌分型和进化中的研究进展[J]. 生物技术通讯,2013,24(3):414-417.
[17] 张立伟,石玉祥,张永英,等. 鸡源大肠杆菌毒力基因检测及分子流行特征[J].微生物学报,2020,60(11):2498- 2510
[18] Clermont O, Bonacorsi S, Bingen E. Rapid and simple determination of the escherichia coli phylogenetic group [J]. Appl Environ Microbiol, 2000, 66: 4555-4558.
[19] Johnson J R, Van Der Schee C, Kuskowski M A, et al. Phylogenetic background and virulence profiles of fluoroquinolone-resistant clinical escherichia coli isolates from the netherlands [J]. J Infect Dis, 2002, 186: 1852-1856.
[20] Hung C S, Bouckaert J, Hung D, et al. Structural basis of tropism of Escherichia coli to the bladder during urinary tract infection [J]. Mol Microbiol, 2002, 44: 903-915.
[21] Sokurenko E V, Feldgarden M, Trintchina E, et al. Selection footprint in the FimH adhesin shows pathoadaptive niche differentiation in Escherichia coli [J]. Mol Biol Evol,2004,21:1373-1383.
[22] 李丹.辽宁部分地区鸡源大肠埃希氏菌分离株毒力基因检测与耐药性分析[D].沈阳:沈阳农业大学,2020.
[23] 刘红玉,王君玮,王娟,等. 禽大肠杆菌毒力因子的研究进展[J]. 中国动物检疫, 2013,30(10):25-29.
[24] Forsyth V S, Himpsl S D, Smith S N, et al. Optimization of an experimental vaccine to prevent escherichia coli urinary tract infection [J]. mBio, 2020, 11(2):e00555-20.
[25] Tan C, Tang X, Zhang X, et al. Serotypes and virulence genes of extraintestinal pathogenic Escherichia coli isolates from diseased pigs in China [J]. Vet J, 2012, 192: 483-488.
[26] 白芷烨,汪雯,吉小风,等. CRISPR在食源性致病菌进化分析、检测分型及毒力耐药调控中的应用进展[J]. 生物工程学报,2021,37(7):2414-2424.
[27] Touchon M, Charpentier S, Clermont O, et al. CRISPR distribution within the escherichia coli species is not suggestive of immunity-associated diversifying selection [J]. J Bacteriol, 2011, 193: 2460-2467.
DISTRIBUTION CHARACTERISTICS OF VIRULENCE GENES AND EVOLUTIONARY TYPING OF MULTIDRUG-RESISTANT UPEC ISOLATES
LIU Bin1, YU Zi-qi1, YAN Meng1, DAI Cai-hua1, ZHANG Wei1, YE Zi-chen1,*FU Qiang1,2
(1. Health Science Center, Jinggangshan University, Ji’an, Jiangxi 343009, China; 2. Institute of Spinal Diseases, Jinggangshan University, Ji’an, Jiangxi 343009, China)
This studyinvestigated the drug resistance, virulence gene distribution and evolutionary typing characteristics of pathogens in clinical cases of urinary tract infection, so as to provide reference for the prevention and control of drug-resistant urinary tract infection pathogens. The collected urine samples were isolated and cultured, and the bacteria were identified by Gram staining and 16S rDNA. The bacteria were tested for drug sensitivity by K-B disk diffusion method, and the virulence genes and phylogenetic and evolutionary typing were analyzed by PCR amplification. In the study, 20 strains of uropathogenic(UPEC) were successfully isolated. The drug sensitivity test showed that most strains had the characteristics of multiple drug resistance. The drug resistance rates of this batch of UPEC to penicillin, erythromycin and vancomycin reached 100%, more than 90% to carbenicillin, ampicillin and azithromycin, more than 80% to tetracycline, norfloxacin, streptomycin and streptomycin, more than 70% to ciprofloxacin, gentamicin, kanamycin, floxacin and ceftriaxone, more than 35% to cefotaxime, nitrofurantoin and chloramphenicol; The resistance rates to amoxicillin and polymyxin B were low, only 10% and 5% respectively. The detection rates of six virulence genes (,,,,,) were 85%, 95%, 85%, 75%, 25% and 10% respectively. In its phylogenetic typing, the bacteria belonging to group B2 are the most, accounting for 40% (n=8), group D takes the second place, accounting for 35% (n=7), the third is group A, accounting for 25% (n=5); Group B1 strains weren’t detected. Through MLST typing analysis, 20 strains ofinvolved 10 ST types, including ST131, ST648, ST744, ST1193. CRISPR sequence amplification results showed that 6 strains of bacteria contained CRISPR sequence, suggesting the evolutionary differences of different strains. This study analyzed the virulence and typing characteristics of clinical multidrug-resistant UPEC, which provided a significant basis for clinical effective prevention and control of urinary tract infection caused by drug-resistant bacteria.
UPEC; multi-drug resistance; virulence factor; distribution characteristics; evolutionary typing
Q936
A
10.3969/j.issn.1674-8085.2022.06.007
1674-8085(2022)06-0040-10
2022-07-06;
2022-10-15
国家自然科学基金项目(31860711, 52163015);国家级大学生创新创业训练计划项目(202210419006);吉安市指导性科技计划项目[吉市科计字(2022)6号6]
刘 斌(1979-),男,江西吉安人,副教授,博士,主要从事病原微生物与免疫方面的研究(E-mail: 1902282875@qq.com);
*傅 强(1988-),男,江西吉安人,讲师,博士,主要从事原微生物与免疫方面的研究(E-mail: fqiang9@126.com).

