番茄基因组DNA不同微量提取方法的比较及适用性分析
王海英,刘航空
(西北农林科技大学 园艺学院,陕西杨凌 712100)
随着分子生物学的研究推进,高通量测序技术的不断完善,大量的园艺作物相继完成基因组测序,极大地推动了遗传基因组学、功能基因组学等方面的研究[1]。科研工作者可以开展种质亲缘关系确立、遗传多样性评估、种子纯度测定、分子标记检测、遗传图谱构建等遗传学试验,还可以进行候选基因的克隆与功能验证、目标基因等位变异的发掘、转基因后代检测等功能基因组试验[2-4],而这些试验都离不开基因组DNA的提取,根据DNA提取原理,基本步骤如下:植物组织破碎(可分为简易破碎和深度研磨);化学试剂或物理加热煮沸法裂解细胞膜释放基因组DNA;化学试剂或离心去除与DNA结合的组蛋白等;预冷的异丙醇、醋酸钠、醋酸钾等试剂沉淀核酸、DNA样品纯化,最后分离获得植物基因组DNA(图1)[5-17]。不同的试验对DNA的浓度和质量要求不同,故提取方法也不尽相同。
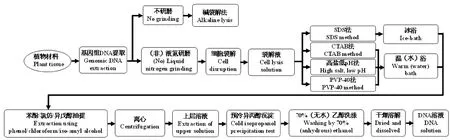
图1 不同DNA提取方法流程示意图Fig.1 Schematic diagram of different DNA extraction methods
本研究比较了当前实验室广泛采用的几种植物基因组DNA提取方法,并通过不同试验检测各方法获得DNA的质量和适用范围,进而提出一套基于不同试验目的的DNA提取方案,为后续研究提供技术支持和参考。
1 材料与方法
1.1 试验材料
番茄材料为Micro-TOM(Solanumlycopersicumcv. Micro-Tom)幼苗(4周)、成株叶片(2个月)和果实,均由西北农林科技大学园艺学院实验教学管理中心提供。
1.2 主要仪器设备
冷冻干燥器(CoolSafeTM110-4 L,丹麦)、QIAGEN研磨仪(Tissuelyzer,德国)、通风橱、台式离心机(eppendorf 5810R,德国)、紫外分光光度仪NanoDrop ND2000/2000C(Thermo ScientificTM,美国)、PCR仪(S-1000 Bio-Rad,美国)、384孔PCR仪(CHEMIDOE XRS+, Thermo ScientificTM,美国)、电泳仪(北京六一DYY-6)、凝胶成像系统(Bio-rad Gel Doc)、酶标仪FLUOstar Omega(BMG LABTECH,德国)等。
1.3 试验方法
采用5种方法提取番茄基因组DNA,即碱裂解煮沸法[16]、SDS(十二烷基肌氨酸钠)冰浴法[18]、改良的CTAB(十六烷基三乙基溴化铵)法[19]、高盐低pH法[20]、PVP-40(聚乙烯吡咯烷酮)法[21]。本研究参考以上DNA提取方法的同时并进行了部分操作环节的改进。
1.3.1 碱裂解煮沸法 该方法样品无需研磨。取番茄嫩叶、成株叶片和去皮果实各50 mg,用剪刀对组织进行简单破碎后分别放入2.0 mL离心管中,加入100 μL 0.4 mol/L NaOH溶液,沸水浴1 min。取20 μL上清液置于1.5 mL离心管中,加入80 μL TE缓冲液,混匀,备用。
1.3.2 SDS冰浴法 取番茄嫩叶、成株叶片和去皮果实材料各200 mg分别装入2 mL离心管,放入两个无水乙醇处理过的直径4 mm钢珠,然后将样品冷冻干燥。利用组织研磨仪对干燥的番茄材料进行研磨,频率30次/s,时间1 min左右。研磨完毕后,按照如下步骤进行:向离心管中加入800 μL、4 ℃预冷的SDS提取液(0.1 mol/L Tris-HCl、10 mmol/L EDTA、2% SDS,总pH 8.0),匀质化后,加入等体积(800 μL)酚∶氯仿∶异戊醇(25∶24∶1,pH=8);充分颠倒混匀30 s后,冰上静置20 min;4 ℃、12 000 r/min离心10 min;吸取650 μL上清液于另一个2 mL离心管中,加入等体积(650 μL)氯仿,轻柔上下颠倒,充分混匀; 4 ℃、12 000 r/min离心5 min;收集上清500 μL,转入新的1.5 mL的离心管中,加入50 μL 3 mol/L NaAc(pH 5.2)去色,然后加入500 mL预冷的异丙醇;轻柔颠倒数次混匀, -20 ℃条件冷冻30 min以上;4 ℃、 12 000 r/min离心3 min;弃上清,加入500 μL 75%酒精洗涤沉淀,4 ℃、12 000 r/min离心3 min;弃上清,加入500 μL无水乙醇洗涤沉淀,4 ℃、12 000 r/min离心3 min;离心后立即倒掉液体,自然风干后加入100~200 μL TE缓冲液或双蒸水(含有10 mg/mL的RNA酶)溶解DNA,置于37 ℃恒温箱约15 h,使RNA消解;置于-20 ℃保存,备用。
1.3.3 改良的CTAB温浴法 该方法样品研磨方式同“1.3.2”,研磨完成后步骤如下:向离心管中加入800 μL 65 ℃预热的2% CTAB抽提缓冲液(0.1 mol/L Tris-HCl,1.4 mol/L NaCl,20 mmol/L EDTA,2% CTAB,2% β-巯基乙醇,总pH 8.0),并匀质化;充分颠倒混匀后,置于65 ℃水浴或恒温箱中40~50 min,期间取出2~3次上下颠倒混匀;冷却2 min后加入等体积(800 μL)酚∶氯仿∶异戊醇(25∶24 ∶1,pH=8),充分振荡2~3 min使其混匀;4 ℃、12 000 r/min离心10 min;缓慢吸取600 μL上清液于1.5 mL离心管中,加入60 μL 3 mol/L NaAc(pH 5.2)去色,然后加入600 μL预冷异丙醇;缓慢摇动离心管30 s,使有机层与水层充分混合至絮状物出现,-20 ℃静置不少于30 min;4 ℃、12 000 r/min离心5 min;离心后立即倒掉液体,切勿将白色DNA沉淀倒出,再加入500 μL 75%乙醇洗涤沉淀,4 ℃、12 000 r/min离心3 min;弃上清,自然风干后加入100~200 μL TE缓冲液或双蒸水(含有10 mg/mL的RNA酶)溶解DNA,置于 37 ℃恒温箱约15 h,使RNA消解;置于-20 ℃保存,备用。
1.3.4 高盐低pH法 该方法样品研磨方式同“1.3.2”,研磨完成后步骤如下:向离心管中加入800 μL 65 ℃预热的高盐低pH提取液(0.1 mol/L NaAC、50 mmol/L EDTA、0.5 mol/L NaCl、2.5% PVP、3% SDS、1% β-巯基乙醇,总pH 5.5),匀质化后65 ℃温育30 min,期间取出 1~2次上下颠倒混匀;室温、12 000 r/min离心10 min;吸取上清液600 μL加入2/3倍体积的 2.5 mol/L KAC(pH 4.8),冰上静置30 min;室温、12 000 r/min离心10 min;吸取500 μL上清液,加入等体积-20 ℃预冷的异丙醇, 轻柔混匀后-20 ℃条件静置30 min; 4 ℃、12 000 r/min离心5 min;立即倒掉液体,用500 μL 75%乙醇洗涤DNA沉淀1~2次,然后4 ℃、12 000 r/min离心3 min;立即倒掉液体,自然风干后加入 100~200 μL TE缓冲液或双蒸水(含有10 mg/mL的RNA酶)溶解DNA,置于37 ℃恒温箱约 15 h,使RNA消解;置于-20 ℃保存,备用。
1.3.5 PVP-40法 该方法样品研磨方式同 “1.3.2”。研磨完成后步骤如下:向离心管中加入800 μL无毒的PVP-40提取液(0.1 mol/L Tris-HCl,1.0 mol/L KCl,20 mmol/L EDTA,2% PVP-40,总pH 9.5),匀质化后置于65 ℃恒温箱内温育30 min,期间取出1~2次上下颠倒混匀;室温、12 000 r/min离心1 min,向管中加入1/3体积(267 μL)的5 mol/L KAC溶液(pH 5.8),室温静置20 min;室温、12 000 r/min离心15 min;吸取600 μL上清液,转移至1.5 mL离心管中,加入等体积-20 ℃预冷的异丙醇, 轻柔混匀,静置20 min;室温、12 000 r/min离心10 min;弃上清液,保留底部沉淀,并加入500 μL 75%乙醇洗涤沉淀1~2次,然后12 000 r/min离心3 min;弃上清液,沉淀自然风干后加入 100~200 μL TE缓冲液或双蒸水(含有10 mg/mL的RNA酶)使DNA溶解,置于37 ℃恒温箱约15 h,使RNA消解; 置于-20 ℃保存,备用。
1.4 DNA纯度与浓度检测
(1)配制0.8%的琼脂糖凝胶,取5种方法提取的DNA样品各10 μL点样,100~120 V恒压下电泳30~40 min,SYBRGreen染色,并在凝胶成像系统(Bio-rad Gel Doc)观察电泳条带。
(2)以TE缓冲液为对照,利用紫外分光光度仪NanoDrop ND2000/2000C测定5种方法提取的DNA样品浓度,利用230 nm、260 nm和280 nm处的吸收值比例(OD260 nm/OD230 nm和OD260 nm/OD280 nm)衡量DNA的质量。
1.5 引物设计与合成
为探索5种方法所提取的基因组DNA的适用范围,分别利用3种PCR扩增技术进行比较,分别为与番茄果实大小连锁的SNP标记solcap_snp_sl_100656[3]、番茄常用的内参基因肌动蛋白(actin)以及R2R3MYB类转录因子设计PCR引物[22]。其中solcap_snp_sl_100656的PCR产物大小为65 bp,内参基因actin的PCR产物大小为126 bp,R2R3MYB类转录因子PCR产物大小为1 359 bp。除了SNP标记设计为AQP引物(等位基因特异的定量PCR 基因分型系统),其他两个均为普通的PCR引物。对于设计好的AQP引物序列,需要在两条正向的引物5′端分别加上FAM或者HEX荧光的接头序列,其中FAM接头序列为5′-GAAGGTGACCAAGTTCATGCT-3′,HEX接头序列为5′-GAAGGTCGGAGTCAACGGATT-3′,用于AQP标记系统的检测[23]。以上引物均由上海生工生物工程股份有限公司合成。合成后的AQP引物经溶解后需配置成引物mix(两条正向引物为12 mmol/L,反向引物为30 mmol/L),其他引物正常溶解。引物序列及其他具体信息见表1。

表1 用于PCR的引物Table 1 Primers for PCR
1.6 AQP-SNP、普通PCR以及基因克隆扩增与检测
AQP的384板PCR反应体系总体积为 5 μL,其中HiGeno 2× Probe Mix B体积为 2.5 μL,引物mix为0.06 μL,DNA模板为2 μL(50~100 ng)。PCR扩增反应在 384孔PCR仪上进行,设置为差减反应程序(Touchdown): 94 ℃变性15 min;94 ℃变性20 s,65 ℃退火 60 s,每循环一次降0.8 ℃,10个循环;94 ℃变性 20 s,57 ℃退火60 s,32个循环[23]。PCR反应完毕后放在酶标仪FLUOstar Omega中读取终端荧光数据,然后将数据导入KlusterCaller软件进行基因分型分析。
内参基因普通PCR体系为15 μL,R2R3MYB基因扩增体系为25 μL,其中DNA模板为2 μL(50~100 ng),2× PCR反应mix 分别为7.5 μL和12.5 μL,ddH2O分别为5.5 μL和10.5 μL。扩增反应在96孔PCR仪中进行,基本程序为:94 ℃预变性10 min;94 ℃变性30 s, 55 ℃退火30 s,72 ℃延伸30 s(其中基因扩增延伸时间为1 min),共35个循环;72 ℃延伸10 min,12 ℃保存。扩增产物分别用6%聚丙烯酰胺凝胶(变性)和1.5%琼脂糖凝胶电泳进行检测,其中聚丙烯酰胺凝胶条件为200 V恒压、1.5 h,然后硝酸银银染显色;琼脂糖凝胶电泳条件为80~100 V恒压、30 min,SYBRGreen染色,然后凝胶成像系统(Bio-rad Gel Doc)观察电泳条带。
2 结果与分析
2.1 紫外分光光度仪比较DNA的纯度和浓度
对于纯DNA溶液,其OD260 nm/OD280 nm比值应为1.80左右,如果OD260 nm/OD280 nm大于 1.90,说明有RNA污染;如果小于 1.60,说明有蛋白质或酚类等杂质。而OD260 nm/OD230 nm一般大于2.0,如果低于2.0,说明DNA溶液中有残存的盐和小分子杂质,如多糖、氨基酸、酚等。从表2可以看出,5种DNA提取方法中,碱裂解蒸煮法提取的DNA浓度最低,质量最差;其他4种方法提取的DNA浓度差异不大,为180~220 ng/μL,SDS冰浴法和CTAB法提取的DNA质量最好,纯度最高;高盐低pH法和PVP-40法提取的DNA有小分子杂质,质量一般。比较样品材料,幼苗叶片提取的DNA质量最好,成株叶和果实之间差异不大。
表2 不同方法提取的番茄基因组DNA的纯度和浓度Table 2 Comparison of purity and concentration of different DNA extraction methods
2.2 琼脂糖电泳比较DNA的纯度
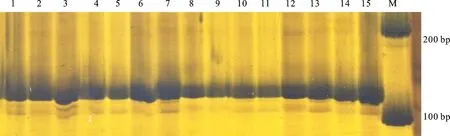
由图2可以看出,5种方法从番茄的幼苗、成株叶片和去皮果实中提取的DNA电泳条带分布较集中。其中碱裂解法提取的DNA样品电泳条带出现拖尾现象,说明样品中存在RNA、蛋白质等杂质,且DNA条带亮度比其他4种方法提取的DNA条带暗;其他4种方法没有明显拖尾现象,条带亮度没有明显差异,说明提取的基因组DNA质量比较理想。
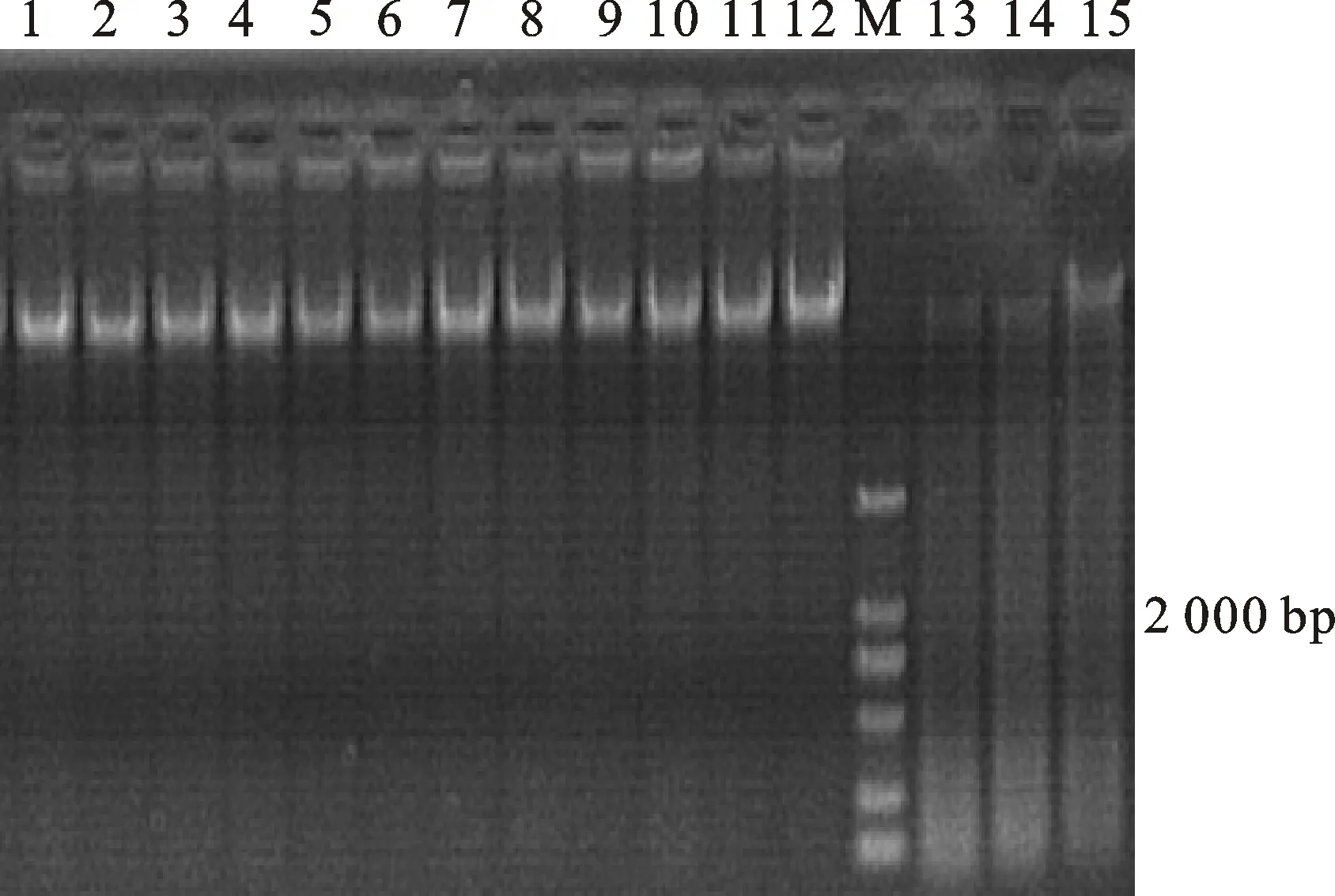
1~3号为PVP-40法;4~6号为高盐低pH法;7~9号为CTAB法;10~12号为SDS冰浴法;13~15号为碱裂解法;每种方法的点样顺序从左至右依次为幼苗、成株叶和果实;M为Marker
2.3 番茄基因组DNA不同PCR检测结果
图3显示,对于AQP标记体系,5种方法提取的各组织DNA样品均能成功扩增出SNP标记solcap_snp_sl_100656的产物,且呈现红色荧光,说明符合AQP检测体系的要求;对5种方法提取不同番茄组织获得的所有样品进行内参基因actin的部分小片段的扩增,并采用6%聚丙烯酰胺(变性)凝胶电泳进行检测,结果表明,所有电泳孔道均扩增出目标条带,完全符合小片段PCR扩增的要求(图4);利用R2R3MYB类转录因子的引物对所有方法提取的DNA进行基因扩增,并利用1.5%琼脂糖凝胶进行电泳检测,结果表明,除了碱裂解方法,其他4种方法提取的DNA样品均能扩增出目的片段(1 359 bp),得到稳定的谱带(图5),符合基因扩增的要求。
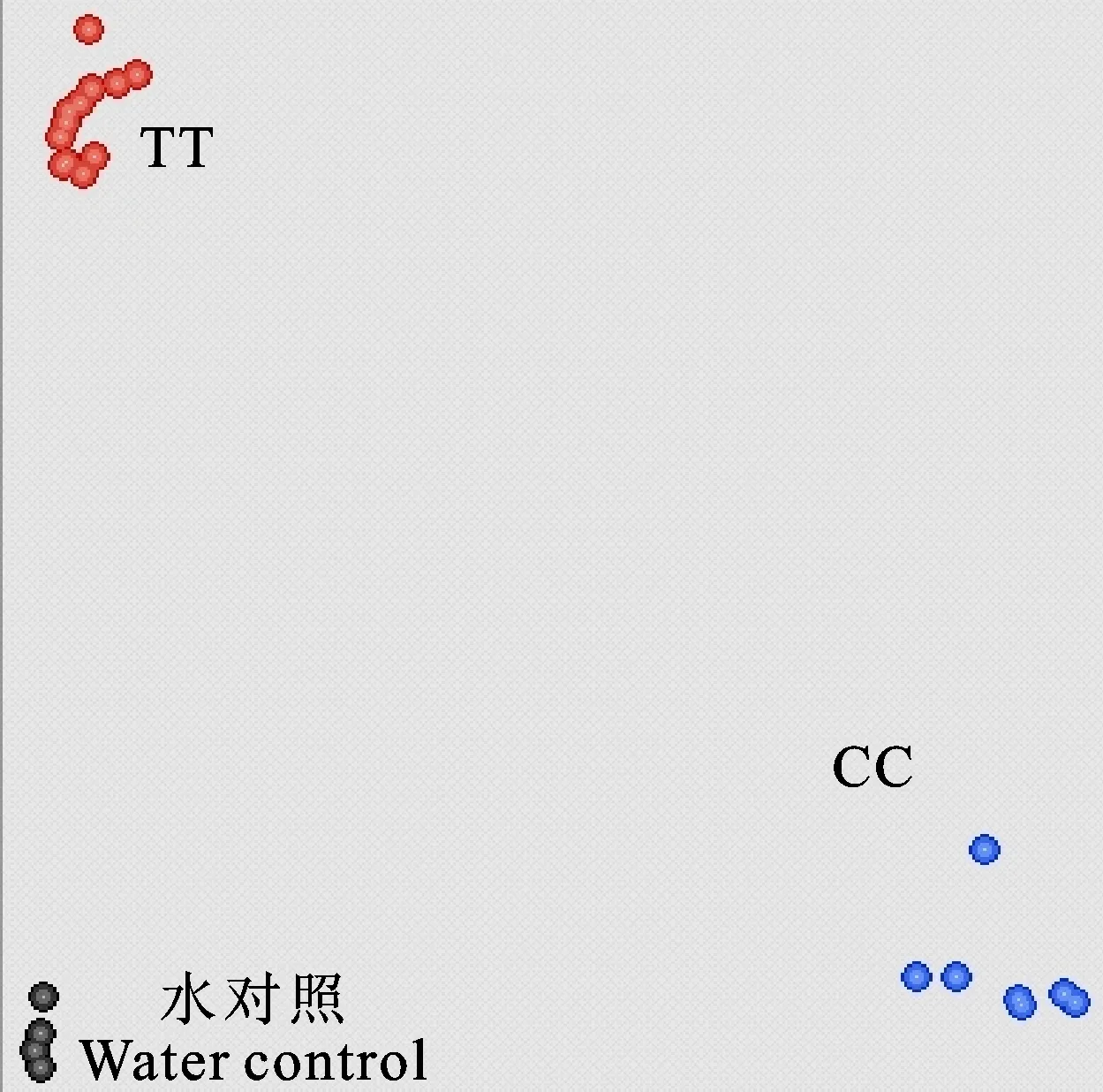
黑色点为水对照,蓝色点为荧光对照,红色点表示用不同提取方法提取的样品
1~3号为碱裂解法;4~6号为SDS冰浴法;7~9号为CTAB法;10~12号为高盐低pH法;13~15号为PVP-40法;每种方法的点样顺序从左至右依次为幼苗、成株叶和果实;M为Marker
3 讨 论
本研究比较了5种DNA提取方法:碱裂解法、SDS冰浴法、改良的CTAB法、高盐低pH法和PVP-40法获得番茄不同组织的基因组DNA质量和适用范围。比较可知,不论采用哪种提取方法,利用番茄幼苗组织获得的DNA质量均好于成株叶片和果实。由于植物成熟组织中DNA的提取有一定的难度,番茄成熟果实中次级代谢产物较多,在研磨后果实内的酚类物质很容易受到氧化而变褐色,进而影响DNA的提取。因此,在条件允许下,应优先选取幼嫩叶片提取基因组DNA。
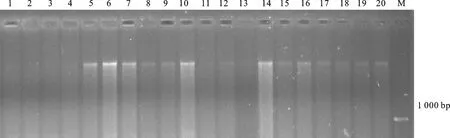
1-4号为碱裂解法;5-8号为SDS冰浴法;9-12号为CTAB法;13-16号为高盐低pH法;17-20号为PVP-40法;每种方法的点样顺序从左至右依次为幼苗、幼苗、成株叶和果实;M为Marker
通过不同试验检测5种提取方法获得DNA的适用范围,得出以下结论:碱裂解蒸煮法成本最低,试剂易得,无需深度研磨,操作简便,提取的番茄DNA浓度最低,质量最差,但能满足一般的分子标记检测试验,如AQP荧光标记和SSR小片段的扩增检测及大规模群体PCR检测的需要。SDS冰浴法和改良的CTAB法成本较高,试剂配制及操作步骤复杂,试验周期较长,但提取的DNA质量最好,更适合要求较高的分子生物学试验,缺点是两种方法均使用了毒性试剂,如酚、氯仿、异戊醇等,污染环境且对人体有害,如在教学试验中采用两种提取方法,容易引起学生的排斥心理,不利用提高学生教学实践的积极性。高盐低pH法和PVP-40法成本低,试剂配制要求低,操作步骤简便,提取时间短,获得的DNA质量虽不及SDS和CTAB法,但也能满足大部分生物学试验,具有一定的通用性,尤其在遗传定位、种质多样性评估、种子纯度检测、转基因植物检测等试验中需要大规模DNA提取时,SDS和CTAB法可有效节约时间成本,且所用的试剂无毒,非常适合于初学者和学生教学试验。建议在以后的教学和科学研究试验中,在满足试验对DNA质量要求前提下优先考虑高盐低pH法和PVP-40法提取基因组DNA。

