芝麻AP2/ERF基因家族生物信息学鉴定与分析
赵冰雪,聂功平,张小惠,黄清俊
(1.上海应用技术大学生态技术与工程学院,上海 201418;2.长江大学园艺园林学院,湖北 荆州 434023)
植物在其生命周期中会受到多种环境条件的影响,主要的极端环境条件,如水分胁迫,盐胁迫和温度胁迫等,限制了全球植物的生长,发育,生产力和地理分布,并降低其潜在价值[1-2]。当植物受到生物和非生物胁迫时,植物会通过一系列调控诱导或抑制基因的表达,进而提高植物对胁迫的耐受性或躲避胁迫损伤,以适应胁迫环境。转录因子是植物体内重要的环境响应调控因子[3],在植物逆境胁迫适应性中发挥重要作用。AP2/ERF(APETALA2/ethylene responsive factor)广泛分布于各种植物体内,是一种能够响应生物和非生物胁迫的重要转录因子,对于植物的生长发育与次级代谢起着重要作用。
AP2/ERF家族成员学包括至少1个AP2结构域,其结构域由60~70个氨基酸残基组成[4]。AP2/ERF家族分为AP2、RAV、Soloist和ERF亚族[5],AP2亚族编码2个AP2/ERF结构域[6-7],在调节植物生长发育中具有重要功能,包括叶表皮细胞特化[8]、花和胚珠发育[9-10]、小穗分生组织确定[11]和种子生长[12-13]等。RAV亚族编码1个AP2/ERF结构域和1个B3结构域[14],在调节靶基因响应乙烯[15]、油菜素内酯调控[16]、生物和非生物胁迫[17]中发挥重要作用。ERF是AP2/ERF转录因子家族的1个最大的亚族[18-19],编码1个AP2/ERF结构域,在生物胁迫[20]、干旱[21]、低温[22]、高盐[23]、热胁迫[24]、多重胁迫[25-26]等调控中发挥重要功能。
芝麻(Sesamum indicumL.)属于胡麻科(Pedaliaceae)胡麻属(Sesamum indicumLinn.),为传统油料作物。目前,对于芝麻AP2/ERF转录因子家族的全基因组分析鲜有报道。本研究基于生物信息学方法对芝麻基因组中AP2/ERF转录因子家族成员进行全基因组鉴定,对其理化性质、结构特征、保守基序、染色体定位、物种共线性、不同组织的表达特异性以及基因表达对胁迫响应等信息进行研究,旨在为AP2/ERF的进一步功能研究提供重要参考,也为芝麻分子遗传改良、抗逆育种研究奠定基础。
1 材料与方法
1.1 芝麻AP2/ERF家族基因的鉴定
从NCBI数据库(https://www.ncbi.nlm.nih.gov/genome/11560)下载芝麻基因组序列、编码基因序列、蛋白序列和注释文件等以供后续分析。从TAIR数 据库(https://www.arabidopsis.org/)下载拟南芥AP2/ERF基因家族蛋白序列作为种子序列,运用本地blast软件(https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/)从芝麻蛋白序列数据库查找芝麻AP2/ERF基因家族成员,通过Excel2000软件去除冗余并整理候选基因家族成员。然后从Pfam数据库(http://pfam.xfam.org/)下 载AP2/ERF基 因 家 族 的AP2(PF00847)和B3(PF02362)保守结构域隐马尔可夫模型,运用HMMER 3.3(http://eddylab.org/software/hmmer/hmmer-3.3.tar.gz)再次提取芝麻AP2/ERF基因家族蛋白序列。结合本地blast和HMMER结果交叉验证,并提交NCBI数据库SMARTBLAST检验序列完整性(https://blast.ncbi.nlm.nih.gov/smartblast/),剔除不相关的假阳性序列、删除重复的转录因子,最终确定芝麻AP2/ERF基因家族候选成员。提取芝麻AP2/ERF基因家族候选成员蛋白序列,运用在线分析工具(https://web.expasy.org/protparam/)预测蛋白理化性质。
1.2 序列比对和进化分析
1.3 保守结构域和motif预测
使 用NCBI在 线 平 台Batch CD-Search(https://www.ncbi.nlm.nih.gov/Structure/bwrps b/bwrpsb.cgi)进行芝麻候选AP2/ERF基因家族成员蛋白保守结构域的预测[27]。保守motif采用
MEME Suite(version 5.1.1)(http://memesuite.org/tools/meme)进行预测[28],motif预测参数设为20。使用Tbtools软件对保守结构域和motif结构预测结果进行可视化[29]。
1.4 染色体定位和共线性分析
从芝麻基因组注释信息中获取AP2/ERF基因家族成员位置信息,然后运用Tbtools软件进行可视化。大麦基因组数据库(https://webblast.ipkgatersleben.de/barley_ibsc/downloads/)和 玉 米基因组数据库(https://www.maizegdb.org/)下载基因组和注释文件,采用MCScanX方法结合Tbtools软件计算芝麻、大麦、玉米的AP2/ERF基因家族成员的共线性关系并进行可视化[30]。
1.5 芝麻AP2/ERF家族响应淹水胁迫的表达分析
芝麻表达谱数据GSE133186从GEO数据库下载(https://www.ncbi.nlm.nih.gov/gds/),根据基因ID从表达谱数据中筛选出AP2/ERF家族成员表达谱数据[27]。运用Excel 2010软件整理数据,分别计算处理组与对照组平均值,采用热图对比淹水胁迫下AP2/ERF家族成员表达差异,热图可视化分析采用Tbtools软件。
2 结果与分析
2.1 芝麻AP2/ERF 转录因子家族成员鉴定及理化性质分析
芝麻基因组中共鉴定出142个AP2/ERF家族基因成员,其编码179条AP2/ERF蛋白序列。在这些基因中,LOC105156293、LOC105177845、LOC105176383、LOC105155699、LOC105165339、LOC105158575、LOC105180195、LOC105180208、LOC105170679、LOC105170345、LOC105177600、LOC105176219、LOC105167013、LOC105162917、LOC105157874、LOC105167791、LOC105168482基 因 分 别 编 码2条AP2/ERF蛋 白 序 列;LOC105161130、LOC105175841基因分别编码3条AP2/ERF蛋白序列;LOC105166874、LOC105170556、LOC105170138基因分别编码4条AP2/ERF蛋白序列;LOC105166361基因分别编码8条AP2/ERF蛋白序列;其他基因分别编码1条AP2/ERF蛋白序列(见表1)。预测结果显示,AP2/ERF蛋白氨基酸数量范围121~697,分子量范围13 428.04~76 020.09 kD,原子数范围1872~10 367。其XP_020554228.1氨基酸数量、分子量和原子数最小,分别为697、76 020.09、10 367;XP_011077145.1氨基酸数量、分子量和原子数最大,分别为697、76 020.09、10 367;等电点范围4.50-10.24,XP_011093871.1等 电 点 最 小,XP_020553968.1等电点最大。脂肪系数反映了蛋白热稳定性,芝麻AP2/ERF转录因子蛋白脂肪系数范围47.98-70.60,总体热稳定性较高,Soloist亚家族的热稳定性均值最低,RAV亚家族的热稳定性均值最高。其中XP_011080300.1脂肪系数最小,XP_011096343.1脂肪系数最大。亲水性范围-1.091~-0.276,XP_011089073.1亲水性值最小,XP_011094016.1亲水性值最大,且平均系数均为负值,表明全部AP2/ERF蛋白均属于亲水性蛋白。
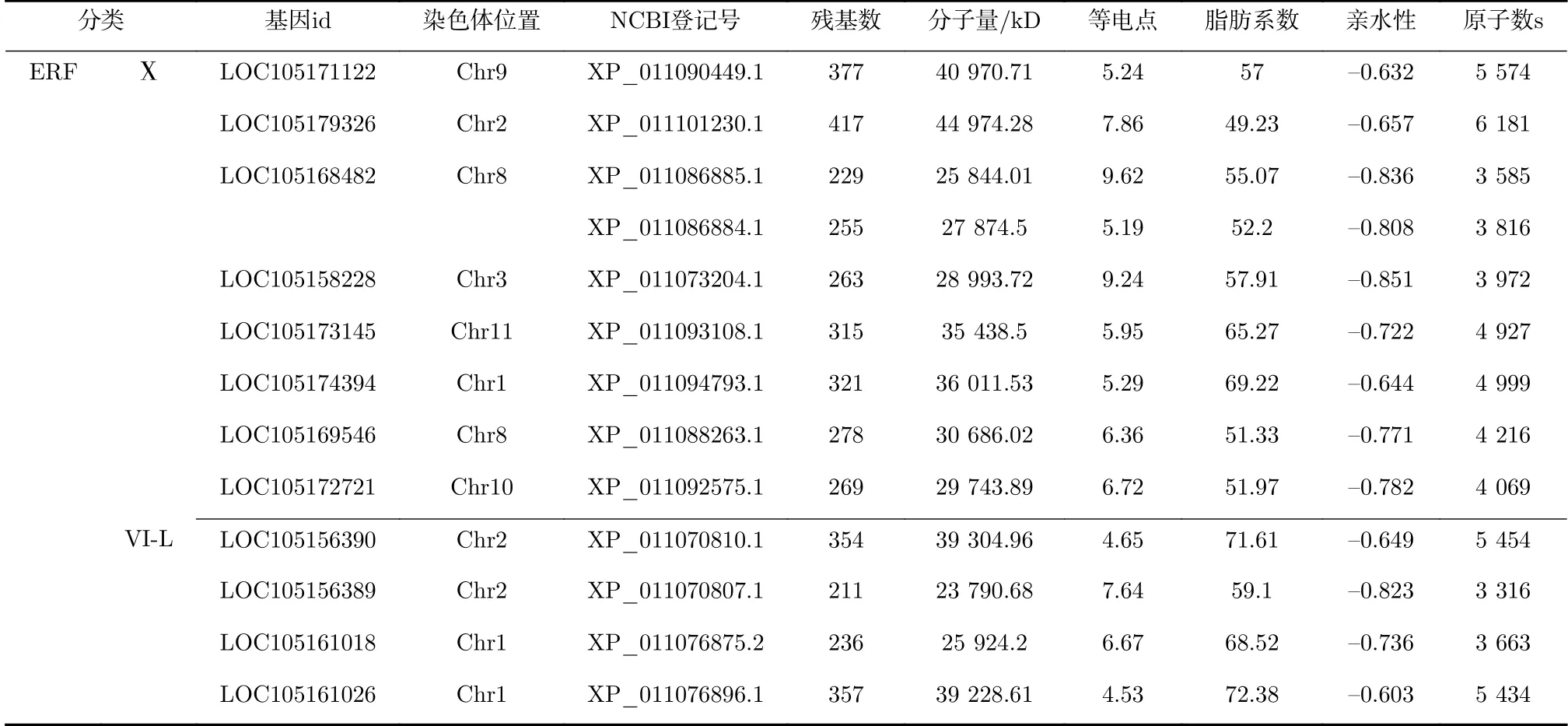
表 1(续)
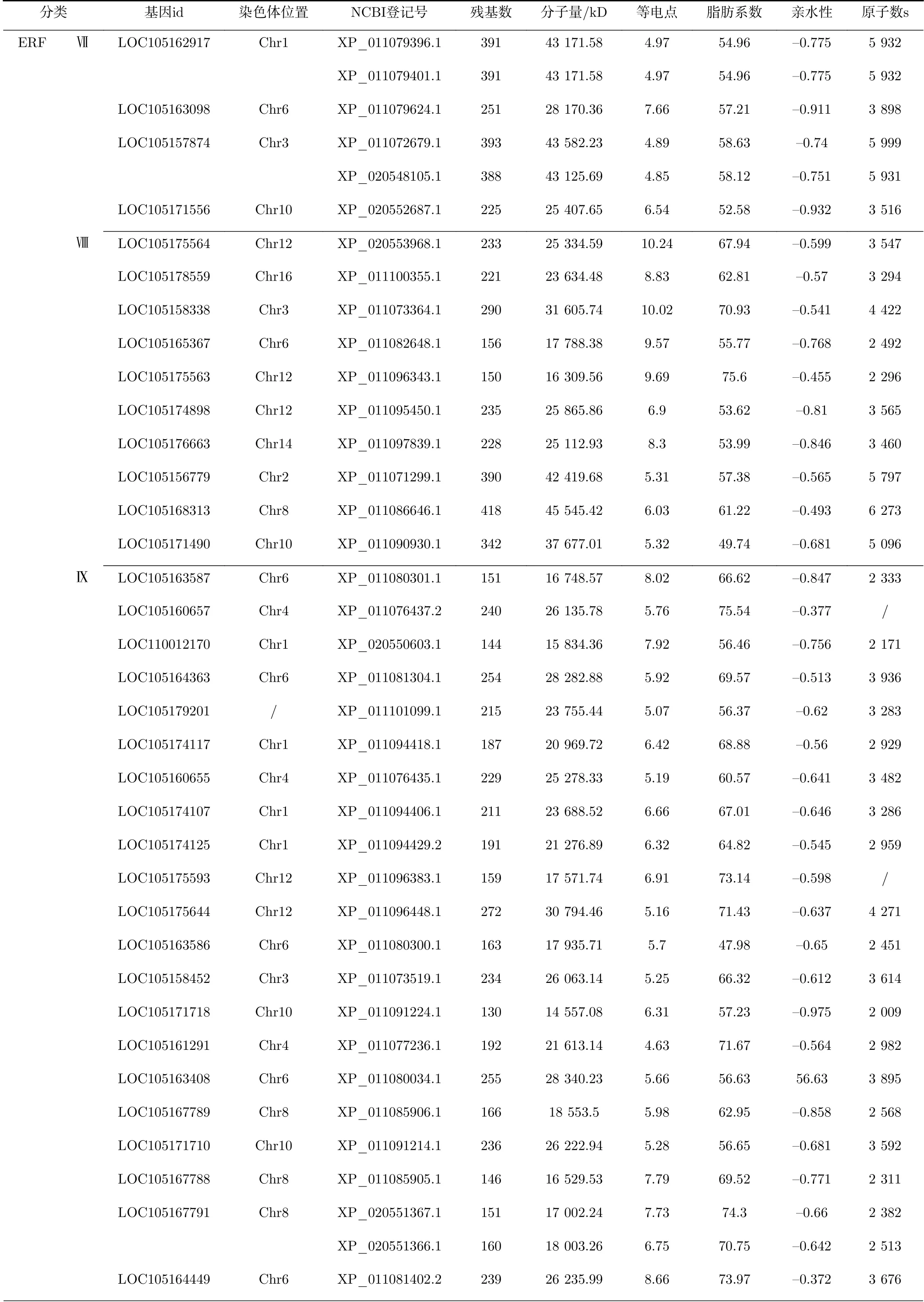
表 1(续)
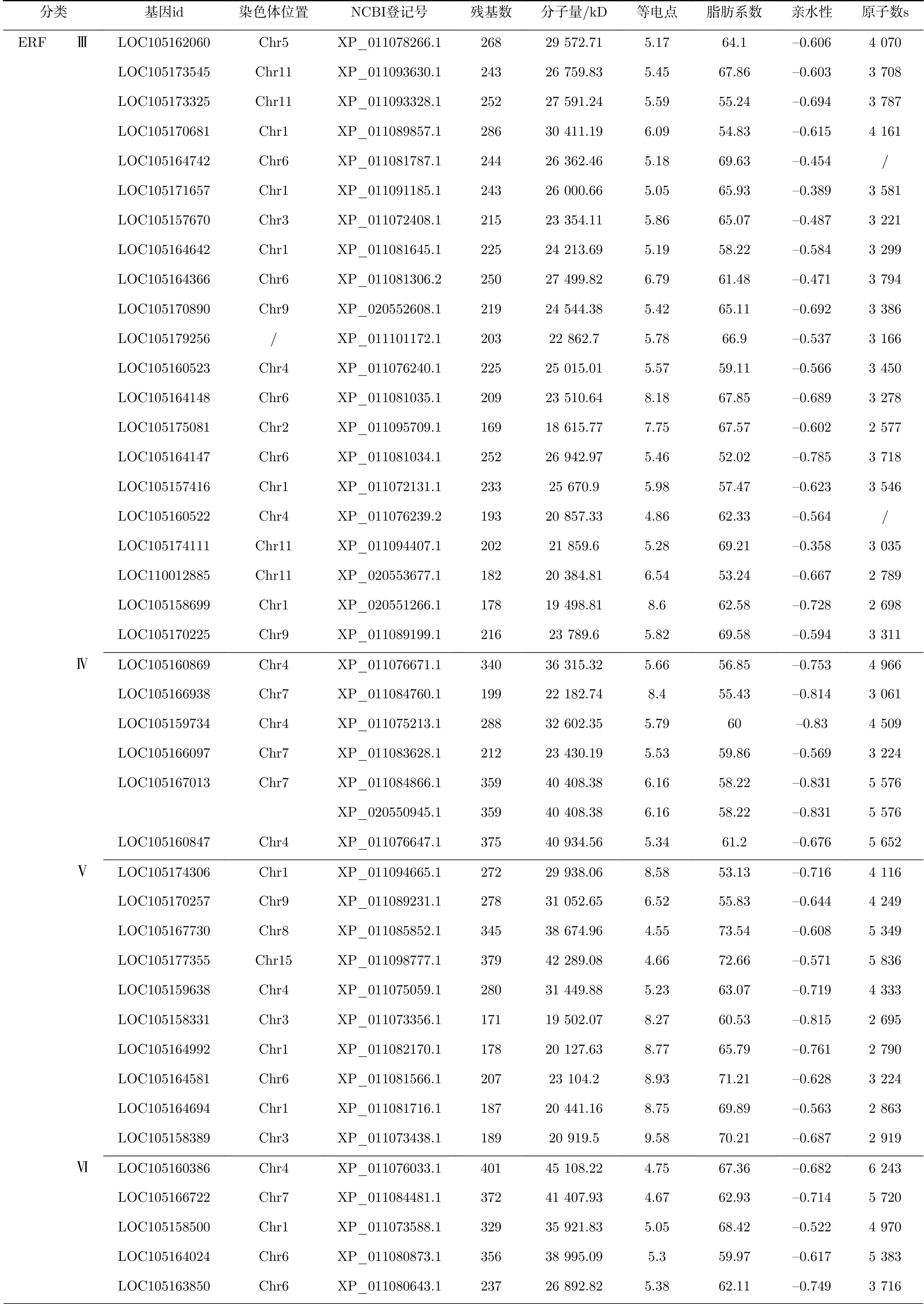
表 1(续)
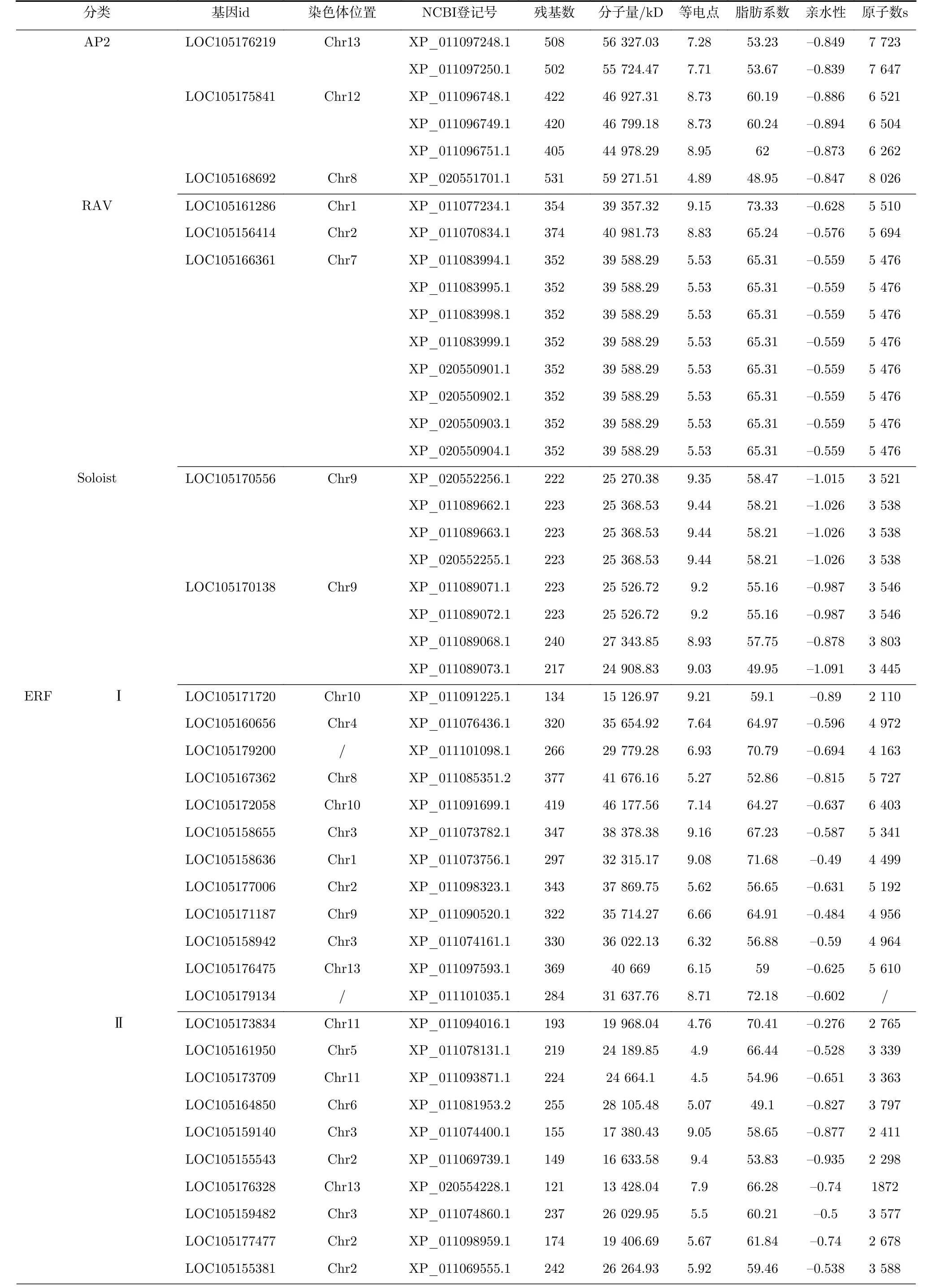
表 1(续)
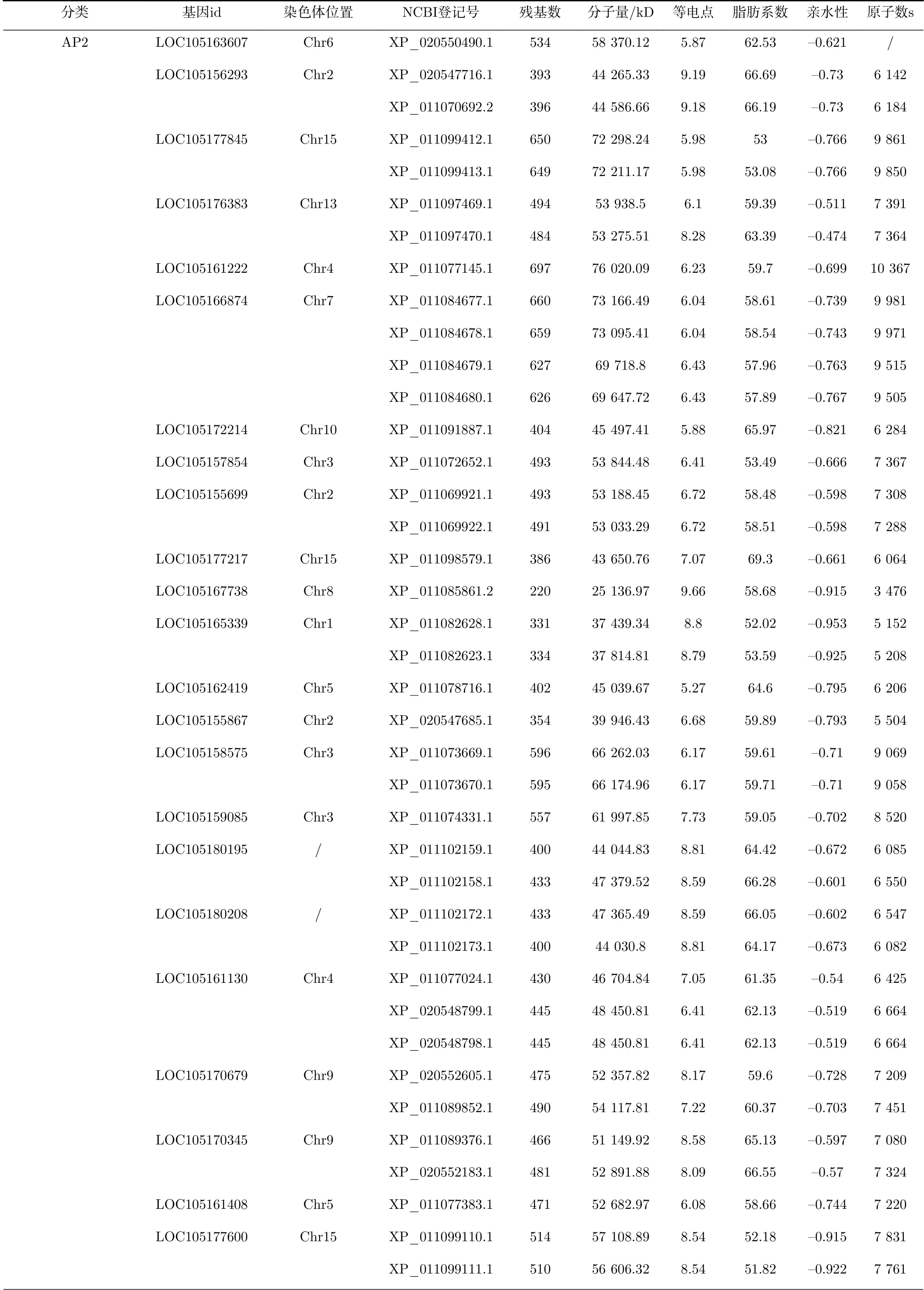
表 1芝麻AP2/ERF基因家族成员分类及性质Tab.1 Classification and properties of protein sequencesof sesame AP2/ERF gene family members
2.2 芝麻AP2/ERF转录因子的进化分析与分类
为对芝麻AP2/ERF家族成员进行精准分类,参考拟南芥分类结果[5],将拟南芥156条AP2/ERF家族成员蛋白序列与芝麻179条AP2/ERF家族成员蛋白序列进行比对并构建系统进化树。进化分析结果显示,鉴定的179条芝麻AP2/ERF蛋白序列聚为4个亚家族,分别为AP2、RAV、Soloist和ERF(见图1)。其中,有45条蛋白序列与拟南芥AP2亚家族聚为一类,属于AP2亚家族,占总数25.14%;10条蛋白序列与拟南芥RAV亚家族聚为一类,属于RAV亚家族,占总数5.59%;8条序列与拟南芥Soloist亚家族聚为一类,属于Soloist亚家族,占总数4.47%;116条序列与拟南芥ERF亚家族聚为一类,属于ERF亚家族,占总数64.80%。进一步将芝麻ERF亚家族与拟南芥ERF亚家族的12个亚组进行进化分析,结果显示,116条芝麻ERF蛋白序列分属于11个亚组,未发现与拟南芥Xb-L亚组同源的蛋白序列。11个亚组分别为Ⅰ、Ⅱ、Ⅲ、Ⅳ、Ⅴ、Ⅵ、Ⅶ、Ⅷ、Ⅸ、Ⅹ、VI-L,分别包含12、10、21、7、10、5、6、10、22、9、4条蛋白序列,分别占总数6.70%、5.59%、11.73%、3.91%、5.59%、2.79%、3.35%、5.59%、12.29%、5.03%、2.23%。ERF、RAV亚家族在芝麻基因组的占比与其在杨树[31]、水稻[32]差异不大,但芝麻中AP2、Soloist亚家族占比相对较高。
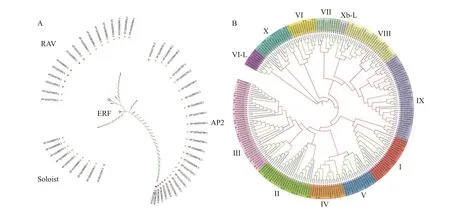
图1 芝麻AP2/ERF基因家族成员蛋白序列进化分析Fig.1 Phylogenetic tree analysis of the protein sequencesof Sesame AP2/ERF gene family members
2.3 芝麻AP2/ERF转录因子的结构分析
利用MEME研究了芝麻AP2/ERF家族蛋白的保守基序,共发现20个保守基序(命名为motif1-20),如图2所示,不同亚家族或亚组中的保守基序和基序数量不同,同一亚家族或同一亚组的蛋白序列含有相似基序,而不同的亚家族和亚组间的基序的种类和数量具有差异。AP2亚家族包含基序种类为motif 1、2、3、4、5、6、11、13、16、17、18、19,其中motif 1、3、4、5为AP2亚家族成员共有基序。RAV亚家族包含基序种类为motif 1、3、4、5、7、8、9、11、12,其中motif 1、3、4、5、7为RAV亚家族成员共有基序,motif 7为RAV亚家族成员特有基序。Soloist亚家族成员包含基序种类为motif 1、3、10、14、20,其中motif 1、10、14、20为Soloist共有基序,motif 14为Soloist特有基序。ERF亚家族成员包含基序种类为motif 3、5、10、14、15,其中motif 3、5为ERF亚家族共有基序。ERF不同亚组间基序差异较小。

图2 AP2/ERF基因家族motif和保守结构域可视化Fig.2 Visualization of AP2/ERFgene family motifsand conserved domains
AP2/ERF基因家族成员蛋白序列保守结构域分析结果显示,AP2亚家族成员蛋白序列包含两个AP2/ERF结构域,RAV亚家族成员蛋白序列具有AP2/ERF结构域和B3结构域,Soloist和ERF亚家族成员蛋白序列均只含有1个AP2/ERF结构域,但Soloist亚家族成员与ERF亚家族成员序列同源性较低。
2.4 芝麻AP2/ERF转录因子的染色体分布
芝麻基因组中共鉴定出142条AP2/ERF家族编码基因成员,通过基因组注释获取AP2/ERF基因在染色体上的位置信息,可视化分析结果显示如图3所示。
针对上述问题,设计了一种基于Arduino的车内儿童防误锁报警系统,系统可以在儿童被家长误锁车内的情况下做出及时反应,保护儿童生命安全,以此避免悲剧发生。
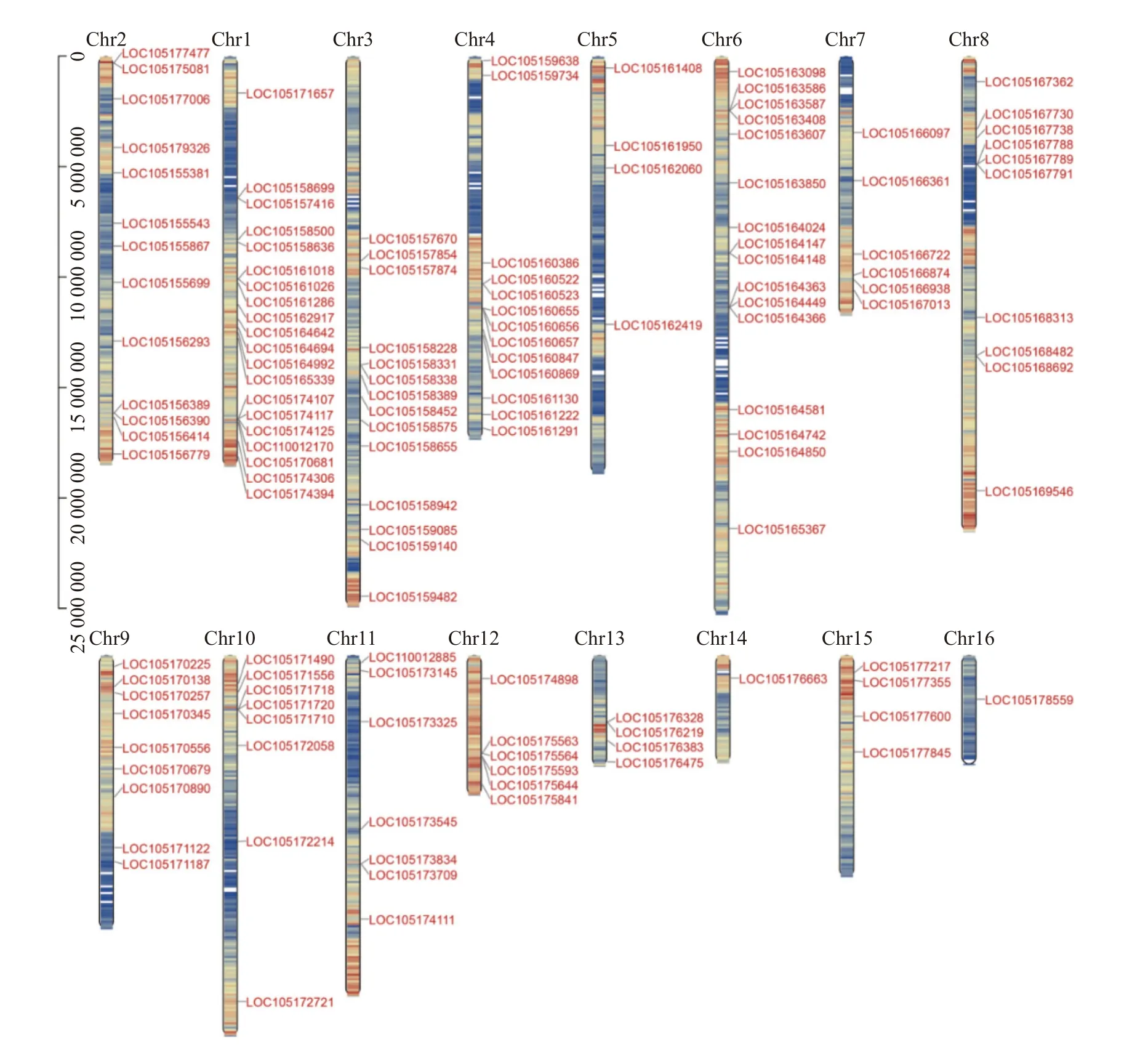
图3 芝麻AP2/ERF基因家族成员染色体定位分析Fig.3 Analysis of chromosome location of Sesame AP2/ERF gene family members
142条编码基因中,136条AP2/ERF基因分布于芝麻16条染色体上,6条分布于测序片段上,未能精确定位到已知染色体上。136条AP2/ERF基因在染色体上分布无规律、不均匀,其中Chr1、Chr6、Chr3、Chr2、Chr4上分布较多,分别有20、16、14、13、13条 基 因,Chr14、Chr16、Chr13、Chr15分布最少,分别有1、1、4、4条基因。各亚家族成员在16条染色体上呈不均匀分布,Soloist亚家族主要分布在Chr9染色体上,RAV分布于Chr1、Chr2、Chr7染色体上,AP2亚家族主要分布于除Chr11、Chr14、Chr16外的13条染色体上,ERF亚家族在16条染色体上均有分布。并且,根据基因在染色体分布密度显示,AP2/ERF家族基因成员多分布于染色体上基因密度较大区域。
2.5 芝麻AP2/ERF转录因子的共线性分析
为探究芝麻AP2/ERF家族基因成员与油料作物及粮食作物在物种演化过程中保守性和变异,将芝麻基因组与油料作物玉米、粮食作物大麦的基因组进行共线性分析,结果如图4所示。芝麻AP2/ERF家族基因成员在演化过程中,与玉米之间的保守性高于大麦,而与大麦的变异性高于玉米,芝麻染色体上共发现49个AP2/ERF基因位点与玉米共线性,32个位点与大麦共线性。AP2/ERF基因共线性位点在染色体上的分布不均匀,芝麻LG1、LG3、LG4上AP2/ERF基因共线性位点最多,LG1上分别有6、10个AP2/ERF基因位点与大麦和玉米共线性,LG3上分别有4、8个AP2/ERF基因位点与大麦和玉米共线性,LG4上分别有5、7个AP2/ERF基因位点与大麦和玉米共线性,LG5、LG12上共线性位点最少,LG5上有1个位点与大麦共线性,LG12上有1个位点与玉米共线性,LG14、LG16两条染色体上没有AP2/ERF基因位点与大麦和玉米共线性。
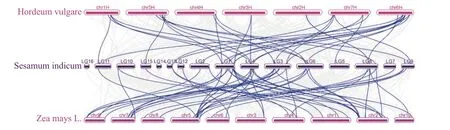
图4 芝麻、玉米、青稞AP2/ERF基因家族成员共线性分析Fig.4 Syntenty analysisof AP2/ERF gene family membersin sesame, maize and highland barley
2.6 芝麻AP2/ERF转录因子响应淹水胁迫表达分析
GSE133186表达谱数据包含‘ZZM2541’和‘Ezhi-2’2个芝麻栽培品种[33],‘ZZM2541’具有较强的耐涝性,‘Ezhi-2’对淹水胁迫较为敏感[34]。芝麻AP2/ERF家族成员中共检测到130条基因的表达,未检测到LOC105157854、LOC110012885、LOC105160869、LOC105159734、LOC105166097、LOC105158389、LOC105156779、LOC105168313、LOC105171490、LOC105167788、LOC105174394、LOC105161018基因的表达。通过热图分析AP2/ERF家族成员表达谱结果显示(见图5),AP2/ERF家族大部分成员在2个芝麻品种中受淹水胁迫影响表达量下调,表明淹水胁迫可能抑制了这部分基因的表达,从而影响芝麻生长发育过程中的其他生理调控过程。少量基因表达量上调,说明AP2/ERF家族这部分成员参与了芝麻淹水胁迫响应调控。AP2/ERF家族中Ⅶ亚组在植物响应淹水胁迫过程中发挥重要作用[35-39],芝麻中基因组中共鉴定出4条Ⅶ亚组成员,分别为LOC105162917、LOC105163098、LOC105157874、LOC105171556,LOC105163098、LOC105171556在2个芝麻品种中均受淹水胁迫诱导下调,LOC105162917在‘ZZM2541’中无显著变化,在‘Ezhi-2’中显著下调,LOC105157874在2个芝麻品种中均上调。Ⅶ亚组中LOC105157874基因可能在芝麻响应淹水胁迫调控中发挥重要作用。

图5 芝麻AP2/ERF基因家族成员响应淹水胁迫表达分析Fig.5 Expression analysis of AP2/ERF gene family members in response to flooding stress in sesame
3 结语
本研究从芝麻基因组中系统整理鉴定出142条AP2/ERF基因,编码179条AP2/ERF蛋白序列,其蛋白氨基酸数量范围为121~697,分子量范围13 428.04~76 020.09 kD,原子数范围1 872~10 367,等电点范围4.50~10.24,脂肪系数范围47.98~70.60,亲水性范围-1.091~-0.276。
将179条芝麻AP2/ERF基因家族蛋白序列与拟南芥进行系统进化分析,根据拟南芥分类结果将芝麻AP2/ERF基因家族划分为4个亚族:AP2,RAV,Soloist和ERF,结果与Nakano等研究结果相同,4个亚族分别包含45、10、8、116条蛋白序列。ERF亚族进一步划分为Ⅰ、Ⅱ、Ⅲ、Ⅳ、Ⅴ、Ⅵ、Ⅶ、Ⅷ、Ⅸ、Ⅹ、VI-L 11个亚组,分别包含12、10、21、7、10、5、6、10、22、9、4条蛋白序列。芝麻AP2/ERF基因家族保守结构域和保守基序预测结果显示,相同亚族或亚组间蛋白序列保守结构域和保守基序数量结构上相似性较高,不同亚族或亚组间保守结构域和保守基序相似性较低。通过比较芝麻与拟南芥间AP2/ERF基因家族蛋白成员数目与序列,得出两者AP2/ERF结构域间具有差异性,但其核心保守结构域相一致,结合前人对多种植物AP2/ERF转录因子的分布与数量研究,发现物种间该转录因子的结构和进化具有相对保守性。
136条芝麻AP2/ERF基因被精确定位到染色体上,6条AP2/ERF基因未定位到染色体上,芝麻AP2/ERF基因家族成员在芝麻16条染色体上分布不均匀,根据基因在染色体分布密度显示,AP2/ERF家族基因成员多分布于染色体上基因密度较大区域,这可能与(非)生物胁迫响应基因的染色体分布状况有关。共线性分析发现芝麻AP2/ERF家族基因成员在演化过程中,与玉米之间的保守性高于大麦,芝麻染色体上共发现49个AP2/ERF基因位点与玉米共线性,32个位点与大麦共线性。Ⅶ亚组中LOC105157874基因在2个芝麻品种中均上调,可能具有与拟南芥等植物Ⅶ亚组转录因子相同的功能。
目前,对芝麻AP2/ERF基因的功能研究还不够深入。本研究基于芝麻全基因组测序数据,对其AP2/ERF基因家族成员进行生物信息学分析,其具体功能仍需利用基因克隆、表达分析等途径进一步验证。AP2/ERF转录因子家族包含多个抗逆相关基因,为研究非生物胁迫下该芝麻家族功能基因的挖掘及鉴定奠定基础,有望在芝麻品种抗逆改良等研究中发挥作用。
——以多重共线性内容为例

