基于KASP技术的SNP标记用于西瓜品种指纹图谱构建和种子纯度检测
刘 欣, 程 瑞, 徐兵划, 白 甜, 许文钊, 张朝阳, 罗德旭, 赵建锋, 张兴平,孙玉东
(江苏徐淮地区淮阴农业科学研究所/淮安市设施蔬菜重点实验室,江苏淮安223001)
随着测序技术的进步,分子标记技术在遗传与育种研究中的应用越来越普及。已有分子标记主要分为四大类:(1)基于DNA-DNA杂交的分子标记,比如RFLP标记;(2)基于PCR的分子标记,比如RAPD标记、SCAR标记和SSR标记等;(3)基于PCR与限制性酶切技术结合的分子标记,比如AFLP标记和CAPS标记;(4)基于单核苷酸多态性的分子标记,比如SNP标记。其中SNP标记分布广、多态性高、可靠性高,在遗传多样性研究、遗传图谱构建和分子标记辅助选择等方面被广泛应用[1-6]。竞争性等位基因特异性PCR (Kompetitive allele specific PCR,KASP) 是一种新型高通量SNP分型技术,该技术准确度高,可以实现自动化操作,简单便捷,并且将使用SNP标记的成本降至最低[7],是目前最理想的基因分型技术。
指纹图谱是指利用DNA分子标记对作物品种进行检测,并根据不同品种的每个标记的基因型而建立的图谱,据此可以有效避免品种的同名异物及同物异名的问题,有利于种业的健康发展。目前,已有许多品种利用基于KASP技术的SNP标记完成了指纹图谱的构建。王富强等[8]利用KASP技术将22个高质量SNP标记用于76份葡萄品种的指纹图谱构建。李志远等[4]利用筛选获得的50个核心SNP标记,构建了59份市场上主要推广的甘蓝品种指纹图谱。魏庆镇等[9]筛选了34个高质量SNP标记,并构建了6个浙茄品种的指纹图谱。目前,在西瓜中,大部分种质资源及品种的指纹图谱由SSR标记构建[10-12],其缺点是多态性低,操作步骤繁琐,不能实现自动化,对于大批量样品检测,耗费时间长,人工成本高。因此,建立基于KASP技术的SNP标记用于西瓜品种鉴定和种子纯度检测是非常必要的。
本研究利用Yang 等[6]开发的32个核心SNP标记,采用KASP技术对79份西瓜杂交种及自交系材料进行基因分型,根据分型数据以及多态性信息,筛选获得30个核心SNP标记,用于构建苏梦和苏创系列西瓜品种的指纹图谱,同时,筛选出具有多态性和分型较好的标记,用于种子纯度检测,为鉴定西瓜品种的真实性提供重要依据。
1 材料与方法
1.1 试验材料
本研究的试验材料如表1所示。每份材料播种5粒于72孔穴盘中,放置于25 ℃、16 h(光)/8 h(暗)环境中,正常水肥管理,保证出芽和成苗。待幼苗长出2~3片真叶时,取嫩叶0.1 g于1.5 ml离心管中,用于提取DNA进行PCR反应。种子纯度检测所用的苏梦5号、苏梦6号、苏梦7号、苏梦9、苏创3号、苏创4号和苏创5号西瓜种子均取自制种棚,制种参考西瓜杂交制种技术[13],随机选取收获的杂交种子90粒播种于穴盘,编号Z1~Z90,待2片真叶展平后取1片真叶于1.5 ml离心管中,用于提取DNA进行PCR反应,待幼苗伸蔓后,按照每个杂交种的编号(Z1~Z90)定植在塑料大棚中,行距2.6 m,株距30.0 cm,爬地栽培,正常水肥管理,待结果后进行田间纯度鉴定。

表1 供试西瓜种质资源
1.2 DNA提取
将取好的样品放入液氮罐中速冻,用组织研磨器[天根生化科技(北京)有限公司产品]研磨至粉末状,采用北京华越洋生物科技有限公司提供的试剂盒(0418-50BB型)提取DNA,最后用蒸馏水洗脱和溶解DNA。取2 μl DNA用1.5 %琼脂糖凝胶电泳检测,无拖带且条带单一即可进行下一步试验。利用Nanodrop 2000测定吸光值OD260/OD280及OD260/OD230,测定值为1.8~2.0时表示DNA质量较好。测定每份DNA样品的浓度,并稀释质量浓度至60 ng/μl进行PCR反应。
1.3 PCR反应
根据Yang 等[6]开发的40个核心SNP标记信息合成引物,在每组引物中的正向引物(Forward 1)的序列5′端加上接头序列GAAGGTGACCAAGTTCATGCT,在正向引物(Forward 2)序列的5′端加上接头序列GAAGGTCGGAGTCAACGGATT,反向引物序列不用改变,用蒸馏水溶解稀释引物浓度至100 μmol/L,按照如下体系配置引物混合物(Primer Mix):正向引物Forward1和Forward2各12 μl,反向引物30 μl,加蒸馏水至100 μl,配置完成后保存于-20 ℃备用。每个PCR反应孔的体系配置如下:DNA模板1.20 μl,Primer Mix 0.14 μl,2×KASP Mix(北京嘉程生物科技有限公司产品)5.00 μl,蒸馏水3.50 μl,配置完成后封膜,每个96孔反应板中加入2个阴性对照(NTC)。PCR反应板和封板膜由爱思进生物技术(杭州)有限公司提供。将加样完成的PCR板放入荧光定量检测仪器(Applied Biosystems /7500型定量PCR仪)中进行荧光定量PCR反应,反应程序如下:94 ℃预变性15 min; 94 ℃变性20 s,61~55 ℃复性和延伸1 min, 10个循环,每循环一次降低0.6 ℃; 94 ℃变性20 s,55 ℃继续扩增60 s ,26个循环。PCR反应结束后读取数据,若分型不充分,则继续扩增,每3个循环查看分型情况,不超过40个循环。
1.4 KASP基因分型
荧光定量PCR结束后,使用Real-Time PCR Software V2.4软件进行分型及分析。
1.5 数据统计及分析
根据引物信息、荧光信号和分型结果,统计每个SNP位点基因型。聚类分析采用MEGA X软件版本,将位点信息转化为序列信息,运用近邻法(Neighbor-Joining method)和JTT矩阵模型来进行计算[14-15],重复计算次数为1 000次。
2 结果与分析
2.1 KASP分型
根据KASP的分型结果进行数据统计,结果(图1 )显示,用32个核心标记对所有材料进行KASP分型,有2个标记分型失败,分别是WmSNP178和WmSNP192。其余30个标记虽然在所有材料中存在分型数据缺失的情况,但每个材料缺失标记数量都不超过3个,因此,这30个标记能够成功分型(图1 ),用于遗传背景分析和指纹图谱构建。标记WmSNP47在大部分材料中是杂合位点,杂合基因型占比87.5%。标记WmSNP151在所有材料中均是纯合位点,基因型为C或者T。

A:标记WmSNP140在79份材料中的基因分型;B:标记WmSNP92在苏创4号种子纯度检测中的基因分型。FAM、VIC表示探针的荧光值。
2.2 遗传背景分析
根据KASP分型结果,我们将荧光信号转换成不同位点的基因型,运用MEGA软件对79份材料进行进化树分析,结果见图2。苏梦系列小果型品种苏梦2号(Wm38)、苏梦5号(Wm21)、苏梦6号(Wm40)、苏梦7号(Wm31)被聚类在一个分支上,属于一个类型,说明这些品种遗传关系相近,而在人工选育时,这些品种选择的育种方向都是红瓤,中心糖度高,果皮韧性较强,耐裂[16],并且苏梦5号、苏梦6号和苏梦7号的父本相同,因此这3个品种在遗传背景上具有相似性。Wm13和Wm14是2个野生种,与杂交种在性状上存在较大差异:野生种瓤色为白色,且具有苦味,种子颜色为灰绿色无覆纹或红褐色有斑点状覆纹,与杂交种种子的棕色和黑色不同。从图2中可以看出,其与苏梦系列品种亲缘关系较远,分属不同分支,属于不同类型。苏梦3号(Wm39)与苏梦4号(Wm16)分别属于不同分支,二者瓤色不同,前者为红瓤,后者为黄瓤,且苏梦4号果肉风味独特,推测二者在某些位点具有遗传多样性。苏梦9号(Wm22)与苏梦6号(Wm40)也属于不同分支,苏梦6号果皮覆纹形状为窄齿条状,而苏梦9号齿条较宽,并且其果形比苏梦6号稍大。苏梦7号亲本Wm29和Wm33的遗传距离为0.43,二者遗传距离较远,农艺性状测定结果显示其杂交一代苏梦7号的品质高于双亲,尤其是在果皮韧性方面,其双亲果皮分别为易裂和中等硬度,但苏梦7号表现出韧性强的特点[16]。
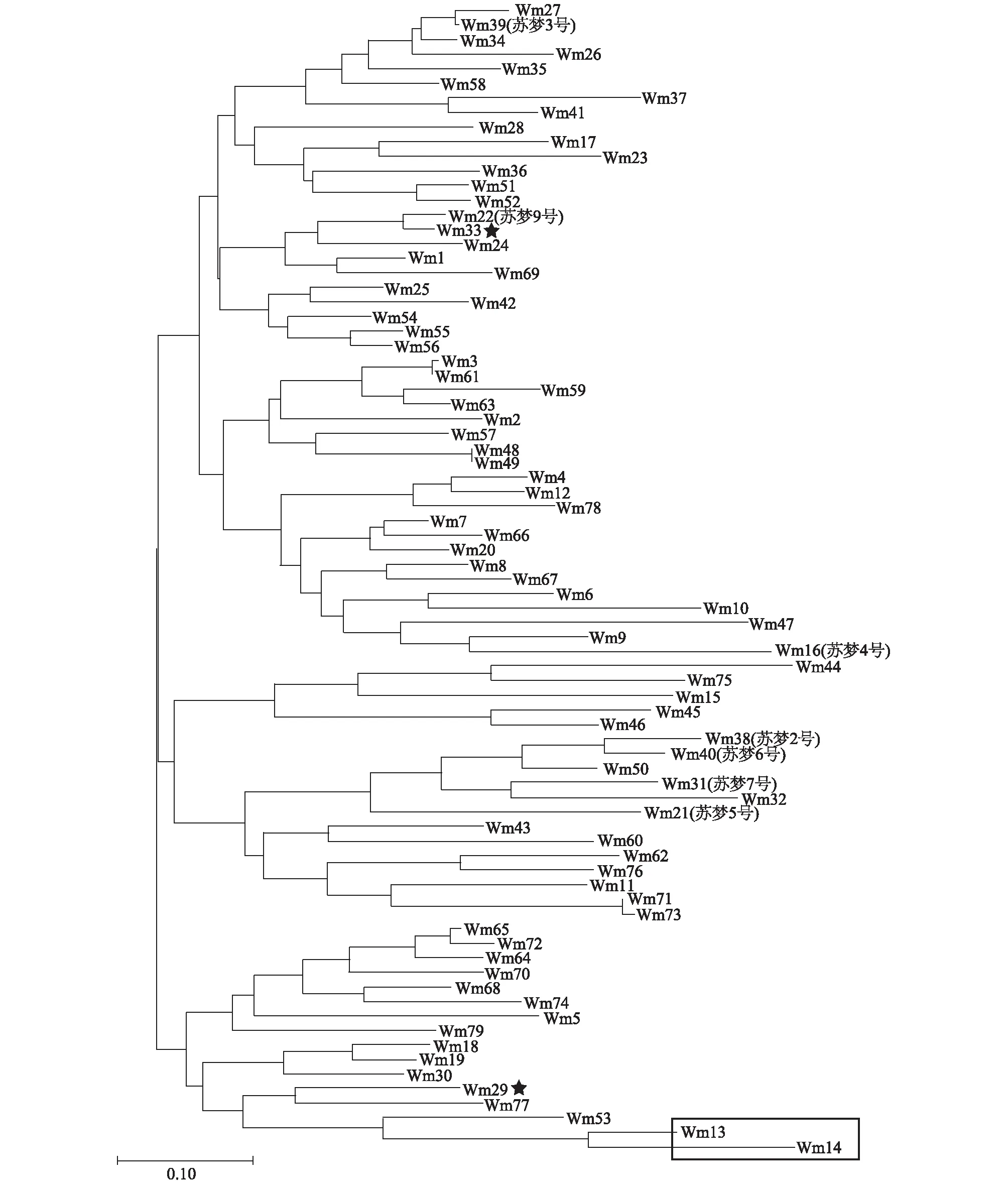
图中的材料见表1。方框中的材料表示2个野生种材料;带星号的材料表示苏梦7号的亲本。
2.3 指纹图谱构建
我们利用KASP分型成功的SNP 标记构建了苏梦和苏创系列西瓜品种的指纹图谱,位点和基因型数据如表2所示。在30个SNP标记中,WmSNP162在15份西瓜品种中基因型(GG)全部一致,为纯合位点。WmSNP226在苏梦8号中的基因型(AG)为杂合,其他品种该位点基因型均为纯合(AA)。WmSNP92在苏梦8号中是纯合基因型(CC),在苏梦1号中未检出,其余品种该位点基因型(TC)均为杂合。通过WmSNP1、WmSNP29、WmSNP60、WmSNP66、WmSNP140和WmSNP156共6个SNP标记即可将所有苏梦和苏创系列西瓜品种区分开。根据位点基因型信息,将每个品种指纹图谱转化为二维码,方便品种的真实性鉴定和推广(图3)。

图3 苏梦和苏创系列西瓜品种指纹图谱二维码

表2 苏梦和苏创系列西瓜品种指纹图谱
2.4 种子纯度检测
根据KASP分型结果,筛选可以对苏梦5号、苏梦6号、苏梦7号、苏梦9号、苏创3号、苏创4号和苏创5号西瓜品种种子进行纯度检测的标记。结果显示,标记WmSNP5在苏创3号的双亲中是纯合位点,且呈现多态性,在苏创3号中是杂合位点,可以用来对苏创3号种子的纯度和真实性进行检测。利用同样的分析方法,我们筛选到标记WmSNP92可以用来检测苏梦5号、苏梦6号、苏梦7号、苏梦9号、苏创4号、苏创5号6个西瓜品种种子的纯度(图1 B)。通过标记WmSNP5和WmSNP92,利用KASP技术对上述7个西瓜品种进行种子纯度鉴定,同时进行田间种植鉴定,鉴定指标包括叶片形态、果皮花色、果型、种子大小及颜色等,结果如表3所示,田间鉴定结果与分子标记鉴定结果一致,因此,标记WmSNP92和WmSNP5可用于苏梦5号、苏梦6号、苏梦7号、苏梦9号、苏创4号、苏创5号和苏创3号种子纯度和真实性的检测。

表3 苏梦和苏创系列西瓜品种种子纯度鉴定结果
3 讨论与结论
对于育种工作者来说,明确种质资源和育种材料的遗传背景,鉴定商品品种种子纯度及种子的真实性非常重要,传统的鉴定方法主要是从植株表型观察鉴定。随着分子生物学和基因组学的发展,人们开发的分子标记可以在基因水平上直接反映植物的遗传多态性,因此分子标记已经成为植物遗传多样性研究的主要方法[17-19]。KASP技术是目前最为经济有效的SNP标记分型技术[20]。利用以KASP技术为基础的SNP标记研究种质资源的遗传背景,构建品种指纹图谱以及鉴定品种的真实性是未来发展的趋势。冯子珊等[21]通过KASP技术,对浙蒲9号父本、母本以及92份F1材料进行基因分型,进而鉴定F1材料的种子纯度,结果与田间种子纯度鉴定结果一致,且该方法准确性高,成本低。Shen等[22]利用SNP标记对372份青花菜种质的遗传多样性和亲缘关系进行了分析与评价,最终筛选出28个KASP标记对青花菜种质进行基因分型,用于种子鉴定和品种鉴定。Mulugeta等[23]利用37个基于KASP技术的SNP标记对48个埃塞俄比亚蚕豆进行了遗传多样性和亲缘关系的分析,发现其遗传多态性较高,有利于培育优良品种。
本研究利用32个核心SNP标记对79份西瓜材料(包括54份自交系和25份F1杂交种)进行KASP分型和遗传背景分析,通过聚类分析及遗传距离分析发现,不同自交系间的遗传距离不同,因此,我们可以通过选择遗传距离较远的自交系进行杂交配组,筛选高优势组合,进而为选育优质和高优势品种奠定基础。通过分析我们还发现苏梦系列不同品种其遗传背景有差异,这也从DNA水平上解释了不同品种间性状差异的原因。但是在进行KASP分型时,有2个标记分型不成功,推测这是因为79份材料不能涵盖西瓜的所有品种,这2个位点基因型不呈现多态性,因此导致分型失败。本研究针对苏梦和苏创系列西瓜,利用筛选出来的30个SNP标记构建指纹图谱,为西瓜品种鉴定和保护提供了技术支撑。通过筛选出来的WmSNP5和WmSNP92标记,对江苏徐淮地区淮阴农业科学研究所培育的苏梦5号、苏梦6号、苏梦7号、苏梦9号、苏创3号、苏创4号、苏创5号7个西瓜品种[24-26]进行种子纯度鉴定,构建了以KASP技术为基础的种子纯度检测体系,具有高效、简单、便捷等特点,方便了种子生产销售中种子纯度的鉴定。
致谢:感谢中国计量大学徐沛研究员对本论文提供指纹图谱构建方面的帮助!

