综合生理学和转录组学揭示芦竹耐盐的候选基因
孙源长, 罗 琳, 林 辉, 林冬梅, 林占熺
(福建农林大学国家菌草工程技术研究中心, 福建 福州 350002)
随着全球人口的快速增长以及人们生活水平的不断提高,对能源的需求也显著增加。但是化石燃料和不可再生能源是有限的,并且在日益减少,因此迫切需要大力发展和利用可再生能源,而生物质能是其重要的组成部分。目前马铃薯(Solanumtuberosum)、甘蔗(Saccharumofficinarum)、小麦(Triticumaestivum)等作物和一些生物量较大的草本植物作为生物燃料应用十分广泛。虽然这些草本植物可以在耕地中正常生长,但是其与农作物竞争生长,不适宜一起种植,因此将适应能力强的草本植物种植到盐渍地、干旱地区和土壤性质差的区域是个不错的选择。其中,土壤盐渍化是一个严重的环境问题,目前世界上有100多个国家的土壤受到影响,面积超过了9亿公顷[1]。它会显著影响作物产量和区域的农业生产。芦竹隶属于禾本科芦竹亚科芦竹属,具有高生物量、高蛋白、高抗逆性等特点,在合适的环境下,芦竹第2年就可以生产出高达40 t·hm-2·a-1的干物质生物质能,对应的能量值约为150 MWh·hm-2·a-1[2],可见芦竹的生产潜力巨大,并且其在高盐度条件下也可实现较为可观的生物产量,因此将它们种植于盐渍地中不仅可以改善生态环境而且它们不与粮食作物竞争,有利于克服粮食安全风险。之前Lloyd L. Nackley等人对芦竹进行过盐胁迫实验,使它们在一系列盐浓度(0~42 dS·m-1NaCl)环境中生长60天,结果显示,在最高盐度下,芦竹整体生长减少了80%以上。然而,死亡率为零,表明芦竹能够耐受较高浓度的盐环境[3]。虽然已知芦竹具有较强的耐盐性,但其分子机制和一些重要的盐响应基因尚未可知,制约了对其耐盐高产的遗传改良。
近10年来,随着高通量测序技术的飞速发展,其成本不断下降,越来越多的研究人员对植物进行测序并通过生物信息学分析筛选到一些重要的基因与信号通路[4-7],为相关的育种工作提供了更加有效的方案。本研究对芦竹采用不同浓度梯度的盐溶液处理,通过生理学与转录组学进行研究,筛选出参与盐响应的重要基因,丰富芦竹的耐盐基因资源,未来可以针对这些基因构建突变体,对这些基因进行深入研究,为耐盐品种的培育提供科学参考。
1 材料与方法
1.1 试验材料
从福建农林大学国家菌草工程技术研究中心菌草圃选取了6个月大的长势一致的芦竹组培苗,移栽到温室用1/5霍格兰营养液培养1个月进行缓苗,使植物在14 h光照/10 h黑暗的光周期下生长,湿度为60%,光照强度为350 μmol·m-2·S-1。待适应环境后分别进行0 mmol·L-1,50 mmol·L-1,100 mmol·L-1,150 mmol·L-1和200 mmol·L-1NaCl盐浓度溶液的处理,处理时间为36小时。
1.2 生理指标的测定
测定盐胁迫后叶片的超氧化物歧化酶、过氧化物酶、过氧化氢酶活性,脯氨酸、可溶性糖、丙二醛含量和Fv/F0指标。每个重复由2~4株芦竹从上至下第3~5片叶片混合得到,共3个重复。数据分析软件采用GraphPad Prism 8。采用索莱宝公司超氧化物歧化酶(SOD)试剂盒测定SOD活性(型号:BC0175),过氧化物酶(POD)试剂盒测定POD活性(型号:BC0095),过氧化氢酶(CAT)试剂盒测定CAT活性(型号:BC0205),脯氨酸(Pro)含量试剂盒测定Pro含量(型号:BC0295),植物可溶性糖(Soluble sugar)含量试剂盒测定可溶性糖含量(型号:BC0035),丙二醛(MDA)含量试剂盒测定MDA含量(型号:BC0025)。用英国Hansatech公司植物效率仪(型号:PocketPEA)测定2-4株芦竹从上至下第3-5片叶片的中部光系统II的最大光化学效率(Fv/F0),每片叶子测定4次,每个重复的测定结果取平均值,共3个重复。
1.3 RNA的提取与转录组数据的获得
收集0,50,100,150,200 mmol·L-1NaCl盐浓度溶液处理过的叶片,叶片中每个重复由2-4株芦竹的从上至下数第2片叶片混合得到,共3个重复。
取各个样品各0.2 g,用Trizol试剂盒提取总RNA。在1%琼脂糖凝胶上评估RNA降解与污染。RNA的纯度通过Nano Drop 2000(Thermo scientific)分光光度计检查。RNA完整性通过Agilent 2100(Agilent Technologies)检测。对合格的样本在Illumina Novaseq 6000平台进行双端测序,读长为150 bp。
1.4 转录组数据的质量控制及De novo组装
为了得到更加完整的转录本,此次组装额外使用了本实验室相同品种的15个芦竹茎的转录组数据。原始数据由FastQC进行质量检测。任何低质量的原始数据和接头序列都通过Trimmomatic[8]软件进行过滤,最终得到高质量的数据(Clean data)。使用Trinity-v2.13.2[9]软件对转录组数据合并进行De novo组装,组装得到的转录组文件利用CD-HIT[10]软件去冗余(相似性设置为0.95)得到最终Unigene序列。
1.5 基因表达量化和差异表达分析
使用Bowtie2比对软件和RSEM进行基因表达定量,以通过Perl脚本align_and_estimate_abundance.pl和来自Trinity软件的-est_method RSEM获得读取计数的数量[11-12]。随后,使用R包DESeq2[13]进行差异表达基因的分析。DEGs的过滤标准是|log2FoldChange|≥1并且p-value adjusted(padj)≤0.05。
1.6 GO和KEGG富集分析
为了研究DEGs的生物学意义,使用clusterProfiler[14]R包对其进行GO与KEGG富集分析。参数设置为校正后的P值小于0.05,基因的注释来源为eggNOG5.0[15]数据库。
1.7 加权基因共表达网络分析
WGCNA(R包中的WGCNA v1.70-3)[16]用于探索基因与生理指标之间的关系,以及基因与基因之间的关系。参照前人方法,通过绝对中值差异筛选出具有4分位数(Q1)表达和TPM值大于2的基因输入WGCNA进行网络的构建[17]。最小模块大小设置为30,最大模块大小设置为20000,merge cut height设置为0.25,合并相似度为0.8的模块,其他参数均为默认值。Cytoscape[18]工具用于构建和可视化交互网络。Turquoise和green模块的权重分别设置为0.18与0.24。
1.8 转录组数据验证
通过qRT-PCR验证转录组的准确性。本研究随机选取5个DEGs进行验证,以证明转录组数据的可靠性。根据Michele Poli等人的实验结果,选取较为稳定的RPN6基因作为内参基因[19],从Phytozome13(https://phytozome-next.jgi.doe.gov/)网站获取高粱的RPN6基因的cds序列(S.bicolor v3.1.1|Sobic.006G092800.2 CDS)。之后从芦竹最长转录本的cds中挑选与高粱RPN6基因相似度最高的作为芦竹的RPN6 cds序列。所有的基因均采用Primer5.0软件设计引物。根据制造商的说明,使用TransScript®Uni All-in-One First-Strand cDNA Synthesis SuperMix for qPCR(One-Step gDNA Removal)试剂盒进行反转录,使用前将各组分别点甩离心。在PCR管中建立20 μL体系,得到的产物直接用于qPCR反应。qPCR反应根据PerfectStart®Green qPCR SuperMIX试剂盒说明书进行。相对定量结果采用2-△△Ct法计算基因的相对表达量[20]。qPCR引物见表1。
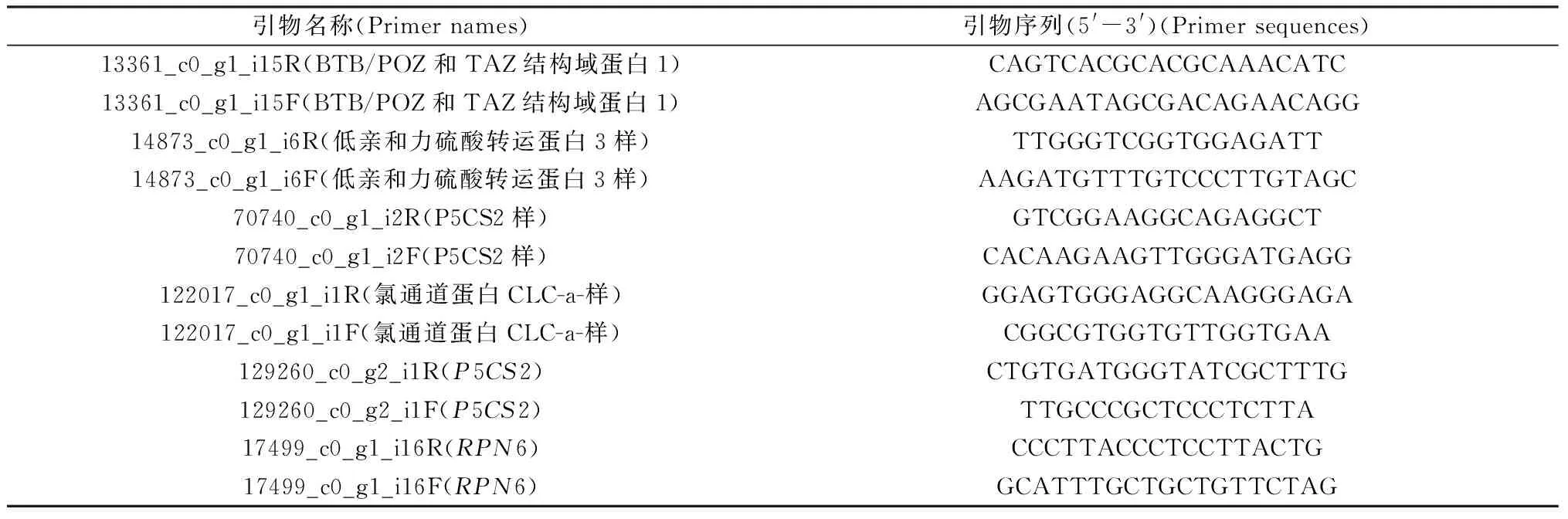
表1 用于qPCR反应的引物Table 1 Primers for qPCR reactions
2 结果与分析
2.1 盐胁迫对芦竹生理生化的影响
随着NaCl盐浓度的改变,芦竹的脯氨酸含量与可溶性糖含量发生了显著变化。其中脯氨酸含量在盐浓度为200 mmol·L-1处上升至最高值(P<0.01)。而可溶性糖含量在150 mmol·L-1处达到了最高值,在200 mmol·L-1处显著下降到最低值(P<0.01)。具体情况如图1所示。

图1 不同盐浓度下芦竹的生理变化Fig.1 Physiological changes of Arundo donax under different salt concentrations
2.2 测序数据的组装结果
使用Trinity-v2.13.2[9]软件对所有Clean data进行从头组装,共产生971 309个转录本,GC含量为47.14%。之后对组装好的转录本用CD-HIT[10]软件去冗余,得到最终的Unigene。表2与表3是对组装结果的统计,transcript contigs的N50达到了1 228 bp,unigene contigs的N50达到了1 126 bp,表明组装效果较好,数据值得进一步分析。

表2 基于所有transcript contigs的统计数据Table 2 Stats based on all transcript contigs
2.3 不同盐浓度胁迫下的基因表达量化和差异表达分析
在R中通过DESeq2包分析了芦竹响应盐胁迫的DEGs。DEGs的过滤标准是|log2FoldChange|≥1并且padj≤0.05。在叶片中检测到的差异表达基因为8 745个,其分布如图2所示。可以看出随着盐浓度的增加,在叶片中无论是上调差异表达基因,下调差异表达基因还是总的差异表达基因数量都是逐渐增加的。不高于100 mmol·L-1盐浓度时都是上调的差异表达基因数目小于下调的差异表达基因数目,当盐浓度大于等于150 mmol·L-1时上调的差异表达基因数目大于下调的差异表达基因数目。这些结果表明芦竹在响应盐胁迫时,随着盐浓度的增加调控过程也越来越复杂。

表3 基于所有unigene contigs的统计数据Table 3 Stats based on all unigene contigs
图3可以较为直观地看出叶片各个组间差异表达基因的重叠数量。结果表明在叶片中有166个基因是不同浓度盐处理下的差异表达基因所共有的,这些基因可能是盐响应核心差异表达基因。并且随着盐浓度的增加,各个组别中特有的差异表达基因数目也逐渐增加。这可能是由于随着胁迫程度的加深,越来越多特定的信号通路受到调控。例如在叶片中,leaf_0 mmol·L-1_vs_50 mmol·L-1到leaf_0 mmol·L-1_vs_100 mmol·L-1到leaf_0 mmol·L-1_vs_150 mmol·L-1再到leaf_0 mmol·L-1_vs_200 mmol·L-1的特有差异表达基因数目从295个上升到653个、1 226个、3 685个。从以上结果可以看出,叶片存在一些较为保守的差异表达基因来响应盐胁迫,并且随着盐浓度的加深有越来越多的特异性差异表达基因参与到表达调控中来。

图2 不同盐浓度下叶片差异表达基因的数目Fig.2 Number of differentially expressed genes in leaves under different salt concentrations
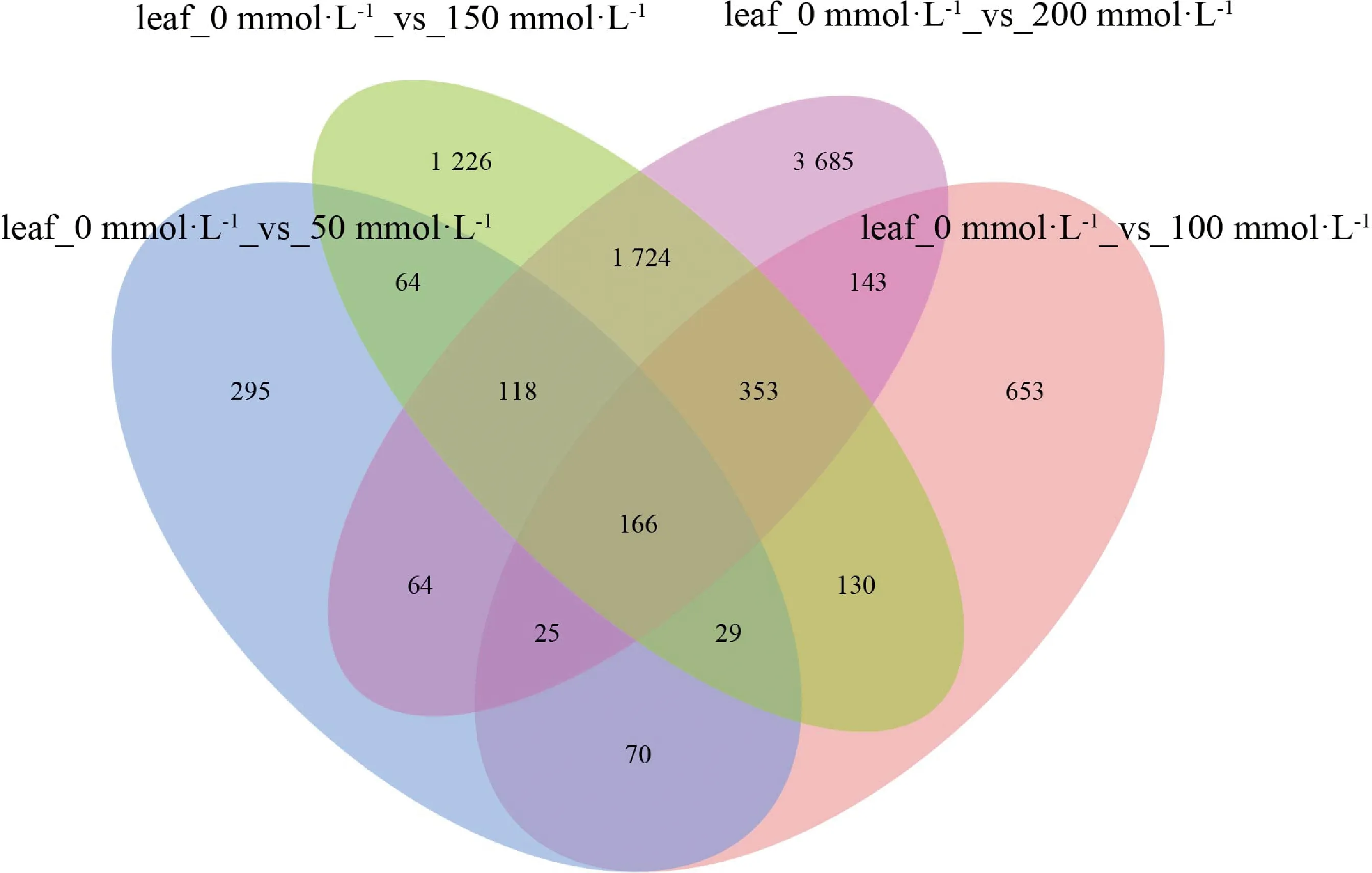
图3 不同盐浓度下叶片差异表达基因的venn图Fig.3 Venn plot of differentially expressed genes in leaves under different salt concentrations
2.4 盐响应核心差异表达基因分析
对166个在叶片中的盐响应核心差异表达基因进行差异倍数分析。在叶片中盐响应的核心差异表达基因大部分是上调的,小部分是下调的;并且这些上调基因相对于下调基因的差异倍数更大(图4)。说明响应盐胁迫主要是通过基因的上调起作用的,这些上调基因相对于下调基因的反应更加强烈。
对叶片中差异倍数最大的前10个核心差异表达基因进行注释,这些基因很可能在芦竹基础盐响应过程中发挥着极其重要的作用。其中差异表达倍数最大的414_c1_g2_i5编码去极化激活的Ca2+通道,与钙离子转运相关;0_c1_g3_i2编码富含亮氨酸的重复蛋白激酶家族成员,与植物的防御相关。具体情况见表4。

图4 叶片中核心差异表达基因的差异倍数分析Fig.4 Fold analysis of core genes expression differences in leaves based on log2FoldChange

表4 叶片中重要的盐响应核心差异表达基因Table 4 Important salt-responsive core differentially expressed genes in leaves
2.5 差异表达基因的GO和KEGG富集分析
对叶片在不同盐浓度下的差异表达基因进行GO与KEGG富集分析。GO富集结果(图5)表明:在叶片响应盐胁迫中,50 mmol·L-1与100 mmol·L-1盐浓度的差异表达基因主要富集到了与硝酸盐、一氧化氮、活性氮和活性氧相关的条目。与之不同的是随着盐浓度的增加达到150 mmol·L-1,一些富集集中在了与光合作用、脯氨酸相关的条目。当盐浓度上升到200 mmol·L-1时,除了富集到了与光合作用相关的条目还富集到了细胞分裂素、支链氨基酸相关途径。KEGG富集结果(图6)表明:在叶片中,50 mmol·L-1盐浓度只富集到了氮代谢通路;而100 mmol·L-1,150 mmol·L-1和200 mmol·L-1盐浓度还富集到了其他条目:例如类黄酮生物合成、芪类与二芳基庚烷类和姜酚生物合成、光合作用、光合作用-天线蛋白、玉米素生物合成和硒化合物代谢等代谢通路。

图5 叶片中差异表达基因的GO富集结果Fig.5 GO enrichment results of differentially expressed genes in leaves
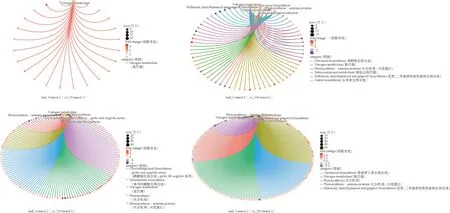
图6 叶片中差异表达基因的KEGG富集结果Fig.6 KEGG enrichment results of differentially expressed genes in leaves
2.6 WGCNA分析
植物在响应盐胁迫时会发生特定基因的表达调控,这些将反应在某些生理特征中。为了探究与特定性状相关的基因集的关键(hub)基因,研究芦竹特异性的盐响应应答过程,采用WGCNA分析芦竹叶片响应盐胁迫的表达谱,检测基因与生理指标以及模块内基因之间的关系。
2.6.1模块的筛选 结合生理指标和叶片的15个转录组数据,通过绝对中值差异筛选出具有4分位数(Q1)表达和TPM值大于2的共14 013个基因输入WGCNA进行网络的构建。通过计算基因间邻接函数确定软阈值为12。并且聚类得到38个模块,其中turquoise模块与Pro的相关性最高,相关性为0.87;green模块与Pro的相关性也较高,相关性分别为0.79。具体见图7。
2.6.2hub基因的鉴定 将2个目标模块的hub差异表达基因导入Cytoscape构建可视化交互式网络,结果如图8所示。Turquoise模块的240249_c0_g1_i1,104188_c0_g1_i3,219926_c0_g1_i1,18316_c0_g1_i4和7477_c11_g1_i1处于网络的中心位置;Green模块的10454_c0_g1_i12,6117_c0_g1_i16,19205_c0_g1_i4,23765_c0_g1_i1和86581_c0_g1_i9处于网络的中心位置。对每个模块Degree程度最强的基因进行注释,结果见表5,这些被认为核心hub基因,其中与Pro相关的turquoise,green模块的hub基因编码核碱基抗坏血酸转运蛋白和外膜孔蛋白等相关蛋白质,还包括一些具有某些特定结构的蛋白例如RING/U-box超家族蛋白和RRM结构域蛋白等等;而对2个模块的核心hub基因进行表达量分析(各组TPM平均值取log2),图9结果显示turquoise与green模块的基因表达量随着盐浓度的加深呈上调趋势。

图7 模块与生理指标关联图Fig.7 Correlation diagram between modules and physiological indicators
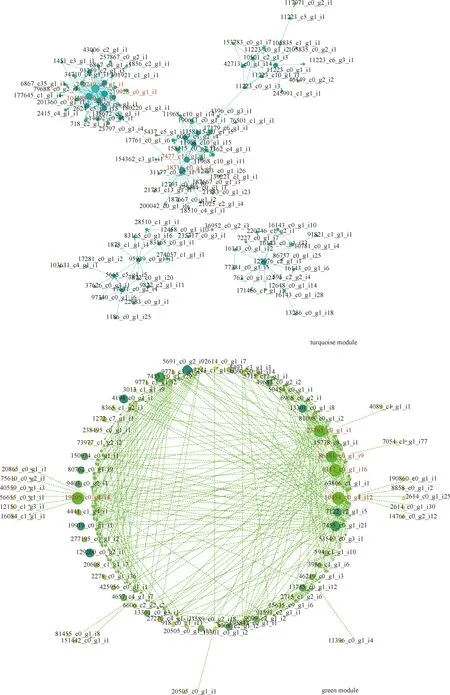
图8 网络分析2个模块中的枢纽基因Fig.8 Net-work analysis the hub genes in two modules

表5 两个模块中前5个hub基因的注释结果Table 5 Annotation results of the top 5 hub genes in the two modules

图9 2个模块中排名前5个hub基因的表达量分析Fig.9 Expression analysis of the top 5 hub genes in the two modules
2.7 qRT-PCR验证转录组数据
由于RNA-seq具有一定的缺陷,可能会得到假阳性的结果,故需要利用qRT-PCR技术进行检验。对叶片的转录组数据随机挑选5个差异基因进行qRT-PCR验证,可以看出这些基因的相对表达量与转录数据的表达模式是基本一致的(图10A~10E),并且基因表达变化数据相关系数R2达到了0.82(图10F),也说明了转录组分析结果与本实验中qRT-PCR所确定的表达模式具有较好的一致性。
3 讨论
3.1 盐胁迫对芦竹的生理影响
植物在盐胁迫的情况下会诱导抗氧化系统的激活,包括抗氧化化合物,例如类胡萝卜素和抗坏血酸盐、谷胱甘肽、α-生育酚和几种参与活性氧(ROS)解毒的酶,例如超氧化物歧化酶、过氧化物酶、过氧化氢酶等[21]。而一些渗透调节物质也在抗逆过程中发挥着重要作用,如脯氨酸[22]与可溶性糖。脯氨酸是一种具有特殊构象的氨基酸,是初级代谢所必需的。压力环境导致植物中脯氨酸的过量产生,进而通过维持细胞膨胀或渗透平衡来赋予压力耐受性;稳定膜,从而防止电解质泄漏;使活性氧(ROS)浓度在正常范围内,防止植物氧化爆发。MDA是脂质过氧化的细胞毒性产物,也是评价自由基产生和组织损伤的指标之一。Fv/F0是Fv与F0的比值,也是Fv/Fm(最大荧光)的一种表达方式,表示光系统II的最大光化学效率。
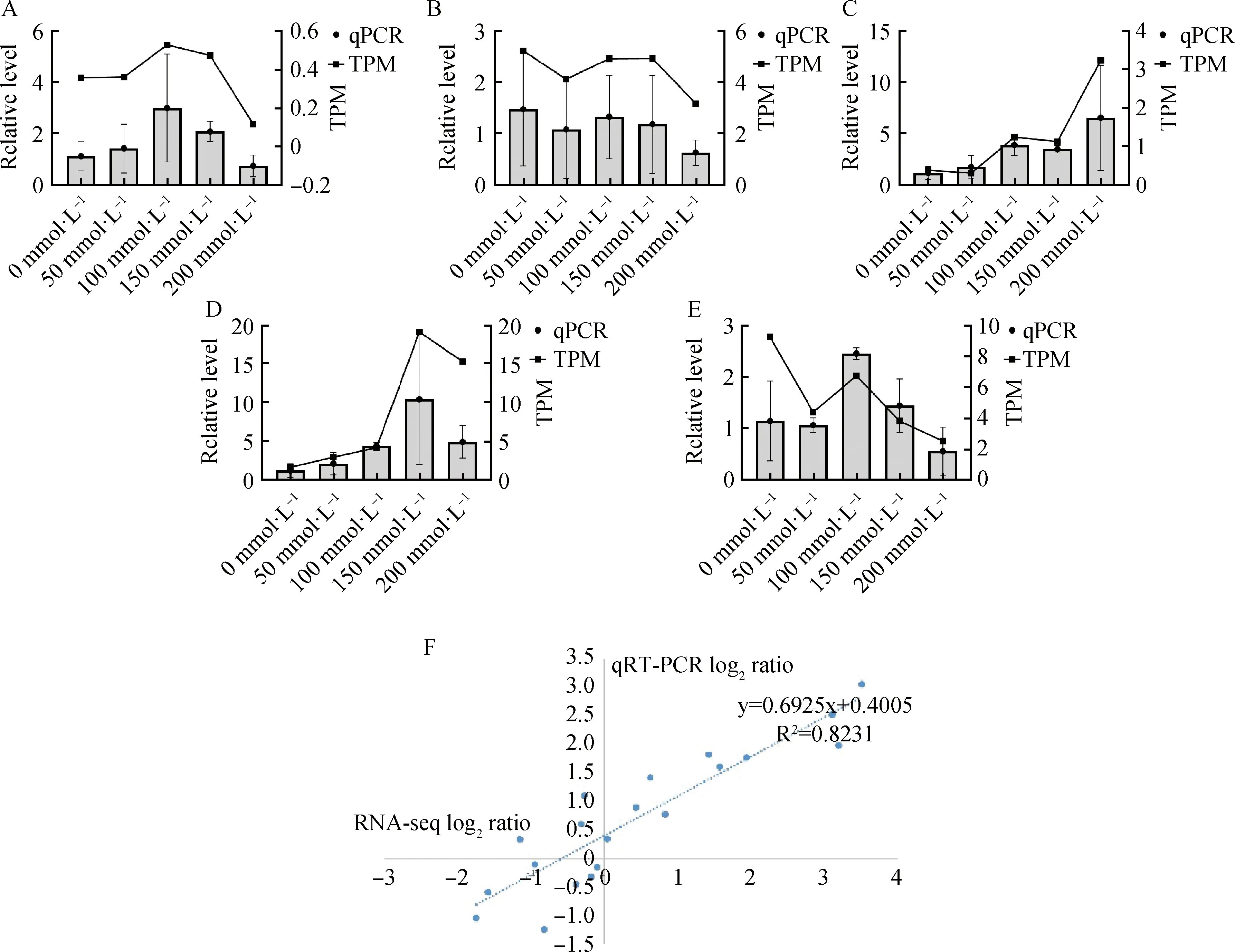
图10 差异表达基因qRT-PCR验证Fig.10 qRT-PCR analysis of differentially expressed genes注:图A~E中左侧纵坐标为qPCR相对表达量,右侧纵坐标为TPM值。A,13361_c0_g1_i15;B,14873_c0_g1_i6;C,70740_c0_g1_i2;D,129260_c0_g2_i1;E,122017_c0_g1_i1;F,相关性分析图Note:In Figures A to E,the left ordinate is the relative expression level of qPCR,and the right ordinate is the TPM value. A,13361_c0_g1_i15;B,14873_c0_g1_i6;C,70740_c0_g1_i2;D, 129260_c0_g2_i1;E,122017_c0_g1_i1;F,Correlation diagram of validated genes
本研究中,对芦竹在0 mmol·L-1,50 mmol·L-1,100 mmol·L-1,150 mmol·L-1和200 mmol·L-1NaCl盐溶液下的超氧化物歧化酶、过氧化物酶、过氧化氢酶活性,脯氨酸、可溶性糖、丙二醛含量和Fv/F0指标进行测定,结果显示随着盐浓度的增加,脯氨酸含量在200 mmol·L-1处达到最高值。可溶性糖含量在150 mmol·L-1处达到了最高,200 mmol·L-1处下降到最低值。而其他生理指标变化并不明显,说明其对盐环境可能并不是十分敏感,并没有对其造成较大的损伤,具有一定的耐受性,也可能是由于胁迫时间与浓度不足导致的。
3.2 芦竹盐响应过程中的关键基因
为了确定芦竹响应盐胁迫的相关基因,利用DESeq2进行基因的差异表达分析。结果显示这些差异表达基因主要与硝酸盐、一氧化氮、活性氮、活性氧、类黄酮生物合成、芪类与二芳基庚烷类和姜酚生物合成、光合作用、玉米素生物合成和硒化合物代谢等条目相关。这些过程可能在芦竹响应盐胁迫的过程中发挥着重要作用。而414_c1_g2_i5(TPC1),4389_c1_g1_i14,4238_c0_g1_i12,0_c1_g3_i2,8776_c0_g1_i37(GSH2)和37109_c0_g1_i12可能是芦竹响应盐胁迫过程中的关键基因。414_c1_g2_i5(TPC1)是从植物克隆的第一个TPC通道[23],定位于液泡膜,负责产生慢速液泡(SV) 电流,参与各种生理过程的调节,例如发芽、气孔开放、茉莉酸的生物合成以及高盐浓度诱导的长距离钙信号传播[24]。4389_c1_g1_i14在细胞分化,细胞壁生物发生,化学稳态,发育生长等方面起作用;4238_c0_g1_i12是与AtMSU81 同源的无功能假基因;0_c1_g3_i2是富含亮氨酸的重复蛋白激酶家族的成员,可以与FRD3相互作用,参与柠檬酸盐外流运输;8776_c0_g1_i37(GSH2)编码谷胱甘肽生物合成酶,三肽谷胱甘肽(GSH)是真核细胞中主要的非蛋白质硫醇化合物,参与细胞氧化还原状态的调节,这在植物暴露于氧化应激期间尤为重要,已被证实能够提高抵抗逆境胁迫的能力[25-26]。37109_c0_g1_i12为HAD超家族,IIIB酸性磷酸酶亚家族成员。
结合生理生化指标对差异基因进行WGCNA分析,以探求与生理变化对应的相关基因,从不同角度研究芦竹的核心盐响应基因。与Pro相关的turquoise,green模块的hub基因编码核碱基抗坏血酸转运蛋白和外膜孔蛋白等相关蛋白质,还包括一些具有某些特定结构的蛋白例如RING/U-box超家族蛋白和RRM结构域蛋白等等。其中核碱基抗坏血酸转运蛋白在植物中运输黄嘌呤和尿酸,已有相关研究证明黄嘌呤和尿酸在缓解盐胁迫方面具有潜在用途,并且过表达核碱基抗坏血酸转运蛋白相关基因可增强植物的耐盐性[27]。而一些与DUF1645[28],RING/U-box[29],RRM[30]和MYB[31]相关的基因家族也被证实在植物应对非生物胁迫过程中发挥着重要作用。
4 结论
本文综合生理学与转录组学来探究芦竹对不同程度盐胁迫的响应变化。结果表明随着盐浓度的增大,芦竹脯氨酸和可溶性糖的含量会发生显著变化。但其对盐环境并不是十分敏感,具有一定的耐受性。而在响应盐胁迫的过程中,差异表达基因主要被富集到了与硝酸盐、一氧化氮、活性氮、活性氧和类黄酮生物合成相关的通路。一些变化倍数较大的核心差异表达基因和一些与脯氨酸含量变化相关的基因可能具有重要作用,可作为候选的耐盐基因。本研究结果可为芦竹耐盐的分子机制研究提供线索,也为今后芦竹的分子育种提供参考。

