大肠杆菌O157:H7与假单胞菌混合菌膜形成时交互作用及其对毒力基因表达的影响
张文栋,张 苏,宓晓雨,程 宇,张 晨,王思亓,王龙凤,江 芸
(南京师范大学食品与制药工程学院,江苏 南京 210023)
大肠杆菌O157:H7作为危害性、致病性和致死性较高的食源性致病菌,其对于食品安全和人群健康具有重大意义,大量文献报道该菌可污染生鲜肉、奶类、蔬菜等农产品。该菌对人体健康的危害与其携带的、、、等毒力基因编码产生的多种毒力因子密切相关。假单胞菌是生鲜肉贮藏末期主要优势腐败菌,也是牛奶、海产品、蔬菜等多种食品中常见腐败菌。研究表明大肠杆菌O157:H7等食源性致病菌及假单胞菌等腐败菌能够黏附于食品加工相关的多种材料表面(如不锈钢、木质、橡胶、塑料、玻璃等)而形成生物菌膜,从而导致食品在屠宰、加工、贮藏、运输、消费各环节受到交叉污染,引发食品安全威胁。生物菌膜对外界应激的抵抗力增强、不易清除,已被证实是最主要的交叉污染源头和传播中心,对食品安全形成重大威胁。
自然环境中,生物菌膜几乎都是由多种微生物形成的,群落内部复杂的相互作用显著影响着生物菌膜的结构与活性。有研究表明混合菌膜比单种菌膜对消毒剂等抗菌物质的抵抗力更强。生鲜肉、奶类等食品中污染的大肠杆菌O157:H7与假单胞菌形成混合菌膜时,将增加食品安全风险。目前,关于大肠杆菌O157:H7与食品中主要腐败微生物混合菌膜形成的研究尚少见报道,尤其是混合菌膜形成时对其毒性的影响报道更少。混合成膜时不同交互作用可能影响混合菌膜的结构、性质,包括致病菌的毒性,进一步影响食品生产加工中生物性危害的防控措施。
因此,本实验结合生产加工实际情况,以不锈钢为生物菌膜形成的载体表面,研究3 株大肠杆菌O157:H7与前期从冷鲜肉贮藏末期分离的3 株假单胞菌及1 株假单胞菌标准菌株为对象,研究两种菌不同菌株之间混合菌膜形成时的交互作用,进一步选取大肠杆菌O157:H7代表菌株,采用反转录实时聚合酶链式反应(reverse transcription real-time polymerase chain reaction,RT-realtime PCR)定量分析其与4 株假单胞菌混合成膜时毒力基因表达的变化。本研究可为揭示食源性致病菌和腐败菌混合菌膜形成的交互作用及进一步风险防控提供科学依据。
1 材料与方法
1.1 菌种与试剂
大肠杆菌O157:H7共3 株:菌株CICC21530,购自中国工业微生物菌种保藏中心;菌株ATCC43895,购自美国菌种保藏中心;菌株95,由南京农业大学江苏省动物源食品生产与安全保障重点实验室惠赠。假单胞菌4 株:荧光假单胞菌ATCC13525(本实验编号为P13525),购自中国工业微生物菌种保藏中心;其余3 株肉源假单胞菌为本实验室前期从腐败猪肉中分离所得,其中荧光假单胞菌1 株P1、莓实假单胞菌2 株P7和P11。
胰蛋白胨大豆琼脂(tryptone soya agar,TSA)、胰蛋白胨大豆肉汤(tryptic soy broth,TSB)、酵母提取物(yeast extract,YE)、山梨醇麦康凯琼脂(Sorbitol macconkey agar,SMAC)、伊红美蓝琼脂(Eosin methylene blue agar,EMB)、假单胞菌CFC选择性培养基基础、CFC基础添加剂、亚碲酸钾、P-09噻孢霉素北京陆桥技术有限公司;利福平、氨苄西林 南京藤春生物科技有限公司;磷酸盐缓冲液(phosphate buffered saline,PBS)(片剂) 南京峰昌生物科技有限公司;氯化钠、乙醇(均为分析纯) 南京化学试剂有限公司;DH5α化学感受态细胞(C502-02) 南京诺唯赞生物科技股份有限公司;质粒提取试剂盒 生工生物工程(上海)股份有限公司;Total RNA提取试剂(D9108A)、Premix(RR901A)、PrimeSTAR Max DNA Polymerase(R045A)、PCR纯化试剂盒(Takara 9761)、pUCm-T质粒、荧光定量试剂盒(RR420A)日本Takara公司。
1.2 仪器与设备
SW-CJ-2F超净工作台 苏州安泰空气技术有限公司;HVE-50立式压力蒸汽灭菌锅 日本Hirayama公司;SG403A生物安全柜 美国Baker公司;HX-4拍打式无菌均质器 上海沪析实业有限公司;QL-200微型涡旋混合仪 海门其林贝尔仪器有限公司;2-16KL高速冷冻离心机 美国Sigma公司;ZQTY-70台式振荡培养箱上海知楚仪器有限公司;DHP-9052型电热恒温培养箱上海一恒科技有限公司;DYCP-32B电泳仪 北京六一生物科技有限公司;Thermal Cycler普通PCR仪 美国Applied Biosystems公司;CFX96实时荧光定量PCR仪、Universal Hood II凝胶成像仪 美国Bio-Rad公司。
1.3 方法
1.3.1 菌液制备
将3 株大肠杆菌O157:H7冻存液划线接种于TSA-YE平板上,37 ℃培养24 h,挑取单菌落接种于3 mL TSB中,37 ℃振荡(220 r/min)培养18 h,再转接1 mL菌液至100 mL TSB中,37 ℃振荡(220 r/min)培养18 h,菌液浓度达到9(lg(CFU/mL))左右。将4 株假单胞菌冻存液分别划线接种于TSA-YE平板上,28 ℃培养48 h,挑取单菌落接种于3 mL TSB中,28 ℃ 振荡(220 r/min)培养24 h,再转接1 mL菌液至100 mL TSB中,28 ℃振荡(220 r/min)培养24 h,菌液浓度达到8(lg(CFU/mL))左右。
1.3.2 大肠杆菌O157:H7和假单胞菌混合培养时的菌体泳动能力分析
将1.3.1节菌液离心,PBS洗涤重悬3 次,调整3 株大肠杆菌O157:H7和4 株假单胞菌的菌液浓度约为6(lg(CFU/mL)),制备接种菌液,其中1 株大肠杆菌O157:H7和1 株假单胞菌包括3 个处理组:大肠杆菌O157:H7与假单胞菌等量混合、大肠杆菌O157:H7与PBS等量混合、假单胞菌与PBS等量混合,备用。
采用软琼脂平板法分析泳动能力(swimming和swarming)。具体方法如下:配制swimming培养基(1%胰蛋白胨、0.5% NaCl、0.3%琼脂粉和0.25%葡萄糖);配制swarming平板培养基(2.5% LB、0.5%葡萄糖和0.5%琼脂粉),于制好的平板中心表面分别滴加3 μL上述备用菌液,分别于25 ℃静置培养8、20 h。用游标卡尺测定细菌扩散菌圈直径,实验重复5 次。
1.3.3 选择性计数平板的筛选
为对混合菌膜中的两种菌进行培养计数,首先需要确定选择性计数平板。分别制备SMAC、CT-SMAC、EMB、TSA-氨苄培养基(100 μg/mL)、TSA-利福平培养基(150 μg/mL)、假单胞菌CFC选择性培养基。将1.3.1节大肠杆菌O157:H7菌液和假单胞菌菌液,经10 倍梯度稀释,选择合适稀释度,各取100 μL分别涂布至上述平板。大肠杆菌O157:H7于37 ℃培养24 h、假单胞菌28 ℃培养48 h,观察菌落形成情况。
1.3.4 大肠杆菌O157:H7与假单胞菌混合菌膜形成时的交互作用
1.3.4.1 混合菌膜制备
食品加工过程中多数接触面为不锈钢材质,因此本实验选用食品级304不锈钢片作为菌膜形成接触面。为防止不锈钢片表面附着化学杂质及油污影响,使用前对不锈钢片进行彻底清洗:丙酮溶液中浸泡3 h,擦拭干净,乙醇溶液浸泡过夜,最后用蒸馏水洗净晾干,实验前于121 ℃灭菌20 min备用。
将1.3.1节大肠杆菌O157:H7菌液和假单胞菌菌液,PBS洗涤重悬3 次,接种至TSB制备两种菌单菌菌液及两种菌混合菌液,其中两种菌浓度均约6(lg(CFU/mL))。分别吸取2 mL加入含有直径1 cm不锈钢片的12 孔板中。封口膜包被,25 ℃静置培养72 h。
1.3.4.2 生物菌膜中细菌的培养计数
培养结束后取出不锈钢片,用无菌生理盐水漂洗3 次以去除未黏附的菌体,放入含有10 mL生理盐水和玻璃珠的离心管中涡旋1 min,梯度稀释,选取合适稀释度涂布于1.3.3节确定的选择性平板上,大肠杆菌O157:H7于37 ℃培养24 h,假单胞菌28 ℃培养48 h后计数。实验重复3 次。
1.3.5 大肠杆菌O157:H7毒力基因表达水平测定
1.3.5.1 生物菌膜和浮游菌的制备与计数
选取1 株大肠杆菌O157:H7代表菌株,将其分别与4 株假单胞菌进行混合培养和菌膜形成,分组同1.3.4节,为了获得更多菌体细胞,于浸没不锈钢片(75 mm×25 mm×1 mm)的TSB中进行,25 ℃静置培养72 h。培养时间结束后,取出不锈钢片,用生理盐水洗涤3 次去除未黏附的菌体,用棉球擦拭法采集钢片上的生物菌膜,将擦拭的棉球置于装有10 mL无菌PBS(pH 7.2)和一定玻璃珠的离心管中,充分涡旋均质3 min,定量吸取一定体积菌悬液12 000×离心15 min(4 ℃),得到生物菌膜的菌体沉淀,以备核酸提取。
取出不锈钢片后,同时对培养液中的浮游菌进行核酸提取。吸取各组浮游菌液4 mL,4 ℃、10 000×离心5 min,PBS重悬洗涤3 次,取菌体沉淀以备核酸提取。
此外,不锈刚片表面形成的菌膜均质后进行选择性平板涂布,浮游菌液经梯度稀释后同时进行大肠杆菌O157:H7选择性平板涂布,37 ℃培养24 h后计数。菌数结果将用于后期基于单位菌数基因表达量的计算。
1.3.5.2 总RNA提取及cDNA合成
按照Total RNA提取试剂(D9108A)说明书进行,用核酸蛋白微量测定仪测量提取的总RNA的核酸浓度和纯度,确定提取的RNA浓度纯度合格(OD/OD在1.8~2.0之间),调整RNA浓度后,根据PrimeScript™RT Master Mix反转录试剂盒进行反转录。反转录的体系(10 μL):2 μL 5×Prime ScriptMaster Mix混合液、2 μL RNA和6 μL Rnase free HO;反应程序为:37 ℃反转录反应15 min,85 ℃反转录酶失活反应5 s,4 ℃,∞。将反转录得到的cDNA于-20 ℃保存进行后续实验。
1.3.5.3 标准检测质粒构建
大肠杆菌O157:H7 4 种毒力基因、、和的引物序列见表1,引物由生工生物工程(上海)股份有限公司合成。反应体系(50 μL):Premix25 μL、上下游引物各1 μL、模板DNA 1 μL、ddHO 22 μL。反应程序为:98 ℃变性10 s,55 ℃退火5 s,72 ℃延伸30 s,反应35 个循环,4 ℃,∞。将上述cDNA分别进行4 种毒力基因普通PCR扩增及验证。验证后利用高保真酶PrimeSTAR Max Premix试剂盒进行PCR扩增以获得目的片段。
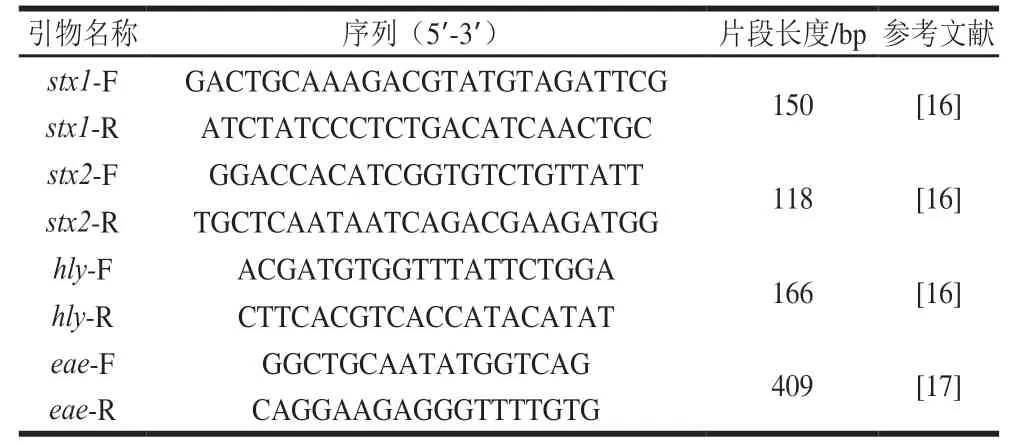
表1 毒力基因real-time PCR的引物Table 1 Primers used for real-time PCR detection of virulence genes
PCR 产物经琼脂糖凝胶电泳和割胶纯化,与pUCm-T vector连接后转入DH5α感受态细胞。蓝白斑筛选阳性克隆子,LB培养基中扩大培养,进一步提取质粒后送生工生物工程(上海)股份有限公司测序,NCBI BLAST比对。比对正确的质粒作为标准检测质粒,采用微量分光光度计测定纯度和浓度,OD/OD值在1.8~2.0之间,表明质粒的纯度较好,利用公式质粒DNA拷贝数/(copies/μL)=(6.02×10)×(质粒浓度/(ng/μL)×10)/(DNA长度×660),计算质粒DNA拷贝数。
1.3.5.4 real-time PCR构建标准曲线
用无菌无酶水对质粒10 倍梯度稀释,以5~6 个梯度的质粒溶液作为cDNA模板,按照SYBR Green Master (ROX) 10 μL、上下游引物各0.4 μL、cDNA 1 μL、ddHO 8.2 μL的反应体系进行real-time PCR。反应条件:95 ℃预变性30 s后,95 ℃变性5 s,60 ℃退火和延伸30 s反应40 个循环,60~95 ℃,熔点分析。以lg copies为横坐标,Ct值为纵坐标,构建一元线性方程,即为标准曲线。
1.3.5.5 菌液和菌膜样品中毒力基因的real-time PCR分析
将1.3.5.2节制备的cDNA作为模板,进行real-time PCR获得毒力基因的Ct值,根据标准曲线分别计算单菌培养、混菌培养的浮游菌中单位体积毒力基因表达量(copies/μL),以及单种菌膜、混合菌膜中单位面积毒力基因表达量(copies/cm)。考虑到单菌培养时、混菌培养时,浮游菌数、菌膜菌数的差异,进一步计算基于单位菌数的毒力基因表达量(copies/CFU),公式如下:

1.4 数据处理与统计分析
利用GraphPad Prism 5软件进行数据处理及图表分析,应用SPSS 17.0软件进行显著性分析,采用ANOVA进行单因素方差分析及Duncan多重比较检验(<0.05),两组之间显著性差异采用检验分析(<0.05)。
2 结果与分析
2.1 两菌混合时的泳动能力变化
大肠杆菌O157:H7菌株CICC21530、ATCC43895和菌株95分别与4 株假单胞菌混合培养时菌体泳动能力见图1~3。结果可见,3 株大肠杆菌O157:H7的泳动能力均显著小于4 株假单胞菌泳动能力(<0.05),两菌混合时的泳动圈均显著大于O157单菌和小于假单胞菌单菌,表明混合培养时假单胞菌的泳动能力受到显著抑制。


图1 大肠杆菌O157:H7菌株CICC21530分别与4 株假单胞菌混合时的菌体泳动能力(n=5)Fig.1 Motility of co-cultures of E.coli O157:H7 CICC21530 with four Pseudomonas strains,separately (n=5)


图2 大肠杆菌O157:H7菌株ATCC43895分别与4 株假单胞菌混合时的菌体泳动能力(n=5)Fig.2 Motility of co-cultures of E.coli O157:H7 ATCC43895 with four Pseudomonas strains,separately (n=5)
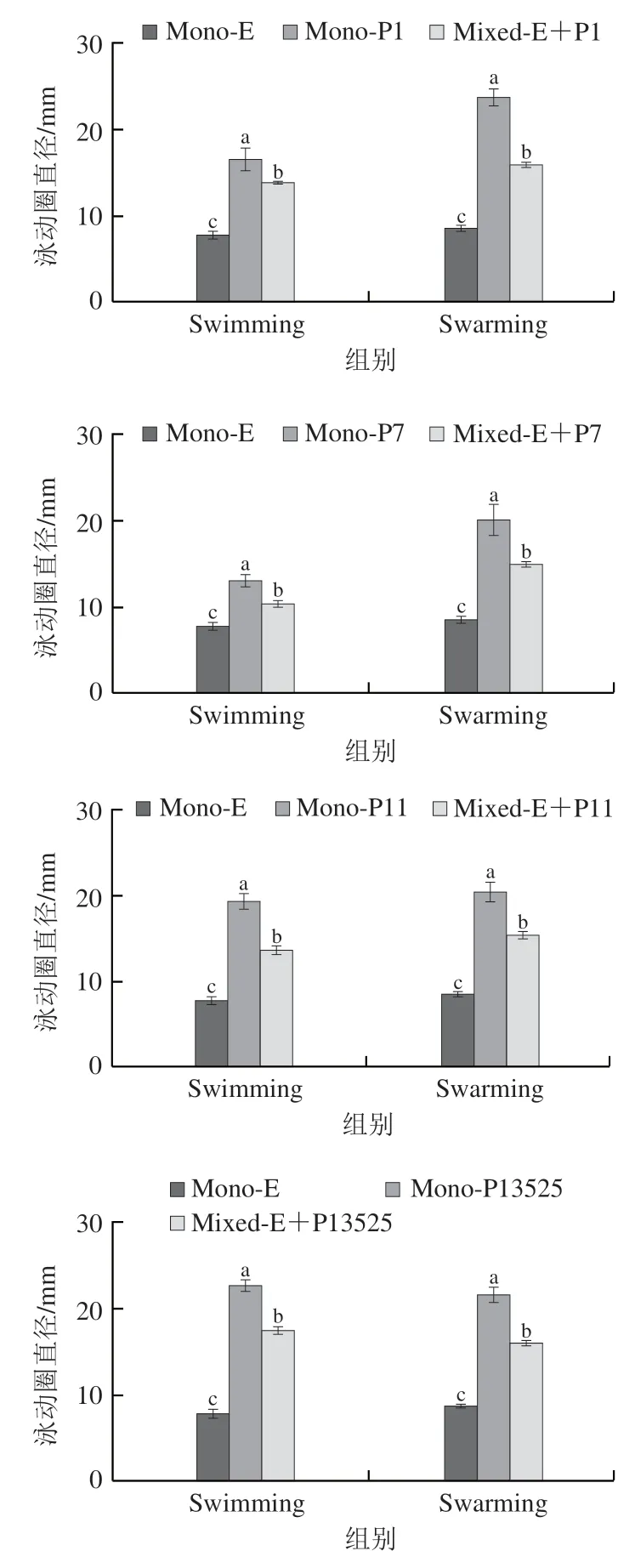
图3 大肠杆菌O157:H7菌株95与4 株假单胞菌混合时的菌体泳动能力(n=5)Fig.3 Motility of co-cultures of E.coliO157:H7 95 with four Pseudomonas strains,separately (n=5)
2.2 选择性计数平板的确定
所用的选择性计数培养基有SMAC、CT-SMAC、SEMB、TSA+利福平平板、TSA+氨苄平板、CFC假单胞菌选择性平板,部分实验结果如图4所示。大肠杆菌O157:H7只在TSA-利福平平板生长,在CFC假单胞菌选择性平板上不生长;假单胞菌在CFC假单胞菌选择性平板上生长,在TSA-利福平平板不生长;两菌在SMAC、CT-SMAC、SEMB、氨苄平板均不生长。因此,本实验将TSA-利福平平板作为混菌中大肠杆菌O157:H7的计数平板,将CFC假单胞菌选择性平板作为混菌中假单胞菌的计数平板。
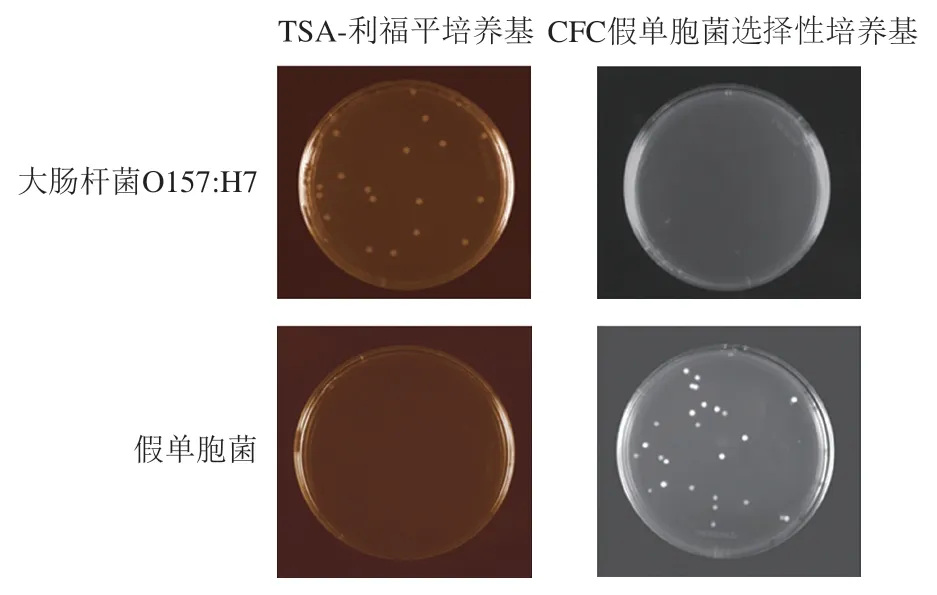
图4 大肠杆菌O157:H7和假单胞菌混合培养计数培养基的确定Fig.4 Determination of counting media for mixed culture of E.coli O157:H7 and Pseudomonas
2.3 大肠杆菌O157:H7和假单胞菌混合菌膜形成时的交互作用
大肠杆菌O157:H7菌株CICC21530、ATCC43895和菌株95分别与4 株假单胞菌25 ℃、72 h菌膜形成情况见图5~7。

图5 大肠杆菌O157:H7 CICC21530和4 株假单胞菌混合菌膜形成Fig.5 Mixed biofilm formation of E.coli O157:H7 CICC21530 and four Pseudomonas isolates
菌株CICC21530与假单胞菌25 ℃、72 h菌膜形成情况如图5所示。CICC21530与假单胞菌P1、P7混合成膜时,混合菌膜中CICC21530与其单种菌膜形成量无显著差异(图5A、B),CICC21530与P11、P13525混合成膜时,显著少于其单种菌膜形成量(<0.05)(图5C、D)。4 种混合菌膜中假单胞菌的量均显著受到CICC21530抑制,少于其单种菌膜形成量(<0.05)。
ATCC43895和P7、P11共培养时,混合菌膜中的量显著低于各自单种菌膜形成量(<0.05),产生了相互抑制(图6B、C)。ATCC43895和P1、P13525共培养时,混合菌膜中ATCC43895菌膜形成未受明显影响,而ATCC43895显著抑制了P1和P13525菌膜形成(<0.05)(图6A、D)。
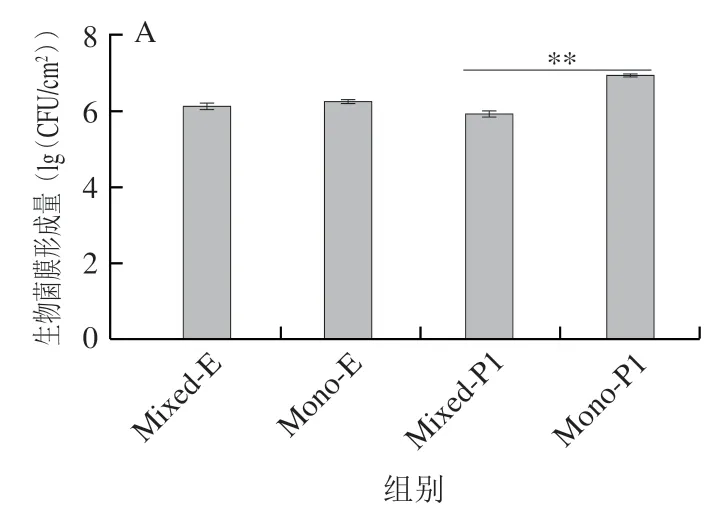

图6 大肠杆菌O157:H7 ATCC43895和4 株假单胞菌混合菌膜形成Fig.6 Mixed biofilm formation of E.coliO157:H7 ATCC43895 and four Pseudomonas isolates
菌株95与P1、P7、P13525会相互抑制菌膜形成(图7A、B、D),混合菌膜中的形成量比其单种菌膜形成量减少(<0.05)。菌株95与P11共培养时,95的菌膜形成未受影响,而95抑制了P11菌膜形成(图7C)。
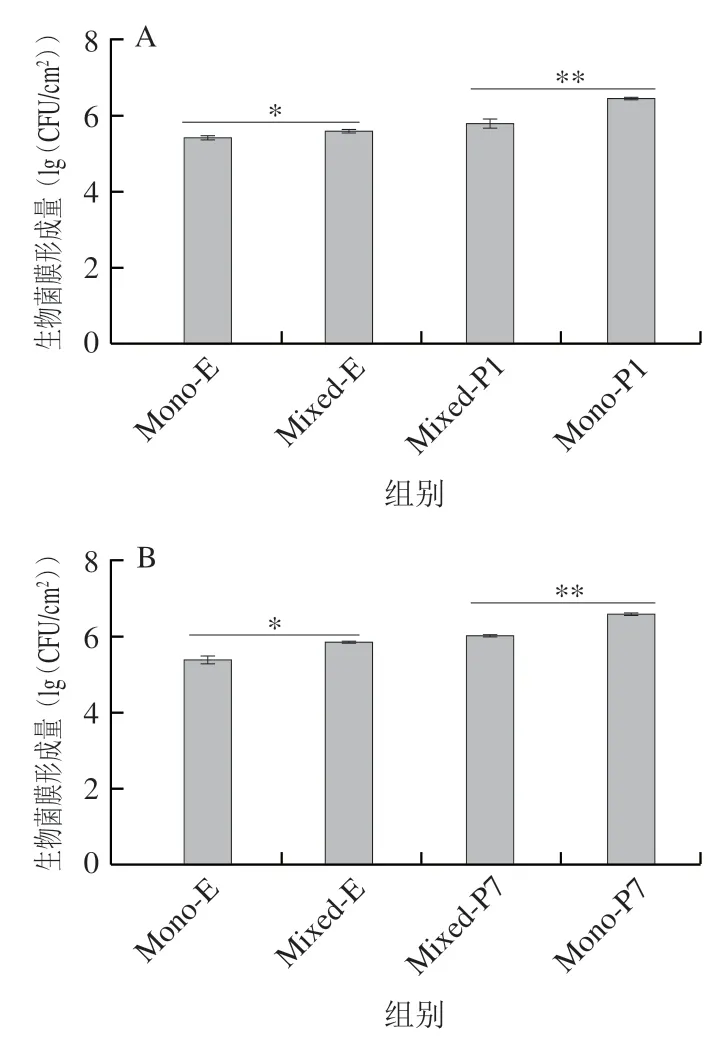

图7 大肠杆菌O157:H7菌株95和4 株假单胞菌混合菌膜形成Fig.7 Mixed biofilm formation of E.coliO157:H7 95 and four Pseudomonas isolates
综上可见,3 株大肠杆菌O157:H7分别与4 株假单胞菌混合菌膜形成时,两种菌未发生相互促进,而4 株假单胞菌菌膜形成均受到了大肠杆菌O157:H7的抑制。
2.4 大肠杆菌O157:H7 CICC21530毒力基因表达分析
2.4.1 4 种毒力基因标准曲线方程
大肠杆菌O157:H7 CICC21530的4 种毒力基因、、和标准曲线方程如表2所示,其中为标准品拷贝数的对数值,为测得的Ct值。由表2可知,相关系数均大于0.99,Ct值在5~40之间,满足标准曲线要求。

表2 毒力基因stx1、stx2、eae和hly的标准曲线Table 2 Standard curves for quantitation of the virulence genes stx1,stx2,eae and hly
2.4.2 浮游菌中单位体积毒力基因表达量的变化
大肠杆菌O157:H7 CICC21530与4 株假单胞菌(P1、P7、P11、P13525)共培养时,CICC21530单菌培养及混合培养时浮游菌中单位体积毒力基因(、、和)的表达量如图8所示。与CICC21530单菌培养相比,混合培养时CICC21530单位体积4 种毒力基因表达量均显著减少(<0.05),4 株假单胞菌产生了不同程度的抑制作用。

图8 单菌培养及混合培养时浮游菌中单位体积毒力基因表达量Fig.8 Virulence genes expression per unit volume of planktonic cells in single and mixed cultures
2.4.3 生物菌膜中单位面积毒力基因表达量的变化
大肠杆菌O157:H7 CICC21530与4 株假单胞菌共培养时,CICC21530单种菌膜及混合菌膜中单位面积毒力基因(、、和)的表达量如图9所示。与单种菌膜相比,混合菌膜中CICC21530单位面积4 种毒力基因表达量均显著减少(<0.05)。
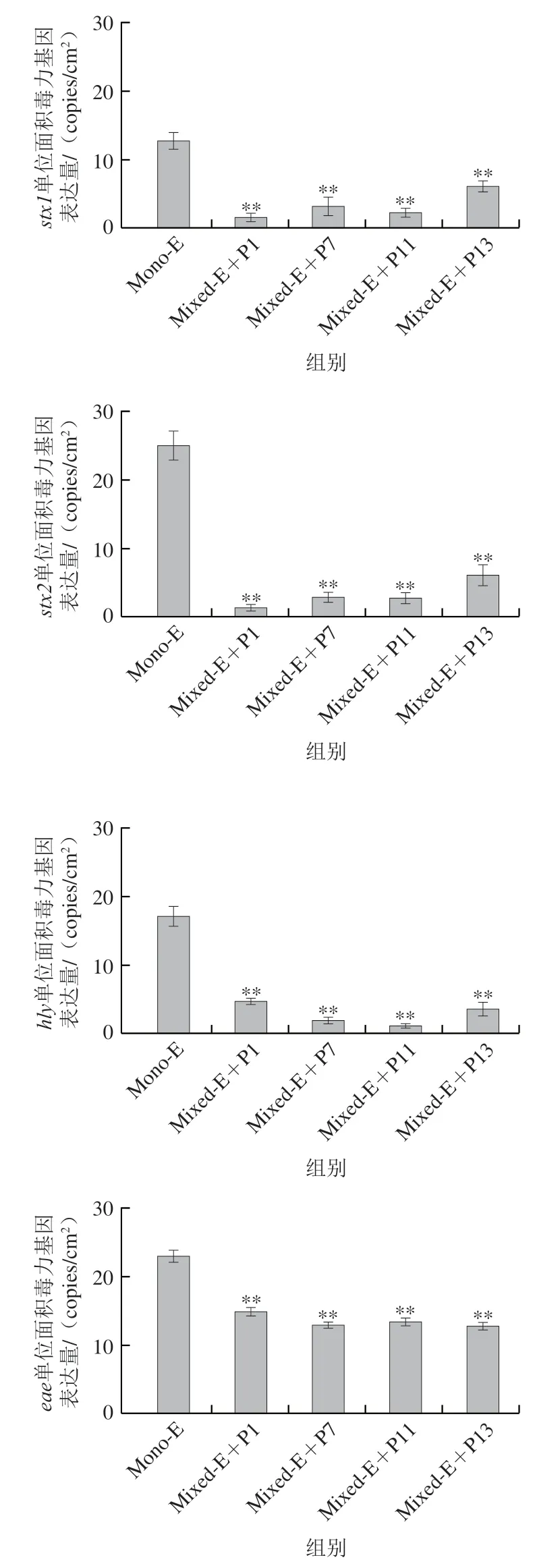
图9 单种与混合生物菌膜中单位面积毒力基因表达量Fig.9 Virulence genes expression per unit area in single and mixed biofilms
2.4.4 浮游菌中单位菌数毒力基因表达量的变化
CICC21530与4 株假单胞菌共培养时,CICC21530单菌培养及混合培养时浮游菌中单位菌数毒力基因的表达如图10所示。与单菌培养相比,混合培养时浮游菌中CICC21530单位菌数4 种毒力基因表达量均显著减少(<0.05)。
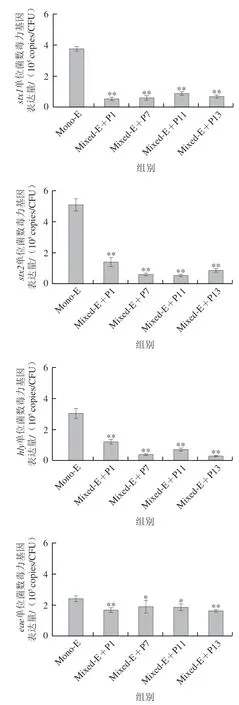
图10 单菌培养及混合培养时浮游菌中单位菌数毒力基因表达量Fig.10 Virulence genes expression per unit cell number of planktonic cells in single and mixed cultures
2.4.5 生物菌膜中单位菌数毒力基因表达量的变化
CICC21530与4 株假单胞菌共培养时,CICC21530单种菌膜及混合菌膜中单位菌数毒力基因表达量如图11所示。与单种生物菌膜相比,CICC21530混合菌膜中单位菌数4 种毒力基因表达量均显著减少(<0.05)。
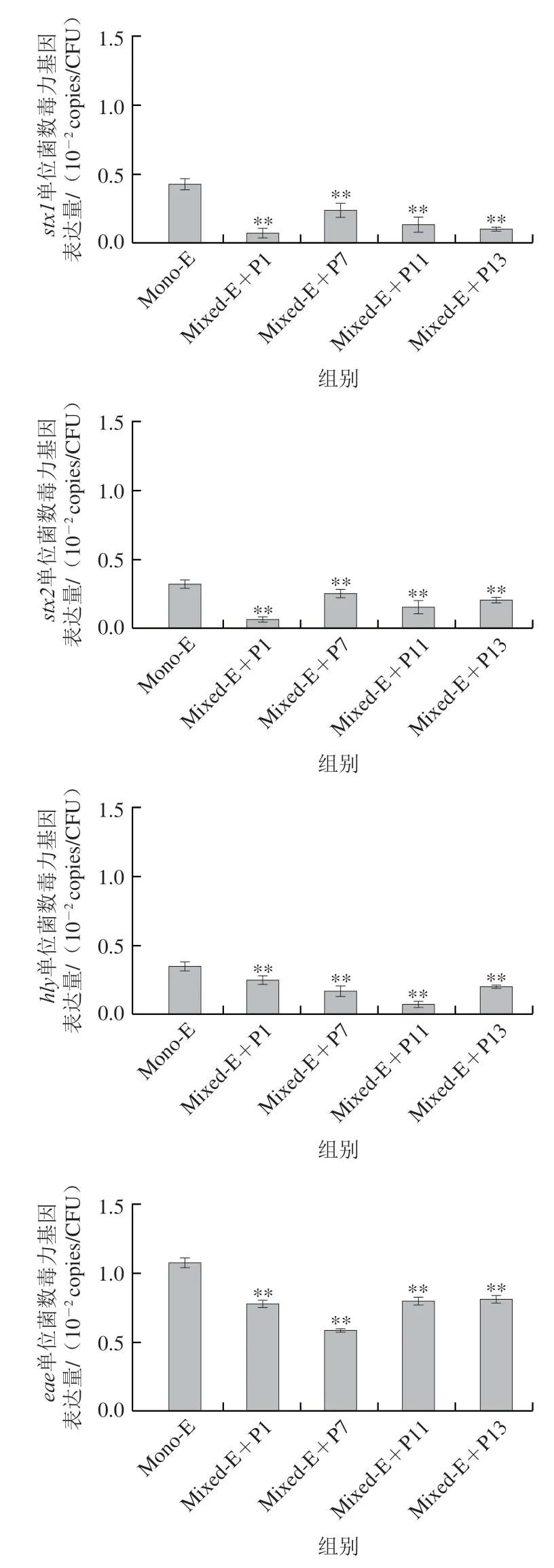
图11 单种与混合生物菌膜中单位菌数毒力基因表达量Fig.11 Virulence genes expression per unit cell number in single and mixed biofilms
综合2.4.4节基于单位菌数的结果,表明CICC21530生物菌膜中4 种毒力基因表达量均显著高于浮游菌中的表达量(<0.05),表明生物菌膜的形成增强了菌体毒力基因的表达。
3 讨论
大肠杆菌O157:H7和假单胞菌可以同时存在于肉类、奶类、蔬菜等食品中,当它们形成混合菌膜时将大大增加食品安全风险。本实验首先从泳动性反映混合培养时两菌之间的相互作用。细菌的泳动能力包括swimming(单一菌体运动行为)和swarming(群体菌体运动行为),研究表明生物菌膜的形成与细菌泳动能力呈现一定相关性,菌体通过泳动到达材料表面并产生附着,在鞭毛/菌毛的作用下黏附固着进一步形成生物菌膜。本实验发现,大肠杆菌O157:H7与假单胞菌各菌株菌体泳动性存在差异,这与铜绿假单胞菌、芽孢杆菌、蜂房哈夫尼菌、沙门氏菌、副溶血弧菌等细菌泳动性的研究报道结论相似。本实验中大肠杆菌O157:H7与假单胞菌相比,假单胞菌的泳动能力更强,这可能与假单胞菌的菌体特性(鞭毛/菌毛及结构)、良好的适应环境与繁殖能力有关。不同细菌混合培养时,细菌间交互作用会影响各自泳动能力。
食品中存在复杂的微生物生态系统,它们之间在不同生存环境下可能产生不同的交互作用。同样,混合菌膜内不同菌种之间也可发生不同交互作用,呈现促进、竞争、或中立效应。关于大肠杆菌或大肠杆菌O157:H7与其他细菌的多菌种混合菌膜形成有不同研究报道。有研究发现粪肠球菌、不动杆菌、恶臭假单胞菌、铜绿假单胞菌、食品加工厂一些分离杂菌等促进大肠杆菌或大肠杆菌O157:H7的菌膜形成;另一些研究发现,铜绿假单胞菌、沙门氏菌、格式假单胞菌等抑制大肠杆菌或大肠杆菌O157:H7的菌膜形成。反之,发现大肠杆菌或大肠杆菌O157:H7可抑制铜绿假单胞菌、成团泛菌的菌膜形成,但未影响沙拉加工厂中分离菌株的黏附。关于假单胞菌与其他细菌混合菌膜的相关研究中也有不同报道。Kives等将原料乳中分离的荧光假单胞菌和乳酸乳球菌在脱脂乳中7 ℃共培养72 h探究混合菌膜的形成,研究发现两种菌的成膜量均显著高于单菌的成膜量;Zhu Junli等利用结晶紫染色法定量荧光假单胞菌和波罗的海希瓦氏菌不同培养温度(4、15 ℃和30 ℃)下单种和混合菌膜形成量差异,结果表明两种菌的成膜量均显著低于单菌;Lapointe等探究了荧光假单胞菌、植物乳杆菌、假肠膜明串珠菌在20 ℃共培养12 d时混合菌膜的形成,结果发现混合菌膜中的明串珠菌、植物乳杆菌的成膜量均显著高于单菌,而荧光假单胞菌与单菌时无显著差异。本实验中3 株大肠杆菌O157:H7和4 株假单胞菌混合成膜时,3 株大肠杆菌O157:H7菌膜形成未受影响或受到假单胞菌的抑制,而4 株假单胞菌菌膜形成均受到了大肠杆菌O157:H7的抑制。上述关于混合成膜时细菌间相互作用的研究报道不完全一致,这可能与不同菌种、菌株及成膜条件差异性相关。
菌膜形成时,由于生存环境和生存状态改变,有可能导致致病菌的毒力发生变化,进而影响食品安全风险。Wang Yang等利用RT-real-time PCR相对定量发现,与浮游菌中毒力基因表达量相比,猪链球菌生物菌膜中、和三种毒力基因表达量下调;谭芳等利用RT-real-time PCR相对定量发现在铜绿假单胞菌生物菌膜形成过程中,其毒力基因表达增强,毒力基因调控的毒素LasA、LasB弹性蛋白酶、鼠李糖脂、绿脓素分泌均增高;Wang Huhu等采用RT-real-time PCR相对定量研究沙门氏菌发现,与浮游菌相比,在TSB中成膜后毒力基因表达量呈现显著上调的趋势,而在肉汁渗出液中成膜后基因在7 d时出现显著降低,和毒力基因表达量在培养周期内持续降低。Sánchez等通过转录组分析比较了牙龈卟啉单胞菌在生物菌膜和浮游菌状态下基因表达差异,发现在生物菌膜中与细菌黏附、侵袭或毒力相关的几个编码蛋白的基因(、、、、)表达上调;Charlebois等通过RNA测序研究了产气荚膜梭菌在生物菌膜和浮游菌之间基因表达差异,发现生物菌膜中编码-氯条蛋白、胶原酶、坏死性肠炎毒素B、全溶素O和磷脂酶C的基因下调,而编码溶血素的基因上调。上述生物菌膜中毒力基因表达变化的研究报道并不完全一致,可能与不同菌种、不同菌株以及不同阶段的生物菌膜等有关。本实验通过绝对定量分析,消除菌数差异后发现大肠杆菌O157:H7生物菌膜中4 种毒力基因(、、和)表达量均显著高于浮游菌,表明生物菌膜的形成不仅导致致病菌抗性增强,不易消除,还可能导致毒力增强,从而增加食品安全风险。
上述是关于单种菌菌膜形成时毒力基因表达变化,实际生产加工中,食品加工接触表面形成的生物菌膜大多是由多种细菌组成的混合菌膜,研究混合菌膜形成时致病菌毒力的变化更具有实际意义。Kart等利用RTreal-time PCR相对定量,以看家基因为内参基因,分析了与变形杆菌混合成膜时白色念珠菌毒力基因表达的变化,发现与单种菌膜相比,白色念珠菌和基因表达显著上调,、和基因表达下调。Cavalcanti等利用相对定量,以为内参基因,研究混合成膜时白色念珠菌毒力基因表达变化,发现与白色念珠菌单种菌膜相比,白色念珠菌和血链球菌混合成膜时、、和显著上调,白色念珠菌和牙龈卟啉单胞菌混合成膜时和表达下调,当3 种菌混合成膜时白色念珠菌上调了、和,白色念珠菌毒力因子表达与混合菌膜中不同的细菌种类有关。Cope等利用相对定量,分别以基因、基因为内参基因,研究肺炎链球菌和流感嗜血杆菌混合成膜时毒力基因表达的变化,发现混合成膜时肺炎链球菌丙酮酸氧化酶基因表达上调,而溶血素、黏附因子A的基因表达下调。陈忠军等采用相对定量,以16S rRNA为内参基因,研究3 种细菌混合成膜时金黄色葡萄球菌毒力基因表达差异,发现与金黄色葡萄球菌单种菌膜相比,混合菌膜中金黄色葡萄球菌毒力基因上调显著的有肠毒素基因、,显著下降的有溶血素基因和脱皮毒素基因。需要注意的是,采用RTreal-time PCR相对定量对混菌中的某一种菌进行基因表达分析时,应该选择该菌特异的内参基因。目前,采用RT-real-time PCR绝对定量方法研究混合菌膜中致病菌毒力基因表达差异,此类报道尚少见。绝对定量不仅可了解接触面或生长环境中因细菌毒力基因表达差异而引起的安全风险变化,亦可通过消除菌数差异来分析菌体毒力基因表达的变化。本实验采用绝对定量发现大肠杆菌O157:H7和假单胞菌混合成膜时,与大肠杆菌O157:H7单种菌膜相比,混合菌膜中单位面积4 种毒力基因表达量均显著减少,表明接触面混合菌膜的风险性比大肠杆菌O157:H7单种菌膜有所降低;消除菌数差异后发现,单位菌数4 种毒力基因表达量均显著减少,表明混合成膜时假单胞菌抑制了大肠杆菌O157:H7菌体毒力基因的表达。值得注意的是,该菌感染剂量极低,致病性较强,故即使毒力基因表达减少,其菌膜的安全风险仍不容忽视。食品生产加工中不同环境条件、不同来源菌种和菌株等可导致它们发挥不同交互作用,在生产加工接触面形成不同结构和性质的混合菌膜,并可进一步影响其抗性及致病性。因此结合实际食品环境,加强对食源性有害分离菌株混合菌膜形成和控制的深入研究具有重要的科学意义。
4 结论
本实验探讨了主要食源性致病菌大肠杆菌O157:H7和食品中常见腐败菌假单胞菌的不同菌株之间混合生物菌膜形成时的交互影响。细菌泳动性结果提示两种菌共培养时假单胞菌泳动能力受到显著抑制。生物菌膜的培养计数发现两种菌混合成膜时,不同菌株之间交互作用不完全相同,两种菌未发生相互促进,其中4 株假单胞菌的菌膜形成均受到3 株大肠杆菌O157:H7显著抑制。大肠杆菌O157:H7 CICC21530与4 株假单胞菌共培养成膜时,采用RT-real-time PCR绝对定量发现,与大肠杆菌O157:H7单种菌膜相比,混合菌膜中单位面积毒力基因表达量均显著减少,表明接触面混合菌膜的风险性比大肠杆菌O157:H7单种菌膜有所降低;消除菌数差异后发现,单位菌数毒力基因表达量均显著减少,表明混合成膜时假单胞菌抑制了大肠杆菌O157:H7菌体毒力基因的表达。此外,菌膜中单位菌数毒力基因表达显著高于浮游菌,表明菌膜形成后大肠杆菌O157:H7毒力增强,安全风险增加。本研究可为揭示食源性致病菌和腐败菌混合菌膜形成的交互作用及进一步风险评估和风险防控提供科学依据。

