84个苹果栽培品种对斑点落叶病的抗性评价和全基因组关联分析
储宝华,曹富国,卞宁宁,钱谦,李中兴,李雪薇,刘泽远,马锋旺,管清美
西北农林科技大学园艺学院,陕西杨凌 712100
0 引言
【研究意义】中国是世界上苹果的生产和出口大国。自2007年以来,中国每年的苹果总产量均占世界总产量的45%以上,2019年中国的苹果产量甚至达到4 100万t,占世界苹果总产量的54.07%(数据来源于苹果产业大数据分析平台(agdata.cn))。早期落叶病是威胁中国苹果生产的主要病害之一,包括褐斑病、斑点落叶病、灰斑病、圆斑病、轮斑病等多种病害,其中斑点落叶病和褐斑病的危害最为严重。斑点落叶病主要通过危害叶片影响树体生长,进而影响苹果的产量和品质。感染斑点落叶病后,苹果叶片表面产生褐色斑点,随后黄化脱落,造成树势早衰;此外,斑点落叶病也会感染幼果从而引发果实腐烂。斑点落叶病严重的果园中叶片感染率能够达到90%左右,落叶率达到80%,给苹果产业造成极大的经济损害。目前关于苹果栽培品种对早期落叶病的抗性研究多集中于褐斑病、腐烂病、枝干轮纹病方面,关于斑点落叶病在苹果生产中的研究则集中于农业防治方面,且关于不同栽培品种对斑点落叶病抗性系统评价的相关研究还较少。目前鉴定到的高抗性苹果栽培品种数量稀少,因此急需挖掘具有高抗病性的苹果栽培品种以满足市场的栽培需求。通过比较国内外苹果栽培品种对斑点落叶病的抗性,从而全面系统地认识不同栽培品种的抗病能力,筛选出稳定的抗性品种,是解决苹果园受斑点病侵害的有效途径。【前人研究进展】全基因组关联性状分析(genome-wide association study,GWAS)是一种用于开展连锁标记开发和关键调控基因挖掘的有效方法,目前在各类粮食作物如水稻、小麦、玉米上广泛应用。PAULINO 等通过GWAS分析发现抗枯萎病相关的关键 SNP位点和关键基因;YANG等整合多种分析方法,如Meta-QTL、GWAS和同源性分析,揭示了一个新的调控小麦粒径大小的关键基因—。目前GWAS在苹果上的应用还很少,且现有的研究主要集中在果实品质方面。如FARNETI等将基因组与苹果挥发性物质相关联,鉴定到9 142个SNP和两个与芳香性物质紧密相关的候选基因和;LIAO等也利用 GWAS发现与苹果果实酸度紧密关联的基因和。【本研究切入点】利用全基因组关联分析挖掘苹果抗斑点落叶病的单核苷酸多态性位点(SNP)及关键候选基因。【拟解决的关键问题】本研究材料的选择基于课题组现有的苹果栽培品种 84份,通过离体叶片接种病原菌的试验评价斑点落叶病抗性,综合多次试验结果分析病斑面积、病斑面积增长率,结合聚类分析的手段,筛选出苹果栽培品种中稳定的抗性品种。
1 材料与方法
1.1 供试材料
试验材料栽植于苹果资源圃。苹果栽培品种共84种,2018年4月将84个栽培品种采用嵌芽接的方式嫁接于砧木‘平邑甜茶’,每个品种均有5个生物学重复。试验分别于2019和2020年进行,于8月中旬采集植物离体叶片,每个品种5个生物学重复,每个生物学重复中采取植株中部健康完整的成熟叶片3—5片。
1.2 接种方法
试验所用的苹果斑点落叶病病菌(f. sp.)来源于西北农林科技大学孙丽英教授实验室,菌种保存,扩繁的方式参考ZHANG等。具体接种方法如下:病菌活化培养4 d后,切取菌块放置在PDA平板中央,黑布遮光,25℃培养4 d,待菌落直径长到2 cm时,用5 mm打孔器取菌落周围新长出的白色菌丝用于叶片接种。接种时将叶片清洗干净,叶柄用湿棉花包裹以防叶片失水,用手术刀刀尖在叶片正面的叶脉左右两侧中部刺伤,于伤口处放置菌块。接完种的叶片放于托盘中,覆盖保鲜膜,黑布遮光培养,于25℃培养4 d和8 d后分别拍照,统计叶背面病斑面积。
1.3 抗性评价的方法
统计接种后4 d和8 d叶背面的病斑面积大小,病斑面积越大,说明植物的抗病能力越弱。此外,通过4—8 d病斑面积增长率,来判断品种抗扩展能力。病斑增长率计算公式:

1.4 全基因组关联分析
使用高效混合模型关联程序EMMAX进行关联分析,截止点=0.0000001(-LgP=7),进行关联位点的检测。本研究利用全基因组深度测序获得的1 243 071个高质量SNP位点,用EMMAX-Kinship程序计算,从所有的SNP中得到亲缘关系矩阵。基于以上SNP信息,用GCTA(1.01版)程序对苹果84个栽培品种进行主成分分析并获得前 10个主成分矩阵。最后,将84份苹果栽培种接种后叶片的病斑面积数据作为表型,全基因组深度重测序获得的1 243 071个高质量SNP位点为遗传标记,结合亲缘关系矩阵和主成分分析结果,采用EMMAX方法进行全基因组关联性状分析。
1.5 候选基因注释
用ANNOVAR软件,结合苹果参考基因组GDDH13(https://iris.angers.inra.fr/gddh13/)对全基因组关联分析得到的SNP进行注释,获得每个SNP上下游约50 kb的突变位点和SNP位点相邻基因的MD号。使用NCBI-Blast-2.9.0+中 Blastp和拟南芥蛋白组 Tair10(https://www.arabidopsis.org/)进行亲缘关系比对。用Python3.7版对数据进行处理,获得SNP相邻候选基因的注释列表。
1.6 数据统计及分析
病斑面积用 Image J软件统计分析,使用 Excel计算平均值和标准差,用SPSS分别对2020年和2021年的病斑面积和病斑面积增长率进行聚类分析,聚类分析方法采用瓦尔德法,聚类距离采用平方欧式距离。
1.7 基因表达分析
根据GDR网站上、、、、、、的mRNA 序列(https://www.rosaceae.org/),在 NCBI网页上设计荧光定量PCR引物(表1),将接种后的GL-3组培苗叶片样品在液氮中充分研磨,用Wolact®Plant RNA Isolation Kit(Hong Kong,China)试剂盒进行 RNA 提取, 使用HiScript Ⅱ Q RT SuperMix for qPCR (+gDNA wiper)进行反转录,定量试剂用ChamQ SYBR Qpcr Master Mix(Q311-02/ 03),反应体系参考试剂使用说明。使用Bio-RadCFX96TM(Bio-Rad)荧光定量 PCR仪进行反应,以接种 0、12、24和 48 h的叶片样品为模板进行基因的相对表达量分析,用作内参基因,使用2计算基因的相对表达水平。

表1 引物序列Table 1 Primer sequence
1.8 苹果叶片的瞬时转化
将的基因编码区(CDS区)进行克隆后构建到入门载体pDONR222,通过LR反应构建至表达载体 PK7-203,引物设计见表1,构建好的表达载体转入农杆菌C58C1中,通过农杆菌介导的苹果叶片瞬时表达系统。试验方法参考ZHANG等,具体步骤如下:将 100 μL 浓度为 100 μmol·L的乙酰丁香酮加入100 mL LB中,加入100 μL的载体转化农杆菌,在28℃培养箱,130 r/min振荡培养12—16 h,直至OD=1,5 000 r/min离心10 min。将农杆菌悬浮于100 mL液体培养基(10 mmol·LMES-KOH(pH 5.2)、10 mmol·LMgCl和 180 μmol·L乙酰丁香酮)中,使用前培养2 h。利用4周大的组培苗进行抽真空,真空压力为0.06—0.08 Mpa,抽真空时间约4—5 h,直至整个叶片出现水渍状,之后将组培苗放回MS培养基进行培养(25±1℃,白天/黑夜16 h/8 h)。在农杆菌侵染4 d后进行离体叶片接种试验,接种3 d后进行病斑面积统计。
2 结果
不同类型的苹果属栽培品种在接种斑点落叶病原菌后,其叶面受侵染的严重度表现出显著多样性。为了系统评价不同栽培品种对斑点落叶病的抗性差异,本研究将叶片坏死的病斑面积作为评价不同苹果属栽培品种对斑点落叶病抗性的标准,病斑面积越大,则植物的抗病能力越差;反之,病斑面积越小,则植物的抗病能力越强。由于单次试验结果具有片面性,本研究连续2年统计了4次试验结果(2次8 d结果,2次4 d结果),最终选择4次数据评价稳定的品种确定为抗性或感病品种。因此,本结果能够更客观地系统评价 84个苹果栽培品种的抗病能力。此外,利用全基因组关联性状分析的手段鉴定抗病性相关的关键 SNP位点,并筛选关键的潜在调控基因。
2.1 84个苹果栽培品种的抗病性差异比较(2020年)
84个苹果栽培品种的离体叶片在接种斑点落叶病菌株后均有不同程度的发病(表2),目前未发现完全免疫的品种。2020年接种8 d后的病斑面积数据显示,病斑面积的分布范围在 0.76—6.02 cm,‘凯密欧’的病斑面积最小,仅为(0.76±0.14)cm;而‘Chanterler’的病斑面积最大,达到(6.02±0.23)cm。另外,为了进一步衡量品种抗扩展能力,进行了4—8 d内的病斑面积增长率的分析。数据显示,‘凯密欧’病斑面积增长率最低,仅为15.00%;而病斑面积增长率最高的‘埃德尔博斯多夫’达 128.25%。综合病斑面积和病斑增长率两类指标,进行聚类分析(图1-a)。84个栽培品种中,‘凯密欧’‘Kiku’‘JA’‘皇家嘎啦’‘秋光’‘蜜脆’‘蓝皮尔曼’‘福拉瑞娜’‘Roho3615’‘艾达红’‘威廉姆斯女士’‘弘前富士’‘Fujiko’‘红盖露’‘Gloster69’‘长富2号’‘早红1号’聚为一类,为抗病品种;‘Chanterler’‘埃德尔博斯多夫’‘比蒂格海姆’‘信浓红’‘赫拉森’‘阿丽亚娜’‘2001富士’聚为一类,为易感品种。

表2 84个苹果属栽培品种离体叶片接种病斑大小及病斑面积增长率(2020年)Table 2 Lesion area and its growth rate of 84 apple cultivars inoculated with Alternaria alternata f. sp. Mali

图1 2020年(a)和2021年(b)84个栽培品种对斑点落叶病抗性的聚类分析Fig. 1 Cluster analysis of resistance of 84 cultivars to Alternaria alternate f. sp. mali in 2020 (a) and 2021 (b)
2.2 不同类型的栽培品种的抗病性差异比较(2021年)
为了进一步证明试验结果的可靠性,于2021年进行了重复试验(表3)。2021年接种8 d后的病斑面积数据显示,病斑面积的分布范围在0.3—2.83 cm。病斑面积最小的是‘蓝皮尔曼’,仅为(0.3±0.11)cm。而接种8 d后病斑面积最大的是‘茜’,达到(2.83±0.47)cm。4—8 d内的病斑面积增长率分析结果显示,‘蓝皮尔曼’的病斑面积增长率最低,仅为 4.75%。而病斑面积增长率最高的品种是‘茜’,其病斑面积增长率高达 56.25%。综合病斑面积和病斑面积增长率,可以看出病斑面积和病斑面积增长率基本保持一致。2021年聚类分析结果显示(图1),84个栽培品种中,‘蓝皮尔曼’‘平成’‘艾达红’‘Yellow Transparent’‘Ce1’‘NJ-90’‘无锈金冠’‘秦冠’‘皇家嘎啦’‘皮诺娃’‘玫瑰光芒’‘夕阳’‘Northfield Beauty’‘信浓甜’‘宝罗红’‘Kiku’‘Gloster69’‘阿莱特’‘蜜脆’‘Challenger’‘凯密欧’‘秦阳’‘美味’‘Ce2’‘Su’聚为一类,说明他们是抗性较强的品种;而‘茜’单独聚为一类,是极其不抗的品种;此外,‘Fujiko’‘埃德尔博斯多夫’‘千秋’‘斯派克’‘K296’‘昂林’‘早红霞’‘甘红’‘王林’‘Redcord’‘富红早嘎’‘长富2号’‘Chanterler’‘新乔纳金’‘荷斯坦’‘Ruby Mac’聚为一类,是低抗性品种。
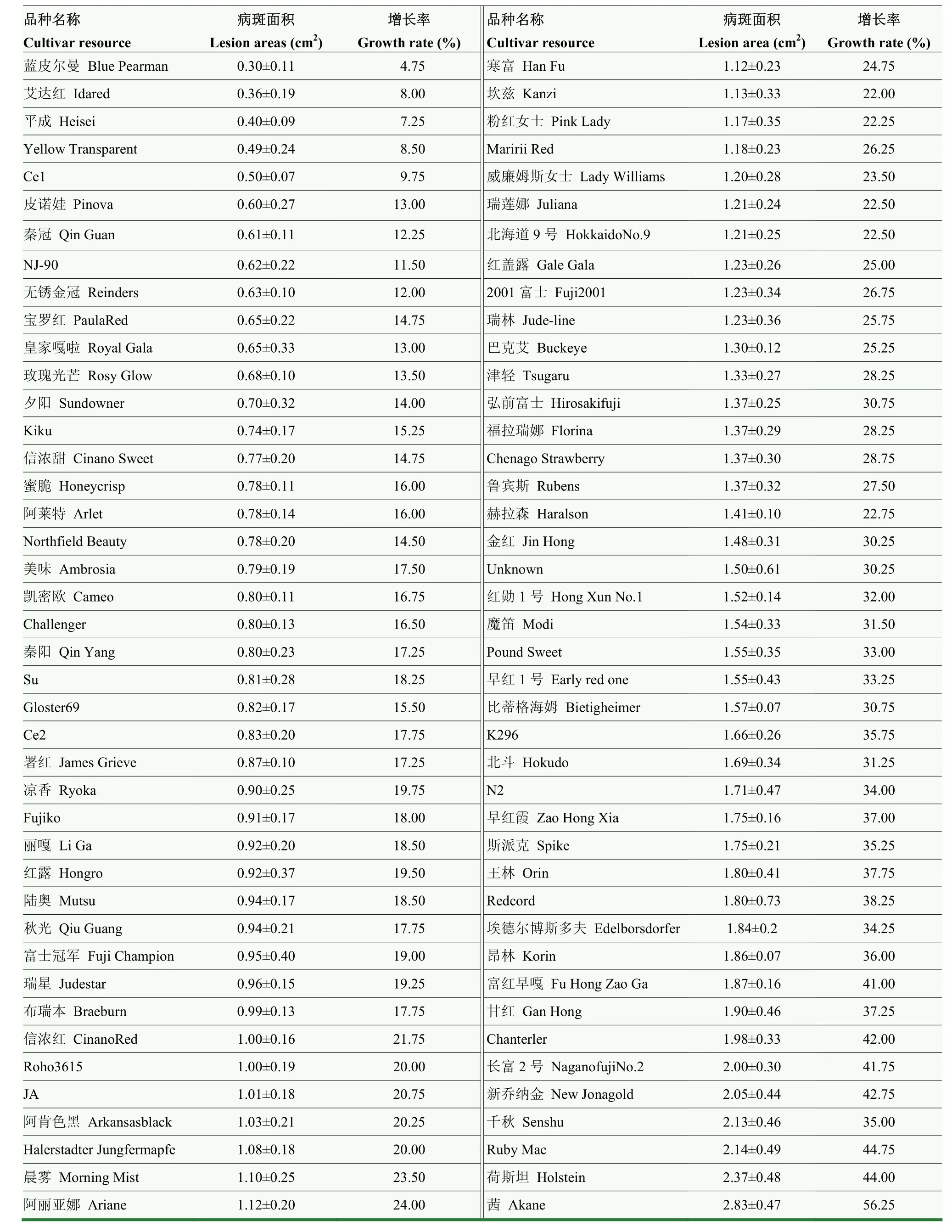
表3 84个苹果属栽培品种离体叶片接种病斑大小及病斑面积增长率(2021年)Table 3 Lesion area and its growth rate of 84 apple cultivars inoculated with Alternaria alternata f. sp. mali
综合两年的聚类分析数据,‘凯密欧’‘Kiku’‘皇家嘎啦’‘蜜脆’‘蓝皮尔曼’‘艾达红’‘Gloster69’在两年的抗性分析中均为抗性品种;而‘埃德尔博斯多夫’‘Chanterler’均为低抗性品种。
2.3 苹果早期斑点落叶病抗病性与 SNP的全基因组关联分析(GWAS)
为了进一步探索苹果抗早期斑点落叶病的遗传基础,对 84份苹果栽培品种在接种苹果斑点落叶病8 d后的病斑面积数据进行GWAS分析。因为2020年8 d接种的病斑面积分布更符合正态分布的趋势,因而选其数据作为表型进行全基因组关联分析。供试群体的病斑面积最小0.3 cm,最大为2.14 cm。病斑面积的峰度和偏度分别为-0.74和0.34,二者的绝对值都小于1,说明苹果早期斑点落叶病抗病性具有正态分布特征,呈现数量性状遗传特征(图2)。

图2 接种斑点落叶病8 d后的叶片病斑面积表型分布图Fig. 2 Phenotypic distribution of lesion area after inoculated with Alternaria alternata f. sp. mali for eight days
从 GWAS结果的分位数-分位数图(quantilequantile plot—Q-Q plot)中看出,Q-Q图左下角观察到的值与预期值一致(图3)。因此,当前统计模型得到的值符合期望值,证明该统计模型的合理性。同时,Q-Q图的右上角为显著性较高的SNP位点,这些候选位点可能与苹果抗斑点落叶病相关联。
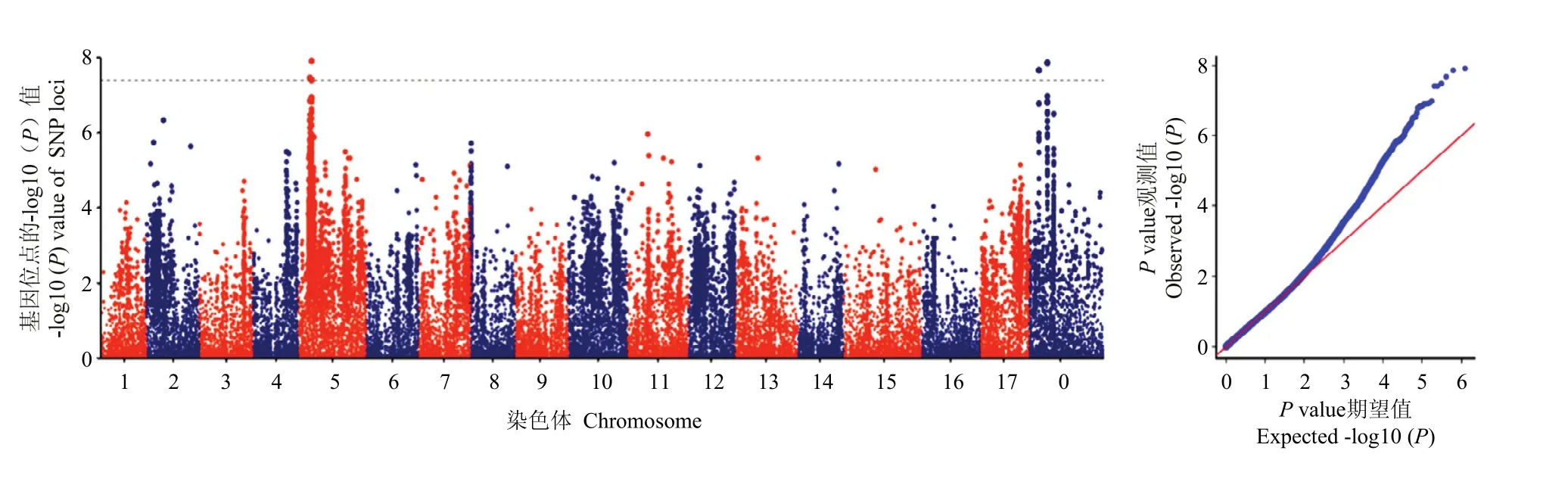
图3 斑点落叶病抗性相关性状全基因组关联分析曼哈顿图和分位数-分位数图(QQ图)Fig. 3 Manhattan plots and Quantile-Quantile Plot (QQ plot) of genome wide association analysis of resistance to Alternaria alternata f. sp. mali in apple cultivars
如图3所示,本研究共检测到6个与苹果早期斑点落叶病抗病性显著关联的位点(<0.0000001,-Lg≥7)(表4),集中分布在苹果的5号染色体(3个)和0号染色体(3个)上。其中位于5号染色体9 336 893 bp位置上的SNP为最显著的关联位点。该位点位于基因和基因之间的基因间区。其中和拟南芥的为同源基因,属于整合素连接蛋白激酶家族中的成员。前人报道该基因通过调控蛋白质氨基酸的磷酸化在植物的先天性免疫应答过程中发挥作用。和拟南芥的同源,编码FMN连锁氧化还原酶。此外,其他5个SNP也关联到7个候选基因(表4、表5),其中基因被 3个 SNP位点关联到,被2个SNP位点关联到,表明二者参与苹果调控斑点落叶病抗性的潜在可能性。其他5个候选基因分别为核糖体结合因子A家族蛋白、B-box型锌指家族蛋白、多蛋白/逆转录转座子、植物特异性GATA型转录因子的一个新家族成员、碱性亮氨酸拉链(bZIP)转录因子家族蛋白。

表4 与抗病性状相关的显著关联SNP位点Table 4 SNPs identified to be associated with Alternaria alternata f. sp. mali resistance (P<0.0000001)
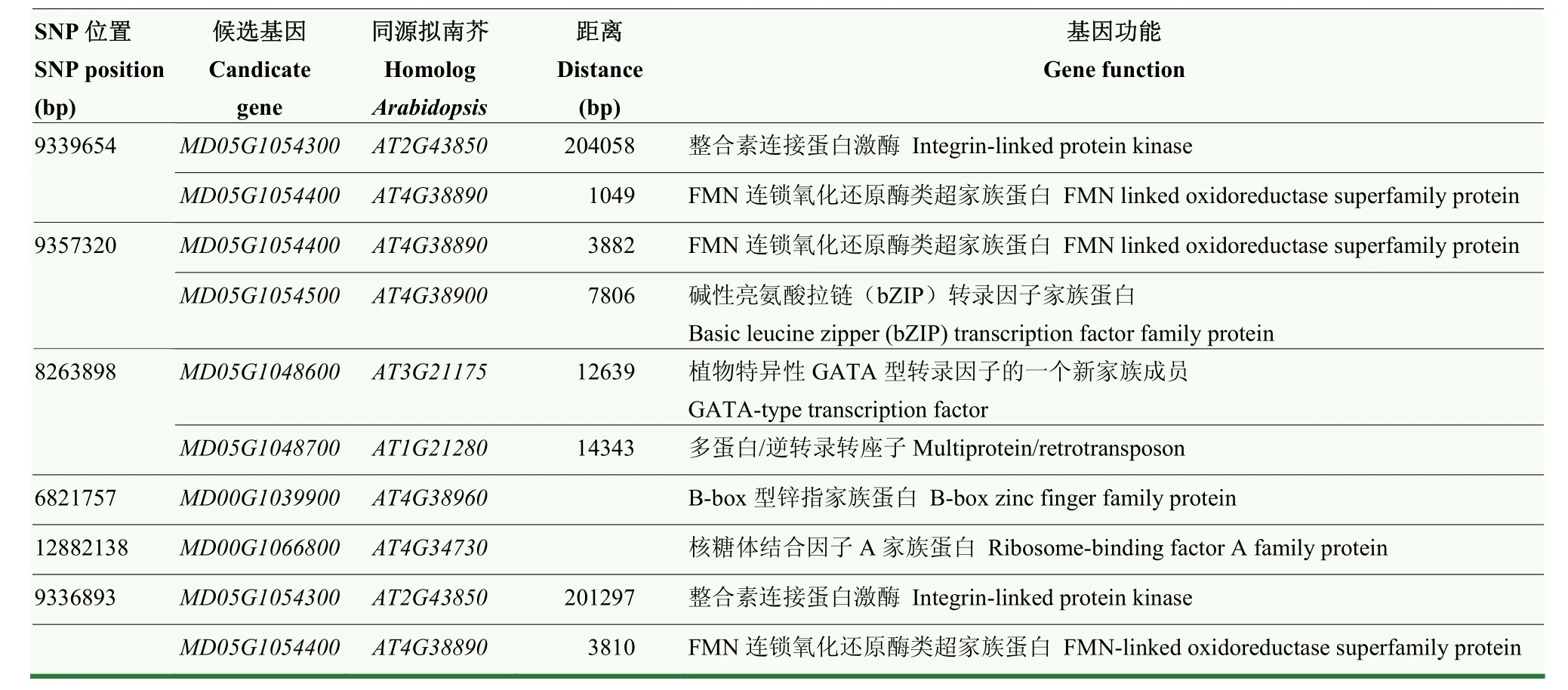
表5 苹果抗斑点落叶病的候选基因功能注释Table 5 Functional annotation of candidate genes
2.4 候选基因抗斑点落叶病的生物学功能验证
为了进一步确认7个关键候选基因、、、、、、在抗斑点落叶病中发挥的生物学功能,首先对 7个关键候选基因在接种斑点落叶病菌 0、12、24和48 h后的基因表达水平进行检测,试验结果显示7个关键候选基因在接种12和24 h后,基因的表达量均显著提高(图4),预示7个关键候选基因均在抗病功能当中发挥着重要作用。其中,在接种12和24 h后的基因上调水平达到 7倍左右,相比于其他 6个候选基因,表达水平响应最为强烈。于是将构建到超表达载体上,对 GL-3(‘嘎啦’苹果实生后代)组培苗苹果叶片进行瞬时转化。随后对瞬时超表达的苹果叶片在第4天和第7天进行表达水平的检测,发现在第7天时仍能过表达2倍。同时对瞬时超表达的苹果叶片在第4天进行斑点落叶病接种试验,结果显示,接种 3 d后在超表达的叶片中的病斑面积明显小于非转基因叶片对照中的(图5)。
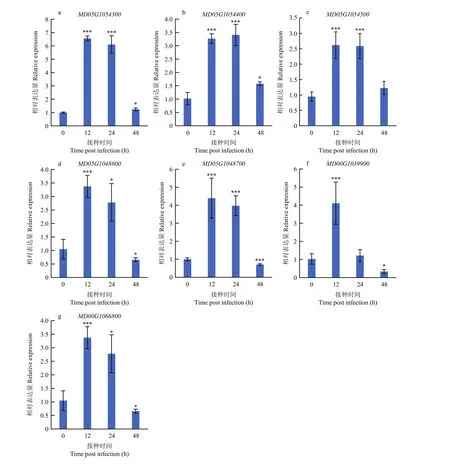
图4 关键候选基因在接种斑点落叶病后的表达水平分析Fig. 4 Expression level of key candidate genes after inoculation with Alternaria alternata f. sp. mali
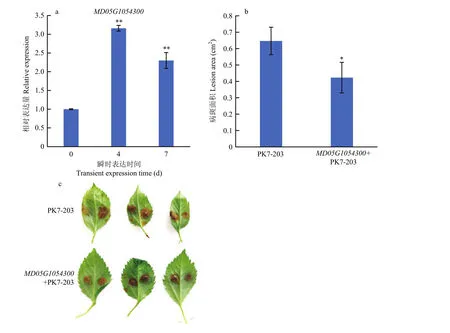
图5 超表达 MD05G1054300 的叶片抗病性Fig. 5 Disease symptoms of GL-3 leaves transiently expressing MD05G1054300
3 讨论
3.1 苹果栽培品种对斑点落叶病的抗性评价
苹果早期落叶病严重威胁苹果园的生产。早期落叶病爆发时,一片叶子上就能产生数十至上百个病斑,严重时病斑甚至连成一片,造成果园早期大面积落叶;同时感染病菌的果实极易因受二次寄生而发生腐烂现象,最终造成苹果园产量和果实品质的急剧下滑,因此,有效防治苹果早期落叶病一直是国内外特别关注的领域。研究学者通过探究斑点落叶病的发生规律,进行抗早期落叶病的基因定位分析,寻找有效的抗病基因,进行分子育种,以期达到苹果树高抗病性的目的。近年来,随着离体叶片的室内接种评价体系的完善,这种评价方式被广泛应用于苹果斑点落叶病、褐斑病等叶片病害的抗性评价研究。这种离体方法可以避免由外界环境因素的多样性和多变性对试验结果造成的干扰,也可以避免由于病原菌的数量、活力不同而造成的差异影响,评价结果具有稳定可重复性的优势。本研究对84个苹果属栽培品种进行斑点落叶病的抗性评价,从中鉴定出7个稳定抗性品种,如‘凯密欧’‘Kiku’‘皇家嘎啦’等。说明苹果属栽培品种中包含丰富的抗性资源,可为生产中引种、培育抗性品种和果园中品种的合理布局提供指导。中国的很多自育品种具有优良的性状特征,如:‘秦冠’具有丰产和抗逆性强的特点;‘秦阳’具有早熟和易结果的特性;‘寒富’具有果实品质优良,抗寒性极强的特征。本研究中,‘秦冠’‘秦阳’和‘寒富’均表现出对斑点落叶病的中等抗性。其中‘秦冠’的抗斑点落叶病能力最强,但相较于本研究中筛选出的7份抗性品种,‘秦冠’的抗病性还略有欠缺。目前,中国大面积栽培‘富士’苹果,但该品种的斑点落叶病抗性极差,因此,亟待进行品种改良以促进苹果产业健康均衡发展。本研究鉴定到7份抗病性的苹果品种,这些栽培种在果实品质和果树长势等方面均有相关报道,如路新创等研究发现美洲苹果‘蜜脆’对斑点落叶病的抗病能力强,这与本研究的结果一致。
3.2 苹果斑点落叶病抗性研究机制
通过比较苹果栽培品种对斑点落叶病的抗性,筛选高抗品种,进而通过品种改良使苹果园免受斑点病侵害。但是由于传统的苹果育种周期长,且杂交育种的不定性较强,近年来通过分子育种手段选育高抗病性苹果种质的研究越来越多。本研究中,通过GWAS关联到显著性候选基因中(附表),多次关联到参与细胞凋亡及防卫反应的含有NB-ARC结构域的抗病相关蛋白基因(NB-ARC domain-containing disease resistance protein)和,其在病害胁迫中发挥着重要作用。目前关于苹果抗斑点落叶病的抗菌机制主要集中在抗菌蛋白与病原菌几丁质的结合、诱导植物木质素的沉积、激素响应、增强植物氧化还原能力等方面。苹果由真菌几丁质诱导,其蛋白可以与几丁质结合,还能通过加强HO积累和胼胝质沉积增加苹果对病原真菌的防御反应。有研究发现能够增厚细胞壁,诱导茉莉酸响应途径,增强抗氧化相关酶活性从而提高植物的抗病性。ZHANG等研究发现两种含有卷曲螺旋、核苷酸结合和富含亮氨酸重复(CCR-NB-LRR)结构域的抗性(R)蛋白MdRNL2和 MdRNL6相互作用形成一种 MdRNL2-MdRNL6复合物,与新型发病机制相关(PR)蛋白MdPR10-1相互作用以抑制真菌生长;在其 2018年的研究中发现‘金冠’()和‘寒富’()的启动子区域中鉴定出单核苷酸多态性(SNP),即-1 186 bp的motif b(G-T)。当突变感病品种中启动子 motif b的G为T时,启动子活性丧失,表明中的 SNP与斑点落叶病抗性相关。因此,该 SNP可作为区分对斑点落叶病具有抗性或易感性的苹果品种的标记。ASR(ABA/water stress/ripening-induced)基因家族在植物应对斑点落叶病过程中存在不同的基因表达模式,暗示 Md-ASR基因家族在病害胁迫中发挥着重要作用。有研究者通过对新疆野苹果的抗斑点落叶病转录组研究发现,其响应斑点落叶病的主要方式为茉莉酸信号途径、乙烯信号途径和水杨酸信号途径。此外,该研究还发现为苹果抗斑点落叶病的主基因之一,该基因能够增强苹果氧化还原酶的能力,降低活性氧含量,从而使苹果更抗斑点落叶病。上述研究结果表明,植物的抗病机制和氧化还原能力相关,而本研究定位到的关键候选基因(FMN,连锁氧化还原酶类超家族蛋白),参与植物以氧化还原为特征的各类生理反应。
目前,GWAS分析是一种有效的复杂性状功能定位的正向遗传学分析策略,可利用群体内所有个体全基因组水平上的等位遗传变异和表型变异进行相关性分析,从而鉴定出与目标性状显著连锁的等位变异位点,最后分析等位基因型对表型的遗传效应,在植物数量性状遗传结构解析中取得了丰硕成果。GWAS分析现已被广泛用于农作物定位,如小麦籽粒品质性状和小麦抗倒伏性状的关联位点和候选基因的预测。但是由于参考基因组杂合度高,性状统计较难等原因,GWAS分析在苹果中的研究还很少。本研究以前期获得的高质量 SNP数据为基础,结合84份苹果栽培品种在接种斑点落叶病8 d后的表型数据进行GWAS分析,最终得到6个显著SNP位点,并随后关联到7个关键候选基因。其中(FMN,连锁氧化还原酶类超家族蛋白)被关联到3次,(ILK1,整合素连接蛋白激酶)被关联到2次,且这两个基因在苹果‘金冠’基因组的5号染色体上紧密相连。前人研究证明核黄素可以直接参与植物以氧化还原为特征的各类生理反应,从而诱导植物对多种病害的防治,而FMN是核黄素在体内具有生物活性的主要形式,说明苹果FMN在参与抗斑点落叶病的潜在可能性。此外,在拟南芥中整合素连接蛋白激酶(ILK1)已被报道有助于提高植物对病原体的防御,本研究在苹果中超表达能提高苹果斑点落叶病抗性,表明其具有和类似的功能。本研究预测的关键候选基因中有 3个转录因子,包括碱性亮氨酸拉链(bZIP)转录因子()、植物特异性GATA型转录因子()和B-box(BBX)家族转录因子()。这三者均被证明对植物的抗逆和生长发育起关键调控作用。如大米的属于bZIP家族蛋白,具有能够直接结合下游抗病基因启动子的能力,进而调控抗病相关基因的表达,也有研究报道辣椒中过表达能够增强植物的抗病性;小麦中过表达GATA型转录因子—TaGATA1则能够显著增强小麦对纹枯病的抗性;甘薯的B-box转录因子—IbBBX24可以通过调控茉莉酸(JA)通路来响应枯萎病的侵害。尽管本研究中鉴定到的候选基因(、和)在苹果的抗病研究中还未见报道,但是根据同源基因的功能预测,推测这些基因可能与苹果抗病性紧密相关。后续研究可将本文鉴定到的这些候选基因作为重点,深入探究其在苹果抗斑点落叶病中的生物学功能及分子机制。
4 结论
在 84份苹果属栽培品种中鉴定到斑点落叶病的抗性品种7份、易感品种2份。此外,鉴定到6个与苹果斑点落叶病抗病性显著关联的SNP位点,并关联到7个关键候选基因,其中位于5号染色体9 336 893 bp位置上的SNP为最显著关联位点,且这个位点上下游的两个基因 FMN连锁氧化还原酶类超家族蛋白()和整合素连接蛋白激酶()为关键的候选基因,其中超表达可显著提高苹果斑点落叶病抗性。

